三维二值掩模零化法结合体素绘制法切割及渲染CT与MRI体积数据
田 野,戴燕飞,季达峰*
(1.南通大学医学院医学信息学系,江苏 南通 226001;2.南通大学附属医院分院影像科,江苏 南通 226001)
CT及MRI体积数据三维可视化已成为当前研究热点[1-2],主要方法包括等值面绘制及体积渲染,后者又分为光线投射算法[3]、错切-变性算法、频域体绘制算法、最大密度投影及抛雪球算法等[4-5]。经强化处理后,如血管、肌肉及神经等解剖结构与病灶等产生与背景不同的灰度特征,结合上述特征可单独予以提取并三维可视化[6]。可视化目标结构后,如何在整体中定位加以显示是目前三维可视化与重建面临的重要问题,主要解决方案为阈值化显示结合等值面重建模型进行定位,而重建模型与阈值化渲染景深则较为棘手[7-9]。Matlab软件有多种三维绘制算法,plot3和scatter3最常用且效果相当,但利用前者所获体素模型的属性控制较少,而后者所获模型可改变体素大小并基于体素(灰度)值预设伪彩。本研究观察三维二值掩模零化法结合体素绘制法对于切割CT及MRI体积数据的作用。
1 材料与方法
1.1 材料 头颈部CT、腹部平扫CT、正常头部MR T1WI及脑胶质瘤T1WI DICOM数据取自南通大学附属医院分院不同患者。头颈部CT扫描范围自颞骨中部至T3,像素512×512×385,层间距0.63 mm,重建核0.49 mm×0.49 mm×0.63 mm;腹部CT扫描范围自T11至髋关节下缘,像素512×512×455,层间距1 mm,重建核0.67 mm×0.67 mm×1.00 mm;正常头部T1WI扫描范围自头顶至下颌,像素256×256×172,层间距0.90 mm,重建核0.95 mm×0.95 mm×0.90 mm;胶质瘤数据T1W1,像素512×512×232,层间距0.60 mm,重建核0.47 mm×0.47 mm×0.60 mm。
1.2 方法 采用Matlab软件(R2016a)。将DICOM数据导入Matlab软件获得体积数据,对其进行阈值分割,形成与更新二值掩模,之后行掩模切割与点积、目标结构再绘制与体素模型更新等。于x(左右)、y(前后)及z轴(上下)方向处理二值掩模,以体积数据左下为原点,向右、后和上方进行。见图1。
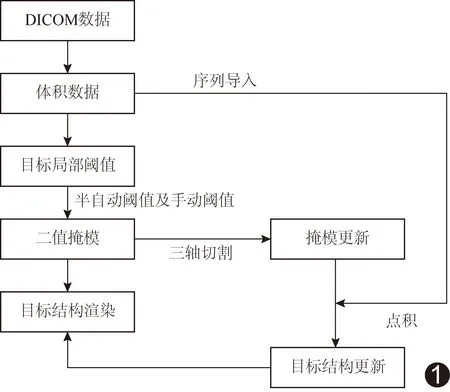
图1 技术路线示意图
1.2.1 生成体积数据 向Matlab软件中导入正常头部T1WI,利用DICOM相关处理函数生成体积数据,结合空间分辨率信息对其进行像素与空间分辨率归一。
1.2.2 设定阈值 截取目标区域断层图像,设置取值像素半径(r)与像素灰度容差比(R);点击目标结构,获得灰度计算起始点(x0,y0),根据公式(1)、(2)计算目标结构阈值:以r均值±R为阈值范围,以掩模最大程度显示目标结构为最佳取值范围。
F=f(x0±r,y0±r)
(1)
(2)
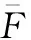
手动对目标结构取值时,使高阈值滑动条始终处于1的位置,调节滑动条设定低阈值;通过掩模与体数据点积得到阈值限制后的体积数据,并以最大密度投影实现体积绘制预览。
1.2.3 获取二值掩模 以阈值上下限为标准对体积数据进行分割,建立体积数据三维掩模,通过逻辑运算得到的目标结构体积数据二值掩模见公式(3),以之为切割与还原的原始掩模。
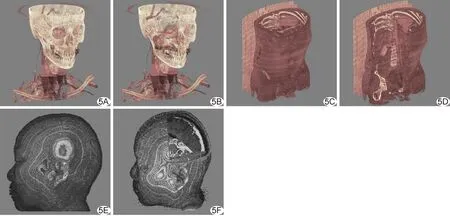
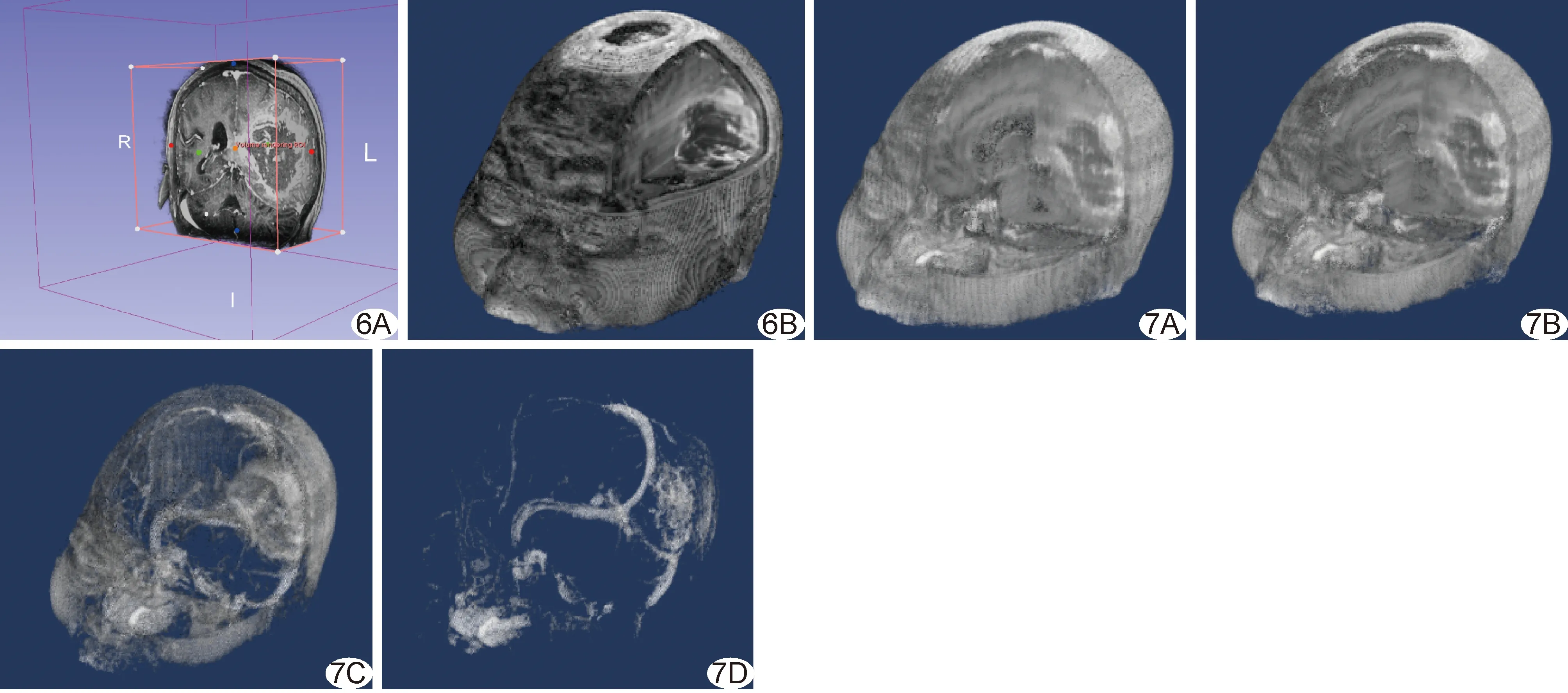
Mask=V>thrLow×V (3) 其中,Mask为所得掩模,V为体数据,thrLow与thrHigh分别代表低、高阈值,可半自动或手动设定。 1.2.4 三轴切割算法 采用正向或反向切割算法进行切割。正向切割算法主要利用单向正轴(x轴左向右,y轴前后向,z轴上向下,图2)零化法对三维二值掩模进行三轴切割,对去除区域进行零化,见公式(4)。 图2 体积数据零化方向示意图 Mask(1∶x1,1∶y1,1∶z1)=0 (4) 其中,x1,y1,z1分别表示掩模在x,y,z轴上的切割点。通过控制交互式滑动条设定切割点,使其位置与体积数据层面数相对应,基于滑动条值实现程序回调。以Mask作为原始二值掩模的更新变量而不覆盖原始掩模,调节不同切割位点,实现任意位点三轴切割。 基于正向切割算法实现反向切割算法,将零化范围限制在切割点至体数据维度末,即以切割点至体积数据在各轴方向重构后的大小进行零化,实现x轴自右向左,y轴自后向前和z轴自下向上切割(图2),见公式(5)。 Mask(x1∶Dx,y1∶Dy,z1∶Dz)=0 (5) 其中,Dx,Dy,Dz分别表示体积数据在x,y,z轴方向上的维度,即重构后冠状轴、矢状轴与水平轴方向的大小。不同轴向的正向零化及反向零化可同步进行,故可利用6种零化法实现正、反向切割。 1.2.5 渲染目标结构 对体积数据与掩模点积后的非零数据进行渲染,以可视化目标结构;通过交互式选色对非零体素灰度值与所选颜色各RGB分量进行点积,以颜色区分不同结构。体素绘制算法见公式(6)。同步更新渲染数据与切割后的掩模。 V(i)=Re(x(i),y(i),z(i),S,color,'Marktype'),i=1,2,…,N (6) 其中V(i)表示第i个层面的体素渲染结果,x(i),y(i),z(i)为非零像素点在第i个层面中的坐标,S为绘制体素大小,color为绘制颜色,'Marktype'为绘制点类型,N为总断层数,Re为体素绘制命令。 1.3 评估模型切割效果 将头颈部CT及腹部平扫CT数据导入Matlab软件,基于正常头部T1WI所获目标结构阈值,尽量减少噪点,手动调整阈值并获得相应掩模,以体素绘制法观察目标结构的完整度,评估模型切割效果。 1.4 比较切割脑胶质瘤效果 将脑胶质瘤患者T1WI数据导入3DSlicer 5.4.0软件(www.slicer.org)进行体积渲染,采用ROI交互法调节ROI手柄进行前后与左右切割,以显示其内部结构,并与三轴切割算法进行比较,观察轴限制切割显示病灶及其毗邻组织的效果。手动输入不同阈值(0.02~1.00、0.1~1.0、0.2~1.0、0.4~1.0),以相同切割参数进行切割,观察其显示不同灰度值范围内解剖结构的效果。 2.1 目标结构二值掩模 基于正常头部MR T1WI数据,设半径(r)=2、容差(R)=2时,显示上矢状窦-窦汇区较充分(图3),此时阈值范围为0.540~0.601,以之计算目标结构原始掩模。 2.2 渲染目标结构体素效果 基于阈值范围对正常头部T1WI体数据进行逻辑运算并渲染非零像素,基于上矢状窦的原始掩模覆盖灰度区较大,基于乙状窦的原始掩模覆盖区小,而基于皮肤原始掩模可较全面显示皮肤。见图4。 2.3 三轴零化法切割模型效果 在排除部分颅骨遮挡前提下,经切割后,头颈部CT可显示上矢状窦、直窦、侧脑室等颅内结构,并保持颈胸部骨骼结构、显示胸锁乳突肌与颈椎及颅骨关系;腹部CT后显示肝脏、肾脏及脊柱、髋关节等结构;正常头部T1WI可显示颅内血管与脑组织及头皮的位置关系。见图5。 图5 以三轴零化法切割CT及MRI体积数据的效果 A.未切割头颈部CT体积数据; B.切割后头颈部CT体积数据; C.未切割腹部CT体积数据; D.切割后腹部CT体积数据; E.未切割正常头部T1WI体积数据; F.切割后T1WI正常头部体积数据 2.4 切割脑胶质瘤效果 以ROI交互法于前后与左右方向全切割可显示胶质瘤内部结构,而以三维二值掩模零化法可行前后与左右方向部分切割、自由采样,提供更多病灶周围组织相关信息。见图6。 图6 以不同切割方法可视化脑胶质瘤效果 A.基于ROI交互法显示胶质瘤内侧面与前面; B.基于三维二值掩模零化法(阈值0.07~1.00,零化断层:x轴自右向左第122个层面,y轴自前向后第157个层面,z轴自下向上第66个层面)显示胶质瘤内侧、后方及下方毗邻 图7 以不同阈值、相同参数(x轴自右向左第107个断层,y轴自后向前第151个断层,z轴自下向上第34个断层反向切割)切割脑胶质瘤效果 A~D.阈值参数分别为0.02~1.00(A)、0.1~1.0(B)、0.2~1.0(C)、0.4~1.0(D) 2.5 以不同阈值切割脑胶质瘤效果 阈值分别取0.02~1.00、0.1~1.0、0.2~1.0、0.4~1.0时,以相同参数切割脑胶质瘤患者T1WI数据,发现阈值下限越高,阈值差间体素量越少,呈现结构越少,切割效果越不明显。见图7。 目前主要通过ROI实现切割以显示体积数据内部结构,如以3DSlicer软件通过调节左右、前后及上下轴的交互球进行光线投影或最大密度投影切割[9-10]。通过非可视化ROI外结构可无需经过二值掩模换算,内存消耗低、实时性较好[11]。基于Matlab软件的轴限制性显示方法以三维视角利用平移工具实现快速切割模型,内存消耗亦少,但平移时仅能于前-后、左-右、上-下进行单方向移动,难以实现量化与自由化切割;欲行双向切割,则需设置6个滑动条对显示轴进行限制,使其交互性弱于方框式ROI调节法。交互式ROI利用立方体6个面显示部分体积数据,但仅能于单轴全切方式下进行切割,其任意切割自由度低于三轴零化法。 本研究基于轴向平等二值掩模体零化与体积数据点积实现了三轴不等比例自由切割以显示体积数据,并将参数结合于滑动条上进行调整,利用1与掩模相减,得到反向切割效果,支持多达8(23)种组合方式;利用不同掩模可对不同目标结构进行三轴不等比例自由切割,具有内部局限切割效果;且所获二值掩模切割为不规则切割奠定了基础。 光线投影算法和最大密度投影算法为目前图形VTK软件的主流渲染方法,前者需对虚拟光线发射点与摄像机进行更新[3,10]以获得三维旋转效果,其优点是渲染速度快、细节处理清晰。本研究采用体素绘制法对体积数据中经掩模点积后的非零体素进行渲染,以互动式颜色选择器对渲染体素进行颜色管理,以区分不同目标结构;其速度稍慢且对电脑硬件依赖性高,但所获自由切割是对光线投影算法的较好补充。 为突出目标结构细节,设体素渲染法后与重采样后数据比例为1∶1,以去除绘制体素时软件本身存在的点边界颜色问题与矩阵稀疏性,使模型尽可能还原目标结构在体积数据中的解剖学特点。相比VTK软件以快速图像插值法对放大图像进行优化,本研究所用方法对体积数据进行空间分辨率重采样时损失的原始信息较多,导致模型仅能在一定范围内呈现较好可视化效果;但像素-空间分辨率重采样可节省绘制内存,提高体素绘制速度。另外,本研究采用具有最大连通性的简单区域生长算法[12-13]、结合操作者选取的r与R以尽可能满足不同目标结构所需阈值,模型可在不同阈值条件下反映不同目标结构,且阈值宽度较大(阈值差>0.06)时所显示的组织结构较全面,切割效果越明显,显现周围组织效果越好。 综上,利用三维二值掩模零化法联合体素绘制法可对CT及MRI体积数据进行三轴任意切割,结合不同层面体积数据显示其内部结构。但本研究采用数据下采样,体素模型结构清晰度不足,有待后续进一步完善。
2 结果
3 讨论

