基于3D路径聚合高分辨率网络的胰腺分割
尹静,刘哲,宋余庆,邱成健
江苏大学计算机科学与通信工程学院,镇江 212013
0 引言
胰腺癌是一种转移早、侵袭性强、发展较快、恶性程度高、预后较差的恶性肿瘤。根据美国癌症学会2021 年报道,自2000 年开始,胰腺癌每年发病率增长约1%,5 年生存率低于10%(Siegel 等,2021)。胰腺癌的早期诊断对于降低死亡率至关重要,是临床医学的迫切需求。胰腺的准确分割是早期胰腺癌检测识别的重要前提,但是由于胰腺在整个腹部CT(computed tomography)图像中所占比例较小,且空间位置和形态多变,如图1 所示,其准确分割一直是一项具有挑战性的任务(Yu等,2018)。

图1 NIH数据集中样本示例Fig.1 An example from the NIH dataset
随着卷积神经网络(convolutional neural network,CNN)(Krizhevsky 等,2012)在图像识别任务上的成功,研究者将其拓展到图像语义分割任务,提出全卷积神经网络(fully convolution network,FCN)(Long 等,2015)。近年来,全卷积神经网络的编码—解码结构一直是胰腺分割网络的主流结构(Zhou等,2017;Xie 等,2020;Zhang 等,2021a)。但是由于其在编码阶段捕获多尺度语义信息识别目标使用连续下采样操作,导致特征位置和细节信息的丢失,进而影响解码器不准确地预测分割边界。编码—解码结构对于形状、位置变化较大的小器官(胰腺)分割来说,影响更为明显。
针对编码—解码结构丢失特征位置和细节信息的问题,研究者提出高分辨率网络(high-resolution net,HRNet)(Sun 等,2019;Wang 等,2021)。通过在主体网络中全程保持一条高分辨率特征分支,避免位置和细节信息的丢失,提升了NIH(National Institutes of Health)数据集上胰腺的分割精度。但是直接使用高分辨率网络分割胰腺存在以下3 个问题:1)高分辨率网络是2D 网络,而CT 图像是3D 输入,2D 网络缺少提取3D 图像特征上下文信息的能力;2)受限于显存,输入图像进入高分辨率网络主体网络之前需要在茎网络中进行下采样,导致图像位置和细节信息丢失;3)在高分辨率网络分割头网络中,将主体网络输出的不同通道数的多尺度低分辨率特征直接压缩到与最高分辨率特征同等的通道数,容易导致低分辨率特征出现通道过度压缩现象(Szegedy等,2016),损失过多的特征信息内容。
针对2D 高分辨率网络缺少提取3D 图像特征上下文信息能力的问题,本文提出3D高分辨率网络(3D high-resolution net,3D HRNet)。将2D网络中的特征提取单元拓展为3D特征提取单元,使网络能够同时利用多个轴的3D特征上下文信息,改善分割结果。
针对输入图像进入高分辨率网络主体网络前下采样导致的位置和细节信息丢失问题,常用方法是使用单层卷积运算将输入图像进行转换,然后与上采样后的主体网络输出特征相融合。虽然此种方式能够弥补输出结果位置和细节信息的不足,但是融合的特征却存在巨大的语义差异,如图2 所示,导致分割结果不佳。因此,本文提出全分辨率特征路径融合模块,通过连续的非线性变换逐渐增加特征的通道数,自适应调整输入与输出之间语义差异的同时,为输出特征补充全分辨率图像输入的位置和细节信息。

图2 单层卷积运算获取的特征图和分割掩码之间的语义差异对比图Fig.2 Comparison of semantic differences between the feature map obtained by single-layer convolution operations and the predicted segmentation mask
针对高分辨率网络分割头网络压缩不同通道数的多尺度低分辨率特征时存在的信息内容损失问题,本文提出多尺度特征路径聚合模块,通过渐进地聚合由低分辨率到高分辨率的特征,以一种更为平缓的方式保证特征的压缩融合过程具有自适应调节能力,减少过度压缩低分辨率特征通道导致的信息内容损失。
根据以上分析,本文针对现有胰腺分割网络采用编码—解码结构会丢失位置和细节信息的问题,提出使用高分辨网络。针对现有高分辨率网络缺少提取3D图像特征上下文信息的问题,茎网络中下采样丢失位置和细节信息的问题以及分割头网络中多尺度低分辨率特征存在通道过度压缩的问题,分别提出了相应的解决方案。集成所有解决方案即本文提出的3D 路径聚合高分辨率网络(3D path aggregation high-resolution network,3DPAHRNet)。提出的方法在公开的数据集上进行了验证,实验结果表明,其有效解决了编码解码分割网络以及高分辨率分割网络存在的问题。
本文的主要贡献如下:1)提出使用高分辨网络分割胰腺,改变了现有胰腺分割网络一直使用的编码—解码结构,同时实现3D 高分辨率网络适应CT图像的3D 特性。2)设计了全分辨率特征路径聚合模块,为输出特征补充位置和细节信息的同时,通过连续的非线性变换缩小全分辨率图像输入和输出特征之间的语义差距。3)提出多尺度特征路径聚合模块,以平缓、渐进的方式压缩融合不同通道数的多尺度低分辨率特征,缓解了特征通道过度压缩导致的信息内容损失问题。
1 相关工作
1.1 胰腺分割
由于胰腺在腹部CT 图像中所占比例较小(< 1.5%)(Yu 等,2018),形状不规则以及位置变化较大的特点,胰腺的准确分割是一项具有挑战性的任务。因此,研究者提出了多种方法。包括基于统计模型的方法(Hammon 等,2013)、基于像素强度的方法(Tam 和Binh,2014)、基于水平集的方法(Jain等,2015)、基于图谱的方法(Oda 等,2016;Karasawa等,2017)、基于超像素的方法(Farag 等,2017)以及基于图的方法(Asaturyan 等,2019)。但是上述传统分割方法要么依赖手工提取特征,限制了特征表示能力;要么分步骤完成分割任务,增加了人工偏置。
随着全卷积网络(Long 等,2015)和U-Net(Ronneberger 等,2015;周涛 等,2021)的出现,胰腺分割任务在性能上取得了巨大提升。毕秀丽等人(2022)提出在U-Net 中使用两个不同深度的解码器,解决了现有单解码器基于较深编码特征解码,丢失胰腺位置和细节信息的问题。但是由于胰腺CT 图像的3D 特性,U-Net 等2D 分割网络难以捕获3D 图像特征上下文信息。因此,Yu 等人(2018)、Zhou 等人(2017)、Xie 等人(2020)和Roth 等人(2018)提出使用横断面、矢状面和冠状面分别训练2D 分割网络,并集成所有2D 分割网络的输出结果。虽然极大地提高了分割准确率,但本质上分割结果的获得只利用了伪3D 图像特征上下文信息。Cai 等人(2017)、Li等人(2021)和王瑞豪等人(2021)则从另一个角度考虑,将横断面切片序列看作循环卷积神经网络(convolutional long short-term memory,CLSTM)的输入,以此达到利用3D 图像特征上下文信息的效果。但是仅从3D 输入的1 个轴状面提取3D 上下文信息,缺乏信息的完整性。Zhu 等人(2018)、Schlemper等人(2019)和Mo等人(2020)从根本上缓解了3D 上下文信息无法完整获取的问题,使用3D卷积网络提取3D 图像输入的上下文信息。考虑到3D 网络虽然能捕获3D 图像特征的上下文信息,但受限于显存,网络输入往往是基于分块(patch)3D 图像,提取的3D 特征上下文信息具有局部性质,容易导致分块表面预测像素的不连续性。因此,Fang 等人(2019)和Zhang 等人(2021b)提出结合2D 网络和3D 网络的胰腺分割方法,在利用2D网络获取单张切片全局信息的同时,使用3D 网络提取的3D 图像特征上下文信息弥补单张切片缺少的上下文信息。但是由于3D网络特征通道的过度压缩,导致丢失过多相邻切片上下文信息。除了上述分割方法外,Liu等人(2022)考虑如何通过定位周围器官缩小胰腺分割区域,再利用2D卷积网络分割胰腺。
考虑到CT 胰腺数据的3D 特性,上述方法逐渐将分割网络从2D 转换为3D,利用提取的3D 特征上下文信息,改善了胰腺分割结果。但缺乏结合胰腺本身特性对分割网络进行深入思考。由于胰腺在腹部CT 图像中所占比例较小,形状和边界不规则,现有胰腺分割网络在编码阶段通过下采样提取强语义信息识别胰腺将导致胰腺特征位置和细节信息丢失,严重影响分割结果边界像素的准确性。因此,提出的分割方法基于高分辨率网络(Sun 等,2019;Wang等,2021),在分割的过程中全程保持高分辨率特征,缓解了由于下采样导致的位置和细节信息丢失问题。
1.2 高分辨率网络
现有胰腺分割方法主要基于编码—解码网络结构,在编码阶段通过连续的下采样获取不断增强的语义信息识别目标,再通过对称的连续上采样预测分割结果。但连续的下采样会导致分割目标位置和细节信息的丢失。因此,研究者提出高分辨率网络(Sun等,2019;Wang等,2021),如图3所示。高分辨率网络包括3个子网络:茎网络、主体网络和分割头网络。

图3 高分辨率网络Fig.3 High-resolution network
茎网络如图3 左边部分所示,负责降低输入图像的分辨率,减少网络占用的内存消耗。包括两个连续的两倍卷积下采样,将输入图像分辨率降低为原始分辨率的1∕16。
主体网络如图3中间部分所示,细节信息如图4所示,包括4 个平行分支和4 个特征提取阶段。其中,第1 个平行分支提取特征的过程中,全程保持茎网络输出特征的高分辨率,完整保留了特征的位置和细节信息;第2 个分支在第1 阶段最后通过2 倍卷积下采样,将特征分辨率变为1∕4,特征通道变为两倍,因此,第2 个分支全程保持1∕4 的特征分辨率。与此方式类似,第3 和第4 个分支分别在第2 和第3 个阶段的末尾进行2 倍下采样,降低特征分辨率,提升特征通道数,在各自分支中全程保持相同的特征分辨率。高分辨率网络还包括4个阶段,除第1个阶段外,每个阶段包括特征提取和特征融合两个模块。特征提取模块由4 个残差卷积单元组成,每个卷积由2 个普通卷积和1 个残差连接构成。特征融合将所有平行分支进行融合。具体来说,高分辨率特征分支通过下采样与低分辨率特征融合,为低分辨率特征提供详细的位置和细节特征信息;而低分辨率特征分支通过直接的上采样与高分辨率特征分支融合,为高分辨率特征提供更强的语义信息。通过后3个阶段重复特征提取和特征融合串联模块,主体网络能够提取多层级、位置和语义信息鲁棒的多尺度输出特征。值得注意的是,由于第1阶段只有1个分支,因此没有特征融合模块。此外,在每个阶段融合模块中,如果分辨率相同,则将前层的特征作为当前层的特征,相当于复制过程,如图中红色箭头所示。
分割头网络如图3 右边部分所示,其将主体网络4 个分支输出的多尺度特征直接进行压缩融合,再通过sigmoid 函数获得最终的分割结果。由于在主体网络始终有1 个分支保持茎网络输出的高分辨率特征,因此整个网络称为高分辨率网络,这不同于现有的先下采样,再上采样的分割网络。本文方法基于高分辨网络。
1.3 特征通道压缩融合
随着反向传播算法的提出和GPU(graphic processing unit)计算性能的提升,基于卷积神经网络的方法在图像领域取得了巨大成功。基于卷积神经网络方法的主干网络,通过逐渐增大感受野,增加提取特征通道数的同时,捕获更多高级语义特征模式识别目标。受限于显存和网络训练的难度,早期的卷积神经网络(Krizhevsky 等,2012;Simonyan 和Zisserman,2014)层数较浅,网络提取的特征通道数较少,很少有研究者关注特征压缩和融合问题。随着GoogLeNet(Szegedy 等,2015)提出多分支特征结构,导致网络层中的特征通道数大幅度增加,进一步导致网络难以训练。由此研究者意识到压缩特征通道并尽可能保留有效特征信息具有重要研究意义。Network in network 提出使用1 × 1 卷积压缩融合特征信息的方式逐渐成为后续方法压缩并保留有效特征信息的标准方法。根据卷积网络设计法则(Long等,2015;Ronneberger 等,2015;Badrinarayanan 等,2017;Zhou等,2020),网络中特征通道应以2倍的方式压缩,而Szegedy等人(2016)在研究多分支结构的实验中,总结出使用1 × 1卷积压缩超过2倍的特征,应以渐进、平缓的方式,即压缩特征过程应是多步骤、有中间过程的,使特征能够进行自适应调整,否则会出现特征通道过度压缩现象,导致特征信息内容损失。本文方法针对高分辨率网络分割头网络中过度压缩不同通道数目多尺度低分辨率特征通道导致的信息内容损失问题,提出了一种渐进的多尺度特征路径聚合模块,从低分辨率逐渐向高分辨率特征压缩融合,中间过程中通过卷积对特征进行自适应调整,缓解了特征过度压缩的问题。本文方法中的多尺度特征压缩融合方式类似3D U-Net(Çiçek等,2016)、AttentionUNet(Schlemper 等,2019)和VNet(Milletari 等,2016)等网络特征压缩融合方式。不同点在于:1)提出的压缩融合方式是为了解决高分辨率网络分割头网络特征通道过度压缩的问题,而上述网络融合是为了恢复图像的位置和细节信息;2)在通道特征渐进压缩融合的过程中,本文方法通过区分语义特征信息及位置和细节特征信息,提供更加准确的语义识别模式和更加精准的分割边界。
2 方法
针对基于编码—解码网络和基于高分辨率网络分割胰腺存在的问题,本文提出基于3D路径聚合高分辨率网络的胰腺分割方法。在分割胰腺的过程中全程保持高分辨特征,提升对于像素位置和细节信息敏感的胰腺分割网络的性能。本文方法分割过程如图5 所示,包含3 个步骤:1)为了增加图像的对比度,降低网络的学习难度,输入图像首先需要进行预处理;2)受限于显存,预处理后的图像被随机切分成160 × 96 × 96 大小的块,输入3D 路径聚合高分辨率网络;3)为获得最终的分割结果,所有基于块的网络分割结果需要经过后处理步骤。图5中n表示横断面切片数量。
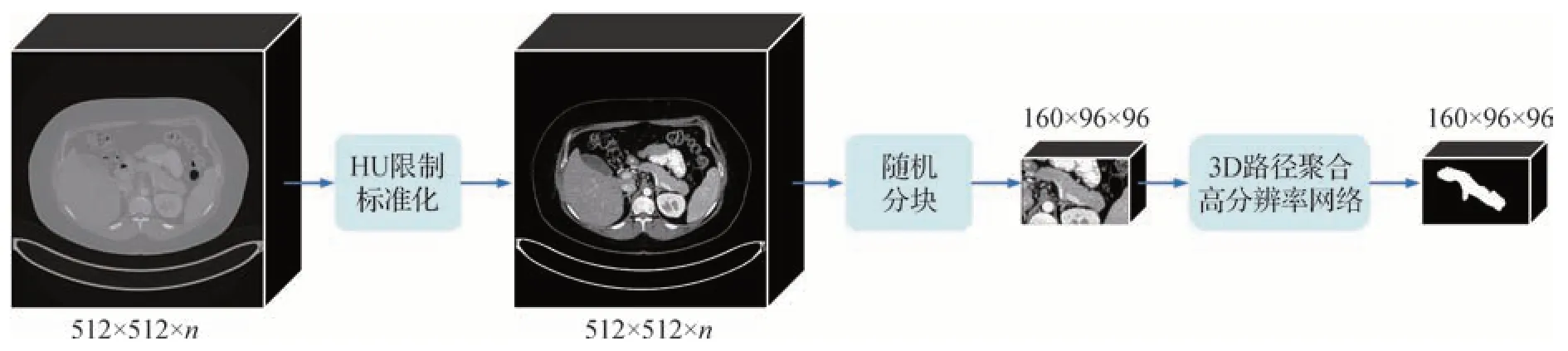
图5 胰腺分割过程Fig.5 The pipeline of pancreas segmentation
2.1 数据预处理
数据预处理的目的是增加胰腺区域的对比度,降低网络的学习难度。主要包括两个步骤:
1)由于CT 图像的HU(hounsfield unit)值分布范围较大,胰腺和周围组织器官边界较为模糊,根据其窗框窗位,将胰腺的HU 值限制在[-100,240]之间,增加胰腺边界和周围组织器官的对比度。HU 限制具体为
2)为了降低分割网络的学习难度,加速收敛,将经过HU 限制的输入图像进行Z-Score 标准化处理。处理过程为
式中,xi和xj分别表示输入CT 图像在i和j位置的HU值,n表示像素的总数目。
2.2 3D路径聚合高分辨率网络
本文提出的3D 路径聚合高分辨率网络如图6所示。通过降低茎网络下采样次数和提出全分辨率特征路径聚合模块减少了高分辨率网络(图3)茎网络连续下采样丢失的位置和细节信息;通过提出多尺度特征路径聚合模块缓解了由于高分辨率网络分割头网络过度压缩低分辨率特征通道导致的信息内容损失问题。3D 路径聚合高分辨率网络和高分辨率网络主体网络结构相似,仅将高分辨率网络中的2D卷积替换为3D卷积,捕获CT图像的3D特征上下文信息;将批量归一化替换为实例归一化,减少输入批次大小对于分割结果的影响。

图6 3D路径聚合高分辨率网络Fig.6 3D path aggregation high-resolution network
2.2.1 全分辨率特征路径聚合模块
在高分辨率网络中,输入图像进入茎网络时会进行连续两次2倍卷积下采样,将特征图减小到1∕16,达到降低GPU 显存消耗的目的。但是由于胰腺在整个输入CT 图像中所占比例较小,4 倍的下采样会导致位置和细节信息的严重丢失。因此,本文方法仅对输入图像进行单次2 倍下采样,减少位置和细节信息的损失。同时为了缓解单次2 倍下采样丢失的位置和细节信息,提出了全分辨率特征路径聚合模块,如图6底部所示。
由图2 可知,由于全分辨输入和输出结果的巨大语义差异,简单将其融合将导致胰腺分割网络性能的下降。因此,需要添加更多的非线性函数拟合输入到输出的变换。但是如果按照3D 路径聚合高分辨率网络茎网络初始设置的通道数开始并以通道递增的方式进行连续的非线性变换将导致显存消耗过大,网络无法训练。考虑到全分辨率特征路径的作用仅是为了补充更加丰富的位置和细节信息,提出方法将全分辨率特征路径聚合模块初始通道设置为3D 路径聚合高分辨率网络茎网络初始通道数的1∕5,大量减少计算和显存消耗的同时,为网络输出弥补精准的位置和细节信息。全分辨率路径聚合模块每层的非线性变换可表示为
式中,Xi∈RW×H×D×2i×C表示茎网络第i层的输出,其中W、H、D分别表示特征图的3 个维度;2i×C表示第i层的特征通道数;Vi=[v1,v2,…,vi×j]表示卷积核集合,其中i×j表示第i层卷积核的个数;X0表示全分辨率图像输入,第0 层卷积核个数为0;F表示ReLU(rectified linear unit)非线性函数;I表示实例归一化;“*”表示卷积。
全分辨率特征路径聚合模块通过对输入的全分辨率CT 图像进行5 层的非线性变换,将特征图通道逐渐增加到网络设置的初始输入通道数,不显著增加显存和计算消耗的同时,获得的输出结果为X5。
2.2.2 多尺度特征路径聚合模块
针对高分辨率网络分割头网络中输出的不同通道数多尺度低分辨率特征融合过程中存在特征通道过度压缩的问题,本文提出多尺度特征路径聚合模块,以一种渐进、平缓的方式自适应压缩融合多尺度特征。
多尺度特征路径聚合模块如图6 右边所示。将主体网络输出的多尺度特征记为Yi,i表示不同尺度分支,i=1 表示最小尺度分支(最高分辨率分支),i=4表示最大尺度分支(最低分辨率分支)。虽然每个尺度分支在主体网络中都融合了其他所有尺度分支的特征,但不同尺度分支输出的特征有不同的侧重点。即最小尺度分支Yi(i=1)侧重保证分割结果位置和细节信息的准确性,较大尺度分支Yi(i=1,2,3)侧重保证通过不同强度的语义信息识别出完整的目标,避免误分割。
为了识别上述较大尺度分支有效语义信息的同时抑制无效的噪声信息,本文提出语义自适应模块(semantic adaptation,SA),对主体网络输出的较大尺度特征Yi(i≠ 1)进行语义信息校准。语义自适应模块由通道注意力机制(Hu 等,2018)实现,如图7 所示。图中,卷积核大小为3 × 3,步长为1,填充为1;ReLU 和sigmoid 分别表示非线性函数和归一化函数。通过为输入特征通道生成不同大小的权重,抑制无关噪声通道,增强有效语义特征。

图7 语义自适应模块Fig.7 Semantic adaptation module
将语义自适应模块的输出记为Si(i=2,3,4),并将多尺度特征路径聚合模块渐进压缩融合过程中每个尺度输出的结果记为Zi(i=1,2,3,4)。Si通过与经过转置卷积的前一层输出结果Zi-1融合,并通过卷积单元自适应调整获得当前层的输出Zi。其过程为
式中,T表示卷积核大小为2 × 2 × 2、步长为2 的转置卷积。C表示卷积单元,包括3 × 3 × 3、步长为1、填充为1 的卷积核,ReLU 非线性函数和实例归一化函数。当i=1时,S1=Y1,“+” 表示特征图拼接;i≠ 1时,S1表示语义自适应模块输出特征,“+” 表示对应元素相加操作。
最终的融合结果为Z1。首先,通过转置卷积获得全分辨率大小的输出特征;其次,将转置后的输出特征与全分辨率路径聚合模块输出特征X5进行拼接融合,补充位置和细节信息;然后,通过sigmoid 函数获得分割结果P。最后,将分割结果P和标准标签G进行损失计算,优化分割结果。分割结果的获得过程为
式中,+表示拼接操作;C表示卷积融合操作;T表示转置卷积。
由于3D 路径聚合高分辨率网络是基于随机分块输入的分割网络,对于输入块中前景和背景区域的比例无法确定,因此,损失函数既要考虑前景对分割结果的影响,也要考虑背景对分割结果的影响。本文采用Dice 损失函数和二分类交叉熵损失函数结合的混合损失函数作为反向传播的损失。这里Dice 损失能够解决前景和背景不平衡的问题,而二分类交叉熵损失在输入块中前景所占比例较大时,通过考虑背景像素改善分割结果。混合损失函数计算为
式中,λ表示不同损失函数所占的权重大小,本文设置为0.5。pi和gi分别表示预测像素和标准标签像素,n表示像素的总个数,Ld、Lb和L分别表示Dice 损失函数、二分类交叉熵损失函数和总的损失函数。
2.3 后处理
后处理步骤只用在测试过程中,包括分块输出结果集成和最大连通区域选取。
由于基于分块的3D 分割方法缺少分块外的3D特征上下文信息,因此,当网络输入块中分割目标不完整时,容易导致预测出错误的块边界分割目标像素。基于此问题,本文方法在测试时以滑动窗口一半的大小作为步长,移动滑动窗口选取测试分块作为网络的输入。从而选取的相邻测试分块之间有一半区域重合,解决了分块边界像素预测不准确的问题。完整的胰腺分割结果是所有分块分割结果的集成。由于基于分块的3D分割方法在测试时,网络的分块输入上下文信息不完整,分块的网络输出结果容易出现假阳性预测像素。因此,通过集成所有分块分割结果获得的完整胰腺分割结果存在假阳性分割区域,本文方法通过选取最大连通区域作为最后的分割结果,减少不相关假阳性噪声的影响。
3 实验和结果
3.1 数据集及实验设置
本文方法在NIH 胰腺数据集(Roth 等,2015)上进行了验证。NIH 数据集由82 例增强CT 卷组成。单张切片扫描具有512 × 512 像素的固定分辨率,所有切片的数目分布在[181,466]之间。CT 卷的3 个轴状面像素之间都存在间距,其中矢状面和冠状面间距大小分布在[0.5 mm,1.0 mm]之间,横断面间距都为1.0 mm。针对卷积网络同等看待不同轴状面间距导致丢失真实物理距离的问题,根据Isensee等人(2021)的方法,本文统计NIH 数据集所有样本3 个轴状面的间距,并将其采样到每个轴状面的中位数,降低真实物理距离对分割结果的影响。采样后冠状面、矢状面和横断面间距分别统一为0.85 mm、0.85 mm 和1.0 mm.本文实验中数据集按照7∶1∶2比例随机划分为训练集57 例、验证集8 例和测试集17例。
训练过程中,提出的3D 路径聚合高分辨率网络,在数据集上迭代了1 000次(epoch),初始学习率设置为0.000 1,在达到训练600 次和800 次的时候分别衰减90%,帮助网络收敛到更低的最小值。除此之外,使用Adam 作为优化器,网络输入大小为160 × 96 × 96,批次大小(batch size)设置为1,网络中最高分辨率分支通道数保持48,其他分支每当分辨率减小为原来的1∕4,通道数就会增加两倍。网络中卷积单元中卷积核大小为3 × 3 × 3,步长为1,填充为1。卷积下采样中卷积核大小为3 × 3 × 3,步长为2,填充为1。在全分辨率特征路径聚合网络中5个卷积核通道数分别为3,6,12,24和48。所有实验使用的显卡为RTX A6000,训练时间为48 h。测试过程中,滑动窗口大小设置为160 × 96 × 96,步长设置为80,48,48。
3.2 评价指标
本文方法使用Dice 系数(Dice similarity coefficient,DSC)衡量正确预测像素的覆盖率,使用Jaccard 系数(Jaccard index,JI)衡量正确的预测像素集合在总集合像素中的比率,使用精确率(precision)衡量正确预测像素在预测像素集合中的比率,使用召回率(recall)衡量正确预测像素在标准标签像素集合中的比率(Huang和Wu,2022)。
从评价指标的定义可以看出,在结果相差不大的前提下,精确率和召回率在分割结果中可能存在相反的关系,这种相反的关系易出现在消融实验中。
3.3 不同基线网络的结果对比
本节通过与3D 编码—解码医学图像分割基线网 络3D U-Net(Çiçek 等,2016)、AttentionUNet(Schlemper等,2019)和VNet(Milletari等,2016)的对比实验以及与3D 高分辨率网络(3D HRNet)的对比实验,验证提出的3D 路径聚合高分辨率网络(3DPAHRNet)的有效性。
分割结果如表1 所示。由于3DUNet,VNet 和AttentionUNet 在编码阶段通过不断地下采样,在更大的感受野上捕获强语义信息识别胰腺,导致获得的特征丢失位置和细节信息。因此,在Dice 系数、Jaccard 系数、精确率和召回率4 个评价指标上都明显低于3D 高分辨率网络(3DHRNet)。提出的3D 路径聚合高分辨率网络,除了全程保持高分辨率特征外,还通过全分辨率特征聚合模块缓解了3D高分辨率网络茎网络丢失特征位置和细节信息的问题,以及通过多尺度特征路径聚合模块以一种更平缓的方式促进多尺度特征自适应调整压缩融合。因此,Dice 系数、Jaccard 系数、精确率和召回率相比于3D高分辨率网络分别提升1.41%,2.09%,2.35%和0.49%。此外,由于本文方法所提取的特征在位置和细节信息上的准确性,最小分割准确率(困难样本)均大幅提升。表1 的最右一列提供了不同基线模型每例样本平均推断时间的对比。从中可以看出,本文方法推断时间消耗小幅度高于基线模型。由于本文方法在网络中全程保持高分辨率,有着更多的计算消耗,所以推断时间有所增加。但对于具有挑战性的胰腺分割任务来说,通过增加少量的时间成本,大幅提升胰腺分割网络的性能,对于实际临床应用是可折中的。并且随着硬件设备的快速更新迭代,推断时间差距将进一步缩小。
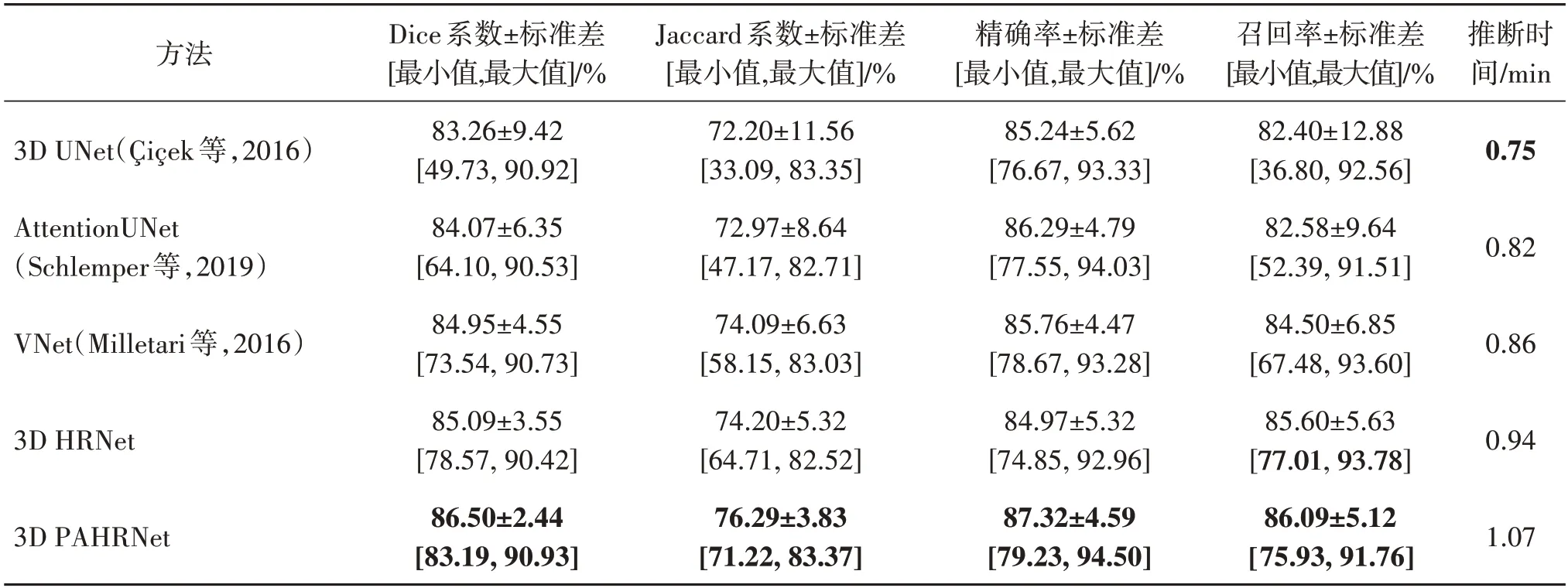
表1 3D基线胰腺分割网络模型对比结果Table 1 Comparison results of 3D baseline pancreas segmentation networks
除了上述分割结果的定量分析,本文通过图8定性展示不同基线网络分割的对比结果。图中用绿色圆圈出了相比于本文方法的分割结果,与标准标签(ground truth,GT)相差较明显的位置。
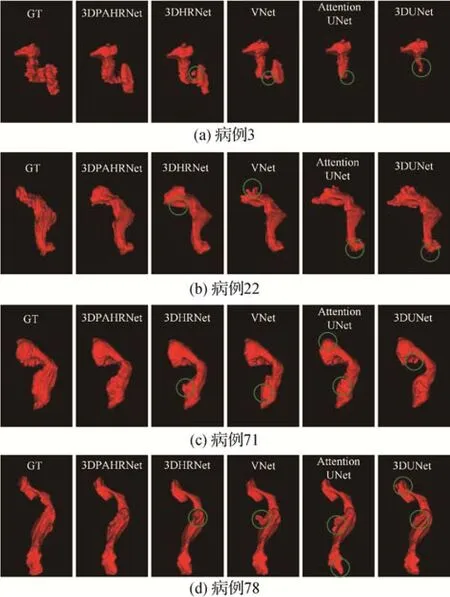
图8 不同基线网络的3D分割对比结果Fig.8 Comparison results of different baseline networks((a)case 3;(b)case 22;(c)case 71;(d)case 78)
3.4 消融实验
通过分析不同茎网络下采样因子对比实验、全分辨率特征路径有效性以及多尺度特征路径有效性分别验证本文方法及各模块的有效性。
3.4.1 不同茎网络下采样因子对比实验分析
本节针对3D 高分辨率网络(3DHRNet)茎网络中使用不同下采样因子对于分割结果的影响进行分析。受限于显存,下采样因子只采用2倍或4倍。实验结果如表2 所示。当采用连续两次卷积进行4 倍下采样时,相比于使用1 次2 倍卷积下采样,胰腺分割在Dice 系数,Jaccard 系数和精确率上分别降低了0.74%,1.12%和2.6%。其主要原因是胰腺器官在腹部CT 中所占比例较小,形状和位置变化较大,在茎网络中使用4 倍下采样会导致特征位置和细节信息的严重丢失,降低分割结果的平均准确率。

表2 采用不同茎网络下采样因子的对比实验结果Table 2 Comparison results using different down-sampling factors in the stem network
图9 展示了测试集中每个样本分别通过2 倍下采样(2×)和4 倍下采样(4×)3D 高分辨率网络获得的平均准确率。可以看出,当茎网络采用更小的2 倍下采样因子时,能够提升大部分样本的平均分割准确率。后续消融实验均基于茎网络采用2 倍下采样因子的3D高分辨率网络。

图9 茎网络中使用不同下采样因子测试样本的对比实验结果Fig.9 Comparison results of test samples utilizing different down-sampling factors in the stem network((a)Dice;(b)Jaccard;(c)precision;(d)recall)
3.4.2 全分辨率特征路径聚合模块有效性分析
为了验证全分辨率特征路径聚合模块的有效性,本节针对3D高分辨率网络添加和未添加全分辨率特征路径聚合模块以及将全分辨率特征路径聚合模块替换为单层卷积模块进行实验分析。如表3 所示。相比于未添加全分辨率特征路径聚合模块的3D 高分辨率分割网络,当添加全分辨率特征路径聚合模块时,在Dice系数、Jaccard系数和召回率3个评价标准上分别提升0.44%、0.64%和1.55%,标准差分别降低0.53%、0.76%和1.85%,最小分割准确率分辨提升1.65%、2.31%和6.43%。从中可以看出,全分辨率特征路径聚合模块提供的全分辨率输入图像的位置和细节信息,显著提升了分割结果的平均准确率,增加了胰腺分割网络的稳定性以及改善了困难样本的分割结果。而当将全分辨率模块替换为单层卷积模块时,Dice 系数,Jaccard 系数和召回率3 个评价标准却出现了显著下降,低于未使用全分辨率特征路径聚合模块的高分辨率网络。这是因为单层卷积模块无法拟合输入到输出之间复杂的非线性变换,缩小输入和输出特征之间的语义差距。反而由于直接融合特征使原始特征信息产生了混乱,导致更低的分割精度。

表3 全分辨率特征路径聚合模块消融实验Table 3 Ablation study of the full-resolution feature path aggregation module
图10 展示了3D 高分辨率网络添加(是)和未添加(否)全分辨率特征路径聚合模块以及将全分辨率特征路径聚合模块替换为单层卷积模块(单层卷积)输出分割结果的总体分布箱线图,以图10(a)Dice 系数最左侧盒子为例加以说明。最左侧盒子底部表示分割结果分布的第1 四分位,盒子顶部表示分割结果的第3 四分位,最底部的水平线表示最小值,最顶部的水平线表示最大值。最左侧盒子中实线表示平均值,虚线表示中位数。平均值、中位数均是反映数据整体分布的评价指标。从图10 可以看出,本文方法在Dice 系数、Jaccard 系数和召回率上有着更高的平均值和中位数,并且结果分布更加集中,进一步验证了提出方法的有效性和稳定性。
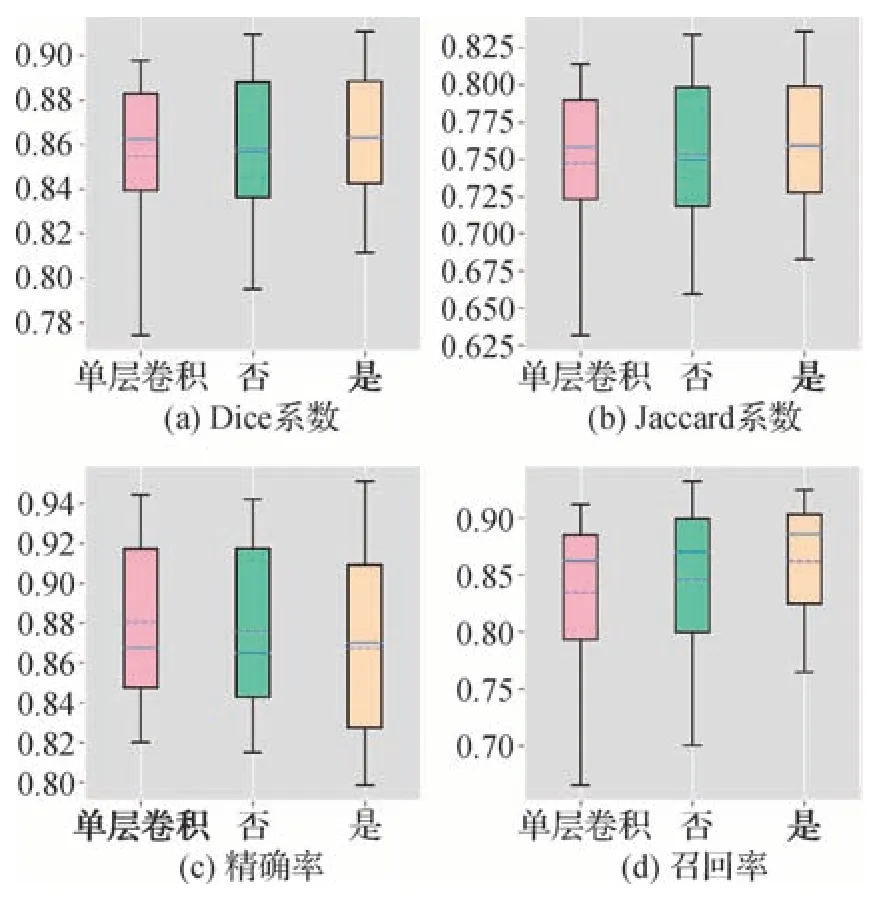
图10 全分辨率特征路径聚合模块对比实验箱线图Fig.10 Boxplot of experimental results using the full-resolution feature path aggregation module((a)Dice;(b)Jaccard;(c)precision;(d)recall)
除此之外,通过图11 定性展示了单层卷积模块输出特征、全分辨率特征路径聚合模块输出特征和分割掩码之间的对比结果。从图11 可以看出,单层卷积模块输出的特征相比于全分辨率特征路径聚合模块输出特征,和分割掩码之间有着更大的语义差距。

图11 不同模块输出特征和分割掩码之间的语义信息比较Fig.11 Semantic comparison between output features of different modules and segmentation masks((a)segmentation masks;(b)output feature maps of full resolution feature aggregation model;(c)output feature maps of multi-scale feature aggregation module)
3.4.3 多尺度特征路径聚合模块有效性分析
为了验证多尺度特征路径聚合模块对于胰腺分割性能的影响,本节针对3D高分辨率网络添加和未添加多尺度特征路径聚合模块进行实验分析,如表4所示。相比于未添加多尺度特征路径聚合模块的3D 高分辨率分割网络,添加多尺度特征路径聚合模块在Dice 系数、Jaccard 系数和召回率上分别提升0.49%、0.71%和1.83%,最小值分别提升1.68%、2.35%和5.85%,标准差分别降低0.59%、0.87%和1.94%。从中可以看出,添加多尺度特征路径聚合模块,以渐进,平缓的方式融合压缩多尺度特征,减少中间过程特征信息内容损失,能够显著提升胰腺分割网络性能。

表4 多尺度特征路径聚合模块消融实验Table 4 Ablation study of the multi-scale feature path aggregation module
图12 展示的是测试样本在3D 高分辨率网络中添加(是)和未添加(否)多尺度特征路径聚合模块输出分割结果的总体分布箱线图。从图中可以看出,添加了多尺度特征路径聚合模块在大部分评价指标上,中位数和平均值均有所提高,并且整体分割结果的平均准确率也都有所改善。
3.5 不同分割模型的结果对比
为了进一步验证本文方法的先进性,本文方法与当前具有代表性的分割方法进行了比较。
不同分割方法定量比较结果如表5所示。相比于编码解码结构的胰腺分割网络(Zhou等,2017;Yu等,2018;Schlemper 等,2019;Xie 等,2020;Zhang 等,2021a,b;王瑞豪 等,2021;毕秀丽 等,2022;Liu 等,2022),Dice系数从最高85.40%提升到86.50%,最小Dice系数从最高70.61%提升到83.19。由于本文提出的3D路径聚合高分辨网络能够全程保持高分辨率并且最大程度地减少分割网络中胰腺特征位置和细节信息的损失,显著提升了胰腺分割网络的性能,改善了困难样本的分割结果。表5的最后一列给出了每例样本平均推断时间的结果对比。由于Zhou 等人(2017)在3个轴状面分别训练模型分割胰腺,并且通过循环迭代的过程进行测试,所以相比本文方法推断时间增加明显。Zhou 等人(2017)、Yu 等人(2018)和Xie等人(2020)将测试过程由分步骤变为端到端,显著减少了推断时间消耗,但测试过程中采用循环迭代的方式并没有改变。因此,推断时间消耗高于本文提出方法。Zhang等人(2021b)通过结合传统和深度学习方法分3步分割胰腺,在所有对比方法中时间消耗成本最大。毕秀丽等人(2022)方法基于2D U-Net,虽然极大减少了推断时间消耗,但由于网络缺少捕获3D上下文特征信息的能力,Dice系数减少了11.4%。
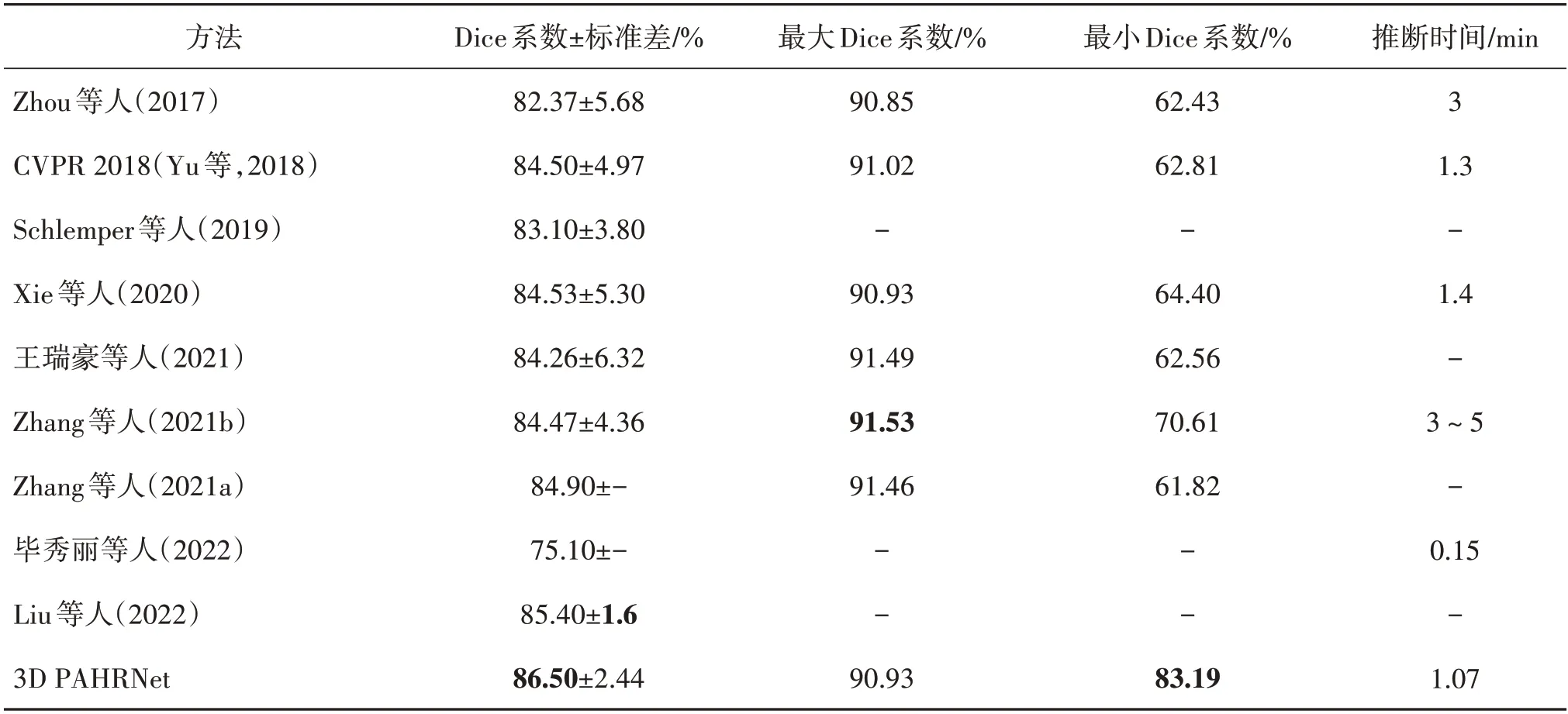
表5 不同分割方法的对比实验结果Table 5 Comparison results of different segmentation methods
为了进一步验证本文方法的有效性,通过图13定性展示分割结果和标准标签分割边界的对比结果。其中,红色实线(GT)表示标准标签边界,蓝色实线(SEG)表示分割结果边界。从图中可以看出,本文方法的分割边界与标准标签分割边界非常接近,进一步验证了本文方法能够保留精确的胰腺位置和细节信息。

图13 本文方法分割结果(SEG)和标准标签结果(GT)的对比展示Fig.13 Comparison of predicted segmentation results of the proposed method and ground truth((a)case 8;(b)case 40;(c)case 52;(d)case 72)
4 结论
针对现有胰腺分割网络编码过程丢失位置和细节信息的问题,本文提出基于3D路径聚合高分辨率网络的胰腺分割方法。通过提出的全分辨率特征路径聚合模块和多尺度特征路径聚合模块,缩小全分辨率图像输入和深层输出特征语义差异的同时,以一种平缓自适应的方式渐进压缩融合多尺度特征。
在NIH 胰腺数据集上的实验结果表明,本文方法是一种高性能的胰腺分割方法。由于提出方法能够始终保持输入图像特征和分割结果的细节和位置信息,因此,在分割结果上显著优于经典分割方法和最先进的胰腺分割方法,除此之外,本文方法具有相对较少的推断时间,实时性强。但本文方法部署于临床应用还存在较高的计算复杂度。因此,未来工作将从轻量级网络模型方面开展,通过剪枝、模型压缩等方法降低模型复杂度的同时保持较高的分割精度。

