基于Scissor算法的肝内胆管癌新型分子预测模型
宋一粟,张惠忠,疏文志,苏仁义,魏绪勇,徐骁
1.浙江大学医学院附属杭州市第一人民医院肝胆胰外科,浙江 杭州 310006;2.浙江省肿瘤融合研究与智能医学重点实验室,浙江 杭州 310006;3.浙江大学医学院,浙江 杭州 310058;4.金华广福肿瘤医院肝胆胰胃外科,浙江 金华 321111
肝内胆管癌(intrahepatic cholangiocarcinoma,ICC)是指肝内二级胆管至肝内最小胆管分支的衬覆上皮及其胆管周腺体发生的恶性肿瘤,并表达胆管细胞标志物[1]。在原发性肝癌中,ICC的发病数量占10%~15%[2]。由于ICC早期症状隐匿,病人往往在肿瘤进展至晚期时才能被确诊,而且目前仍缺乏针对ICC的有效治疗方法,从而导致预后较差[3]。近年来,免疫治疗已成为综合治疗ICC等胆道肿瘤的重要手段之一[4]。由于肿瘤异质性以及肿瘤微环境的复杂性,免疫治疗效果在不同ICC个体间差异巨大,部分病人存在免疫治疗抵抗[5]。目前针对ICC的预后模型较为多样,但仍然缺乏根据肿瘤微环境单细胞构建的与免疫治疗相关的预后预测模型。高通量单细胞测序技术是探索ICC肿瘤微环境及其治疗响应的有力工具,而目前仍缺乏与ICC预后相关联的临床大样本的单细胞测序研究。Scissor算法是近年开发的可用于将单细胞数据和临床表型相关联的新兴技术[6],可以从大量单细胞中筛选出与临床预后相关的细胞。肿瘤免疫功能障碍和排斥(tumor immune dysfunction and exclusion,TIDE)算法则可以通过病人肿瘤样本的转录组数据预测病人的免疫治疗抵抗[7-8]。本研究整合分析了美国国立生物技术信息中心基因表达(GEO)数据库中高质量ICC单细胞数据集(GSE151530)和癌症基因组图谱(TCGA)数据库内ICC的转录组测序数据及临床数据,通过Scissor算法识别ICC肿瘤免疫微环境密切相关的细胞,并分析差异表达基因(differentially expressed genes,DEGs)及其关键信号通路。建立蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络并将模块基因与肿瘤TIDE评分进行相关性分析,筛选出与免疫治疗效果相关的关键基因。最后使用多因素Cox回归分析得到3个枢纽基因并构建了一个预后预测模型,同时通过外部数据集验证该模型的预测效能。
材料与方法
一、数据集
从GEO数据库下载ICC单细胞数据集(GSE151530),该数据集包含12例病人的14个肿瘤样本,共计6 698个细胞。从TCGA数据库下载ICC包含38个肿瘤样本的RNA高通量测序数据集。从既往公开研究中[9]获取ICC测序数据集,该数据集包含集255例ICC病人临床信息和转录组数据。
二、预后相关细胞及对应DEGs鉴定
通过细胞过滤、归一化、标准化以及降维聚类和注释等方法处理单细胞数据集[10],明确肿瘤微环境细胞,应用Scissor包[6]整合单细胞及组织转录组学数据,并筛选与样本存活表型相关的细胞,采用Seurat包[11]中的FindMarkers功能按照|lg2(差异倍数)|>1.5筛选DEGs。
三、DEGs功能富集分析及蛋白互作网络构建
运用Cluster profiler[12]和ReactomePA[13]进行MSigDB、KEGG和Reactome通路富集分析,进一步使用基因相互作用检索工具(search tool for the retrieval of interacting genes,STRING)在线生物数据库[14]构建DEGs的PPI网络,置信度评分设置为≥0.9。然后,通过Cytoscape软件可视化PPI网络[15],并通过应用分子复合物检测插件(MCODE)筛选PPI网络中的重要模块。此外,通过clusterprofiler包对前4个模块中的DEGs进行KEGG通路富集分析。
四、肿瘤标本TIDE评分及基因相关性分析
TIDE是一个根据转录组学数据评价免疫治疗抵抗的在线算法工具[7-8],本研究使用TIDE算法计算了TCGA数据集中组织样本的免疫治疗响应性评分,并分析DEGs与免疫治疗响应评分的相关性。
五、多因素Cox回归分析和预后预测模型构建
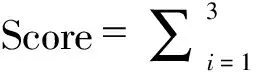
使用surviveminer包中的“surv_cutpoint”功能得到最佳截断值,并根据该截断值将病人分为高、低表达组,同时使用TCGA和外部数据集进行验证。
六、统计学方法
本研究数据应用R(4.2.2版)软件进行分析,DEGs采用t检验,DEGs与免疫治疗响应评分相关性采用Pearson相关性检验,生存资料采用log-rank检验,P<0.05为差异具有统计学意义。
结 果
一、肿瘤相关巨噬细胞和T细胞与ICC预后不良相关
来源于GEO数据库的单细胞数据集(GSE151530)通过降维注释可分为5类细胞亚群,包括肿瘤相关巨噬细胞(tumor associated macrophages,TAMs)、T细胞、B细胞、肿瘤相关成纤维细胞(cancer associated fibroblast,CAFs)和肿瘤相关内皮细胞(tumor-derived endothelial cells,TECs)(图1A)。随后,利用Scissor算法整合该单细胞数据集和TCGA的ICC组织转录组以及临床预后信息,分析并筛选出604个与肿瘤不良预后相关的细胞(图1B)。在各细胞亚群进行统计后发现其中有311个为TAMs,180个为T细胞,85个为B细胞,22个为CAFs,6个为TECs(图1C),它们分别占各自细胞亚群的56.2%、8.1%、19.4%、5.5%和2.0%。

注:UMAP.统一流形逼近与投影;CAFs.肿瘤相关成纤维细胞;TAMs.肿瘤相关巨噬细胞;TECs.肿瘤相关内皮细胞。
二、预后相关DEGs高度富集于程序性死亡受体1(programmed death1,PD-1)等信号通路
运用FindMarkers方法在单细胞数据集中鉴定得到366个DEGs,其中281表达上调,85个表达下调(图2A)。进一步富集分析发现上调基因富集于PD-1信号通路(P=1.76×10E-6)、白细胞介素10(IL-10)信号通路(P=3.51×10E-6),见图2B。部分DEGs在预后相关细胞亚群中的分布见图2C。
注:PD-1.程序性死亡受体1;IL.白细胞介素;NF-κB.核转录因子-κB;TNF-α.肿瘤坏死因子-α。
三、模块基因与免疫代谢通路高度关联
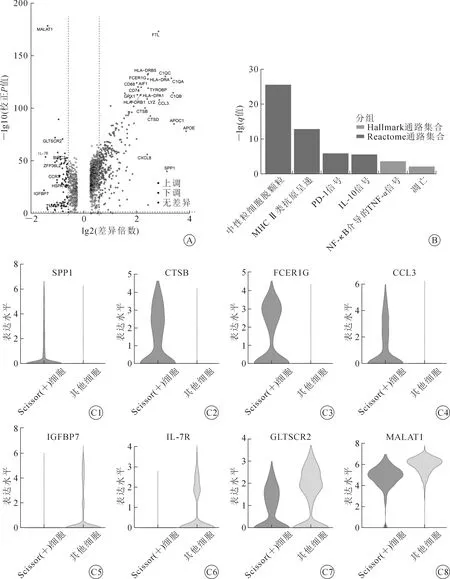
应用在线数据库STRING按置信度评分>0.9构建上述DEGs的PPI网络,并通过Cytoscape的MCODE插件(参数设置degree cut-off=2,node score cut-off=0.2,k-core=2,max depth=100)筛选得到4个基因模块(图3A~D),共囊括了53个模块基因。KEGG通路分析显示模块基因与吞噬小体(P=6.74×10E-13)、辅助T细胞分化(P=9.81×10E-9)、细胞因子互作(P=1.11×10E-4)以及碳代谢(P=8.98×10E-7)等免疫代谢通路显著相关(图3E)。
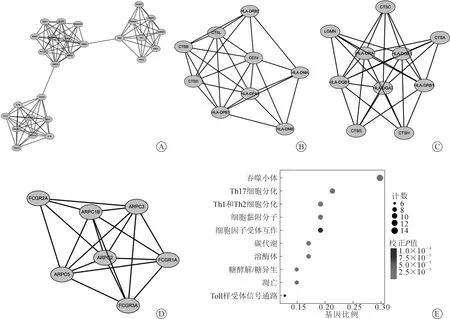
图3 差异表达基因(DEGs)的蛋白质相互作用网络和通路富集分析 A~D.DEGs的基因模块;E.模块基因的富集通路
四、运用TIDE算法得到免疫相关的18个关键基因
运用TIDE算法得到TCGA的ICC病人的免疫响应性评分,将上述模块内的基因在TCGA中的表达量分别与TIDE进行相关性分析,发现CTSD与TIDE评分存在负相关(r=-0.386,P<0.05);FCGR2A、FCGR1A、ARPC5、CTSC、ARPC1B、KIAA0101、IL-18、BIRC5、TOP2A、CDK1、MKI67、ALDOA、PTTG1、ARPC2、NUASP1、UBE2C、CENPF等17个基因与TIDE评分存在正相关(均P<0.05,r值见图4)。
图4 筛选得到的关键基因与肿瘤免疫功能障碍和排斥(TIDE)评分的相关性
五、基于CDK1、CTSD和FCGR2A构建的模型可有效预测ICC预后
通过多因素Cox回归分析,从上述18个关键基因中筛选得到3个枢纽基因CDK1、CTSD、FCGR2A。其中CTSD、FCGR2A主要分布在TAMs,CDK1主要分布在T细胞(图5A~C)。根据回归系数和基因的表达量构建如下模型:Score=0.517×expression ofCTSD+0.006×expression ofFCGR2A+0.121×expression ofCDK1,并计算得到TCGA病人评分,随后根据surminer包计算得到最佳截断值,将病人按模型评分分为高、低组,本研究发现评分高的病人预后较差(P=0.016),同样在外部数据集中进一步验证也得到了相同的结果(P<0.000 1)。计算该模型预测效能,发现在TCGA数据集中,1年生存曲线下面积(area under the curve,AUC)为0.672,2年生存AUC为0.692,3年生存AUC为0.742。在外部数据集中,1年生存AUC为0.584,2年生存AUC为0.651,3年生存AUC为0.668(图5D~G)。
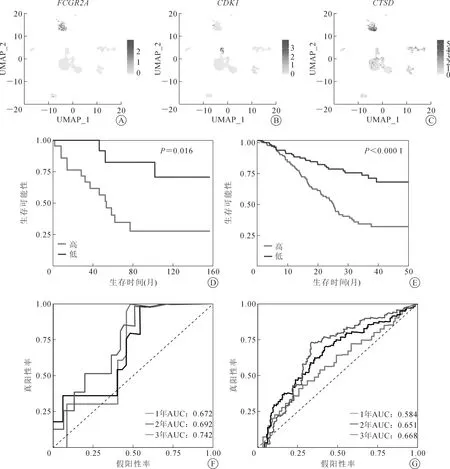
注:UMAP.统一流形逼近与投影;AUC.曲线下面积。
讨 论
ICC是一种高度异质性的恶性肿瘤,由于其在早期缺乏特异临床表现,大多数病人在出现症状时已处于肿瘤终末阶段,失去了手术切除的机会。除此之外,只有肿瘤长径小于2 cm的极早期ICC才能从肝移植中受益[16]。虽然靶向治疗联合化疗被认为是终末期胆道肿瘤的有效治疗手段[17],但对病人的预后改善仍不明显。而近年来,众多学者对肿瘤微环境进行了广泛研究,发现肿瘤微环境与肿瘤病人预后以及肿瘤病人的治疗抵抗性高度相关[18-19]。目前针对ICC的预后模型较为多样,但仍然缺乏根据肿瘤微环境单细胞构建的与免疫治疗相关的预后预测模型。本研究应用R软件分析GEO数据库的ICC单细胞数据集以及TCGA的组织转录组数据集,鉴定在肿瘤微环境中与病人不良预后相关的细胞,进而分析得到281个上调基因和85个下调基因,其中有3个基因被最终认定为在肿瘤微环境中与ICC不良预后相关的枢纽基因。
本研究发现,在肿瘤微环境中与ICC不良预后相关的细胞主要为TAMs和T细胞(图1C)。已有文献报道,表达CD86、CD206、SIRPα或PD-1的TAMs浸润程度能够独立预测ICC病人的生存和复发预后[20-22]。本研究在肿瘤微环境中鉴定到与预后不良相关的细胞并得到DEGs,同时进行了富集分析,发现上调的DEGs生物学功能主要富集在中性粒细胞脱颗粒、抗原呈递、PD-1信号通路、IL-10信号通路以及肿瘤坏死因子(TNF)和凋亡相关通路。有研究表明,TAMs可以联合肿瘤相关中性粒细胞激活ICC细胞中的STAT3信号通路,促进肿瘤进展[23]。程序性死亡受体配体1(programmed death-ligand 1,PD-L1)阳性的TAMs可以被IL-10信号通路激活,从而通过PD-1/PD-L1信号通路抑制T细胞免疫反应[24]。然而,也有研究人员证实当缺乏磷脂酰肌醇3-激酶γ (phosphatidylinositol 3-kinase,PI3Kγ)激活时,TAMs可以通过上调MHC Ⅱ和促炎因子同时降低抑炎因子如IL-10的分泌来改善免疫响应[25]。这提示TAMs存在着异质性,与本研究中56.2%的TAMs与不良预后相关的结果相互印证。TAMs也可通过产生TNF-α来减少肿瘤细胞的凋亡从而促进肿瘤进展[26]。这群肿瘤细胞的凋亡通路的增强也提示,TAMs也可能存在与肿瘤细胞类似的“凋亡悖论”情况[27]。
通过将DEGs中的模块基因与TIDE评分进行相关性分析以及多因素Cox回归分析筛选得到的3个关键基因当中,CDK1是一个丝氨酸/苏氨酸的蛋白激酶,在物种中高度保守,在细胞周期中起着重要作用。联合共济失调毛细血管扩张突变基因和Rad3相关蛋白(ATR)抑制剂和抗PD-L1制剂可以通过激活CDK1-SPOP信号通路来提高前列腺癌的免疫应答[28]。CTSD,即组织蛋白酶D,是一种溶酶体天冬氨酰蛋白酶。在三阴性乳腺癌当中,针对CTSD靶点的免疫治疗已经在人源性组织异种移植模型中表现出了抗肿瘤效果[29]。同时在乳腺癌中,CTSD也被鉴定为抗转移治疗的潜在靶点[30]。FCGR2A所编码的蛋白是一种存在于巨噬细胞等吞噬细胞表面的受体分子,已有相关的临床证据表明,在中国人群中病人FCGR2A的表达与胃癌的发病率存在相关性[31]。抗原呈递是抗肿瘤免疫发生和肿瘤免疫逃逸的关键机制之一[32],FCGR2A介导免疫复合物吞噬和清除是抗原呈递的起始步骤,已有报道表明FCGR2A也与肿瘤中的免疫浸润相关[33]。以上基于其他肿瘤类型的泛癌分析结果在ICC中也可能发挥相同的作用。因此,通过在ICC病人中这3个基因的表达情况对肿瘤病人进行筛选,可能增加免疫治疗的受益人群。最后,在本研究中,通过生存分析验证了由3个关键基因构建得到的预后预测模型对病人生存预后的预测性能,然而仍需要较大的队列对该模型的效能进行确认。
综上所述,通过单细胞数据和组织转录组数据进行生物信息学分析,本研究鉴定了3个可能在ICC肿瘤微环境中以及免疫治疗至关重要的基因和重要信号通路,同时根据这些基因构建了一个ICC病人的预后预测模型。该模型的建立可能为今后在ICC病人中筛选免疫治疗受益人群提供参考价值。
利益冲突所有作者均声明不存在利益冲突

