MALDI-TOF质谱技术对猪肠道菌群中携带mcr-1细菌的鉴定与聚类分型
高 芸,轩慧勇,姚晓慧,魏建超,刘 珂,邵东华,邱亚峰,马志永,李蓓蓓,夏利宁
(1.新疆农业大学动物医学学院,乌鲁木齐 830052;2.中国农业科学院上海兽医研究所,上海 200241)
随着抗生素的广泛应用,细菌耐药已经成为21世纪人类健康的最大威胁之一[1]。动物肠道菌群长期经受高水平的抗生素选择性压力,是耐药性发生发展的重要场所和耐药基因的贮藏库,一些严重危害人类健康的耐药基因,如质粒介导的多粘菌素耐药基因mcr-1[2]、替加环素耐药基因tet(X3/4)[3]以及利奈唑胺耐药基因optrA[4],在养殖动物肠道来源的细菌中不断被发现和报道。然而,对于特定耐药基因在动物肠道菌群中的发生发展和传播扩散机制却知之甚少。
细菌种属鉴定以及亲缘关系分析是耐药流行病学研究的基础。目前细菌的鉴定方法主要有生化反应试验、16S rRNA测序比对和细菌特异基因检测等,菌株同源性分析则多采用脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)、随机多态性DNA(random amplified polymorphic DNA,RAPD)、多位点序列分型(multilocus sequence typing,MLST)、全基因组测序(whole genome sequencing,WGS)等分子生物学方法[5]。上述细菌鉴定及分子分型方法存在步骤繁琐,操作过程耗时较长或仪器昂贵等缺点。
近年来发展起来的基质辅助激光解析飞行质谱(matrix assisted laser desorption/ionization-time of flight mass spectrometry,MALDI-TOF MS)技术,通过检测菌体蛋白质指纹图谱实现对不同细菌属、种的区分和鉴定[6]。与其他传统的分子生物学技术相比,它的操作方法更简单和快速。目前MALDITOF广泛用于细菌鉴定,而基于MALDI-TOF的分型方法也在快速发展当中[7]。如Egli等[8]通过MALDITOF MS方法对产超广谱β-内酰胺酶的大肠杆菌进行分型,结果显示,使用MALDI-TOF MS的聚类树状图与PFGE的分析非常相似。Ueda等[9]通过MALDITOF MS方法对24株临床分离的耐甲氧西林金黄色葡萄球菌质谱分析显示,蛋白质指纹图谱成功用于分型,且分型结果与PFGE结果无差异。
本研究以猪肠道菌群为研究对象,以多粘菌素耐药基因mcr-1为目的基因,获取猪肠道菌群的mcr-1阳性菌株库,在此基础上,应用MALDI-TOF MS技术,对携带mcr-1菌株进行快速鉴定和亲缘关系分析,为解析mcr-1基因在肠道菌群中的分布扩散特征和遏制耐药菌的传播提供科学依据和技术支撑。
1 材料和方法
1.1 样品来源与细菌分离 2019年1月,从上海某屠宰场采集一头肥猪的新鲜肠道内容物约1 g,将其置于冰盒内低温运输至实验室。将上述粪便样本稀释至合适浓度,直接涂布于含有多粘菌素E(3 mg/L)+利奈唑胺(20 mg/L)+万古霉素(20 mg/L)药物的TSA固体培养基上,倒置于37℃恒温培养箱中培养48 h。挑取培养皿上所有单菌落,经二次纯化和细菌增殖后,制成20%的甘油菌进行保种,于-80℃冻存备用。
1.2 主要试剂 胰蛋白胨大豆琼脂(Tryptone Soya Agar,TSA)、胰蛋白胨大豆肉汤(Tryptone soya broth,TSB)购自上海少辛生物科技有限公司;2×TaqPCR MasterMix、琼脂糖、50× TAE和核酸染料均购自上海翊圣生物科技有限公司;α-氰基-4-羟基肉桂酸基质液(CHCA)购自西格玛奥德里奇(上海)贸易有限公司;多粘菌素E、万古霉素、利奈唑胺均购自上海源叶生物科技有限公司。
1.3 DNA模板的制备 将多粘菌素耐药菌株划线接种于TSA固体培养基上,37℃培养16~24 h,刮取菌苔于50 μL TE中,100℃煮沸10 min,冰浴5 min,12 000 ×g离心10 min,取上清液作DNA模板,-20℃保存备用。
1.4 耐药基因mcr-1的检测 根据相关参考文献[10]由上海派森诺生物科技股份有限公司合成mcr-1耐药基因引物,引物序列为F:5'-CGGTC AGTCCGTTTGTTC-3';R:5'-CTTGGTCGGTC TGTAGGG-3'。反应体系(20 μL)为:2×TaqMaster Mix 10 μL,上、下游引物各0.5 μL,DNA模板0.5 μL,ddH2O 8.5 μL。扩增条件为:95℃预变性5 min;95℃变性45 s,55℃退火45 s,72℃延伸1 min,共30个循环;72℃再延伸10 min。PCR扩增产物进行1%琼脂糖凝胶电泳。
1.5 菌株鉴定及分型
1.5.1 上样前准备 将待测菌株接种于5%血平板,经37℃过夜培养。用无菌接种环挑取单个菌落均匀涂抹于靶板的样本孔,滴加1 μL CHAC基质液覆盖菌苔,质控大肠杆菌ATCC8739涂于靶板的质控孔,自然晾干后上机检测。仪器参数为:激光强度47 Hz,质量范围m/z:2~20 kDa,有效检测范围2~15 kDa,线性操作模式,激光点击数为100。
1.5.2 MALDI-TOF MS菌株鉴定 在IVD模式下进行目标菌株数据采集,检测结束后,通过Myla软件查看鉴定结果。鉴定结果可信度判定标准为:>99.00%表示鉴定结果极可靠,>90.00%表示鉴定结果可靠,80.00%~90.00%为鉴定结果较可靠,有待进一步判别,其中<80%的认为鉴定结果极不可靠。
1.5.3 MALDI-TOF MS聚类分析 在RUO模式下进行目标菌株数据采集,通过SARAMIS Target Manager软件,在Results界面下选择需要进行同源性分析的菌株,将蛋白指纹图谱导入SARAMIS分类树,用SARAMIS软件自带的TAXMONY功能模块进行聚类分析。菌株亲缘关系分析参照PFGE标准,以相似性≥80%作为同一克隆的划分依据[11]。
2 结果
2.1mcr-1阳性菌株分离鉴定结果 本研究以同一猪肠道内容物样本为研究对象,利用添加抗生素平板进行多粘菌素耐药菌株的筛选和收集,共计获得325株细菌。利用VITEK MS微生物检测系统对上述325株细菌进行种属鉴定。鉴定结果显示,325株细菌包括70株大肠杆菌、19株肺炎克雷伯菌、47株放线杆菌、122株产气巴斯德菌和67株奇异变形杆菌(表1)。据文献报道,奇异变形杆菌对多粘菌素为天然耐药[12],因此本试验分离的67株奇异变形杆菌未列入后续研究中。对上述除奇异变形杆菌外的其他菌株利用PCR进行mcr-1基因的筛查,共得到136株mcr-1阳性菌株,包括大肠杆菌70株、肺炎克雷伯菌19株和放线杆菌47株(图1)。此外,122株多粘菌素耐药产气巴斯德菌均为mcr-1阴性,对其进行了其余mcr基因的检测[13],结果也均为阴性,提示可能存在其他对多粘菌素耐药的机制。以上结果表明,多粘菌素耐药基因mcr-1在动物肠道菌群中存在种属水平的广泛分布。
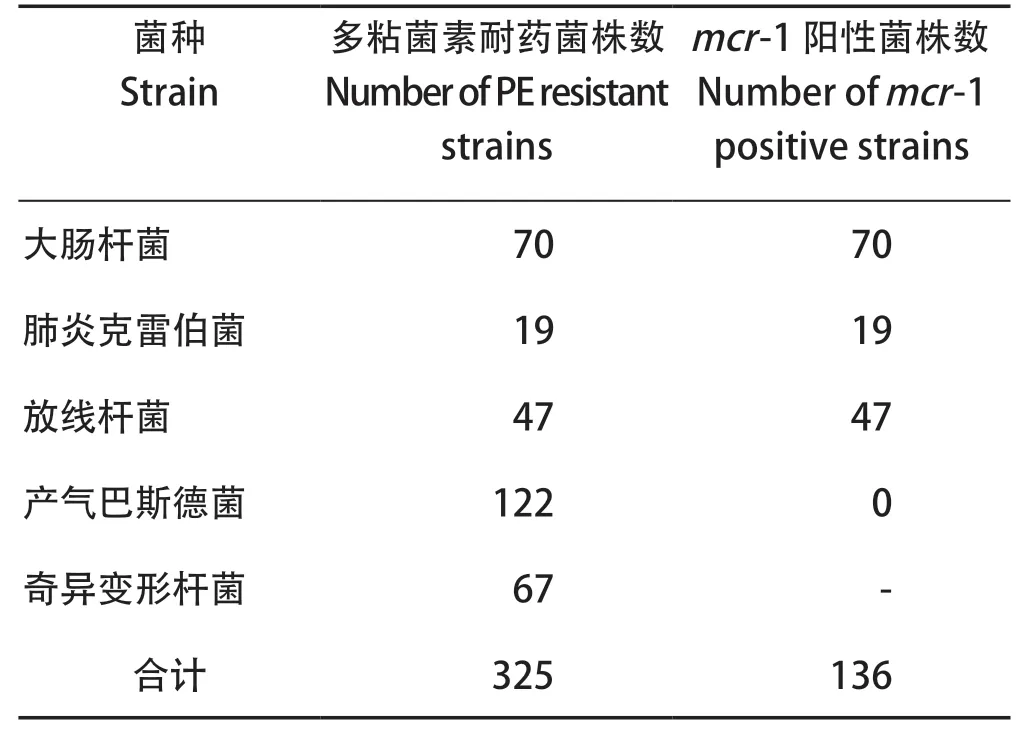
表1 细菌分离鉴定结果Table 1 Bacteria isolation and identification results
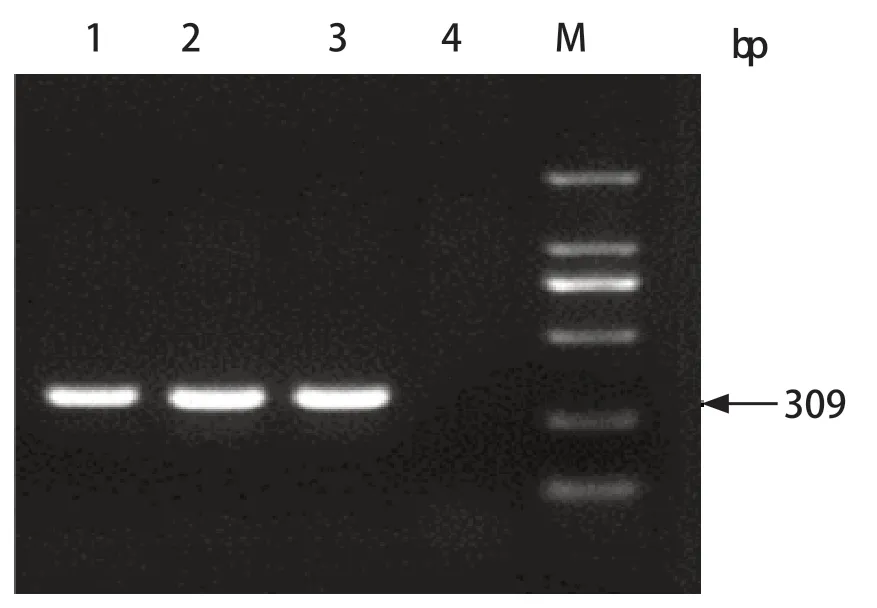
图1 部分mcr-1阳性菌株基因检测电泳图Fig.1 Electrophoresis of PCR detection of mcr-1 gene
2.2 MALDI-TOF MS对mcr-1阳性菌株的分型
2.2.1mcr-1阳性大肠杆菌的分型 在RUO模式下,对70株mcr-1阳性大肠杆菌的蛋白指纹图谱进行聚类分析(图2),结果显示,70株大肠杆菌相似度为65.0%~95.0%,可分为a~m共13个亚型。a亚型克隆群菌株数量最多,包含40株菌(占57.1%),是mcr-1携带大肠杆菌的优势克隆菌株。而l型和m型菌株数最少,各为1株,其他亚型包含的菌株数为3~4株。丰富的克隆群提示mcr-1基因在猪肠道大肠杆菌菌群中同时存在克隆传播和水平转移。
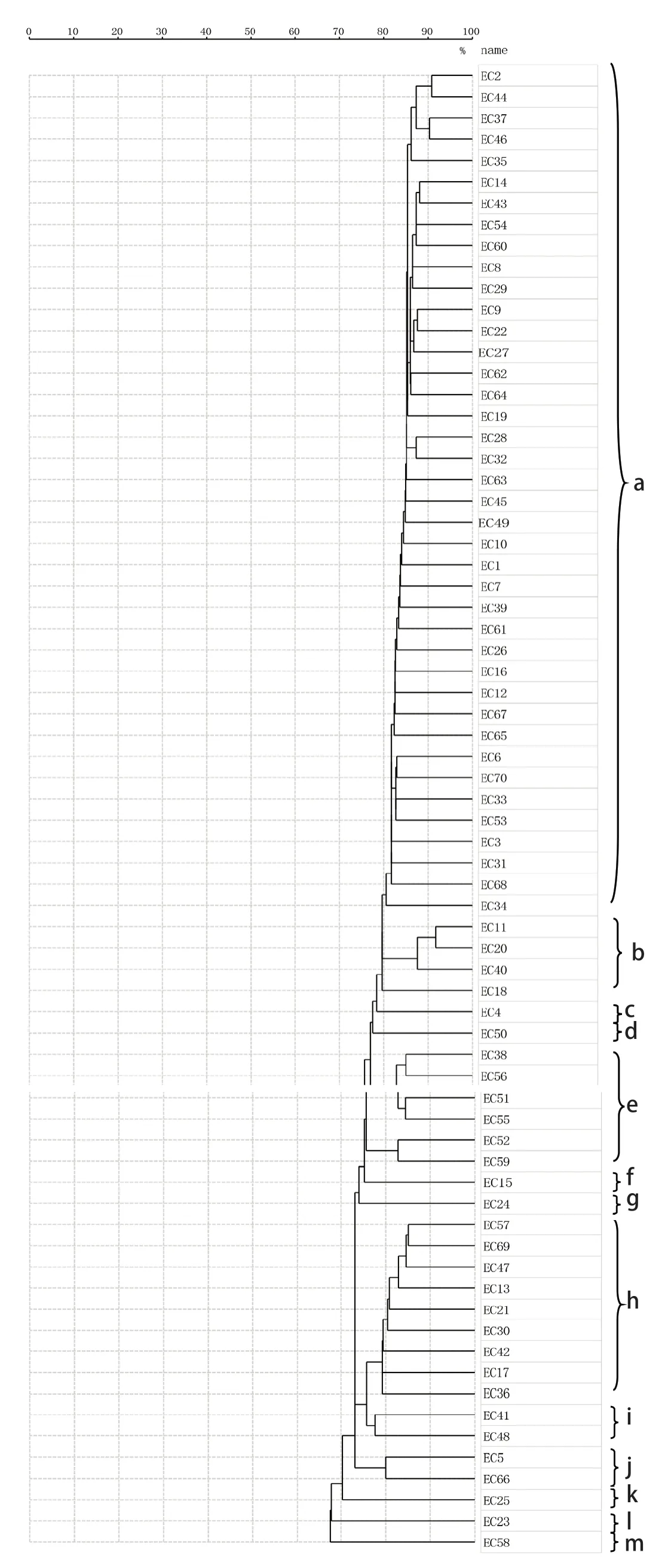
图2 70株大肠杆菌的MALDI-TOF-MS分型聚类图Fig.2 Dendrogram based on MALDI-TOF-MS of the 70 mcr-1-positive E.coli isolates
2.2.2mcr-1阳性肺炎克雷伯菌的分型 运用RUO模式,对19株肺炎克雷伯菌的蛋白指纹图谱进行同源性分析(图3),结果显示,19株肺炎克雷伯菌相似度为70.0%~90.0%,可分为a、b、c 3个亚型,其中有17株肺炎克雷伯菌属于a亚型,占比89.5%。另外两个亚型均只包含一个菌株。上述结果提示,mcr-1在猪肠道中肺炎克雷伯氏菌中以克隆传播为主。
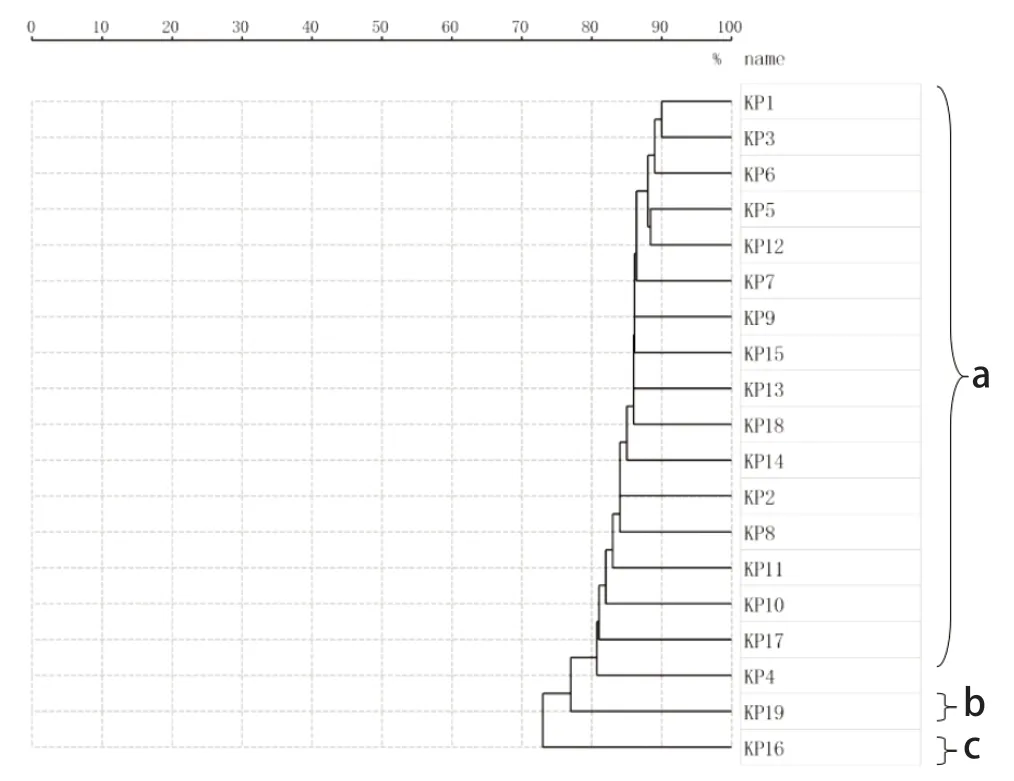
图3 19株肺炎克雷伯菌的MALDI-TOF-MS分型聚类图Fig.3 Dendrogram based on MALDI-TOF-MS of the 19 mcr-1-positive K.pneumoniae isolates
2.2.3mcr-1阳性放线杆菌的分型 在RUO模式下,对47株放线杆菌的蛋白指纹图谱进行聚类分析(图4)。结果显示,47株放线杆菌的蛋白指纹图谱的相似度均在80%以上,表明这些菌株均为同一克隆。
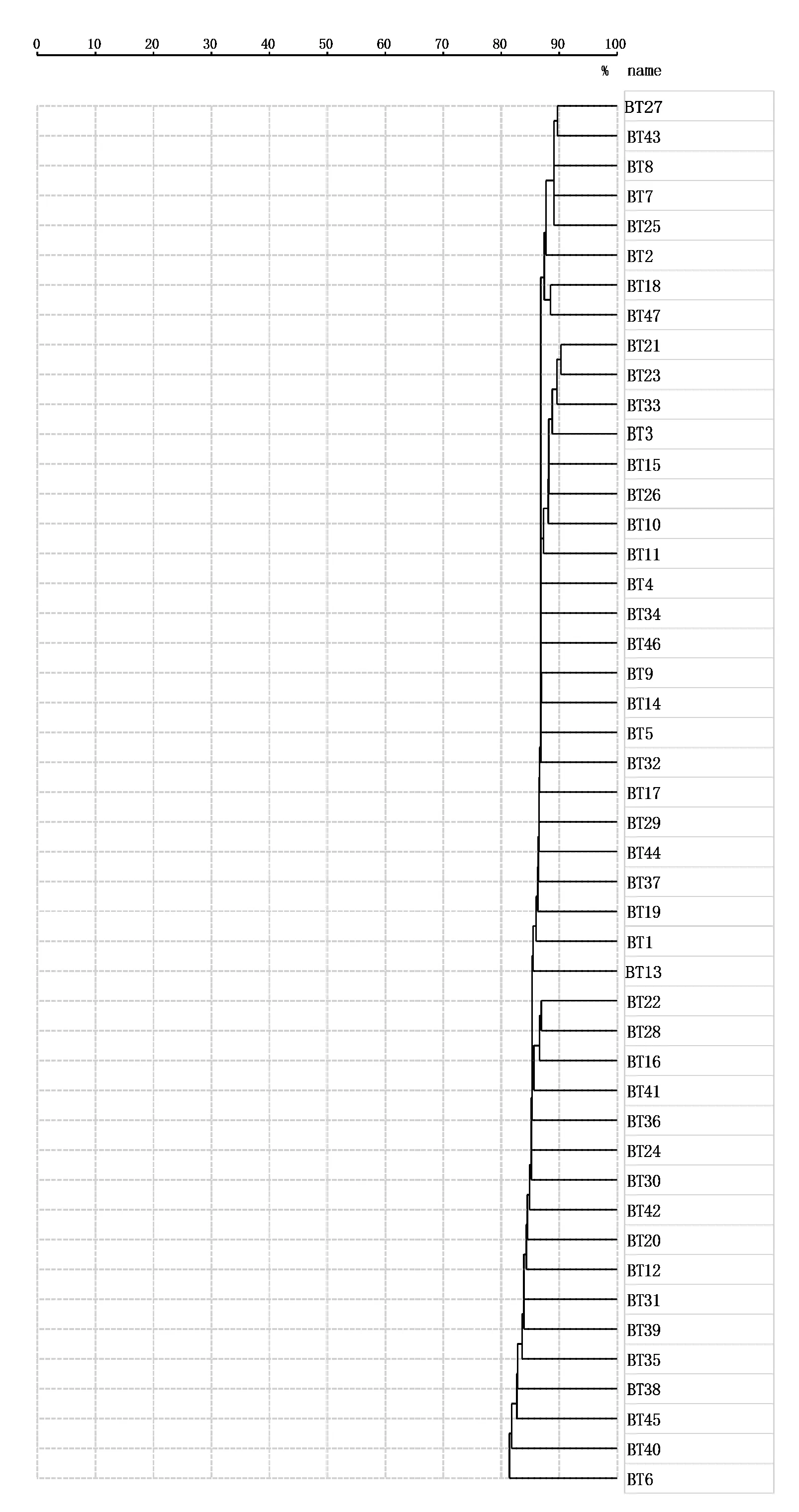
图4 47株Actinobacillus sp.的MALDI-TOF-MS分型聚类图Fig.4 Dendrogram based on MALDI-TOF-MS dendrogram of the 47 mcr-1-positive Actinobacillus sp.isolates
3 讨论
多粘菌素被认为是治疗多重耐药肠杆菌以及其他革兰氏阴性菌感染的“最后一道防线”[14],质粒介导的可水平转移的多粘菌素耐药基因mcr-1的发现严重威胁该药的临床应用。mcr-1在养殖动物源细菌中有极高的流行率。一项涵盖1611株禽源大肠杆菌的研究表明,80年代之前几乎不存在mcr-1基因,该基因的阳性率从2009年的5.2%上升至2014年的30%[15]。仝慧娴等[14]调查了2016—2017年我国18省市猪源粪便中mcr-1的携带率,结果表明,我国猪粪便中mcr-1的携带率高达76.2%。肠道菌群存在细菌种类多、数量大和密度高的特点,为耐药基因的转移和扩散创造便利的环境条件,是耐药菌生存和繁殖的理想场所。探明特定耐药基因在肠道菌群中的分布模式和传播扩散机制对于理解耐药性的发生发展具有重要意义。
本研究以同一猪肠道样品为研究对象,从中共获得136株mcr-1阳性菌株,包括70株大肠杆菌、19株肺炎克雷伯菌和47株放线杆菌。研究结果显示,多粘菌素耐药菌中mcr-1携带率高,且可在不同种属的细菌中分布。在本研究的mcr-1阳性菌株中,大肠杆菌的占比达到51.5%(70/136)。Chen等[16]分离鉴定出的54株猪源mcr-1阳性菌株,均属于大肠杆菌;黄腾[17]报道的735株猪源粘菌素耐药大肠杆菌中,有610株检测到mcr-1基因,检出率高达83.0%。上述结果均说明大肠杆菌是mcr-1基因的主要宿主。此外,本研究还检测到47株携带mcr-1基因的放线杆菌属细菌,这些菌株的蛋白指纹图谱一致,说明它们是同一种属的细菌。本实验室的另一项研究对其中一株菌进行全基因组测序[18],发现它是一种携带mcr-1基因的新型放线杆菌。进一步对该菌中mcr-1基因环境进行分析发现,该基因位于一个新的整合性接合元件(ICEAsp1)上,mcr-1与其他9个耐药基因共同组成了一个多重耐药基因簇。对mcr-1基因环境深入分析发现,其由一个复合转座子Tn6330(ISApl1-mcr-1-pap2-ISApl1)携带,这种结构增加了mcr-1的可转移性,有利于其在不同种属的细菌间进行水平传播。
本研究MALDI-TOF MS聚类分析显示,大肠杆菌被分为13个亚型,肺炎克雷伯菌分为3个亚型,而放线杆菌均为同一克隆,同一种属细菌分型的多样性显示出耐药基因mcr-1在同一动物肠道菌群中既存在水平传播也存在克隆传播。目前,MALDI-TOF技术已被应用于多种常见细菌的分型检测中,如赵贵明等[19]利用MALDI-TOF MS对克罗诺杆菌进行分型,在RUO模式下32株克罗诺杆菌可分为a~f共6种亚型,该方法丰富了现有的克罗诺杆菌分型方法;宗凯等[20]应用MALDI-TOF MS对采集的44株芽孢杆菌进行蛋白指纹图谱及亲缘关系分析,建立了蜂蜜中芽孢杆菌溯源的方法。目前PFGE是细菌分子分型的金标准[21],通过获得菌株的DNA指纹图谱进行同源性分析,具有很高的辨析度,但该方法不能对细菌进行种属鉴定,同时存在耗时长、仪器昂贵和成本高等缺点。而MALDI-TOF-MS通过收集蛋白质指纹图谱,可以对细菌进行初步的分型研究,只需要简单的样品制备,就可以在短时间内获得结果。目前已有多项研究通过该方法对常见细菌进行分型研究,也表明该方法具有较好的可靠性[7]。如Meng等[22]应用MALDI-TOF MS对碳青霉烯类耐药肺炎克雷伯菌和黏质沙雷菌进行分型,分型结果与MLST和全基因组测序具有较好的一致性;王耀等[23]应用MALDI-TOF MS方法建立单增李斯特氏菌数据库,通过对数据库中该菌的主要图谱进行比对,构建系统进化树,进而将37株单增李斯特菌分离株划分为9个亚型,以此建立MALDI-TOF MS技术对单增李斯特菌的分型。
综上,本研究利用MALDI-TOF MS技术对同一猪肠道菌群中分离的325株多粘菌素耐药菌株进行鉴定,并对136株mcr-1阳性菌株进行同源性分析,结果表明mcr-1在猪肠道菌群中既存在水平传播也存在克隆传播。基于MALDI-TOF MS的分型方法具有快速、高通量和分析成本低的优势,可作为现有分子分型方法的补充,在细菌溯源和亲缘关系分析中可起到重要作用。

