锦鲤疱疹病毒胸苷激酶基因的生物信息学分析
陈 莉,周广彪,郑耿东,温尔英,陈文婉,吴松浩,利光辉*
(1.汕头海关技术中心,广东汕头 515041;2.汕头海关技术中心/汕头大学理学院水产品联合实验室,广东汕头 515041;3.汕头海关动植处,广东汕头 515057)
锦鲤疱疹病毒(Koi Herpesvirus,KHV)又称鲤鱼间质性肾炎及鳃坏死性病毒(carp interstitial nephritis and necrosis virus,CNGV),被归类为疱疹病毒目(Herpesvirales)疱疹病毒科(Herpesviridae)鲤疱疹病毒属(CyprinidHerpesviridae),该病原被我国列入动物疫病病种名录中的二类疾病,世界动物卫生组织(WOAH)也将其列为必须申报的动物疫病之一[1-4]。锦鲤疱疹病毒是一种dsDNA疱疹病毒的病原体,它是由31种病毒粒子多肽和8种糖基化蛋白组成,成熟的病毒粒子包含一个松散的包膜,病毒粒子总直径为170~230 nm,具有一个线性双链DNA基因组[5-8]。Aoki等[9]描述了锦鲤疱疹病毒的全基因组序列,并鉴定了156个独特的蛋白质编码基因,为后续科学研究奠定基础。鱼类感染锦鲤疱疹病毒,最明显的特征之一是鳃弓内血管充血,鳃耙变细,其次该病易发生在肾,肾小管周围出现炎性浸润,并伴随血管充血;发病中后期,病鱼行动缓慢,鱼眼严重凹陷,一般情况下,锦鲤患病死亡率高达90%~100%,因此应引起各国高度重视[10-14]。有研究表明,疱疹病毒增殖的主要毒力基因是胸苷激酶基因[15],胸苷激酶基因编码胸苷激酶,该酶可将核苷类似物无毒性抗病毒物丙氧鸟苷磷酸化为一磷酸化形式,继而在细胞的一磷酸鸟苷激酶或细胞内其他激酶的作用下形成二磷酸化产物和三磷酸化产物,三磷酸化产物能整合到细胞DNA上,抑制DNA聚合酶的活性,从而抑制蛋白的合成,阻断DNA的合成,使分裂细胞被杀伤。锦鲤疱疹病毒中ORF140基因编码胸苷激酶,是致病基因中的一种,在病毒吸附、穿入、复制合成及细胞中发挥重要作用[16]。
生物信息学(Bioinformatics)是随着人类基因组计划发展而不断发展的一门联合计算机和信息科学中的技术、方法[17-18]。生物信息学在分子生物学领域取得重大进步,加上基因组技术的进步,突显生物信息学的重要性。随着一代测序、二代测序及三代测序的快速发展,测序成本不断降低,促使更全面、更深入地对基因组进行分析。目前基于生物信息学的方法多种多样,笔者通过相关软件对锦鲤疱疹病毒胸苷激酶基因进行生物信息学分析,以期为锦鲤疱疹病毒的分子生物学研究提供方向,为下一步研究的开展奠定基础。
1 材料与方法
1.1 序列信息锦鲤疱疹病毒(KHV)登录号:DQ177346.1,可由登录号在NCBI中查阅完整基因序列。胸苷激酶基因(登录号:AB375391)是编码其中一段以ATG为起始密码子,在NCBI(https://www.ncbi.nlm.nih.gov/nuccore/AB375391)中可以查看TK基因的完整基因序列。
1.2 方法
1.2.1BioXM基因序列的组成分析。BioXM是进行DNA序列的常规分析,包括ORF查找、序列格式化、翻译、限制酶切位点分析等功能,通过对基因组分析,确定序列的基本信息。利用NCBI数据网站获得TK基因的序列,以FASTA格式保存至相关文件夹。将TK基因序列运行至BioXM软件中,获得核酸序列的组成分析。
1.2.2TK蛋白质理化性质分析、亲/疏水性、信号肽及跨膜区预测。根据TK基因登录号,从NCBI中获得的TK基因序列,利用NCBI ORF Finder软件寻找序列中潜在开放阅读框(open reading fraction,ORF),并获得TK基因的氨基酸序列。通过获取的氨基酸序列和在线软件Expasy进行蛋白质的理化性质分析,将获得蛋白质的一般信息;使用在线分析软件ProtScale(http://web.expasy.org/cgi-bin/protparam)预测TK蛋白的亲水性和疏水性;利用在线分析软件TMHMM server(http://www.cbs.dtu.dk/services/TMHMM)和SignalP sever(http://www.cbs.dtu.dk/services/signalP)预测TK蛋白的信号肽和跨膜结构,获得相关数据。
1.2.3TK蛋白结构域分析。利用在线软件NCBI-CDD分析TK蛋白的结构域,并结合HMMER和SMART对TK蛋白同时进行预测。
1.2.4TK蛋白的二级结构和三级结构预测。利用SOPMA软件对已获得的氨基酸序列进行蛋白质二级结构预测的综合分析。根据SWISS-MODEL软件进行蛋白质三级结构预测,为丰富TK基因的蛋白数据提供支持。
1.2.5利用MEGA构建蛋白系统发育树。从数据库中获得不同病毒中TK蛋白的氨基酸系列,利用MEGA6软件的NJ法构建蛋白质系统进化树,分析亲缘关系。
2 结果与分析
2.1 TK基因序列的组成根据TK基因登录号,从NCBI数据网站获得TK基因序列,将序列输入BioXM软件,可知TK基因序列长度224 bp,其中腺嘌呤核苷酸(A)共24个,占10.71%;鸟嘌呤核苷酸(G)共15个,占总核苷酸序列6.70%;胞嘧啶核苷酸(C)共8个,占总核苷酸序列3.57%;胸腺嘧啶核苷酸(T)共9个,占总核苷酸序列4.02%;其中腺嘌呤核苷酸和胸腺嘧啶占14.73%,较鸟嘌呤核苷酸和胞嘧啶核苷酸(10.27%)少4.46%,TK基因分子量为17 378 Da。
2.2 TK蛋白质理化性质预测分析TK基因共编码224个氨基酸,编码蛋白质的分子质量为24 623.70 Da,蛋白质的等电点(pI)值为6.31,其中氨基酸的组成见表1。该蛋白中含量前4的为丙氨酸、缬氨酸、亮氨酸和脯氨酸,占比分别为10.7%、9.4%、8.0%和8.0%。酸性氨基酸总数为(Asp+Glu)23,碱性氨基酸总数为(Arg+Lys)22,分子式为C1096H1733N289O316S19,原子总数为3 453。由TK基因编码的蛋白质不含任何色氨酸残基,表明这可能导致计算的消光系数约有10%以上的误差。不稳定系数为44.11,其中不稳定系数大于40,则表示该蛋白不稳定;脂肪指数为84.91,总平均亲水性为0.030。通过ProtScale软件分析结果可知,纵坐标越大,蛋白疏水性就越强。如图1所示,在氨基酸序列第45个位点,亲水性最高;在第5个位点,TK蛋白的疏水性得分最高,综合理化性质分析,说明该蛋白为亲水蛋白。SignaIP是一个信号肽预测服务器,它的功能是预测给定的氨基酸序列中是否存在潜在的信号肽剪切位点及其所在位置。如图2所示,每个氨基酸对应1个S值,信号肽区域的S值较高;同时每个氨基酸有1个C值,在剪切位点的C值是最高的。综合考虑S值、C值、Y值,该蛋白不含有信号肽。蛋白结构决定蛋白功能,利用生物学软件工具TMHMM Server来预测蛋白质跨膜螺旋(图3)。由TMHMM分析结果可知,TK蛋白长度为224,该蛋白不存在跨膜螺旋,同时跨膜螺旋氨基酸残基数量的期望值远远低于18,该蛋白不存在跨膜螺旋和信号肽,且位于膜外。
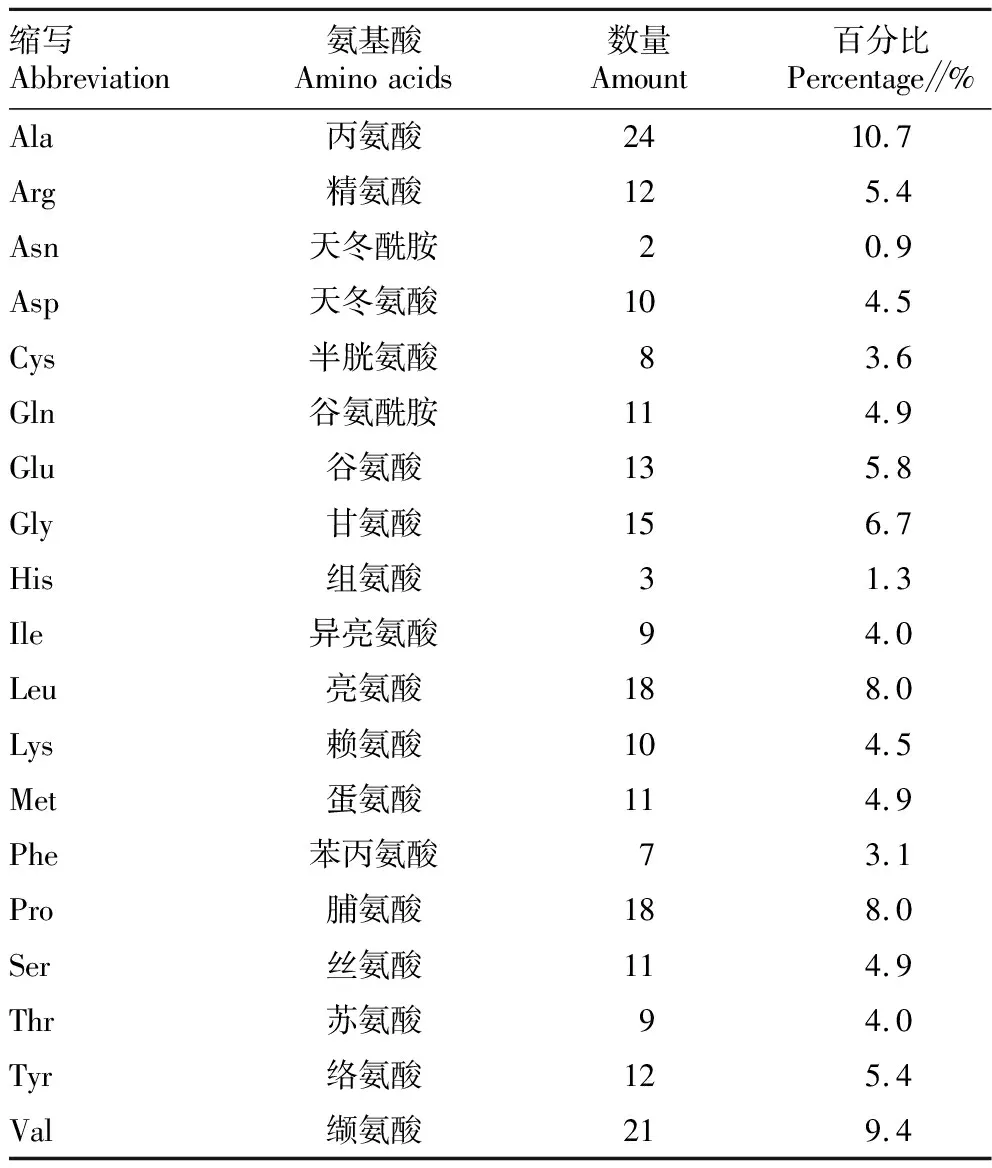
表1 TK基因编码蛋白质数量及百分比
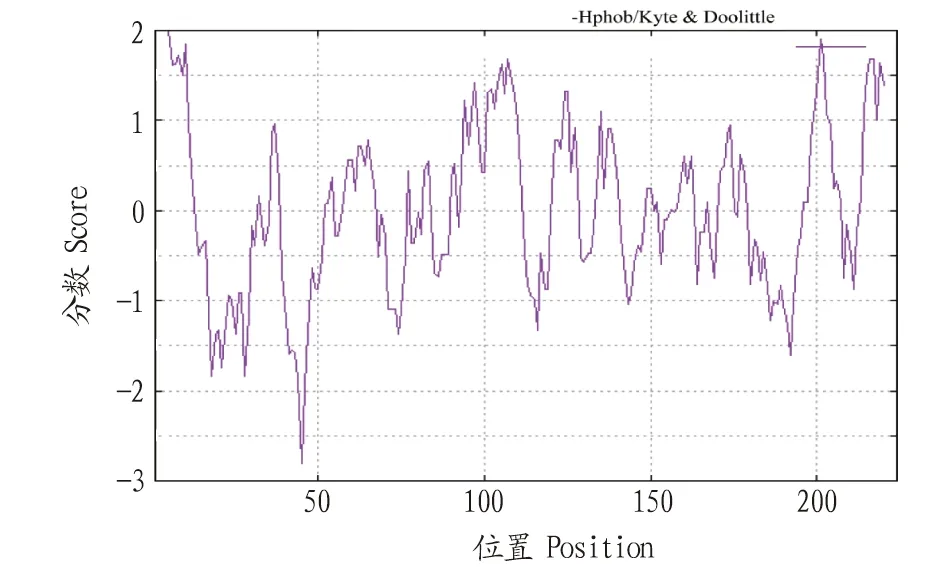
图1 TK蛋白亲水性、疏水性预测Fig.1 Predictive analysis of hydrophilicity and hydrophobicity of TK protein
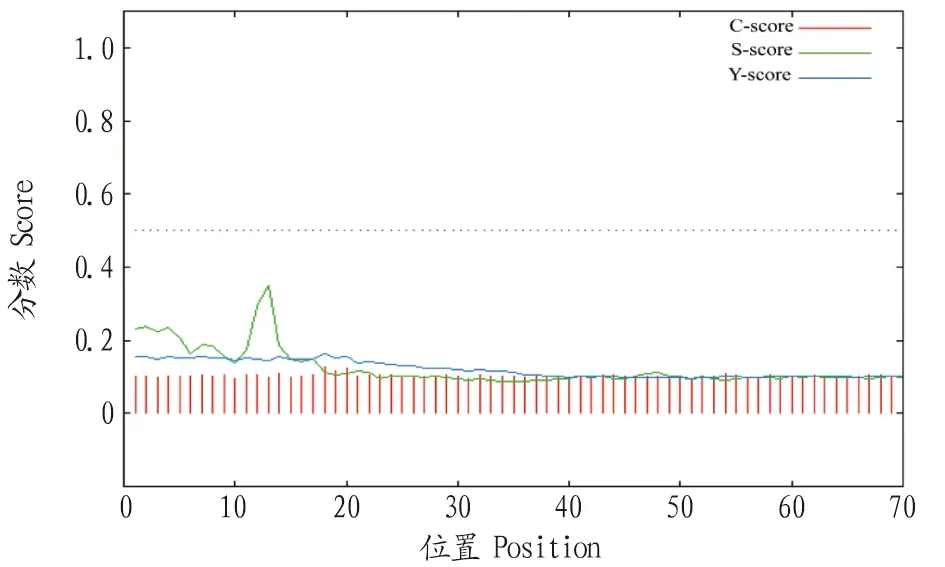
图2 TK蛋白信号肽预测Fig.2 Predictive analysis of TK protein signal peptide
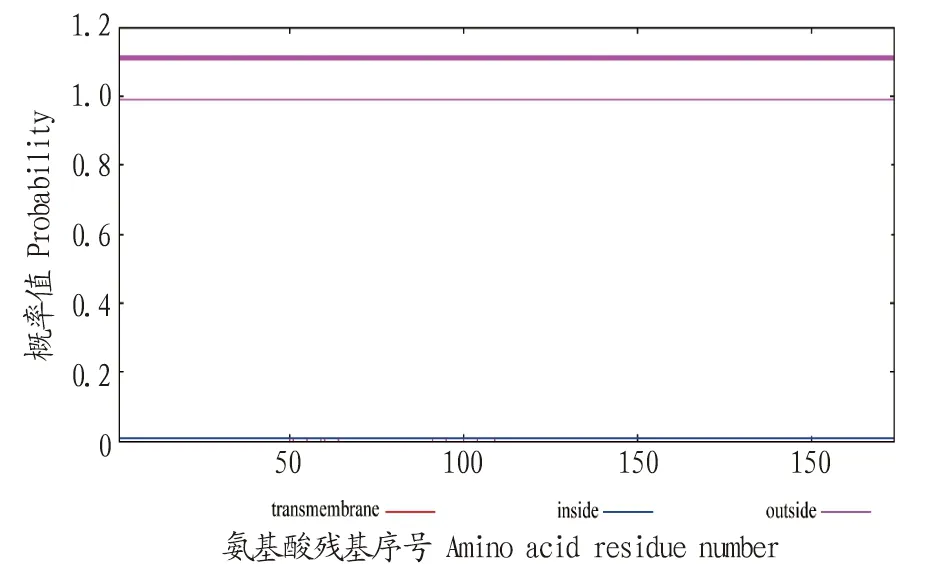
注:横坐标表示提交蛋白序列对应的氨基酸残基序号,纵坐标表示为横轴上每个氨基酸位于膜内侧、膜外侧和跨膜螺旋的概率值。图中蓝色线段是位于膜内的结构,红色线段是位于膜外的结构。Notes:The horizontal coordinates indicate the amino acid residue numbers corresponding to the submitted protein sequences,and the vertical coordinates are the probability values of each amino acid located in the inner membrane,outer membrane and transmembrane helix on the horizontal axis.The blue line segment in the figure is the structure located inside the membrane,and the red line segment is the structure located outside the membrane.
2.3 TK蛋白结构域分析根据NCBI-CDD进行蛋白结构域分析,结果可知,该蛋白有一个TK超家族结构域,在序列中的位置是4-185(图4a)氨基酸残基区域,图4b则为该蛋白结构域区间;其中E值越小随机性越低,结果在统计学中越显著。HMMER(图4c和4d)和SMART(图4e)结果表明,TK蛋白结构域位于2-175氨基酸残基区域。综合分析可知,TK蛋白结构域位于2-175氨基酸残基区域的可能性较大。
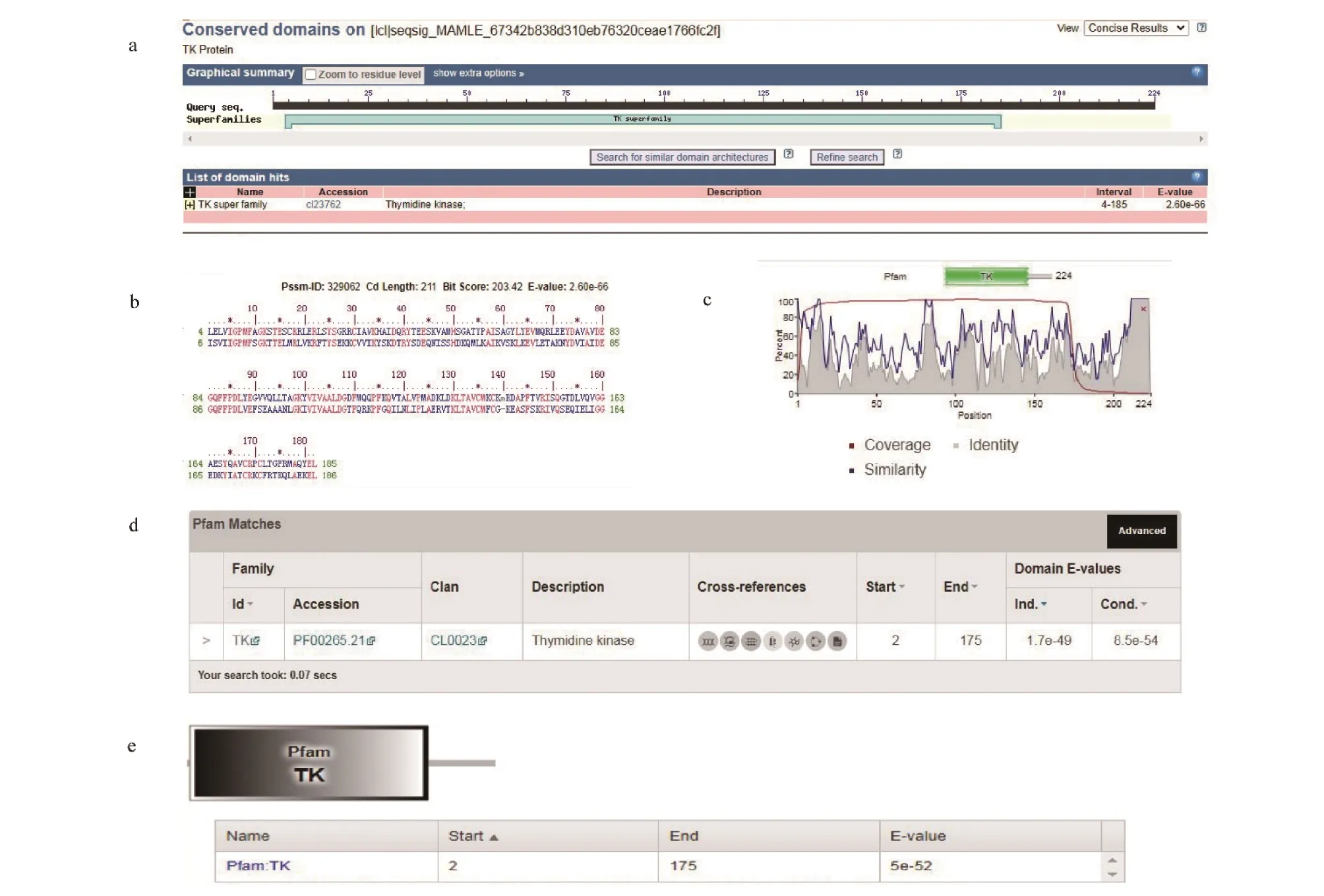
注:a、b.NCBI-CDD蛋白结构域分析及蛋白结构域区间;c、d.HMMER蛋白结构域分析;e.SMART蛋白结构域分析。Note:a,b.NCBI-CDD protein structural domain analysis and protein structural domain interval;c,d.HMMER protein structural domain analysis;e.SMART protein structural domain analysis.
2.4 TK蛋白质二级结构预测分析蛋白质二级结构的预测通常被认为是蛋白结构预测的第1步,二级结构在蛋白质分析、酶活性残基分析、蛋白结构预测等方面都是不可缺少的。SOPMA在线软件对TK蛋白进行二级结构分析,结果如图5所示,在TK蛋白编码的224个氨基酸中α-螺旋共72个,占32.14%;β-转角17个,占7.59%;无规则卷曲86个,占38.39%;延伸链49个,占比21.88%。
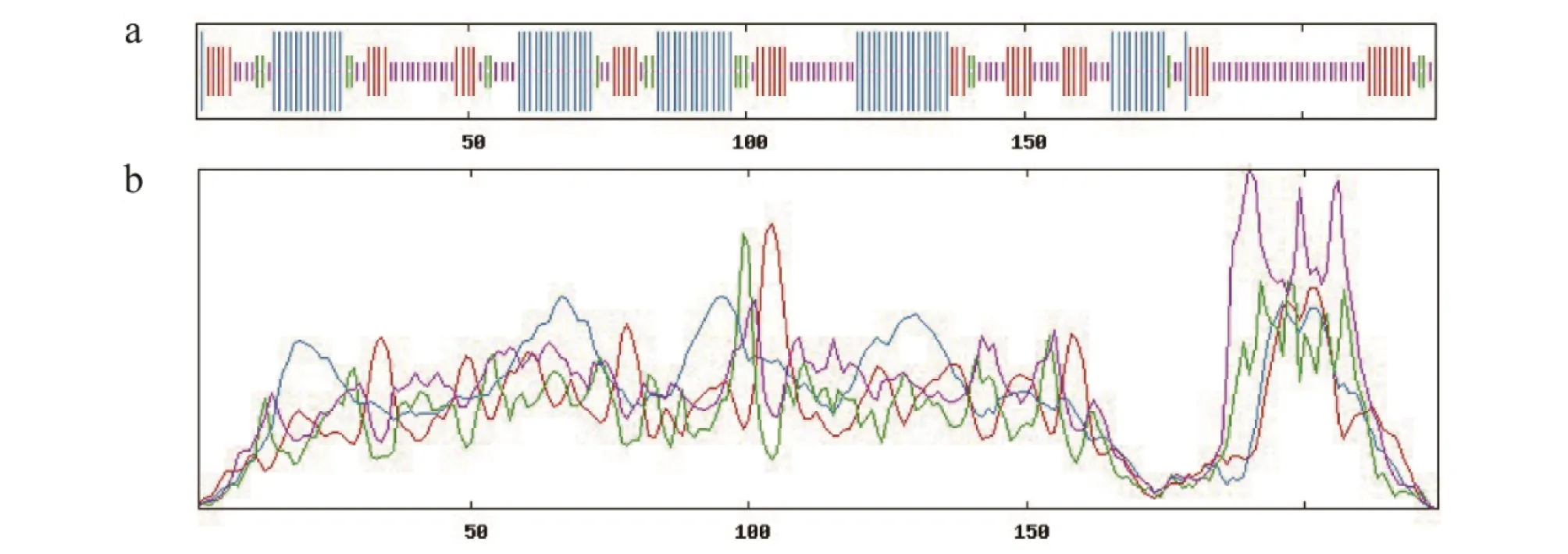
注:蓝色.α-螺旋;绿色.β-折叠;黄色.无规则卷曲;红色.延伸链。Note: Blue.α-helix;green.β-fold;yellow.Irregularly curled;red.Extended chain.
2.5 TK蛋白质三级结构预测分析采用同源建模的方法,根据SWISS-MODEL对TK蛋白进行三级结构预测,结果如图6所示。TK蛋白的序列相似度与布氏锥虫胸苷激酶(SMTL ID:5fuv.1.A)模板序列较为相似。
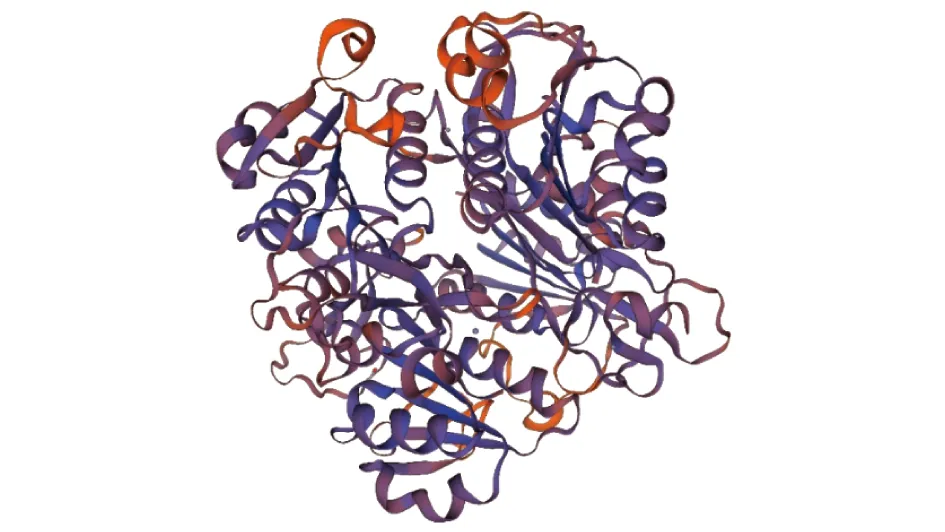
图6 TK蛋白质三级结构预测分析Fig.6 Prediction analysis of tertiary structure of TK protein
2.6 TK蛋白MEGA构建系统发育树分析利用TK蛋白的氨基酸序列在NCBI进行blast,得到骆驼痘病毒、沙鼠痘病毒、马痘病毒、鲤科疱疹病毒1、鲤科疱疹病毒2、鲤科疱疹病毒3、牛痘病毒、火鸡痘病毒和斑马鱼病毒等的TK蛋白序列,与锦鲤疱疹病毒氨基酸进行同源序列比对分析,表明锦鲤疱疹病毒TK氨基酸序列与鲤科疱疹病毒亲缘关系较近(图7)。
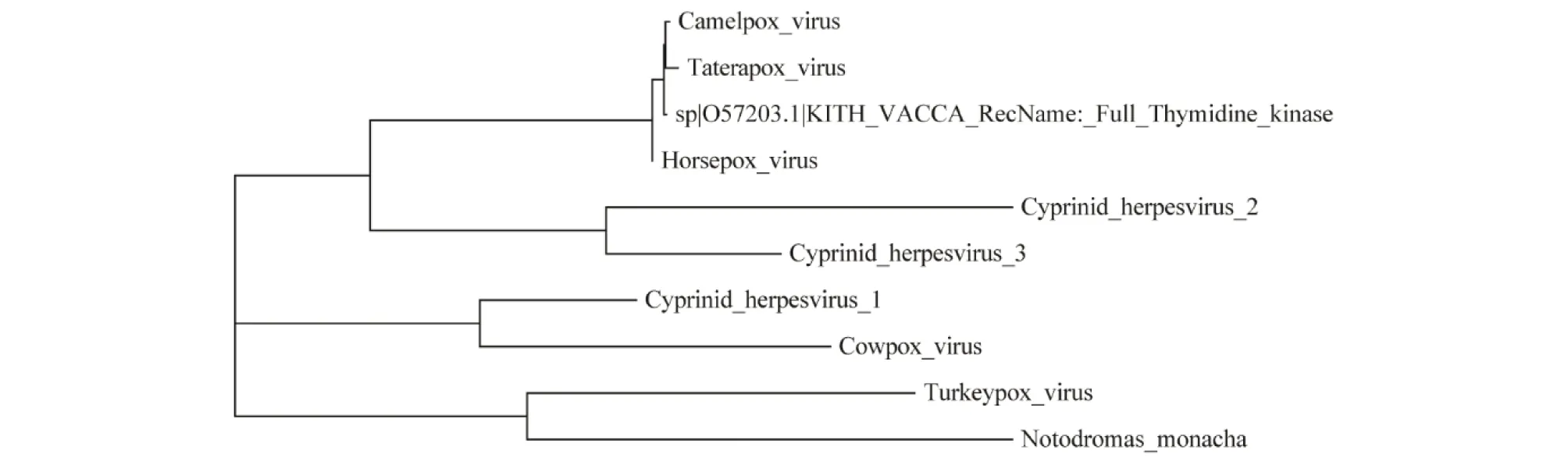
图7 TK蛋白系统发育树分析结果Fig.7 Results of TK protein phylogenetic tree analysis
3 结论
随着生物学技术的不断发展提高,生物信息学的应用也逐渐丰富[19]。胸苷激酶基因是大多数疱疹病毒的主要毒力基因之一,也是病毒复制的非必需基因,胸苷激酶蛋白是疱疹病毒中一种高度保守性蛋白,且具有较高的抗原性[20]。前人研究表明,胸苷激酶是DNA合成的关键酶之一,痘病毒在复制过程中需要借助胸苷激酶作用形成高浓度核酸以完成子代的顺利复制[21]。胸苷激酶基因是猪伪狂犬病病毒的主要毒力基因和非必需基因,也是病毒化学治疗和缺失致弱疫苗的主要靶基因[22],因此对TK基因的生物信息学分析,在一定程度上为各种病毒的研究提供了科学基础。
锦鲤疱疹病毒病是我国农业农村部规定的二类传染病,目前针对该病毒的研究主要集中在检测方面。该研究进行了相关基因的生物信息学预测,为今后的研究方向提供基础。综合表明,锦鲤疱疹病毒中胸苷激酶基因编码的蛋白是不稳定蛋白,理化性质分析该蛋白为亲水蛋白、不含有信号肽;其蛋白结构域位于2-175氨基酸残基区域内;该蛋白二级结构主要为无规则卷曲和α-螺旋。综上,锦鲤疱疹病毒胸苷激酶基因的生物信息学分析为进一步挖掘该基因的功能提供了理论基础。

