扁刺蛾线粒体基因组全序列特征及系统发育分析
江宏燕,陈世春,廖姝然,陈亭旭,杨普香,谢小群,王晓庆*
扁刺蛾线粒体基因组全序列特征及系统发育分析
江宏燕1,陈世春1,廖姝然1,陈亭旭1,杨普香2,谢小群2,王晓庆1*
1. 重庆市农业科学院茶叶研究所,重庆 402160;2. 江西省经济作物研究所,江西 南昌 330203
扁刺蛾()具有分布广、多食性、危害大等特点,是我国重要的农林业害虫。为报道采自江西的扁刺蛾线粒体基因组,了解其线粒体基因组的多样性与差异,探究刺蛾科昆虫线粒体基因组进化规律。通过Sanger测序后拼接、校正、注释获得扁刺蛾的线粒体全基因组序列,并基于蛋白质编码基因序列构建了鳞翅目17个科26种蛾类昆虫的系统发育树。结果显示,扁刺蛾线粒体基因组是1个大小为15 540 bp的闭合环状双链DNA分子,共编码37个基因,包括13个蛋白质编码基因,2个核糖体RNA基因和22个转运RNA基因,还有1个425 bp的控制区,基因排列与鳞翅目双孔类(Ditrysia)昆虫相同。通过与其他刺蛾的全序列和蛋白质编码基因序列对比相似度,结果显示,扁刺蛾与茶刺蛾()的相似度最高,与褐边绿刺蛾()相似度最低。系统发育分析结果表明,扁刺蛾与茶刺蛾的亲缘关系最近,其次为龟形小刺蛾(),鳞翅目昆虫各科均聚为一支。本研究为深入研究扁刺蛾的起源、遗传多样性、迁移和分化,以及对农药的抗性提供科学依据。
刺蛾科;扁刺蛾;线粒体基因组;系统发育
扁刺蛾()俗称洋辣子、火辣子和刺虫等,属鳞翅目(Lepidoptera)刺蛾科(Limacodidae),分布广泛,在我国的台湾、福建、广东、广西、海南、云南、贵州、四川、湖南、江西、浙江、江苏、安徽、湖北、河南、甘肃、山东及陕西等地均有分布[1]。扁刺蛾的寄主植物丰富多样,主要危害茶树、油茶树、樱花树等植物,幼虫取食叶片,轻则影响树势,重则导致植株死亡[2]。幼虫具毒刺,触及皮肤,轻者红肿、疼痛,严重时威胁生命,极大地妨碍了采茶与田间作业。近年来,围绕乡村振兴和茶旅融合发展的产业需求,为推动茶产业转型升级,着力打造观光茶园。而在一些主产茶区的林间和林-茶结合地带的茶园中出现了该虫不同程度的为害,其暴发为害的风险大大提升,不仅影响茶叶的质量和产量,更影响了茶园的正常生产管理[3-4]。
昆虫线粒体基因组具有许多共同特征,包括基因组较小、基因数目少、基因组成稳定、基因排列相对保守、重组率低、碱基突变率高和母系遗传性等特点,在昆虫的种类鉴定、种群遗传结构、系统发育研究和外来入侵物种管理中得到广泛应用[5-6]。鳞翅目是昆虫纲中第二大目,包括蛾(Moths)和蝶(Butterflies)两类,种类分布极广,以热带地区最为丰富,目前已描述的约有16万个物种[7],刺蛾科是鳞翅目中相对较小的类群,全世界已记载301属1 672种[8]。与其他科昆虫相比,刺蛾科线粒体基因组全序列的测序工作开展较晚,2016年才首次报道了黄刺蛾()的线粒体全基因组数据[9];Bian等[10]报道了扁刺蛾的完整线粒体基因组,确定了刺蛾科在鳞翅目中的系统地位。目前GenBank上已报道的刺蛾科线粒体基因组仅6种11个(截止2023年3月1日),相比现有的刺蛾科物种数量,刺蛾科线粒体基因组的测序工作有待加强,刺蛾个体变异丰富、近缘种间相似性较高,不易形态分类鉴定,易造成同物异名。昆虫线粒体DNA存在适应性演化[11],本研究报道的扁刺蛾采自江西省南昌市茶树和樱花树间作的茶园中,扁刺蛾食光樱花树叶片后转移至茶园取食危害,近几年持续暴发成灾,在当地适应能力极强[12-13],线粒体基因组信息可为评判种群对周围环境的适应能力提供依据。本研究利用Sanger测序获得扁刺蛾线粒体基因组全序列,对扁刺蛾的基因组全序列特征进行分析,综合比较分析已报道的刺蛾科线粒体基因组的结构特点,并构建鳞翅目双孔类(Ditrysia)17个科26种蛾类昆虫的系统发育树,以期为鳞翅目刺蛾科昆虫的精准鉴定、分子生物学及综合防控等研究打下基础。
1 材料与方法
1.1 试验材料
试验用虫于2018年8月采集自江西省南昌市茶园,采集到的样本带回实验室,将活体昆虫样本放入无水乙醇中,放置于–20 ℃冰箱保存备用。
1.2 DNA提取、PCR扩增、测序
DNA的提取:挑选乙醇浸泡后保存完好的刺蛾样品,经处理后,采用快速DNA提取检测试剂盒(北京天根生化科技有限公司)按照说明书完成提取,使用1.5%琼脂糖凝胶电泳和Nanodrop 2000分光光度计检测DNA的质量与浓度。
PCR扩增:利用鳞翅目其他昆虫的线粒体基因组序列设计PCR引物,使用Taq HS DNA聚合酶(TaKaRa)进行扩增。扩增条件:94 ℃预变性3 min;94 ℃变性30 s,55~60 ℃退火30~50 s,72 ℃延伸1~3 min,共35个循环;72 ℃延伸10 min,总反应体积为50 μL。用1.5%琼脂糖凝胶检测PCR产物质量,然后将产物送至转导精进(武汉)生物技术有限公司进行Sanger双向测序。
1.3 线粒体基因组的注释与分析
PCR扩增产物序列通过CLC Genomics Workbench 8.5软件进行校正和拼接,获得了扁刺蛾的完整线粒体基因组,线粒体基因组序列通过NCBI网站(https://www.ncbi.nlm. nih.gov)上的ORF Finder和MITOS WebServer程序[14]进行注释,并利用blastn和blastp与其他刺蛾科线粒体基因组进行鉴定比对,确定控制区和37个基因的位置及大小。tRNA基因及其三叶草二级结构通过ARWEN和tRNAscan-SE软件进行预测[15-16],并手动校正。通过DAMBE软件分析蛋白质编码基因的氨基酸使用情况和同义密码子使用度(Relative synonymous codon usage,RSCU)[17]。使用DNAMAN软件对扁刺蛾(MN661155、MK1226244)、黄刺蛾(KY628213)、茶刺蛾(,MK250437)、龟形小刺蛾(,MH675969)、双齿绿刺蛾(,MK122617)、褐边绿刺蛾(,KX108765)6种刺蛾全序列及其蛋白质编码基因的核苷酸进行序列比对分析。
1.4 系统发育分析
从GenBank数据库(https://www.ncbi.nlm.nih.gov/nucleotide)下载已报道的鳞翅目线粒体全基因组序列进行系统发育分析。使用MEGA 7.0[18]的默认设置对13个蛋白编码基因的序列进行比对,氨基酸序列直接比对,DNA序列以无脊椎动物线粒体基因组密码子(Invertebrate mitochondrion)翻译成氨基酸序列之后比对,再反翻译获得比对后的DNA序列。将比对后的每个基因序列分别使用Gblocks程序中的condon/protein识别保守区域[19],完成之后进行序列组装,组装完整的氨基酸序列和核苷酸序列利用最大似然法(Maximum likelihood method,ML)构建系统发育树[20-21],采用Bootstrap(1 000次重复)检验系统树各分支节点的置信值。
2 结果与分析
2.1 扁刺蛾基因组全序列分析
扁刺蛾线粒体基因组序列全长15 540 bp(GenBank登录号:MN661155),由1个控制区(Control region,CR)、2个核糖体RNA(rRNA)、13个蛋白质编码基因(Protein-coding genes,PCG)和22个转运RNA(tRNA)组成,呈双链闭合环状(图1),基因排列与鳞翅目双孔类昆虫相同。的长度最长(1 725 bp),最短仅(64 bp)。2个rRNA()、4个PCG(、、、)、控制区和8个tRNA(、、trnL)位于基因组N链,其他的9个PCG和14个tRNA位于J链。
在扁刺蛾线粒体基因组的37个基因中,相邻基因之间存在着基因间隔和基因重叠现象,其中基因间隔区有20处,共270 bp。和之间间隔最长,为54 bp;其次是和,为27 bp。基因重叠较少,仅有3处,共16 bp,分别位于和、和、和之间,重叠序列分别为7、1、8 bp;既无重叠又无间隔的区域共有14处。蛋白编码基因中AT含量最高的为,达92.73%;最低的为,为71.33%(表1)。
扁刺蛾线粒体基因组的A、T、C、G的碱基含量分别为39.40%、41.54%、11.52%和7.54%,AT含量为80.94%,GC含量为19.06%,具有明显的AT偏好性。AT偏斜和GC偏斜均为负值,表明基因组全序列中碱基A和G的含量分别低于T和C;蛋白质编码基因中AT含量为79.07%,比全序列AT的含量稍低,碱基A含量小于T,而G的含量高于C;tRNA和rRNA中碱基A和G的含量分别高于T和C,控制区则相反(表2)。
2.2 扁刺蛾蛋白编码基因
13个PCG的长度在165~1 725 bp,共计11 163 bp,占总基因组的71.83%。13个PCG中,除以CGA起始,其他均以ATN(ATA、ATC、ATG、ATT)起始,10个以TAA为终止密码子,、和以T终止,这种不完全终止密码子T比较常见。蛋白质编码基因的氨基酸使用频率(图2A)和同义密码子使用度(图2B)的分析结果显示,扁刺蛾13个PCG中使用频繁的氨基酸依次为Leu(14.52%)、Ile(12.12%)、Phe(10.70%)、Asn(8.20%)、Met(7.39%),Cys的使用频率最低(0.91%);进一步分析发现,以A和U结尾的同义密码子使用度高于以G和C结尾的,与昆虫线粒体基因组的AT偏好性特征一致。
2.3 tRNA和rRNA基因
扁刺蛾线粒体基因组包含22个tRNA(图3),长度在64~71 bp。22个tRNA中,21个tRNA能构建典型的三叶草二级结构,而trnS缺失了DHC臂,该现象在昆虫线粒体基因组中较常见[22]。同时,部分tRNA的二级结构还存在G-U、U-U和A-C等非经典配对,G-U弱配共19处,U-U错配共6处,A-C错配1处,这种错配常出现在其他昆虫类群tRNA二级结构中,可维持tRNA二级结构的稳定性。位于trnL与之间,位于与CR之间,长度分别为1 419 bp和783 bp,AT含量分别为85.55%和85.70%,具有AT偏向性。
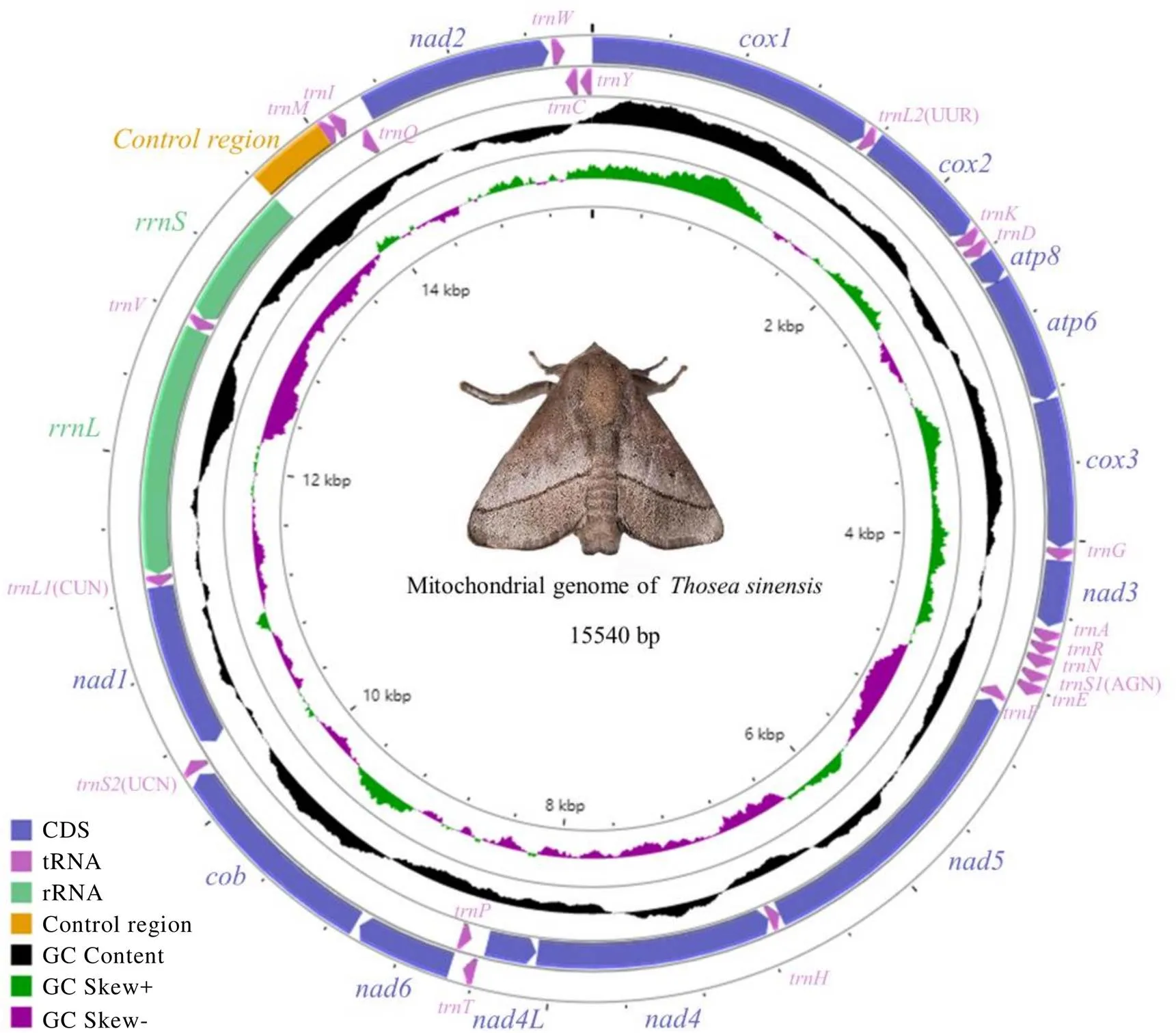
图1 扁刺蛾线粒体基因组结构

表1 扁刺蛾线粒体基因组的基因组成
注:AT-偏斜=(A-T)/(A+T),GC-偏斜=(G-C)/(G+C)
Note: AT-skew=(A-T)/(A+T), GC-skew=(G-C)/(G+C)
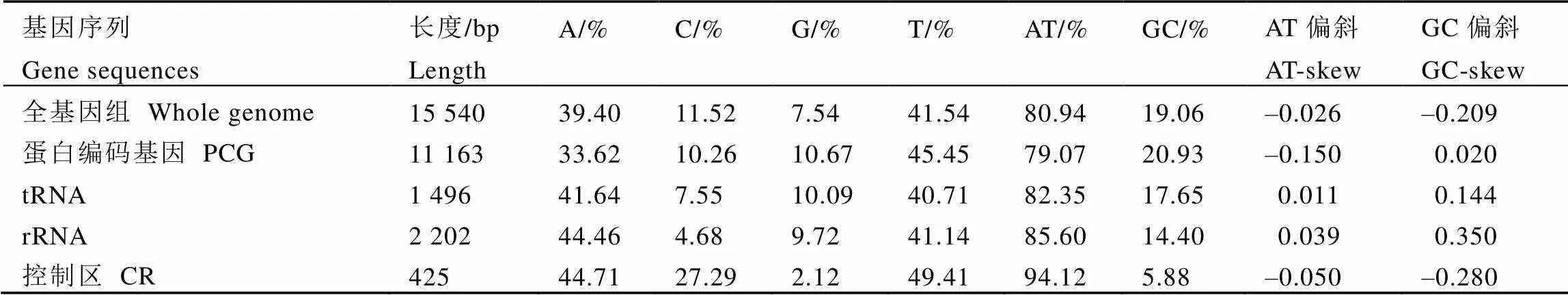
表2 扁刺蛾线粒体基因组的核苷酸组成
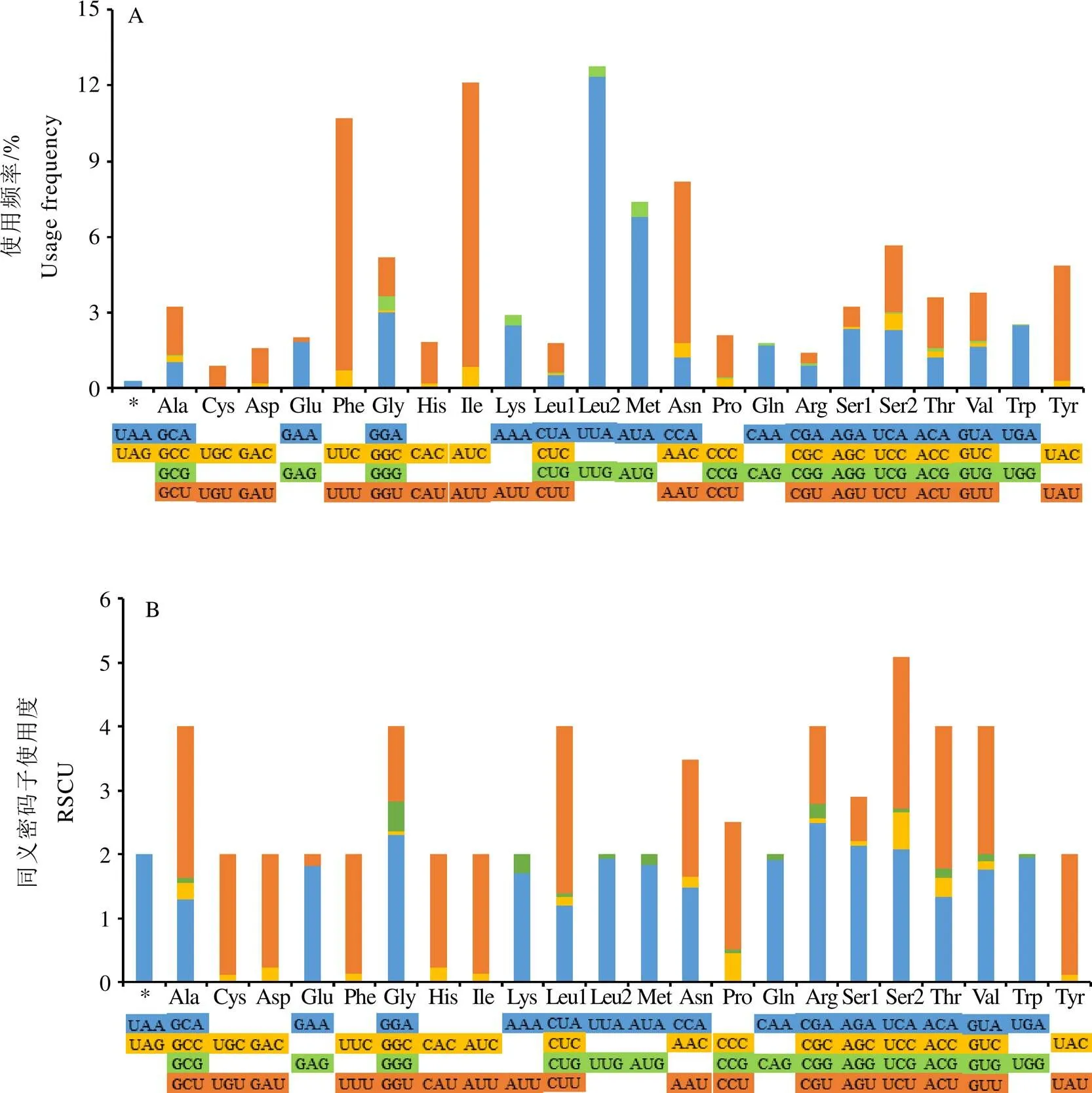
图2 扁刺蛾线粒体基因组中蛋白质编码基因的氨基酸使用频率(A)和同义密码子使用度(B)
2.4 控制区
由图4可知,扁刺蛾线粒体基因组的控制区位于和之间,长度为425 bp,AT含量是基因组各区域中最高的(94.12%),在基因的下游有1段基序“ATAGA”和19 bp的poly T结构,末端有1个9 bp的poly A结构,扁刺蛾控制区发现了3个微卫星(AT)n元件,已报道的扁刺蛾线粒体基因组的控制区微卫星(AT)n元件仅有1个微卫星(AT)10元件[10],但没有发现串联重复。这些特征在鳞翅目昆虫线粒体基因组的控制区较为常见[23-25]。
刺蛾科昆虫线粒体基因组的控制区中,通常有1段20~26 bp的引导序列,除龟形小刺蛾的poly T结构仅有16 bp,扁刺蛾、黄刺蛾、茶刺蛾、龟形小刺蛾、双齿绿刺蛾和褐边绿刺蛾的poly T长度一致,微卫星(AT)n元件有1~3个,除双齿绿刺蛾的poly A结构有突变外,其他刺蛾均比较保守,通常为8~11 bp。
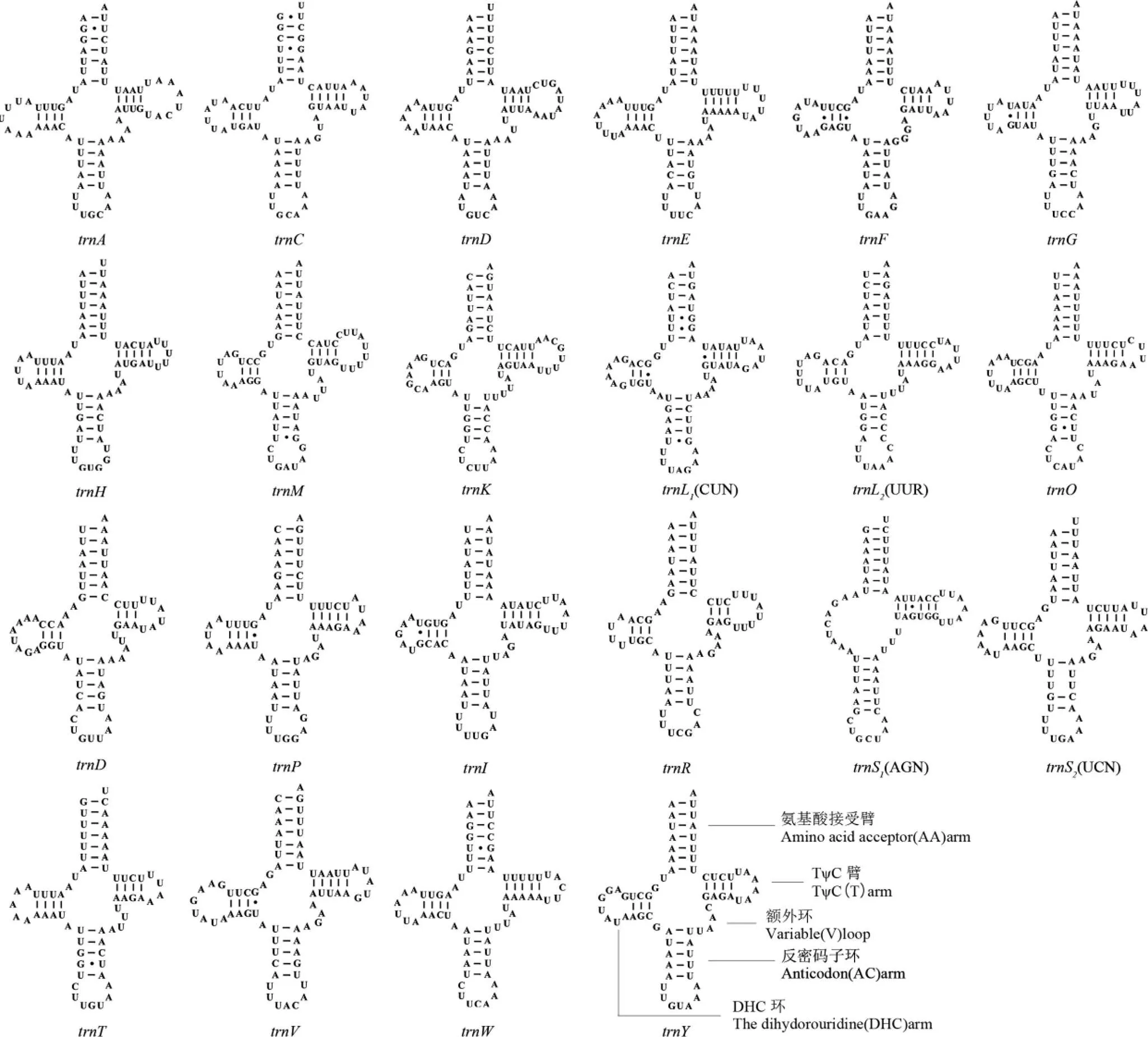
图3 扁刺蛾线粒体基因tRNA预测二级结构
2.5 6种刺蛾的基因组比较及序列比对
对6种刺蛾线粒体基因组核苷酸组成进行分析(表3),6种刺蛾线粒体基因组长度范围在15 292~15 645 bp,AT含量均大于80%,扁刺蛾线粒体基因组较大,仅次于茶刺蛾。由表4可知,本研究的扁刺蛾1与其他5种刺蛾相比,基于线粒体基因组全序列和PCG序列对比的相似度均大于82%,基于线粒体全序列对比结果可以看出,与茶刺蛾的相似度最大,为84.96%,与黄刺蛾、龟形小刺蛾和双齿绿刺蛾相似度差距不大,与褐边绿刺蛾的相似度最小为82.29%;基于PCG的对比结果中,扁刺蛾1与茶刺蛾相似度最大为90.66%,与褐边绿刺蛾的相似度最小,说明扁刺蛾与茶刺蛾的相似度最高,与褐边绿刺蛾相似度最低。
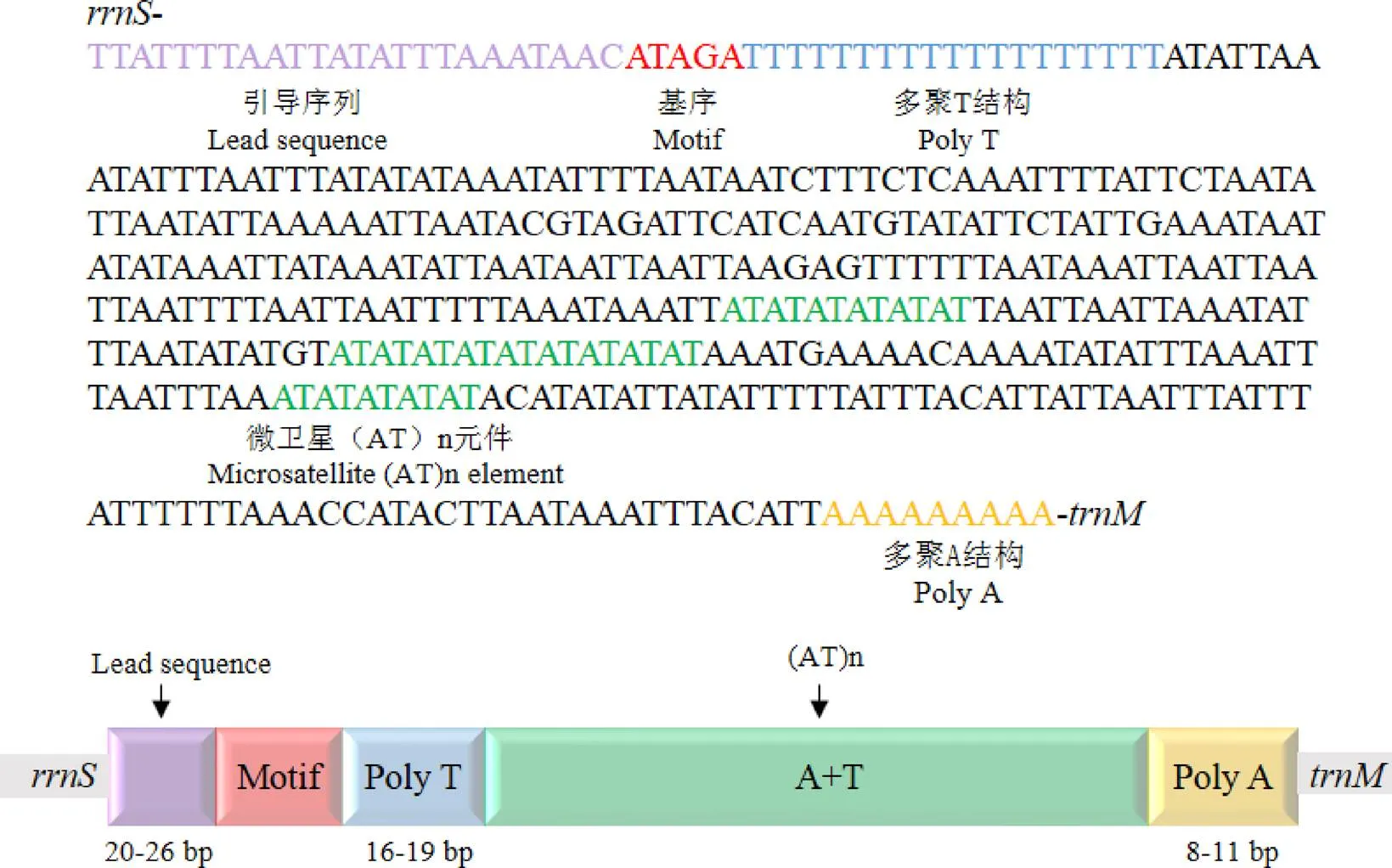
图4 扁刺蛾和刺蛾科线粒体基因组控制区的结构
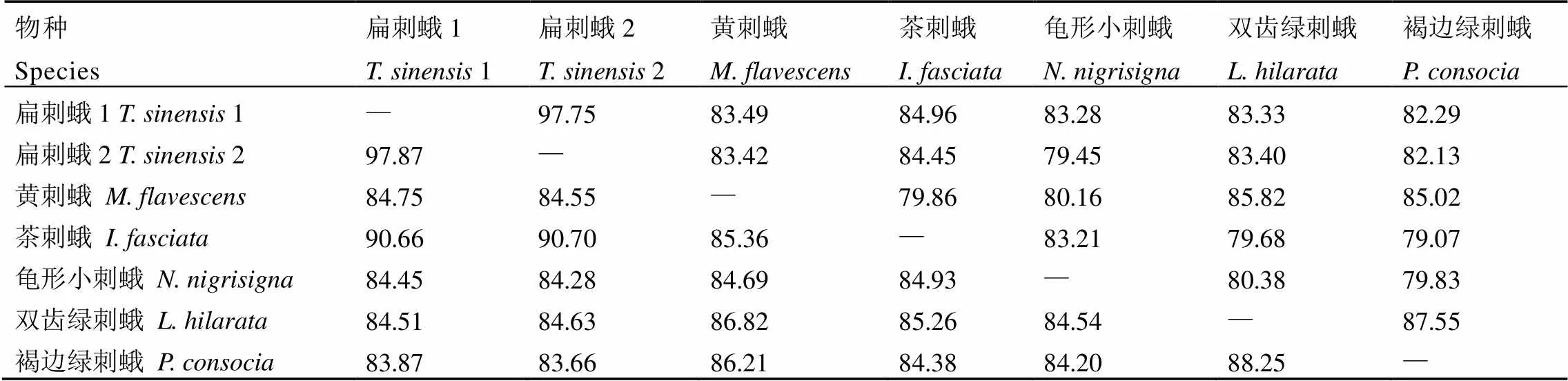
表4 6种刺蛾线粒体基因组相似度比较
注:扁刺蛾1的GenBank登录号为MN661155,扁刺蛾2的GenBank登录号为MK122624。上三角基于全序列对比的相似度,下三角基于编码蛋白基因比对的相似度
Note:1 GenBank accession number is MN661155,2 GenBank accession number is MK122624. The number above the diagonal is based on the alignment of complete sequence, the number below the diagonal is based on the alignment of PCG
2.6 系统发育分析
以黑腹果蝇(,GenBank登录号:DMU37541)为外群,构建了基于26种鳞翅目双孔次目蛾类昆虫13个PCG的核苷酸与氨基酸序列的系统发育树(图5)。结果显示,扁刺蛾、茶刺蛾、黄刺蛾、龟形小刺蛾、双齿绿刺蛾和褐边绿刺蛾同属于刺蛾科,以100%的支持率聚为一支(图5)。结果表明,扁刺蛾与茶刺蛾的亲缘关系最近,其次为龟形小刺蛾,鳞翅目各科均聚为一支。

图5 26种蛾类昆虫的系统发育树
3 讨论
鳞翅目害虫是农林业主要害虫之一,目前GenBank上鳞翅目昆虫的线粒体基因组全序列有1 000多个,长度一般为15~16 kb,AT含量在76%~80%,而刺蛾科线粒体基因组碱基AT含量达80%以上,表现出典型高AT含量。多数刺蛾科昆虫蛋白编码区除以CGA起始,其他均以ATN起始,以TAA和T为终止密码子,不完整的终止密码子会在转录
形成mRNA后转变成完整的终止密码子[26],这种现象普遍存在于大多数鳞翅目线粒体基因组中[24]。tRNA是发生基因重排的热点区域,但刺蛾科昆虫基因重排现象不常见,除褐缘绿刺蛾外,其他5种刺蛾线粒体基因排列顺序相同,褐缘绿刺蛾发现1处基因重排,即与的位置发生了置换[27],rRNA的位置则比较固定。已报道的刺蛾线粒体基因组的trnS的二级结构都缺少DHU臂,但后期可被修复形成典型的三叶草结构[28-29]。
刺蛾科昆虫控制区均有ATAGA基序、Ploy-T结构、微卫星(AT)n元件和Ploy-A结构这4个保守元素,尤其是poly-T结构没有发生碱基突变,表明刺蛾科昆虫物种演化较稳定。在许多鳞翅目昆虫中并无微卫星(AT)n元件[24],有学者认为n≥8[5],但马尾松毛虫()和云南松毛虫()控制区都仅有1个微卫星(AT)7元件[30-31],Bian等[10]报道了江苏扁刺蛾控制区有1个微卫星(AT)10元件,但我们发现还存在1个微卫星(AT)9元件,目前n并没有统一的标准,本研究将n≥5的AT重复定为微卫星(AT)n元件,关于控制区微卫星元件的特点还有待进一步研究。鳞翅目的原始小蛾类物种的控制区中,基序ATAGA结构和poly-T结构发生的突变较为频繁[24],刺蛾科中除双齿绿刺蛾的poly A结构有突变外,其他均比较保守。不同物种的线粒体基因组控制区长度差异较大,甚至同一物种不同地理种群之间的差异也较大,如已报道的重庆茶网蝽与陕西安康茶网蝽,控制区相差1 463 bp[32-33],刺蛾科昆虫也有这种现象,如GenBank中的褐边绿刺蛾(登录号:OK149235、KX108765)的控制区相差232 bp。控制区AT含量高、含有大量重复序列以及能形成稳定的二级结构,使得控制区测序难度较大[34-37],是由测序差异引起的还是控制区本身的特点,有待进一步研究。
有研究者证实灰飞虱自然种群中存在两类线粒体DNA,且Ⅱ类由Ⅰ类演化而来,灰飞虱线粒体DNA的变异带来了生殖和耐寒力方面的优势,使其能够在种群中得到扩散[11]。通常情况下昆虫同一物种的两个地理种群的线粒体基因组全序列相似度达99%,Bian等[10]报道的扁刺蛾采自江苏省,与本研究的江西扁刺蛾线粒体基因组全序列相似度为97.75%,13个PCG的相似度为97.87%,差异较大,说明这两个扁刺蛾地理种群的线粒体基因组存在着一定程度的差异。这种差异可能是由于在进化过程中不同地区的扁刺蛾经历了不同的地理环境或不同寄主的生态压力,从而导致了线粒体的蛋白表达和合成基因的变化。同时,近几年有报道扁刺蛾在江西南昌果园和茶园泛滥成灾[13],推测扁刺蛾线粒体DNA可能也存在地理和寄主环境方面的适应性演化,但后续还需更多样本的测序和生物学研究。
基于26种鳞翅目蛾类昆虫13个PCG的核苷酸与氨基酸序列构建系统发育树较为一致,刺蛾科昆虫均聚为一支,自展值为100%,其系统发育关系为[龟形小刺蛾+(扁刺蛾+茶刺蛾)]+[(双齿绿刺蛾+褐缘绿刺蛾)+黄刺蛾],由此可见,刺蛾科在鳞翅目蛾类昆虫中的系统发育关系较稳定,在许多研究中均被证实[9-10,27,38-40],两个刺蛾在系统发育树上聚为一支,进一步明确了扁刺蛾的线粒体基因组信息。将分子与传统形态分类学结合,不仅能验证形态分类学的准确性,还可以较为准确地鉴定在形态上难以分辨的相似种,是今后昆虫分类研究的主要方向[41]。Li等[42]收集了昆虫纲多个目的形态种信息、核基因和线粒体基因数据,评估已知形态种与包含隐存种的分子种数量,发现每个昆虫形态种平均对应3.1个物种。性信息素和昆虫病毒专一性强,鳞翅目害虫的精准鉴定,可为后期对其进行性信息素诱杀和病毒制剂等精准绿色防控技术的开发打下基础。如茶尺蠖()和灰茶尺蠖()是茶树害虫尺蠖类的2个近缘种,长期以来都把它们看作茶尺蠖,但在实际使用茶尺蠖病毒和性诱剂时发现,不同茶区尺蠖对茶尺蠖病毒和性诱剂的敏感性存在差异[43-44],利用线粒体基因组序列正确鉴定害虫种类对精准指导田间防治意义重大。线粒体基因组信息也被广泛应用于害虫捕食性天敌摄食分析,在生物防治中,需明确天敌的食物谱并对其捕食量进行估算,基于形态学的传统研究方法不仅耗时耗力,且无法满足农业生态系统中重要的捕食性天敌蜘蛛类的食谱分析,利用害虫的DNA条形码与PCR技术可对蜘蛛的摄食进行精确定量分析,实际应用价值极大[45-46]。
本研究通过测定江西省南昌市茶园扁刺蛾线粒体全基因组序列,分析其全序列特征、与其他刺蛾昆虫线粒体基因组的差异以及与26种鳞翅目物种间的系统发育关系,既丰富了刺蛾科线粒体基因组数据库,也为刺蛾科物种精准鉴定、系统进化分析和防控研究提供基础。
[1] 肖强, 唐美君, 周孝贵. 茶树病虫和天敌名录[M]. 北京: 中国农业出版社, 2020: 104. Xiao Q, Tang M J, Zhou X G. List of tea pests and natural enemies [M]. Beijing: China Agriculture Press, 2020: 104.
[2] 张汉鹄, 谭济才. 中国茶树害虫及其无公害治理[M]. 合肥: 安徽科学技术出版社, 2004. Zhang H H, Tan J C. Chinese tea pests and their pollution-free control [M]. Hefei: Anhui Science and Technology Press, 2004.
[3] 谢小群, 贺望兴, 石旭平, 等. 三种微生物农药对扁刺蛾幼虫的毒力试验[J]. 茶叶通讯, 2020, 47(4): 617-622. Xie X Q, He W X, Shi X P, et al. Toxicity test of three kinds of microbial pesticides onWalker [J]. Journal of Tea Communication, 2020, 47(4): 617-622.
[4] 崔林, 刘月生. 茶园扁刺蛾的发生及防治[J]. 中国茶叶, 2005, 27(2): 21. Cui L, Liu Y S. Occurrence and control ofin tea garden [J]. Chinese Tea, 2005, 27(2): 21.
[5] 王维, 孟智启, 石放雄, 等鳞翅目昆虫比较线粒体基因组学研究进展[J]. 科学通报, 2013, 58(30): 3017-3029. Wang W, Meng Z Q, Shi F X, et al. Advances in comparative mitochondrial genomics of Lepidoptera [J]. Chinese Science Bulletin, 2013, 58(30): 3017-3029.
[6] 魏书军, 陈学新. 昆虫比较线粒体基因组学研究进展[J]. 应用昆虫学报, 2011, 48(6): 1573-1585. Wei S J, Chen X X. Advances in comparative mitochondrial genomics in insects [J]. Journal of Applied Entomology, 2011, 48(6): 1573-1585.
[7] 古丽扎尔·阿不都克力木, 张秀英, 苏比奴尔·艾力, 等中国鳞翅目新物种2021年年度报告[J]. 生物多样性, 2022, 30(8): 37-45. Gulzar A, Zhang X Y, Subinur E, et al. Annual report of new taxa for Chinese Lepidoptera in 2021 [J]. Biodiversity Science, 2022, 30(8): 37-45.
[8] Zhang Z Q. Animal biodiversity: an outline of higher-level classification and survey of taxonomic richness [J]. Zootaxa, 2011, 3148: 1. doi: 10.11646/zootaxa.3148.1.10.
[9] Liu Q N, Xin Z Z, Bian D D, et alThe first complete mitochondrial genome for the subfamily Limacodidae and implications for the higher phylogeny of Lepidoptera [J]. Scientific Reports, 2016, 6(1): 35878. doi: 10.1038/srep35878.
[10] Bian D D, Ye W T, Dai M L, et al. Phylogenetic relationships of Limacodidae and insights into the higher phylogeny of Lepidoptera [J]. International Journal of Biological Macromolecules, 2020, 159: 356-363.
[11] Sun J T, Duan X Z, Hoffmann A A, et al. Mitochondrial variation in small brown planthoppers linked to multiple traits and probably reflecting a complex evolutionary trajectory [J]. Molecular Ecology, 2019, 28(14): 3306-3323.
[12] 贺望兴, 谢小群, 杨普香, 等. 茶园扁刺蛾生防菌筛选、鉴定及其发酵条件优化[J]. 福建农业学报, 2022, 37(11): 1463-1469. He W X, Xie X Q, Yang P X, et alIdentification and culture optimization of effective biocontrol agent onfor tea plantations [J]. Frujian Journal of Agricultural Sciences, 2022, 37(11): 1463-1469.
[13] 王金昌, 类承凤, 陈俊晖, 等. 扁刺蛾核型多角体病毒新分离株的基因组测序与分析[J]. 病毒学报, 2023, 39(1): 185-198. Wang J C, Lei C F, Chen J H, et al. Genome sequencing and analyses of a new strain ofnucleopolyhedrovirus (OxocNPV-Ts) [J]. Chinese Journal of Virology, 2023, 39(1): 185-198.
[14] Bernt M, Donath A, Jühling F, et al. MITOS: improved de novo metazoan mitochondrial genome annotation [J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 313-319.
[15] Laslett D, Canbäck B. ARWEN: a program to detect tRNA genes in metazoan mitochondrial nucleotide sequences [J]. Bioinformatics, 2008, 24(2): 172-175.
[16] Lowe T M, Eddy S R. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence [J]. Nucleic Acids Research, 1997, 25(5): 955-964.
[17] Xia X H. DAMBE7: new and improved tools for data analysis in molecular biology and evolution [J]. Molecular Biology and Evolution, 2018, 35(6): 1550-1552.
[18] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[19] Castresana J. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis [J]. Molecular Biology and Evolution, 2000, 17(4): 540-552.
[20] Guindon S, Dufayard J F, Lefort V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0 [J]. Systematic Biology, 2010, 59(3): 307-321.
[21] Lefort V, Longueville J E, Gascuel O. SMS: smart model selection in PhyML [J]. Molecular Biology and Evolution, 2017, 34(9): 2422-2424.
[22] Cameron S L. Insect mitochondrial genomics: implications for evolution and phylogeny [J]. Annual Review of Entomology, 2014, 59(1): 95-117.
[23] Kong W Q, Yang J H. The complete mitochondrial genome of(Lepidoptera: Bombycidae) [J]. Journal of Insect Science, 2015, 15(1): 48. doi: 10.1093/jisesa/iev032.
[24] 陈鲁. 鳞翅目昆虫线粒体基因组的结构特征分析[D]. 长沙: 湖南农业大学, 2021. Chen L. Structural characteristics of mitochondrial genome in Lepidoptera [D]. Changsha: Hunan Agricultural University, 2021.
[25] 张敏, 赵盼, 尹洁, 等. 小红珠绢蝶线粒体基因组特征及基于线粒体基因组的蝶类高级阶元系统发育关系分析[J]. 昆虫学报, 2017, 60(11): 1324-1338. Zhang M, Zhao P, Yin J, et alMitochondrial genome characteristics and phylogenetic relationship analysis of the higher order members of the butterflies based on mitochondrial genome [J]. Acta Entomoloica Sinica, 2017, 60(11): 1324-1338.
[26] Cao S Y, Wu X B, Yan P, et alComplete nucleotide sequences and gene organization of mitochondrial genome of[J]. Mitochondrion, 2016, 6(4): 186-193.
[27] Liu Q N, Xin Z Z, Zhu X Y, et al. A transfer RNA gene rearrangement in the lepidopteran mitochondrial genome [J]. Biochemical and Biophysical Research Communications, 2017, 489(2): 149-154.
[28] Segovia R, Pett W, Trewick S, et alExtensive and evolutionarily persistent mitochondrial tRNA editing in velvet worms (Phylum Onychophora) [J]. Molecular Biology and Evolution, 2011, 28(10): 2873-2881.
[29] Lavrov D V, Brown W M, Boore J L. A novel type of RNA editing occurs in the mitochondrial tRNAs of the centipede[J]. Proceedings of the National Academy of Sciences, 2000, 97(25): 13738-13742.
[30] 杜会聪, 王瑶, 方加兴, 等马尾松毛虫线粒体全基因组的测定与分析[J]. 林业科学, 2019, 55(12): 162-172. Du H C, Wang Y, Fang J X, et al. Sequencing and analysis of the complete mitochondrial genome of(Lepidoptera: Lasiocampidae) [J]. Forestry Science, 2019, 55(12): 162-172.
[31] 王瑶, 孔祥波, 张苏芳, 等云南松毛虫线粒体基因组全序列测定和分析[J]. 林业科学研究, 2019, 35(5): 11-20. Wang Y, Kong X B, Zhang S F, et al. Sequencing and analysis of complete mitochondrial genome ofLajonquiere (Lepidoptera: Lasiocampidae) [J]. Forestry Science Research, 2019, 35(5): 11-20.
[32] 杨金宏, 谢满超, 文欣茹, 等茶网蝽线粒体基因组全序列测定及系统发育分析[J]. 茶叶科学, 2022, 46(6): 839-850. Yang J H, Xie M C, Wen X R, et alThe complete mitochondrial genome sequence and phylogenetic analysis of the[J]. Journal of Tea Science, 2022, 46(6): 839-850.
[33] Li P W, Wang X Q, Chen S C, et al. The complete mitochondrial genome of the tea lace bug,(Hemiptera: Tingidae) [J]. Mitochondrial DNA Part B, 2017, 2(2): 607-608.
[34] Hua J M, Li M, Dong P Z, et al. Comparative and phylogenomic studies on the mitochondrial genomes of Pentatomomorpha (Insecta: Hemiptera: Heteroptera) [J]. BMC Genomics, 2008, 9: 610. doi: 10.1186/1471-2164-9-610.
[35] Li H, Liu H Y, Song F, et alComparative mitogenomic analysis of damsel bugs representing three tribes in the family Nabidae (Insecta: Hemiptera) [J]. Plos One, 2012, 7(9): e45925. doi: 10.1371/journal.pone.0045925.
[36] Wang Y, Huang X L, Qiao G X. Comparative analysis of mitochondrial genomes of five aphid species (Hemiptera: Aphididae) and phylogenetic implications [J]. Plos One, 2013, 8(10): e77511. doi: 10.1371/journal.pone.0077511.
[37] 郭仲龙, 袁明龙. 半翅目昆虫线粒体基因组学研究进展[J]. 中国科学: 生命科学, 2016, 46(2): 151-166. Guo Z L, Yuan M L. Research progress of mitochondrial genomes of Hemiptera insects [J]. Scientia Sinica (Vitae), 2016, 46(2): 151-166.
[38] Jiang H Y, Chen S C, Peng P, et al. The complete mitochondrial genome of a slug moth,(Lepidoptera: Limacodidae) [J]. Mitochondrial DNA Part B, 2019, 4(1): 320-321.
[39] Jiang H Y, Chen S C, Hu X, et al. Characterization of the complete mitochondrial genome of the tea slug moth,(Lepidoptera: Limacodidae) [J]. Mitochondrial DNA Part B, 2022, 7(8): 1545-1547.
[40] Peng S Y, Zhang Y, Zhang X C, et alComplete mitochondrial genome of(Lepidoptera: Limacodidae) [J]. Mitochondrial DNA Part B, 2017, 2(2): 534-535.
[41] 陈晓晓, 袁周伟, 苑晓伟, 等. 叶蝉线粒体基因组全序列结构研究进展[J]. 基因组学与应用生物学, 2020, 39(6): 2565-2577. Chen X X, Yuan Z W, Yuan X W, et al. Advances in mitochondrial genome complete sequence structure of leafhopper [J]. Genomics and Applied Biology, 2020, 39(6): 2565-2577.
[42] Li X, Wiens J J. Estimating global biodiversity: the role of cryptic insect species [J]. Systematic Biology, 2023, 72(2): 391-403.
[43] 肖强. 茶园害虫“双胞胎”—茶尺蠖和灰茶尺蠖的识别[J]. 中国茶叶, 2019, 41(11): 11-12. Xiao Q. Identification of "twins" of pests in tea garden-tea inchworm and grey tea inchworm [J]. China Tea, 2019, 41(11): 11-12.
[44] 张家侠, 孙钦玉, 葛超美, 等. 4种性诱剂诱芯对茶园尺蠖的引诱与预测效果[J]. 江苏农业科学, 2018, 46(20): 86-88. Zhang J X, Sun Q Y, Ge C M, et al. The lure and prediction effect of four kinds of sex attractants on inchworm in tea garden [J]. Jiangsu Agricultural Sciences, 2018, 46(20): 86-88.
[45] Furlong F M. Knowing your enemies: integrating molecular and ecological methods to assess the impact of arthropod predators on crop pests [J]. Insect Science, 2015, 22(1): 6-19.
[46] 颜亨梅, 钟文涛. 动物捕食性天敌摄食分析方法的研究进展[J]. 生命科学研究, 2021, 25(1): 1-8. Yan H M, Zhong W T. Research progress of feeding analysis methods for predatory animals [J]. Life Science Research, 2021, 25(1): 1-8.
The Complete Mitochondrial Genome Sequence and Phylogenetic Analysis of
JIANG Hongyan1, CHEN Shichun1, LIAO Shuran1, CHEN Tingxu1, YANG Puxiang2, XIE Xiaoqun2, WANG Xiaoqing1*
1. Tea Research Institute of Chongqing Academy of Agricultural Sciences, Chongqing 402160, China; 2. Jiangxi Cash Crops Research Institute, Nanchang 330203, China
is an important agricultural and forestry pest in China with characteristics of wide distribution, polyphagy, and high damage. The purpose of this study was to report the mitochondrial genome ofcollected from Jiangxi, investigate its diversity and difference, and explore the evolutionary characteristics of Limacodidae insects. After Sanger sequencing, thecomplete mitochondrial genome sequence ofwas obtained by splicing, correcting and annotating, and the phylogenetic tree of 26 moth species in 17 families of Lepidoptera was constructed based on the protein sequences. The complete mitochondrial genome sequence was 15 540 bp in size, encoding 37 genes, including 13 protein-coding genes, 2 ribosomal RNAs, 22 transfer RNA genes, and 1 control region of 425 bp. The gene arrangement is the same as that of the Ditrysia moths. By comparing the similarity of the full sequence and protein-coding genes of the mitochondrial genomes with other moths, the results show that the similarity betweenandwas the highest, and that betweenandwas the lowest. Phylogenetic analysis shows that the closest relationship ofwas with, followed by, and all the moths from Lepidoptera were clusteredinto one branch. This study provided a scientific basis for further research on the origin, genetic diversity, migration, and differentiation of, as well as its resistance to pesticides.
Limacodidae,, mitochondrial genome, phylogeny
S571.1;S435.711
A
1000-369X(2023)04-460-13
2023-04-13
2023-05-30
国家茶叶产业技术体系(CARS-19)
江宏燕,女,助理研究员,从事茶树害虫综合防控研究,jianghy925@sina.com。*通信作者:wangxiaoqing2891@126.com

