医院重症医学专病库的建设与应用
□ 武亚琴WU Ya-qin 卢晟晔LU Sheng-ye 赵慧颖ZHAO Hui-ying 闫华YAN Hua刘丽红LIU Li-hong 赵翔宇ZHAO Xiang-yu
Objective To construct a specialized database of critical care medicine with scientific research and clinical application significance. Methods Based on the clinical research data platform constructed by the hospital, the business system data related to critical care medicine were mined. The standard data set model of specialized diseases in critical care medicine was established, so as to realize the automatic collection and integration of data in specialized disease database and form a critical care medicine database and application platform with data analysis and retrieval capabilities. Results Through data management, the hospital's 10-year-old critical care medical data was reconstructed into a specialized database of critical care medicine that met the needs of critical care research, and the system automatically and continuously generated high-quality, standardized and structured critical care patient diagnosis and treatment data. Conclusion The database and application platform of critical care medicine can realize the functions of case retrieval, course browsing and patient data export of critical care medicine, support the research of related topics of critical care medicine, assist researchers to establish clinical prediction models, and apply them in clinic to verify their auxiliary diagnosis and treatment effects.
重症医学科收治的均为危重症患者,由于其疾病的复杂性,需要记录的信息量非常巨大。很多医生想通过挖掘临床数据进行科学研究,但是因数据存储分散、时间精力有限等原因无法获得高质量的临床数据,导致研究工作无法开展。在国际上,美国、欧洲、日本等国家在疾病数据库领域起步较早,已经建立了不同疾病的国家级数据库,美国麻省理工学院与贝斯以色列女执事医疗中心联合研发的重症监护医学信息数据库(MIMIC),已有MIMIC-II、MIMIC-III、MIMIC-IV三个版本,为重症疾病研究与发展奠定了基础。其中,MIMIC-IV包含2008年至2019年(含)的数据,其数据源来自两个院内数据库系统:一个是定制的医院范围的数字电子健康记录(EHR)、一个是重症监护室(ICU)特定的临床信息系统[1],其为真实世界研究 (RWR)提供了数据基础[2]。一个较好的专病数据库,应可以为科研人员提供大量的优质的真实世界数据用于疾病预测模型搭建、疾病预后情况分析等[3-4]。本文研究建立的重症医学数据库,实现以重症患者为中心,将重症患者所有临床诊疗数据的整合与集中展现,为重症医学临床诊疗、科研、决策提供数据支持。
医院重症医学专病库技术架构与实践
重症医学专病库基于医院临床科研数据库搭建,其应用的技术架构能够解决数据资源的海量化、异构化,应用需求多样化、复杂化等现实问题。技术架构包含ODS数据层(ODS,operational data store,数据存储)、数据处理及存储层、数据应用层、数据安全层,如图1所示。
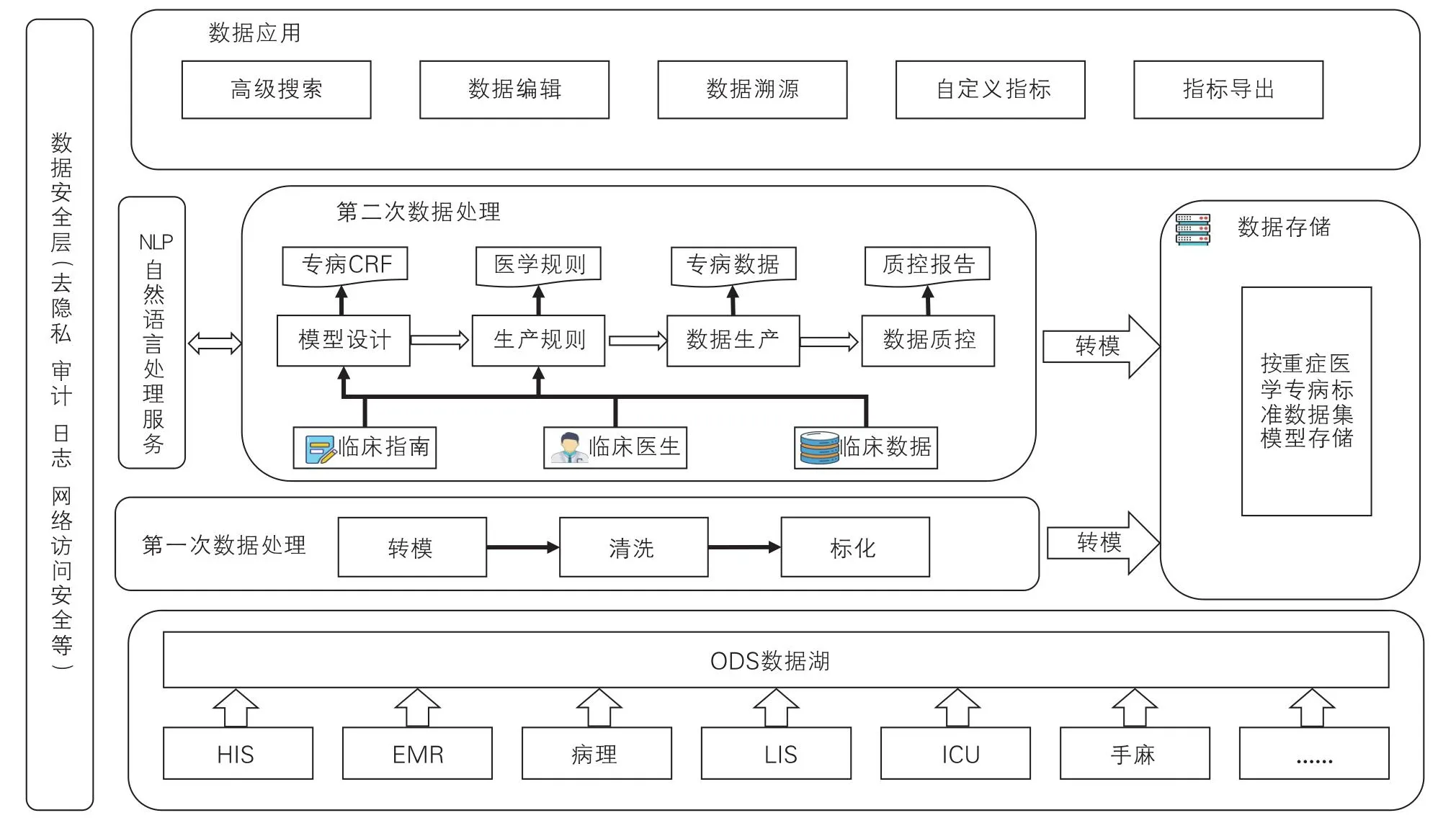
图1 重症医学专病库技术架构
1.ODS数据层。ODS数据层存储了从业务系统获取的最原始的数据,也称之为医院的临床科研数据湖。汇集了18个临床业务信息系统的业务数据,超过1100张业务表,以及近450万份数据文件,总数据量达到1500GB。为了不影响业务系统的正常运行,通过建立备份库的方式,并制定了增量同步机制,实现了“T+1”天准实时的数据同步。
医院依照科研及临床需求,建立了以临床研究通用模型为标准的数据库(common data model,CDM)。从患者历次就诊的维度,共计70余类患者相关的人口学、临床、诊断、检查、检验、用药、手术、症状等面向临床研究的医院真实世界数据[5]。由于不同专病数据模型的差异,比如模型数据项的多少、数据项精细度的要求等,临床研究通用数据模型并不能满足各个专病数据需求,所以需要针对于不同专病建立单独的数据库,但其数据源均来自于ODS数据层,对不同的业务数据集进行不同程度的进一步处理。
2.数据处理及存储层。主要任务是以医院CDM库为基础,按照重症医学专病库标准数据集,对多源异构数据进行采集、治理、入库,将符合纳排条件的重症医学患者的原始数据按照制定的重症医学标准数据集模型映射、重构。包括按照专病模型需要,对专病库所需数据的归一化和结构化处理;对现有数据中存在的不完整、不准确和不标准的“脏”数据进行清洗;基于医学自然语言处理技术对非结构化数据进行语义解析和抽取;对通用CDM模型数据项二次计算存储等。简要生产方式如图2所示。
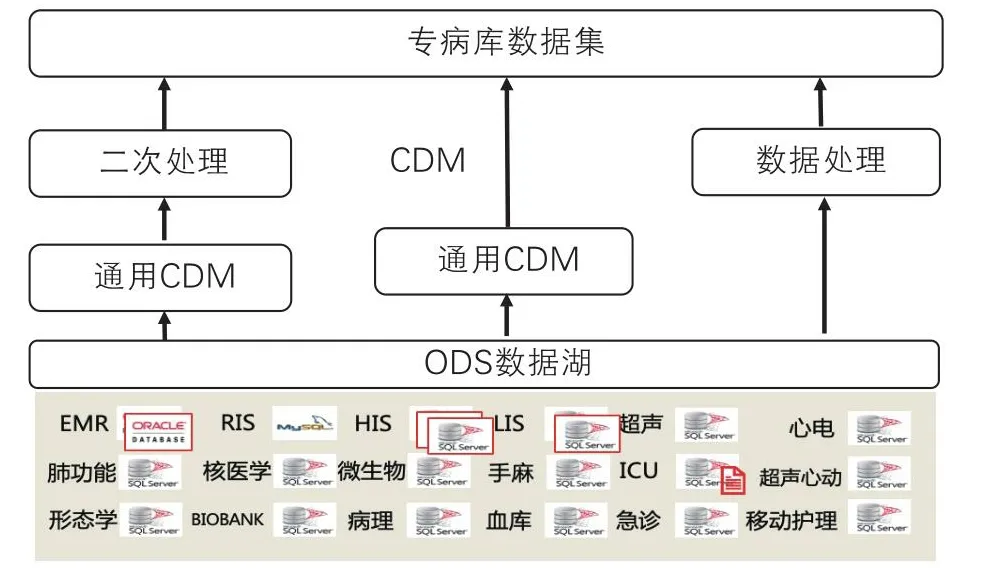
图2 专病库数据集生产过程
2.1 重症医学标准数据集模型。参考国内外行业标准、疾病诊疗指南等规范,调研重症医学研究方向课题科研数据需求,共同制定面向科研与临床的重症医学专病标准数据集模型,本文也参考了MIMIC数据库的数据集模型,包括元数据、数据结构和相关术语标准。重症医学统一标准数据集模型见表1,梳理出共13个大类25个子类235个字段的结构化数据集。在实践过程中,与临床专家明确各个数据项提取的医学规则,定义了数据格式、字段长度、值域、内容约束等,为重症临床患者数据的标准化、规范化收集、利用与数据共享奠定了坚实的基础。
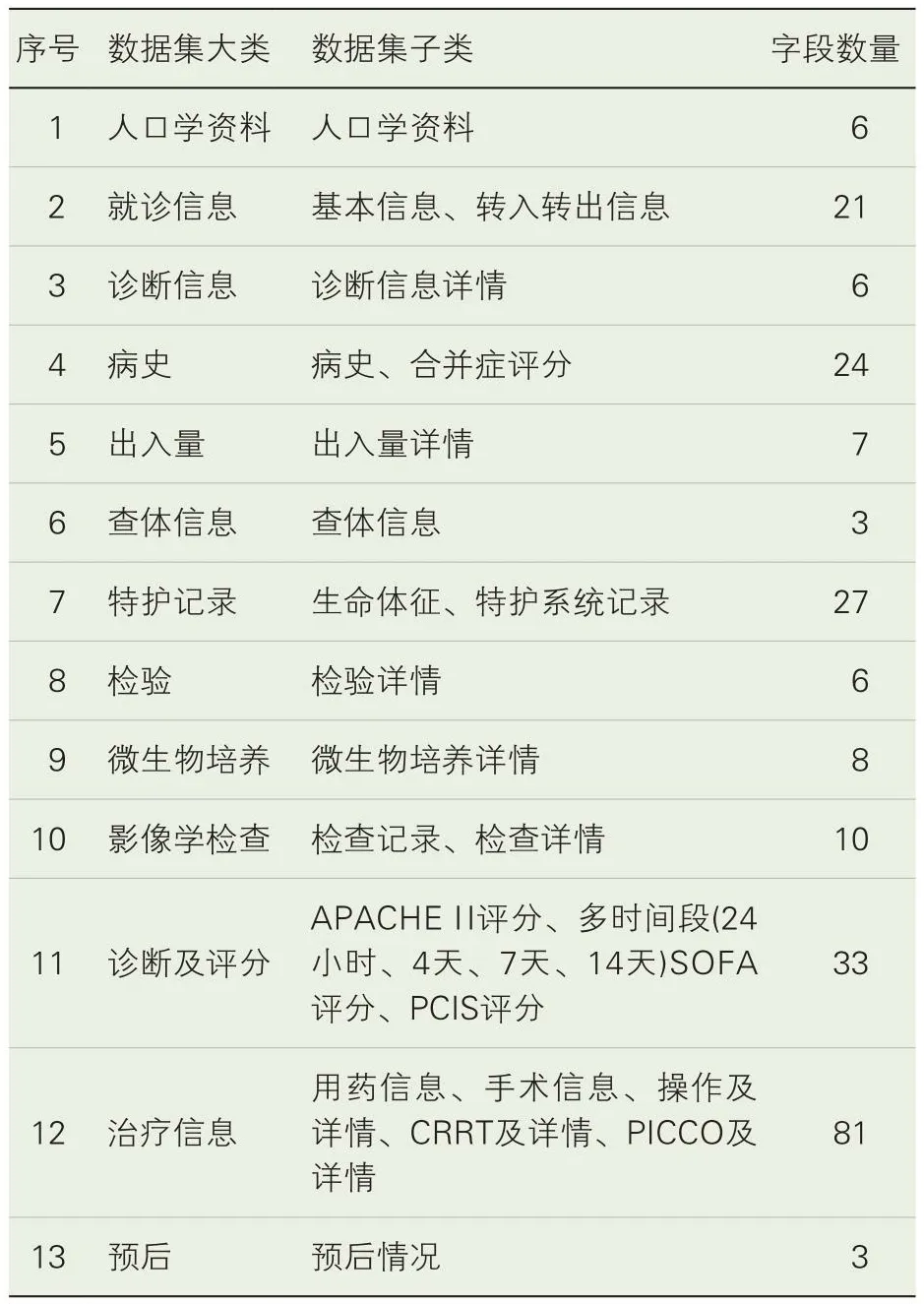
表1 重症医学标准数据集模型
2.2 重症医学专科数据处理。重症医学专科数据大部分存储在医院重症监护信息系统(ICU系统)中,但是医院ICU系统建设较早,系统架构老旧,主要存在的问题有:(1)非常多的关键数据未实现结构化存储,比如通气模式、微量泵用药、出入量等,是以字符串形式存在于文件中,用“$”“|”“@”“^”等特殊符号代表特定含义,通过系统特有的逻辑,转换为前台记录单展示出来。当有导出或者分析的需要时,非结构化存储就成了关键的制约因素,大量宝贵的临床科研数据被“封锁”在特殊格式的文件中,急需解锁出来。(2)有些关键的评分数据系统中完全没有,依赖于大数据平台自动计算,比如不同时段SOFA评分等。(3)源数据存在修改不规范,导致数据有缺项、错项,数据质量较差。
针对以上问题,在建设过程中进行逐一击破,以通气模式规则解析作为突破点,对历史数据库各类文件的规则进行梳理,利用nlp自然语言处理技术,并编写自动解析脚本,有效提升了解析效率,将沉淀了10年的宝贵的历史数据进行了深度清洗、挽救,实现了专病数据与医院其他系统数据的整合利用,真正地成为有研究和临床应用价值的医疗数据,提升了专病库数据质量。
2.3 通用CDM模型数据项二次处理。在重症医学标准数据集模型中,需要对通用CDM模型数据项进行二次处理,将已初步清洗入库到通用CDM模型数据库的数据项依据重症医学标准数据集模型进行二次加工、生产,并通过严格的数据质控,以满足专病数据库要求。这个环节主要是解析解释医学规则,将符合临床医生需求的医学规则转换为前文所提到的标准模型,并将互相依赖的相关规则生成数据库查询语言,生产出专病数据,经过多维度的数据质控,存储到医学专病数据库。
2.4 数据核查与质控。通过对源系统的改造,数据抽取规则的制定,实现自动、持续性地生成高质量、标准化、结构化的重症患者诊疗数据。专病库数据生产后,需经过严格的质控环节,包括自动数据质控、质控异常数据修复、周期全量数据更新、人工质控、课题质控、随机业务质控等,确保专病数据库的完整性、及时性和可用性。见表2。
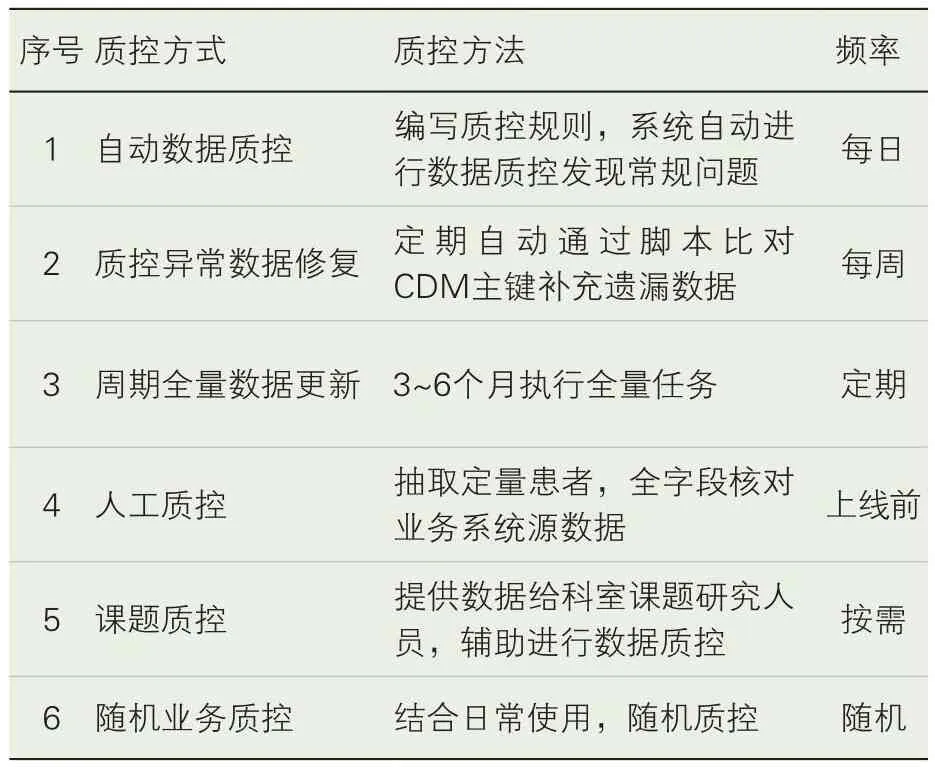
表2 数据质控方法和频率
3.数据应用层。数据应用层与院级临床研究数据平台的数据应用层深度融合,具备数据搜索、数据编辑、数据溯源、自定义指标、数据导出等功能,更好的服务于科研与临床。
3.1 数据检索。据检索功能涉及ElasticResearch、Spark等各种技术的组合。ElasticResearch是一个基于Lucene构建的分布式、高扩展、高实时的搜索与数据分析引擎,支持通过HTTP使用JSON进行数据索引。可以用于全文搜索、结构化搜索及分析。支持and、or、排除等多种关系,字段支持全匹配、模糊匹配等条件,且支持大数据平台及专病库字段联合检索。联合检索的功能之所以能够实现,是因为专病库是基于医院临床科研数据湖搭建,可用数据和检索范围大大扩展。
除数据库已存在的数据项外,还支持科研人员根据研究需要自定义指标项,选择检索结果范围,通过结果处理函数来具体限制条件命中的位置,比如第一次、最后一次、第n次、倒数第n次等,以及递归引用自定义指标形成第2次、第3次引用等形成新的自定义指标,可以更加灵活、精确的定义纳排条件,获得更具有科研意义的数据结果。例如,在系统中,可定义患者的血糖在某时间段内的峰值出现时间,作为时间类型的自定义指标,系统能够基于数据库存储的时间序列数据,根据前后数据值对比计算出来,该指标可以继续用于联合检索及数据导出。
3.2 科研数据导出。让临床医生或研究者先期了解数据库中的数据内容是提高专病数据利用率的重要手段,所以需充分的对用户进行反复多次的培训、讲解、试用,并设计足够灵活的数据表格,方便科研所用,保持持续的科研数据产出,为科研项目、发表论文等提供数据支撑。通过模块化数据整合和分析,从而辅助科室开展真实世界临床研究。围绕临床诊疗难点及相关基础研究,提升重症医学相关科研能力。专病库的数据导出支持按纵表导出,能够更适合作为深度学习的数据源。
3.3 科研与临床应用案例——临床预测模型。重症医学专病库上线后,支持了两项临床预测模型的建立,包括脓毒血症死亡风险预测模型、有创通气拔管时机预测模型。重症医学科科研人员采用深度学习的方法,深入挖掘数据在不同形式下的特征,使用专病数据库提供的医疗时序数据参数集进行学习、训练,形成了具有较好预测效果的临床预测模型。医院重症医学专病库提供的数据集合有173项静态数据,包括人口学信息、并发症信息等,294项时序数据,包括重症监测体征、实验室指标等,确保了真实医疗场景下的可用性。
4.数据安全层。应用数据加密技术,对数据信息系统中的敏感数据字段(病人联系方式、病人姓名、病人住址等)进行脱敏、加密处理。从应用层、数据库维护层、存储层、网络传输等层面对数据安全进行管控,实现字段级的访问控制,以保证信息数据安全。
制定数据安全管理制度,管理流程由科研归口管理部门和数据归口管理部门联合管理,包括科研处、医学信息中心、伦理办公室等,对用户权限进行层级授权管理。
总结
形成了较完整的结构化、标准化的重症医学数据库。通过数据治理,将医院沉淀10年的重症数据重构为符合重症临床与科学研究需要的医院重症医学数据库,形成了13个大类25个子类219个数据项的结构化数据集。建立的重症医学数据库共纳入了2012年11月至 2022年10月的4万余例住院患者的诊疗数据。经过验证,数据库数据质控满足临床及科研要求,并实现了自动、持续性地生成高质量、标准化、结构化的重症患者诊疗数据。重症医学数据应用平台操作便利,实现了病例检索、患者全病程浏览和患者数据集导出等功能。目前,已支持了院内多项课题研究,辅助科研人员建立两项临床预测模型,并应用于临床,促进科研成果的临床转化,对科研应用及临床业务应用具有重要意义[6-7]。

