澳洲坚果种质资源的遗传多样性分析
李志强, 吴 超, 耿建建, 马 静, 宫丽丹
(云南省热带作物科学研究所, 云南 景洪 666100)
澳洲坚果(Macadamiaspp.)是山龙眼科(Proteaceae)澳洲坚果属(Macadamia F. Muell)植物,是重要的经济作物[1]。澳洲坚果属有4个种(M.integrifoliaMaiden &Betche,M.tetraphyllaL.A.S.Johnson,M.ternifoliaF.Muell 和M.janseniiC.L.Gross &P.H.Weston)。其中M.integrifolia,M.tetraphylla及其杂交种被广泛用于商业生产中[2-3],M.jansenii和M.ternifolia的果仁较小且因氰化物含量高而尚未直接用于澳洲坚果育种及生产中[4]。作为一个外来引进树种,我国大陆地区于20世纪80年代开始澳洲坚果的引种试种[5],目前已是全世界澳洲坚果种植面积最大的国家,种植区域主要分布在云南、广西、广东及贵州等省份。
种质资源的遗传多样性分析是种质资源创新与利用的基础。国内外学者对澳洲坚果的分子遗传多样性已经开展了大量的研究。同工酶最早被用于澳洲坚果的遗传变异分析[6-7]。随后,Vithanage等[8]通过8对STS(Sequence Tagged Sites)引物分析了76份澳洲坚果种质的亲缘关系,结果表明,A 16和A 4的遗传距离较近,拥有共同的母本。Peace等[9]认为,相对于同工酶、RAPD标记和STMS标记,RAMiFi(Randomly Amplified Microsatellite Fingerprinting)标记在澳洲坚果遗传分析中更有优势。Steiger等[10]利用AFLP标记分析了澳洲坚果属的3个种及1个近缘种,对三叶澳洲坚果是全缘叶澳洲坚果变种的观点提出怀疑。AFLP标记亦被用于肯尼亚的26份澳洲坚果变异分析[11]。RAF标记被用于分析代表南非澳洲坚果遗传多样性的38份种质,更正了个别混淆的品种[12]。郭凌飞[13]利用 ISSR和SRAP标记分析了123份澳洲坚果种质的遗传多样性。Machado等[14]利用RAPD标记对巴西和澳大利亚两个群体的遗传变异进行分析,发现大多数的变异发生在群体内部。基于高通量测序技术silicoDArT和SNP标记亦被用于澳洲坚果遗传多样性和群体结构研究[15-17]。
SSR标记被认为是遗传研究中最好的方法之一[18]。基于SSR标记的澳洲坚果遗传分析亦有报道[19-21]。本研究利用本课题组开发的22对SSR引物,通过毛细管电泳检测,分析前期收集保存的85份澳洲坚果种质资源的分子遗传多样性,这85份澳洲坚果种质资源材料主要包含从美国和澳大利亚引进的种质,还包含国内通过实生选种及杂交育种方法获得的优异单株。本研究通过揭示供试群体种质的遗传背景,以期为澳洲坚果的杂交亲本选择及遗传改良提供理论依据。
1 材料与方法
1.1 材 料
供试的85份澳洲坚果种质叶片采自农业部景洪澳洲坚果种质资源圃和云南省热带作物科学研究所澳洲坚果品种试验基地(表1)。
1.2 DNA提取及SSR扩增检测
采用CTAB法提取DNA,提取后的DNA经琼脂糖凝胶电泳检测后用于试验。22对SSR引物由本课题组设计并筛选所得(表2),在通用生物系统有限公司合成FAM标记的荧光引物。PCR反应体系:10×Buffer 2 μL,2.5 mmol/L dNTP 0.4 μL,正反引物各0.3 μL,DNA模板2 μL(20 ng/μL),5 UTaq0.2 μL,ddH2O 14.8 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,65 ℃到50 ℃降落复性30 s,72 ℃延伸40 s,35个循环;最后72 ℃延伸3 min。PCR产物经琼脂糖电泳检测后使用3730 XL测序仪进行毛细管电泳检测。
1.3 数据分析
利用Genemarker V 2.2.0软件中的Fragment(Plant)片段对原始数据进行分析,比较泳道内分子量内标的位置与样品峰值的位置,得到样品的片段大小。将每个样品在各引物的片段大小录入到Excel中。使用POPGENE 32软件进行统计分析,计算多态位点百分率(P)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)等参数。通过POPGENE 32计算出群体的基因型频率,再通过PIC_CALC计算多态性信息含量(PIC)。基于遗传距离,使用MEGA 5软件中UPGMA法进行聚类分析,构建系统发育树。使用STRUCTURE 2.3.3软件分析群体遗传结构,参数设置如下:Length of Burn-in period: 200 000;Number of MCMC Reps after Burnin:1 200 000;Set K:2-10;Number of Iterations:8。
2 结果与分析
2.1 SSR标记分析
利用22对SSR引物对85份澳洲坚果种质进行毛细管电泳检测。共检测到269个等位基因(见表3),平均每对引物12.2个,引物P 202等位基因数最低(6个),引物P 33等位基因数最高(18个),有15对引物检测到10个以上的等位基因。有效等位基因数为1.987 9(P 15)~8.794 9(P 11),平均5.087 2个。Shannon指数(I)为1.020 9(P 15)~2.327 3(P 11),平均值为1.854 3。观测杂合度(Ho)为0.117 6(P 238)~0.882 4(P 35),平均值为0.582 6。期望杂合度(He)为0.499 9(P 15)~0.891 5(P 11),平均值为0.784 5。Nei’s指数范围为0.497~0.886 3,平均0.779。多态性信息含量为0.461 8(P 15)~0.875 4(P 11)。22对引物中,有21对引物属于高多态位点(p>0.5),说明该批高效SSR引物可用于澳洲坚果遗传多样性分析。
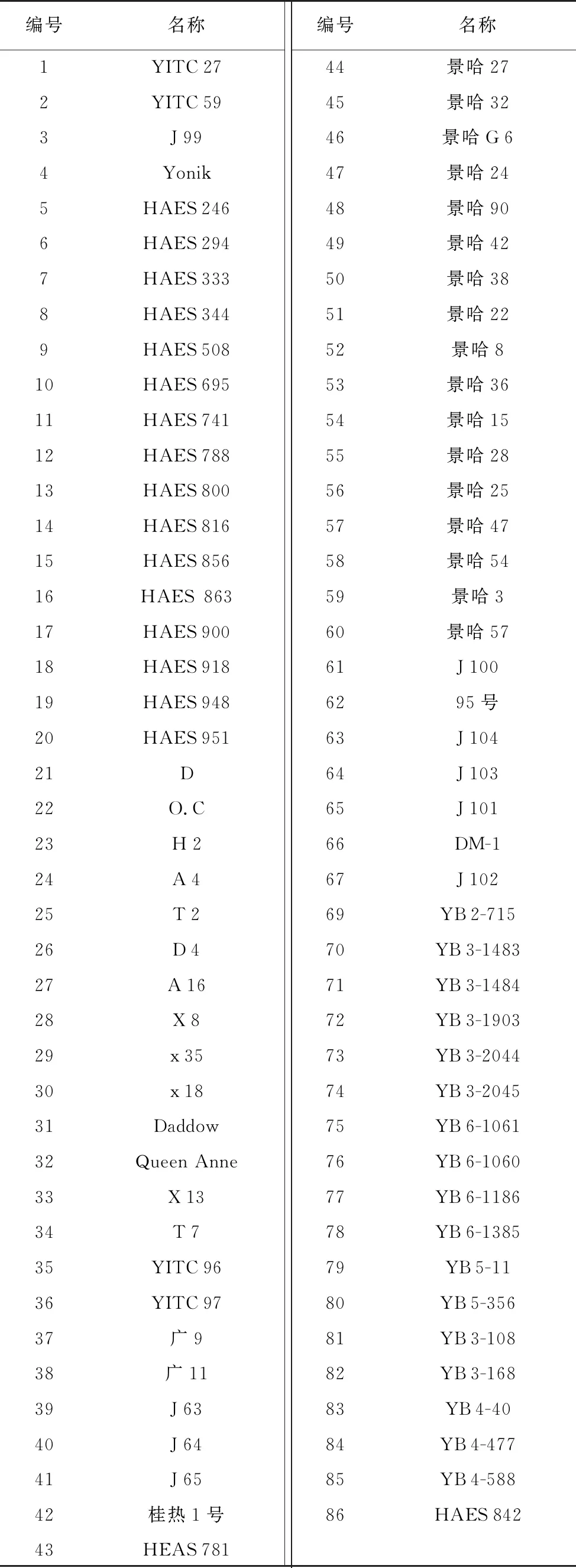
表1 试验材料Table 1 The trial materials
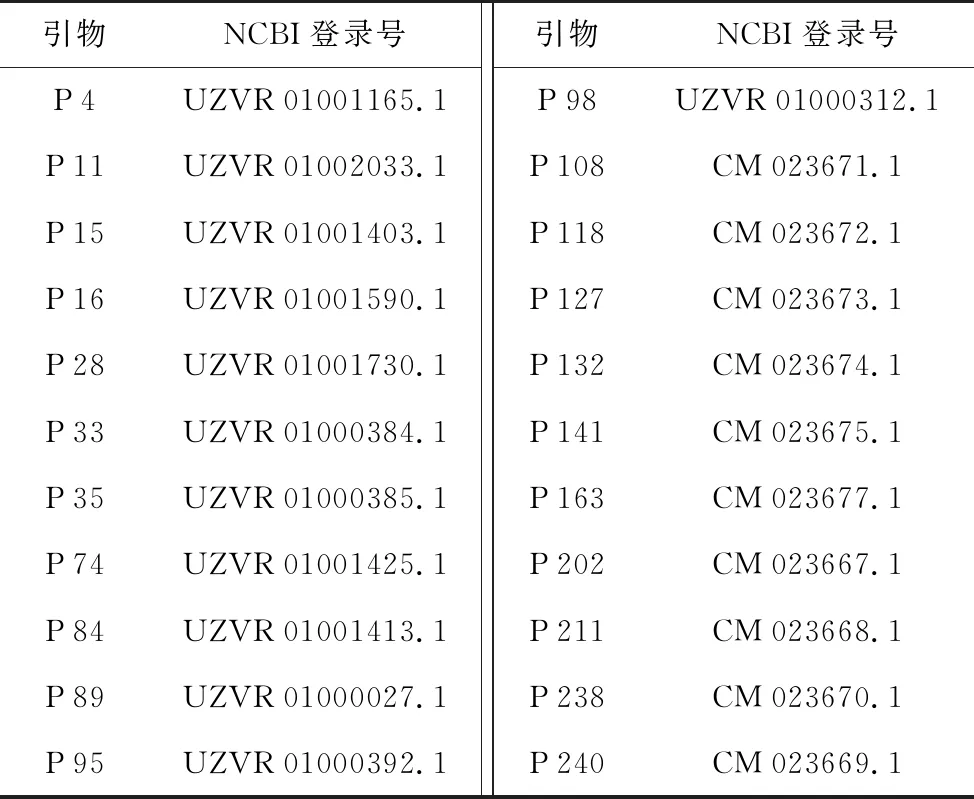
表2 22对SSR引物Table 2 22 SSR primers
2.2 群体间遗传分析
将85份材料大致划分为4个群体:从美国引进种质(pop A)、从澳大利亚引进种质(pop B)、国内实生选种种质(pop C)和国内定向杂交育种种质(pop D)。4个群体的观测等位基因数为5.454 5~7.954 5,有效等位基因数为3.313 3~4.799 8,Shannon指数为1.342 6~1.732 8,Nei’s指数为0.659 3~0.770 6(表4)。相对于其他3个群体,澳大利亚种质在观测等位基因数、有效等位基因数、Shannon指数及Nei’s遗传多样性均为最高。说明澳大利亚群体的遗传多样性最为丰富。分析4个群体的遗传距离和遗传一致度(表5)。遗传距离的变化范围为0.157 5~0.298 6,遗传一致度变化范围为0.741 9~0.854 2。pop B与pop D的遗传距离最小(0.157 5),遗传一致度最高(0.854 2),表明两个群体的遗传关系最近。
2.3 UPGMA聚类及遗传结构分析
基于UPGMA 对85份材料进行聚类(图1),材料间未见有明显的划分规律。澳大利亚种质及美国种质没有各自聚类在一起,分别被划分到不同的组群中,表明聚类划分与地理来源无明显相关性。国内实生选种种质亦被划在不同的组群中。定向杂交种质多数与其亲本之一划为一组,如73号是以10号为母本的杂交优株,26号是69号、83号、84号和85号的亲本之一。
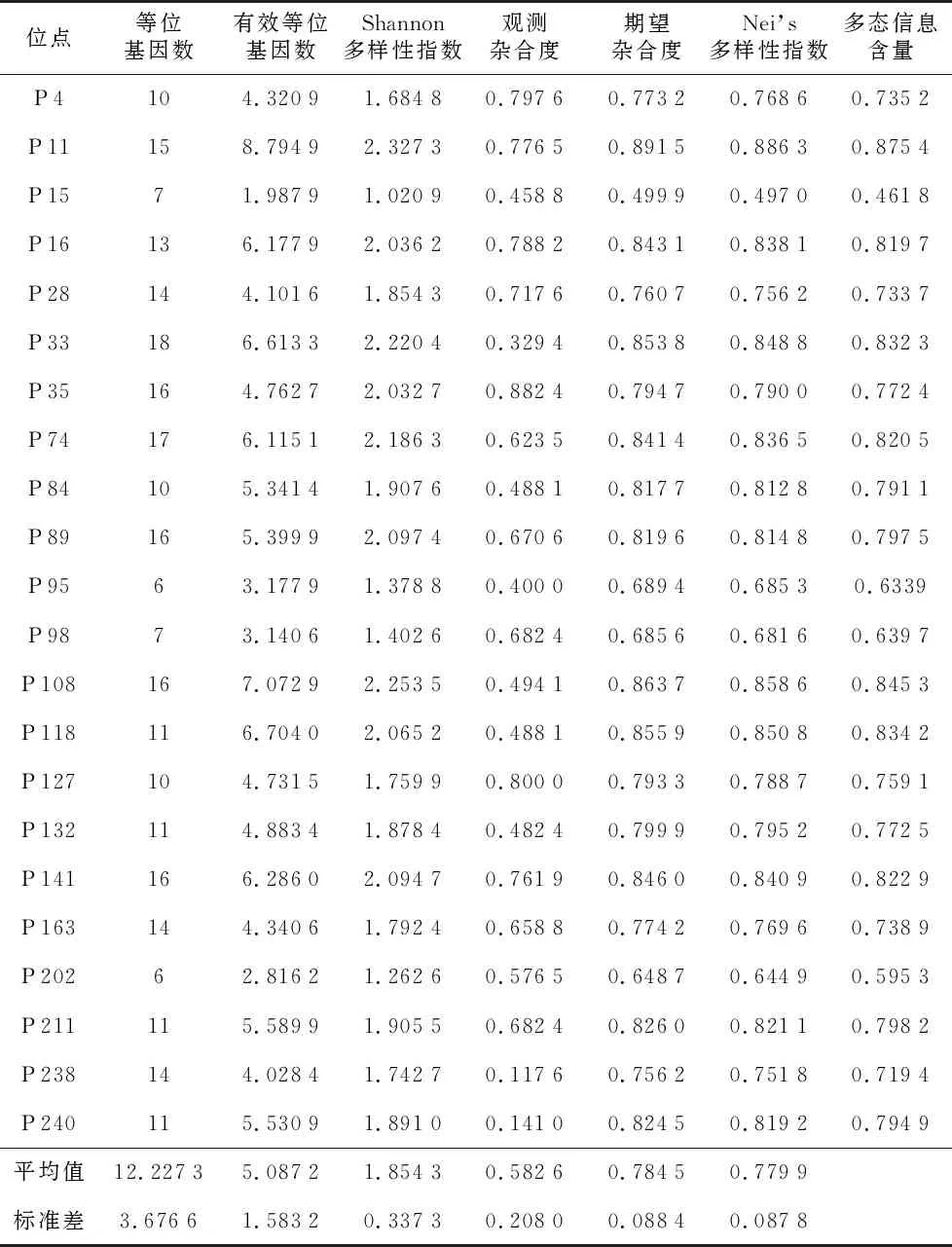
表3 SSR 标记的遗传多样性参数Table 3 Genetic diversity parameters of SSR
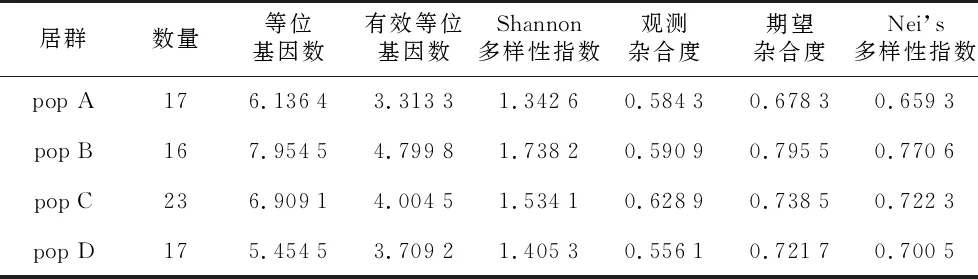
表4 群体遗传多样性参数Table 4 Genetic diversity parameters of populations
应用Structure软件对85份澳洲坚果种质进行群体结构分析(图3)。用ΔK来确定K值,当K=6时,ΔK取得最大值(图2),即群体结构分析将85份种质分为6个类群。类群1至类群6分别包含12、19、10、17、9、18份种质。17份美国种质被分在类群1、类群3、类型4、类群5中,16份澳大利亚种质在6个类群中均有分布,23份国内实生种质被分在类群1、类群2、类群3和类群6中,杂交种质被分在类群2、类群3、类群4中。刘丽华等[22]认为,在群体遗传结构分析中,当遗传组分(Q值)≥0.6时,可认为该材料的遗传背景较为单一。分别统计分析各澳洲坚果种质在各类群中的Q值,85份材料中有51份(60%)种质Q值大于等于0.6,34份(40%)供试种质遗传背景较为复杂。供试的17份杂交优株中,有4份种质显示遗传背景单一,分别为71号、72号、78号和82号。
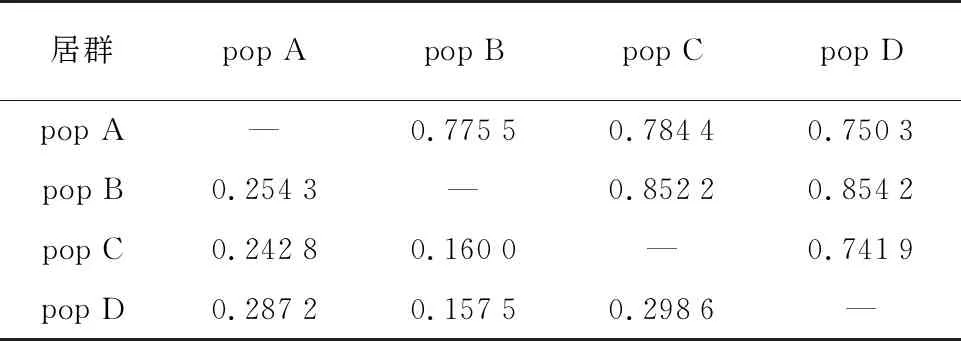
表5 群体间遗传相似系数和遗传距离Table 5 Genetic similarity coefficient and genetic distance between populations
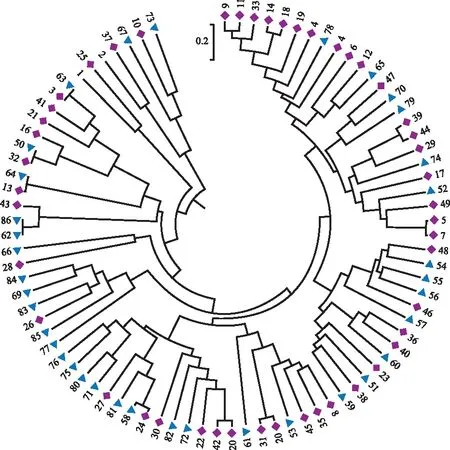
图1 85份澳洲坚果种质的UPGMA 聚类图Fig.1 UPGMA dendrogram of 85 Macadamia ssp. accessions
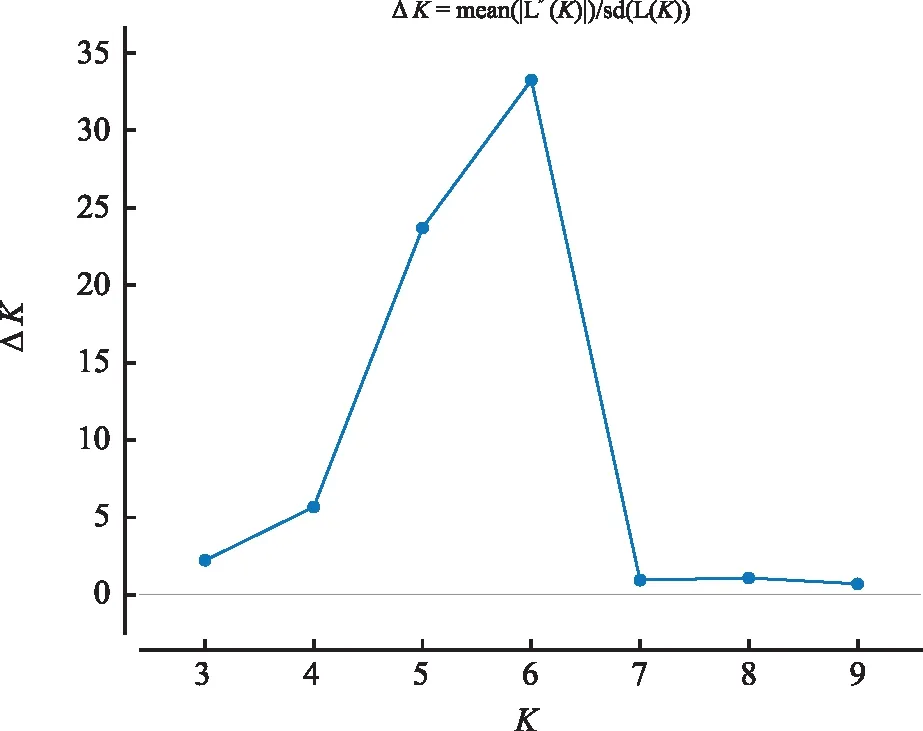
图2 ΔK随 K值的变化趋势Fig.2 Trend of ΔK with K value
3 讨论与结论
聚丙烯酰胺凝胶电泳银染检测和毛细管电泳荧光检测是SSR标记常见的检测方法,两种方法的检测结果理论上是一致的。但在数据的读取和检测的效率上,毛细管电泳都更具优势[23-24]。该研究利用毛细管电泳及已筛选的高多态性SSR引物进行检测,保证了检测的准确性和快捷性。
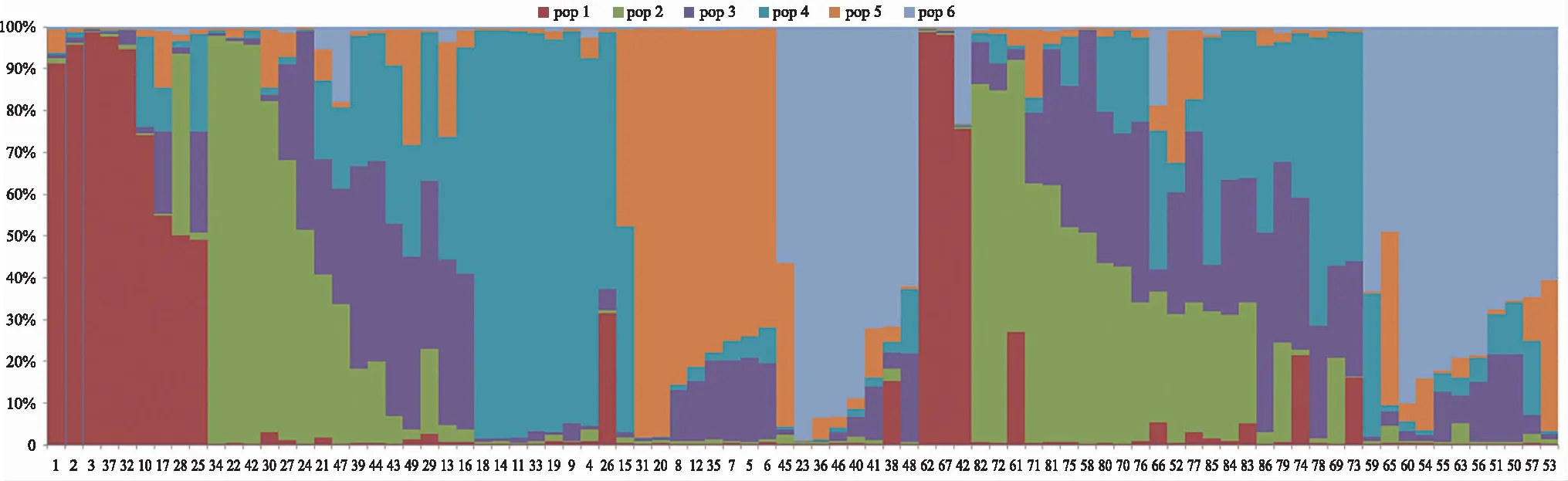
图3 85份澳洲坚果种质资源群体结构图Fig.3 Population structure diagram of 85 Macadamia ssp. germplasms
作为一个外来引进物种,目前我国的澳洲坚果种植面积居世界第一。优良品种是澳洲坚果产业稳定发展的前提,而种质资源收集是品种改良的物质基础。初期我国澳洲坚果种质资源收集以从国外引种为主,随着各国对种质资源保护工作的重视,目前从国外引进新种质十分困难。现在国内澳洲坚果种质资源收集方法逐渐转为筛选国内优异单株和种质创新。本次研究通过比较现有的澳大利亚引进种质、美国引进种质、国内实生特异种质及国内定向杂交优异种质4个群体,研究结果表明,澳大利亚作为澳洲坚果的发源地,其种质群体有最丰富的遗传变异,国内实生特异种质和定向杂交种质均比美国引种种质的遗传多样性高。表明今后对澳洲坚果种质资源的评价,侧重点应在澳大利亚种质及国内优异单株种质群体上。遗传多样性分析有助于了解各种质间的亲缘关系。本次研究的聚类分析中,澳洲坚果种质246(5号)与种质333(7号)、种质A 4(24号)和种质A 16(27号)划分在一起,亲缘关系较近,这与Aradhya等[7]和Peace等[9]结果一致,而种质800(13号)未能与其聚类到一起。种质741(11号)与种质344(8号)亲缘关系较远,与Vithanage等[8]结果一致。不同标记方法所揭示的遗传多态性信息不同,可能得出不同的分析结果。SSR标记以其共显性、稳定性被广泛用于遗传多样性研究。本次研究利用22对高效SSR引物对85份澳洲坚果种质进行评价分析,有助于了解不同群体种质的遗传背景,对澳洲坚果种质资源收集利用及品种改良有指导意义。

