玉米灰斑病病原菌胁迫下玉米转录组测序的SNP和InDel分析
蒋 滔, 张晓梅, 杨 梅, 张 志, 邱红波
(1.贵州大学农学院, 贵阳 550025; 2.云南省德宏州农业技术推广中心, 云南 芒市 678400)
玉米灰斑病(Gray leaf spot,GLS) 是玉米十大常见病害之一,它是由病菌Cercosporazeae-maydis和Cercosporazeina引起的,是全球玉米种植区最严重的玉米叶病害之一[1-2]。2009年玉米全基因组测序的完成促进了玉米分子标记辅助育种研究,也推动了玉米新品种的培育。同时,玉米灰斑病抗性属于数量性状,Pedro等[1]、Nsibo等[2]通过不同类型的分子标记对其展开了研究,其中图位克隆的方式克隆未知抗病基因成为当下研究热点,而SNP和InDel分子标记作为图位克隆的重要工具,通过对转录组测序结果进行SNP和InDel标记分析,可为开发筛选玉米抗病种质资源标记奠定基础。
SNP/InDel作为新基因或等位基因变异驱动因素的宝贵来源,当结果表型表现出有利特征时,可以通过自然或人工方式选择这些优良变异[3]。利用下一代测序技术进行基于转录组测序的标记搜索,可以快速发现专注于编码区域的多态性,避免了高度重复的基因组区域,使其成为检测变异/突变的强大工具[4-5]。尽管从转录组测序数据中获得了大量的研究成果和丰富的信息,但其在识别SNP和InDel等分子标记方面的应用仍然很少[6]。SNP/InDel是植物基因组中最丰富的DNA变异,因其高效性而被广泛应用于植物改良和作物育种[7-8]。与其他分子标记相比,SNP和InDel便于基因组序列或转录组序列的比较[9-10]。单核苷酸多态性 (SNP) 标记是单个碱基发生变异的多态性序列,具有突变率低、遗传稳定性高及可以自动化检测等特点,使标记在广泛应用时更为有效[11-12]。插入/缺失多态性(InDel)标记是根据核苷酸片段的插入或缺失而开发的,有分布广、可重复性高、密度高、成本较低、变异率低、多态性强且易于检测等优点[13-14]。目前,SNP和InDel已应用于研究许多优良物种的遗传多样性分析、遗传图谱构建、关联作图分析和标记辅助选择(MAS)育种[15-16]。随着使用新一代测序技术收集越来越多的基因组或转录组数据,开发SNP和InDel变得越来越容易。到目前为止,已经使用新一代测序技术获得了很多与玉米抗病过程相关的转录组数据,并在NCBI数据集中发布[17-18]。本研究使用可用的玉米转录组数据集识别和评估玉米灰斑病病原菌胁迫下SNP和InDel标记,以玉米灰斑病抗、感自交系T 32和J 51为材料,通过高通量测序技术,获得了大量转录组信息,对SNP和InDel标记进行分析,旨在开发出更丰富的分子标记,为抗灰斑病的SNP 和InDel标记的后续开发利用,为玉米抗灰斑病分子标记辅助育种以及候选基因功能分析提供理论参考。
1 材料与方法
1.1 试验材料
选用玉米(ZeamaysL.)灰斑病抗、感自交系T 32和J 51为试验材料,均由贵州大学玉米研究所提供,于2020年在云南省芒市(98.58°E,24.43°N)玉米灰斑病重发区种植玉米材料作为玉米灰斑病病菌自然接菌处理,在芒市无灰斑病发生地种植作为不接菌处理。在抽雄期进行穗位叶取样并送往深圳华大生物公司进行转录组测序。
1.2 试验方法
使用擎科生物科技有限公司的植物总RNA提取试剂盒提取RNA,详细步骤参照说明书。对用于转录组测序的6组样本材料(T 32的3个生物学重复,命名为T 32_Repeat_1、T 32_Repeat_2和T 32_Repeat_3;J 51的3个生物学重复,命名为J 51_Repeat_1、J 51_Repeat_2、J 51_Repeat_3)的总RNA进行mRNA纯化、反转录、转录组测序(在深圳华大基因有限公司完成)等。
测序后的数据通过测序平台的软件将其转换为FASTQ格式的原始数据,再对每个样品的原始数据分别进行整理统计,包括样品名称、Q 20、Q 30和GC等。使用过滤软件SOAPnuke对原始数据 (Raw reads) 进行过滤处理,把获得的高质量数据 (Clean reads) 比对到参考基因组 (B 73_Ref Gen_V 4) 上,使用GATK检测样品中的SNP和InDel信息,利用HISAT 2软件进行数据预处理;利用Haplotype Caller对SNP和InDel变异位点进行检测[19]。最后统计SNP和InDel的变异类型及分布情况,采用Excel 2010软件进行数据分析与作图。
1.3 玉米转录组测序的SNP 和InDel分析
经过处理后的转录组数据通过Varscan软件获取SNP和InDel位点,再对得到的SNP和InDel进行统计分析,包括: 1) 样品转录组测序质量分析; 2) SNP特征分析; 3) InDel分析; 4) SNP/InDel在玉米基因组中Up 2 k,Exon,Intron,Down 2 k,Intergenic 等5种功能元件上的区域分布; 5) 差异表达基因GO 和 KEGG 功能分类分析。
2 结果与分析
2.1 样品转录组测序质量分析
测序后6组样品共获得39.68 G的可用数据,经过滤得到T 32和J 51最终可用的reads 数量分别是130 369 594个和134 221 272个,占原始数据的95.8%和96.7%。碱基质量值Q 20大于94.5%,Q 30大于88.15%(表1)。两组样本通过triniy组装得到61 947条转录本,平均长度为1 639个核苷酸,用Tgicl 软件去除冗余得到37 268条Unigene,平均长度为1 728个核苷酸。将所有的Unigene 按照从长到短排序并依次相加,当相加长度等于 Unigene总长度的一半时,所对应的Unigene的长度即为N 50值。本实验所得转录本N 50等于2 307 nt,Unigene的N 50等于2 040 nt(表2)。Unigene长度在500~2 100 nt范围的数量最多,占69%,随着Unigene长度的增加,数量呈梯度下降,小于300 nt的数量最少(图1)。
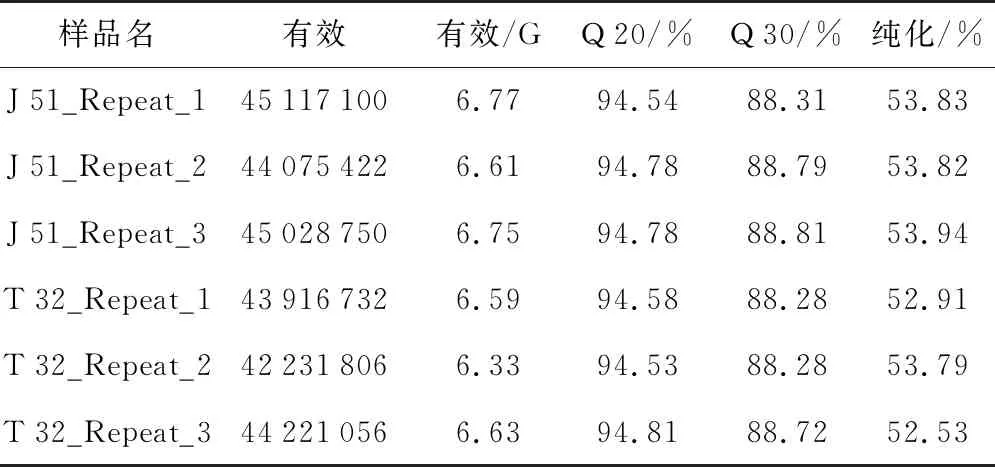
表1 样品转录组测序质量统计Table 1 Sample transcriptome sequencing quality statistics

表2 转录本和Unigene的组装结果Table 2 Assembly results of transcripts and Unigene
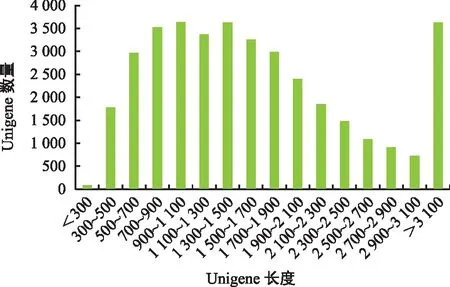
图1 Unigene长度统计Fig.1 Unigene size statistics
2.2 SNP特征分析
使用GATK软件对两组样品中的SNP进行分析,共获得109 977个SNP。为保证SNP位点的准确性,筛选SNP应保证两个转录本的覆盖率大于20个重叠群之和,候选SNP位点两侧至少有5 bp的保守序列。根据上述条件,筛选出A/C,A/G,A/T,C/T,C/G,G/T六种碱基突变类型,其中转换类型71 352个,颠换类型38 625个,转换类型是颠换类型的1.85倍。在转换位点中,C/T 和A/G转换分别有37 894个和33 458个位点;在颠换位点中,A/T、C/G、G/T和A/C颠换分别有8 125个、10 652个、10 896个和8 952个位点(图 2)。
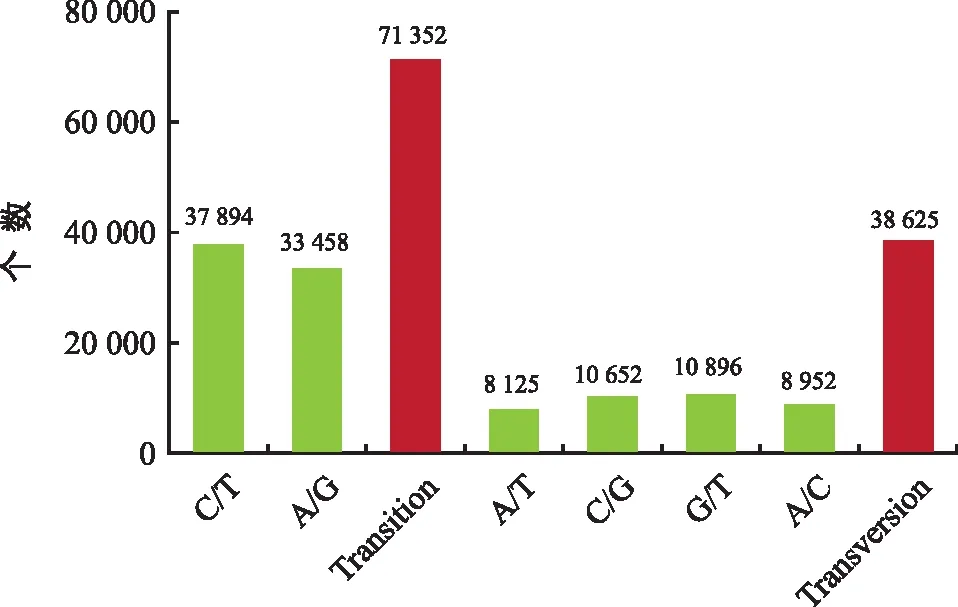
图2 基于转录组测序的玉米不同SNP基因型统计Fig.2 Statistics of different SNP genotypes of maize based on transcriptome sequencing
SNP跨染色体和基因的分布对于评估其基因组覆盖度和标记密度特别重要。 因此,分析了所有玉米染色体的SNP分布(图3)。结果表明,在转录组数据中鉴定出的109 977个SNP位点分布在10条染色体中,每间隔585 bp出现1个SNP。位于第1染色体上的SNP数量最多,占16.11%,其次是第5染色体上,占12.78%,第10染色体分布最少,占7%,平均每条染色体上有10 997.7个SNP。
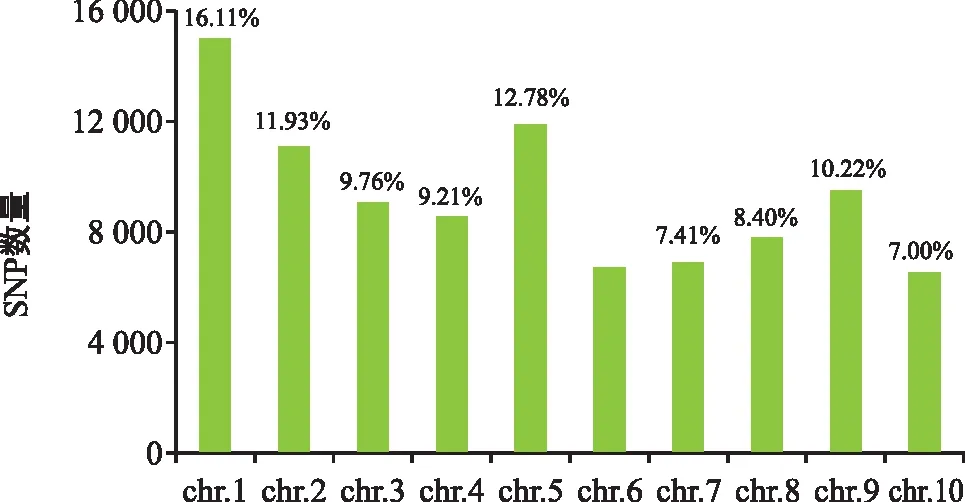
图3 不同染色体转录组 SNP 位点密度分布频率Fig.3 Distribution frequency of SNP site density in different chromosome transcriptomes
2.3 InDel分析
T 32共有12 147个插入位点,13 141个缺失位点,J 51共有9 964个插入位点,11 130个缺失位点。在T 32的10条染色体上,InDel位点在第1染色体最多,为4 214个,第9染色体最少,为1 620个,平均每条染色体有2 528个。在J 51的10条染色体上,InDel
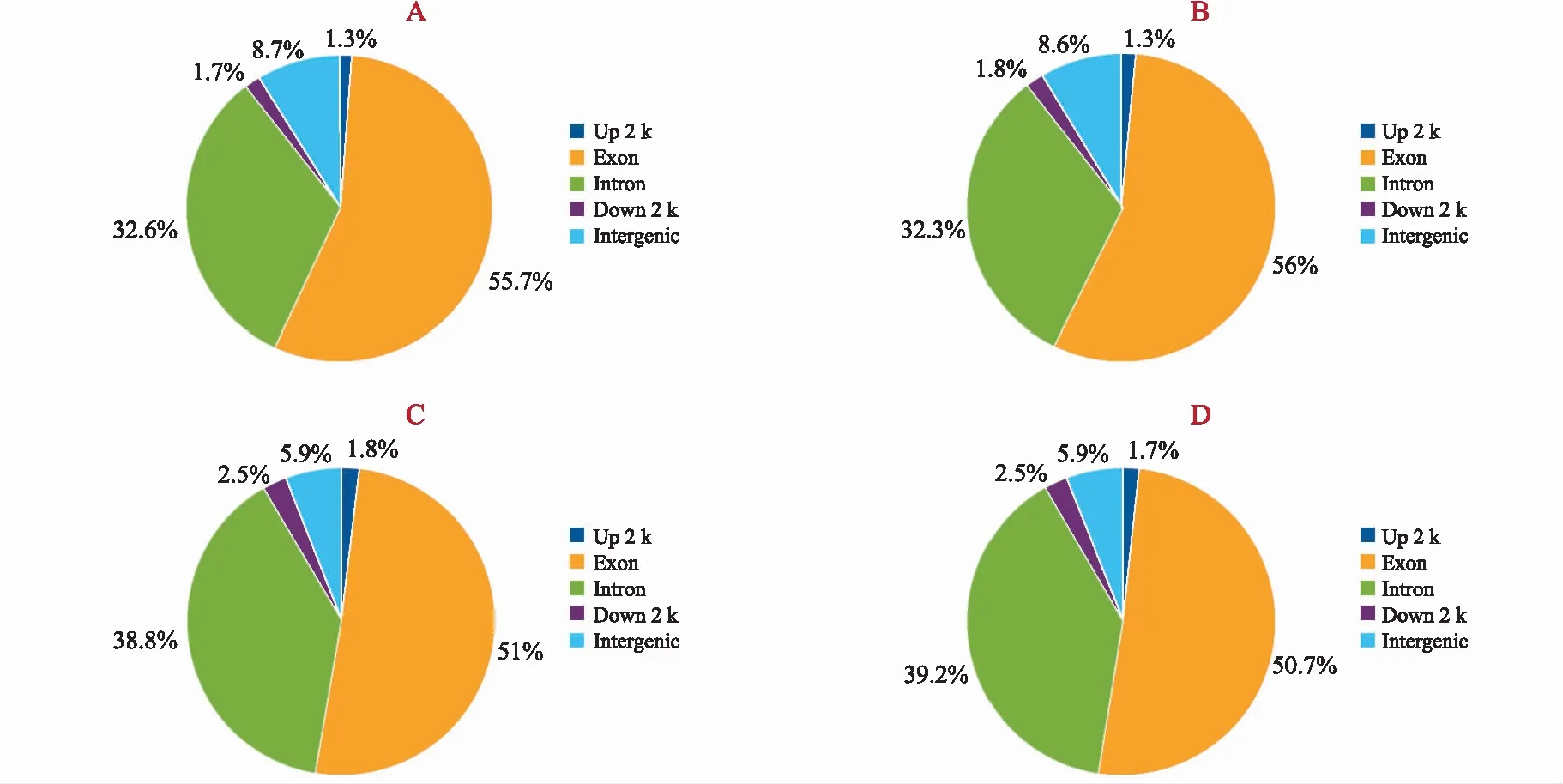
注:A为J 51的SNP位点在玉米基因组上的区域分布情况;B为T 32的SNP位点在玉米基因组上的区域分布情况;C为J 51的InDel位点在玉米基因组上的区域分布情况;D为T 32的InDel位点在玉米基因组上的区域分布情况(Up 2 k表示基因上游2 kbp;Down 2 k表示基因下游2 kbp;Intron表示内含子区;Exon表示外显子区;Intergenic表示基因间隔区)。图5 SNP/InDel在玉米基因组中的区域分布Fig.5 Regional distribution of SNP/InDel in the maize genome
位点在第1染色体最多,为3 467个,第10染色体最少,为1 389个,平均每条染色体有2 111个(表3)。InDel插入/缺失片段以1~3 bp长度为主,片段从小到大数量依次减少,均仅在6 bp长度时有小幅度上升,在大于11 bp长度之后逐渐下降,长度大于20 bp的数目占比不超过1%(图4)。
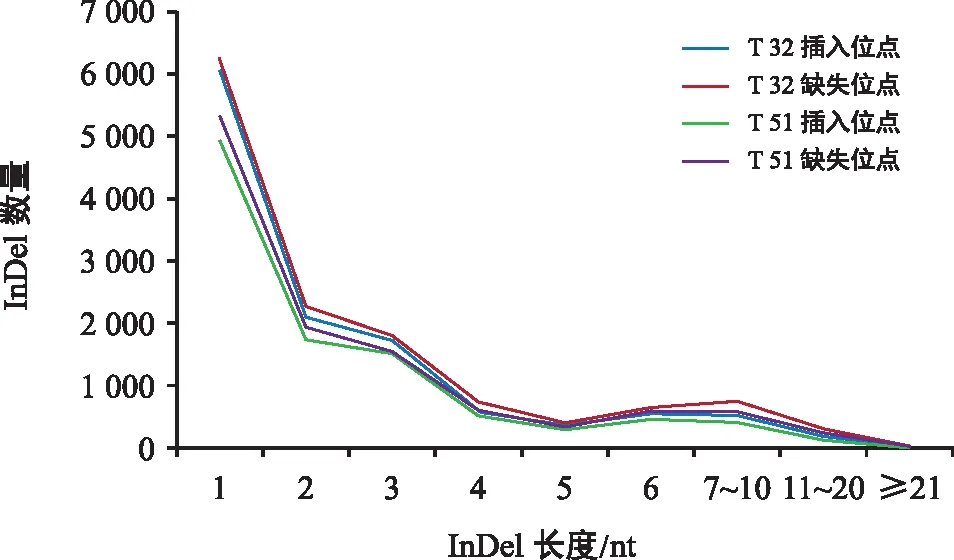
图4 InDel 类型统计Fig.4 InDel type statistics
2.4 SNP/InDel的区域分布
SNP/InDel 位点在基因组的5个位置区域(Up 2 k,Exon,Intron,Down 2 k,Intergenic)的分布(图5),在外显子(Exon)区域分布最多,占比50.7%~56%,超过了SNP/InDel位点总数的一半,其次是内含子(Intron)区域,占比分别为32.6%、32.3%、38.8%、39.2%;在基因的间隔区(Intergenic),两材料SNP位点的占比分别为8.7%、8.6%%,InDel位点的占比均为5.9%;其余基因上下游(Up 2 k,Down 2 k)区域内SNP/InDel位点数量最少,不超过总量的5%。
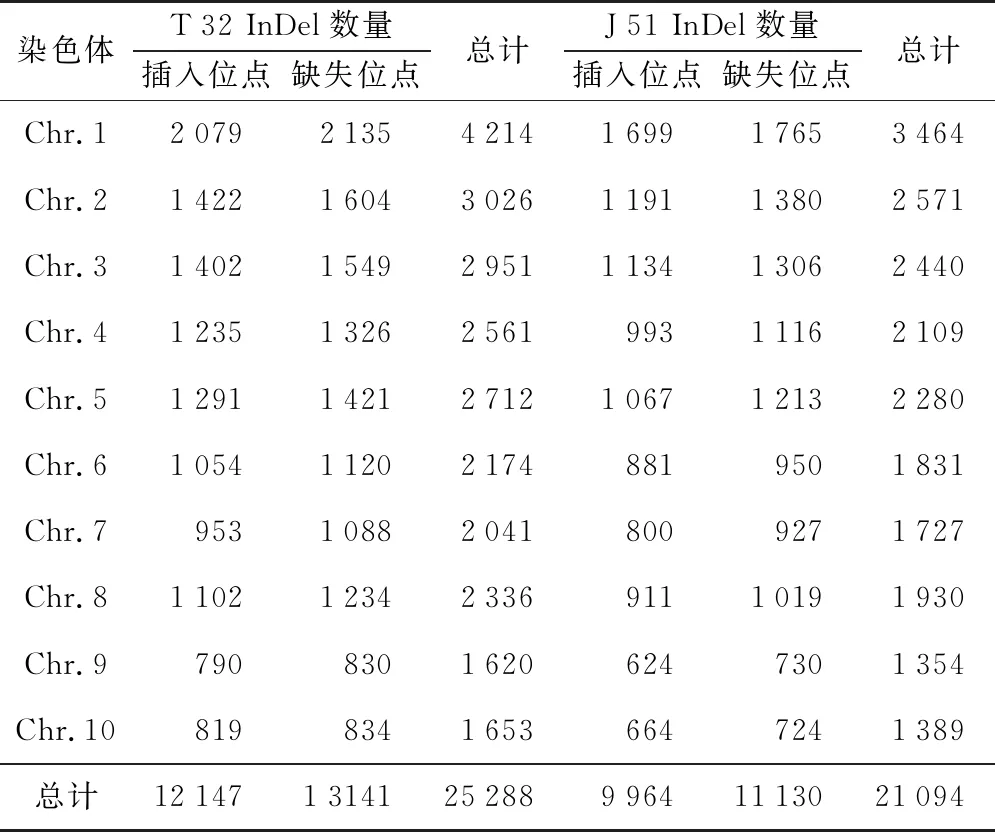
表3 T 32、J 51两自交系InDel在染色体上的分布Table 3 Chromosome distribution of InDel,T 32 and J 51 inbred lines 单位:个
2.5 差异表达基因GO和KEGG 功能分类分析
通过使用BLAST针对NCBI参考序列蛋白质数据库的序列相似性搜索,对得到的最终组装转录本进行验证和注释。基于序列同源性获得的61 947条转录本的功能注释,在分析中转录本被分为3个标准类别:生物过程通路、细胞成分通路和分子功能通路。在生物过程中,差异表达基因富集在代谢过程和细胞过程,而免疫系统过程、细胞增殖、碳利用、细胞杀伤、氮利用和硫的利用等没有差异表达基因富集。细胞成分通路分为14个小类,在细胞成分中,差异表达基因也富集在细胞和细胞部分,而超分子复合物、细胞外区域部分和类核素等只有几个或没有差异表达基因富集。在分子功能中,差异表达基因与催化活性和黏合相关性显著,而与营养库活性、分子载体活性、蛋白标记和翻译调节活性相关性不显著(图6)。玉米转录组数据库中含SNP/InDel标记基因经KEGG数据库注释后,差异表达基因被定位到KEGG中的规范参考通路途径。所有独特的差异表达基因被分配到5个第一层级通路途径和第二层级34个KEGG通路途径。其中全局和概览图在代谢相关通路途径中占主导地位;翻译和折叠、分类和降解在遗传信息过程中占主导地位。此外,大多数差异表达基因由环境信息处理途径中的信号转导表示,而运输和分解代谢、细胞生长和死亡在细胞过程途径中占主导地位,有机系统途径中环境适应占主导地位(图7)。
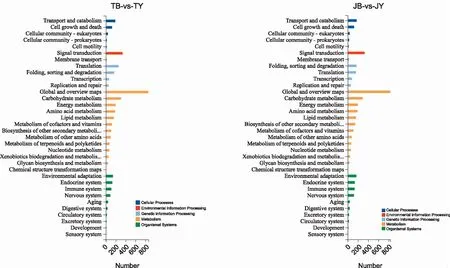
图7 玉米转录组序列中SNP/InDel基因的KEGG功能分类Fig.7 KEGG functional classes of SNP/InDel genes in the maize transcriptome sequence
3 结论与讨论
在下一代测序数据中发现的SNP和InDel已广泛用于人类和许多其他动物物种以及植物的基因组和转录组分析, 如在棉花[20]、水稻[21]和大麦[22]等植物中大量应用。然而,在玉米病原菌胁迫中的应用很少,以往的应用研究主要局限于SSR标记的开发[23],SNP 和 InDel 标记很少研究,特别是在玉米灰斑病病原菌胁迫下,玉米中用于转录组测序的SNP和InDel分子标记的开发未有报道。
本研究探索了6组样品在玉米灰斑病病原菌胁迫下获得的39.68 Gb高质量转录组序列数据,并对转录组测序数据进行SNP和InDel分析。收集了玉米灰斑病抗病自交系T 32和感病自交系J 51的转录组数据集,并得到61 947条转录本,平均长度为1 639个核苷酸。从转录组数据集中生成了37 268条非冗余转录本,结果表明,单个转录组不包含玉米中所有基因的表达图谱,这在小麦[24]、油菜[25]和水稻[26]中也观察到。此外,在探索基因表达图谱的过程中,可能不会检测低表达水平的转录本,一些不足以产生全长组装的转录本也会影响分析[26]。由于转录组代表基因的时间和空间表达图谱,因此参考转录组由不同的基因型对于使用转录组数据开发SNP和InDel 至关重要[27]。同时,在开发标记之前评估转录数据的质量是必要的。
SNP是植物基因组中最丰富的DNA标记,已广泛用于遗传研究和育种计划。在本实验中,两种玉米材料样品统计分析共检测到17 499个Unigenes有SNP位点,共检索到109 977个SNP位点,平均每条Unigene大约有2.9个SNP,每间隔585 bp 出现1个SNP;其中转换类型71 352种,占64.88%,颠换类型38 625种,占35.12%,转换类型约为颠换类型的1.85倍,转换类型明显大于颠换类型,因为碱基替换是产生遗传变异和推动进化的机制之一,表明转录组序列碱基的转换变异存在特定的作用机制[28]。此外,本研究共有6种SNP类型,并且2种转换类型的SNP位点数量远大于4种颠换类型,这是由于基因组中的不同碱基突变频率存在差异造成的[29]。高频率的转换可能反映了甲基化后高水平的C/T突变[30];同时,转换/颠换的高比率表明基因组比较中的遗传差异水平较低[31-32]。
InDel 是动物、植物和细菌中密切相关的DNA序列之间序列差异的主要来源。本研究InDel分析结果表明,在11 346条Unigene上发现29 849个InDel位点,InDel在每条Unigene上的出现频率为48%,其中InDel缺失位点数量略大于插入位点数量,大部分InDel插入/缺失片段以1~3 bp长度为主,长度大于20 bp的数目占比不超过1%。在一定程度上,较长的 InDel 在基因组上的分布密度相对较低,从而导致较低的多态性产生,这在番茄[33]中得到了证实。此外,InDel可能会导致移码突变,使mRNA 在翻译过程中出现错误的终止密码子造成遗传变异。
SNP和InDel的分布也因序列区域类型而异,在基因间区域的分布频率相对低于基因区域[34-35]。刘小红[35]研究表明,超过60%的总SNP和InDel存在于玉米的基因区域,其中编码区的外显子区分布超过50%。本研究中,筛选了基因组的所有区域也得到相同结果,发现两材料SNP/InDel 位点在外显子区域分布最多(超过50%),其次是内含子区域(超过35%),其他区域内分布不超过总量的5%。此外,在包括外显子和内含子序列在内的基因区的SNP和InDel分布数量,内含子区域约占小麦[36]和大豆[37]等作物中总SNP和InDel的一半。
在本研究中,61 947个转录本被分配到3个主要的GO类别。Hufford等[38]研究表明,大多数 GO 代表的转录与催化活性和黏合(在分子功能中)、细胞和细胞部分(在细胞成分中)以及代谢和细胞过程相关的转录物(在生物过程中)在玉米基因型中显示相似的基因功能(图 6)。通过KEGG通路的分析有助于进一步了解基因功能的生物学相关性[39],基于KEGG通路数据库,遗传信息过程和代谢被表示为优势通路(图7)。遗传信息处理途径由折叠、分类和降解、翻译、转录、复制和修复等4个亚类组成,同时RNA家族在生物活动中起着最重要的作用。在转录水平、遗传信息过程和代谢途径确保其组成基因精确同步操作,以最大限度地减少错误,这个过程对细胞生长和增殖很重要[40]。此外,代谢途径包含13个不同的亚类,包括全局和概览图、碳水化合物代谢、能量代谢和聚糖生物合成和代谢等,并且参与玉米生长发育[41]。因此,可以强调遗传信息处理和代谢过程在玉米生长发育中的重要性。
近年来,SNP/InDel 标记越来越多地用于QTL定位研究,因为其在基因组中的丰度避免了高度重复的区域,并且与其他标记系统相比可以提供较高的图谱分辨率[42-43]。 因此,目前转录组数据中已识别的SNP 和InDel标记及其在GO和KEGG富集的差异表达基因将有助于功能多样性分析,因为它们直接参与代谢和细胞过程的生长和调节。此外,这些标记将丰富玉米中已有的基因组资源,并可用于饱和现有的连锁和关联图谱。

