Streptococcus thermophilus IMAU20551胞外多糖基因簇及其表达分析
乔少婷,代安娜尔,解 敏,孙思霖,聂佳莹,丹 彤
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室,内蒙古 呼和浩特 010018)
嗜热链球菌(Streptococcus thermophilus)作为唯一通过欧洲安全认证的链球菌[1-2],因其活力强、产酸快、安全性高等特点,已被广泛应用于发酵乳制品的生产中[3-5]。乳酸菌胞外多糖(exopolysaccharide,EPS)是由乳酸菌通过代谢碳源物质得到的一种高分子聚合物,分子质量一般在104~6×106Da之间[6-7],包括游离于细胞之外的黏液多糖和附着于细胞壁的荚膜多糖[8]。根据化学成分,EPS可分为同型多糖和异型多糖(heteropolysaccharide,HePS)两种,就目前的研究成果而言,乳酸菌的EPS大多均为HePS[9]。EPS对微生物细胞机械损伤有一定的保护作用,可减缓微生物细胞水分流失,在恶劣的生存环境中也可作为微生物的营养[10],同时具有提高发酵乳制品黏度、持水性及稳定性,改善产品口感的优良特性[11-13]。
近年来,随着乳酸菌基因组学研究的飞速发展,国内外对于乳酸菌eps基因簇的研究也有了全新的理解。EPS的合成共包括两个阶段,分别为前体物质糖核苷酸的合成和eps基因簇指导合成EPS[14-15]。糖核苷酸的合成主要依靠糖酵解途径完成,而eps基因簇则负责参与EPS的重复单元合成,控制EPS链长、聚合以及将EPS运输到细胞外[16-17]。eps基因簇对于EPS的生物合成必不可少[18]。Dertli等[18]对约翰逊乳杆菌(Lactobacillus johnsonii)FI9785的EPS结构和基因簇进行分析,发现与野生菌株相比,缺失epsE的突变体只能产生EPS-1而无法产生EPS-2,而当整个eps基因簇被敲除后,菌株则无法再产生EPS。Gankhuyag[19]通过研究德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckiisubsp.bulgaricus)和副干酪乳杆菌(Lactobacillus paracasei)的eps基因簇后发现不同菌种的eps基因簇存在显著差异,进一步研究发现,来自同一菌种的不同菌株eps基因簇也存在明显差异,例如L. delbrueckiisubsp.bulgaricus2038和ATCC 11842具有翻转酶基因,而其他L. delbrueckiisubsp.bulgaricus中未发现此基因。Dipti等[20]通过对比来自27个物种106个乳酸杆菌(Lactobacillus)的eps基因簇后发现糖基转移酶(glycosyltransferase,GT)、翻转酶(O-antigen flippase,Wzx)和聚合输出酶(O-antigen polymerase,Wzy)的特异性较强,且不同生长环境下菌株gt、wzx、wzy基因编码的蛋白质家族无法高度共享,这些结果表明菌株eps基因簇具有较高的可变性,可以适应不同的生存环境。
S. thermophilusIMAU20551分离自蒙古国的传统发酵乳,具有产酸速度快、遗传性状稳定以及EPS产量高((268.25±5.36)mg/L,数据暂未发表)等特点,是1 株特性优良的发酵剂菌株。本实验以S. thermophilusIMAU20551为研究对象,采用Illumina HiSeq技术对该菌株进行二代基因组重测序,找出和EPS生产相关的eps基因簇,并利用实时聚合酶链式反应(real-time polymerase chain reaction,real-time PCR)技术对eps基因簇的表达进行验证,旨在为S. thermophilusEPS的研究提供更多参考信息,为之后乳酸菌EPS的应用奠定理论基础。
1 材料与方法
1.1 材料与试剂
S. thermophilusIMAU20551分离自蒙古国扎布汗省奥特跟苏木的传统酸牛奶,由内蒙古农业大学乳酸菌菌种资源库提供,菌株保藏号MGB80-7,GenBank序列号HM058270。
M17肉汤培养基 青岛海博生物技术有限公司;Wizard Genomic DNA Purification Kit(1120)、Invitrogen TRIzol RNA提取试剂盒 美国Promega公司;HiScript Q RT SuperMix for qPCR (+gDNA wiper)反转录试剂盒、2×ChamQ SYBR COLOR qPCR Master Mix 南京诺唯赞生物科技有限公司。
1.2 仪器与设备
AR2202CN型电子天平 奥豪斯仪器上海有限公司;SX-700型全自动高压蒸汽灭菌器 日本HIRAYAMA公司;HWS28型电热恒温水浴锅 上海一恒科技有限公司;DHP-9272型电热恒温培养箱 上海坤天试验仪器有限公司;SJ-CJ-2FDQ型超净台 上海智城分析仪器制造有限公司;BX 50型光学显微镜 日本奥林巴斯公司;Mix Max型旋涡振荡器 合肥艾本森科学仪器有限公司;5810 R型高速控温离心机 德国Eppendorf公司;Nano-Drop 2000型微量分光光度计 美国Thermo Scientific公司;ABI 7500型荧光定量PCR仪 美国Applied Biosystems公司。
1.3 方法
1.3.1 菌株活化
参照陈海燕等[21]方法,将保藏于-80 ℃的S. thermophilusIMAU20551接种于脱脂乳培养基中,接种量为2%(m/m),37 ℃培养24 h后,以2%(V/V)的接种量接种于M17液体培养基中,37 ℃培养24 h,连续培养2 代,使菌株活力达到最大(活菌数>109CFU/mL)。
1.3.2 菌株DNA提取
将活化后的IMAU20551以2%(V/V)接种量扩培至50 mL的M17液体培养基中,37 ℃培养24 h后,离心除去上清液,用磷酸盐缓冲溶液清洗2 遍得到干净菌体,利用Wizard Genomic DNA Purification Kit提取DNA,提取方法参考试剂盒说明书。提取出的DNA通过微型紫外分光光度计测定浓度,并利用1%(m/m)琼脂糖凝胶电泳判断其纯度和完整度,选取出质量合格的DNA进行重测序。
1.3.3 基因组重测序
将通过1.3.2节得到的DNA采用Illumina HiSeq 4000进行基因组二代重测序,构建Illumna PE文库。
1.3.4 基因组组装
将原始数据raw data通过Illumina HiSeq 4000过滤得到clean data,利用SOPAdenovo软件对重测序完成后的基因数据进行组装。选取合适的Kmer值组装拼接基因组数据,并对单碱基进行矫正。以基因组大小、scaffold数量、GC含量、N50值、N90值为指标,筛选与参考值接近的基因组拼装结果作为最终组装结果进行SOAP验证及校验。
1.3.5 基因组预测及注释
利用prokka软件预测基因组基因,获得开放阅读框信息。结合RAST(Rapid Annotation using Subsystem Technology)Server-RAST Annotation Server(http://rast.nmpdr.org/)网上在线工具、基因本体论(Gene Ontology,GO)(http://www.geneontology.org/)、直系同源集(Clusters of Orthologous Groups,COG)(http://www.ncbi.nlm. nih.gov/COG/)以及京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)(http://www.genome.jp/kegg/)等数据库完成基因的预测及功能注释。
1.3.6 生物信息分析
采用CGview(http://stothard.afns.ualberta.ca/cgview_server/)软件绘制S. thermophilusIMAU20551基因组扫描图,核酸序列相似性比对采用BLAST(http://www.ncbi.nlm.nih.gov)软件在线分析。
1.3.7 总RNA提取
将活化后的S. thermophilusIMAU20551以2%(V/V)接种量接种于M17液体培养基中,37 ℃培养,分别于0、6、12、18、24 h取出离心(4 000 r/min,10 min)收集菌体。利用Invitrogen TRIzol RNA提取试剂盒提取菌体RNA,提取方法参考试剂盒说明书。利用1%(m/m)琼脂糖凝胶电泳对RNA提取质量进行检测。
1.3.8 cDNA合成
根据HiScript Q RT SuperMix for qPCR (+gDNA wiper)反转录试剂盒说明书进行DNA去除反应及cDNA模版合成反应,方法参考试剂盒说明书,逆转录反应条件为50 ℃,15 min;85 ℃,2 min。
1.3.9 real-time PCR引物设计及反应体系
使用Primer 5软件设计real-time PCR引物,引物由上海美吉生物公司合成。具体引物如表1所示。

表1 real-time PCR引物Table 1 Primer sequences used for real-time PCR in this study
以上述设计的引物为模版,采用gapdh部分序列作为内参基因进行real-time PCR。反应体系为20 µL:10.0 µL 2× ChamQ SYBR Color qPCR Master Mix;Primer F(5 µmol/L)、Primer R(5 µmol/L)各0.8 µL;0.4 µL 50× ROX Reference Dye 2;cDNA模板2.0 µL;补水至20.0 µL。参照Livak等[22]的方法使用2-ΔΔCt法分析数据,计算基因表达量并绘图。
1.4 数据分析
利用Origin 5、Excel、OMNIC等软件对实验数据进行处理,所有实验均重复3次以减少误差。
2 结果与分析
2.1 S. thermophilus IMAU20551基因组基本特征
对S. thermophilusIMAU20551进行DNA提取后采用Illumina HiSeq 4000测序平台进行测序,采用CGView软件绘制菌株基因组扫描图,结果如图1所示。从外到内第1圈和第4圈为正链、负链上的蛋白质编码区(coding sequence,CDS),以不同颜色表示不同的COG功能分类;第2圈和第3圈分别为正链、负链上的CDS、tRNA和rRNA;第5圈为GC含量,向外的部分表示该区域GC含量高于全基因组平均GC含量,反之则表示该区域GC含量低于全基因组的平均GC含量,峰值越高表示与平均GC含量差值越大;第6圈为GC偏移值,具体算法为(GC)/(G+C),可以辅助判断前导链和后滞链,一般前导链GC偏移值>0(绿色部分),后滞链GC偏移值<0(紫色部分),也可以辅助判断复制起点(累计偏移最小值)和终点(累计偏移最大值),尤其对环状基因组最为重要;最内一圈为基因组大小标识。
基因组全长1 725 107 bp,无质粒基因,共56 条scaffold,GC含量为39.08%,N50长度为105 617 bp,N90长度为37 646 bp,包含33个tRNA、3个rRNA和26个重复单位。基因组共编码1 884个基因,基因长度为1 432 380 bp,占总基因组长度的83.03%。各项数据均显示符合S. thermophilus基本特征,表明基因组测序组装结果良好,可进行后续生物信息分析。
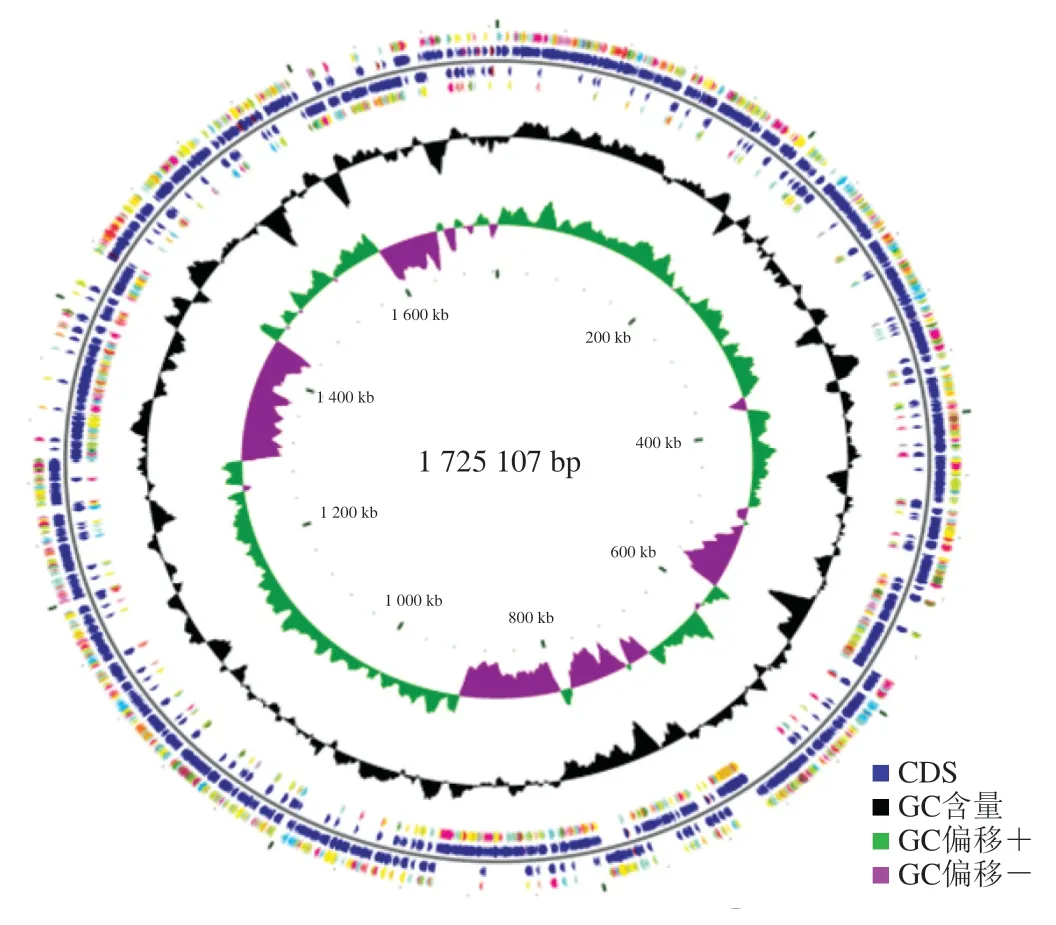
图1 S. thermophilus IMAU20551基因组扫描图Fig. 1 CGView results of S. thermophilus IMAU20551
2.2 S. thermophilus IMAU2055基因组COG分析
如图2所示,比对蛋白COG数据库后发现,在所有的1 884个基因中,有1 357个基因被分别注释到19个COG类别中,占总基因数的81.58%。除去功能未知的345个基因外,被注释到E类别(氨基酸运输与代谢)中的编码基因占比最大,共有195个,其次是J类别(翻译、核糖体结构和生物起源,144个基因)、L类别(复制、重组与修复,127个基因)以及G类别(碳水化合物运输与代谢,94个基因)。另外,eps基因簇中大部分功能基因均被注释到M类别(细胞壁/细胞膜/细胞被膜生物合成)中。
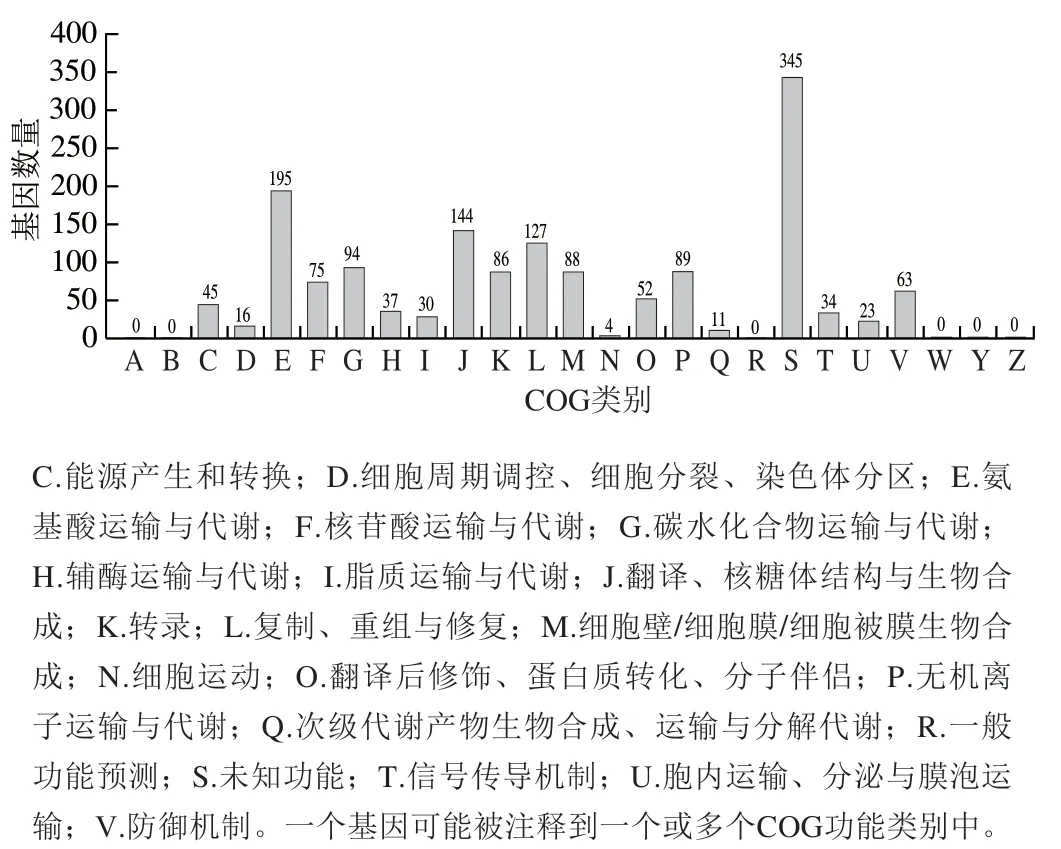
图2 S. thermophilus IMAU20551基因组COG功能分类统计分析Fig. 2 COG function analysis of genomic DNA from S. thermophilus IMAU20551
2.3 S. thermophilus IMAU2055基因组GO分析
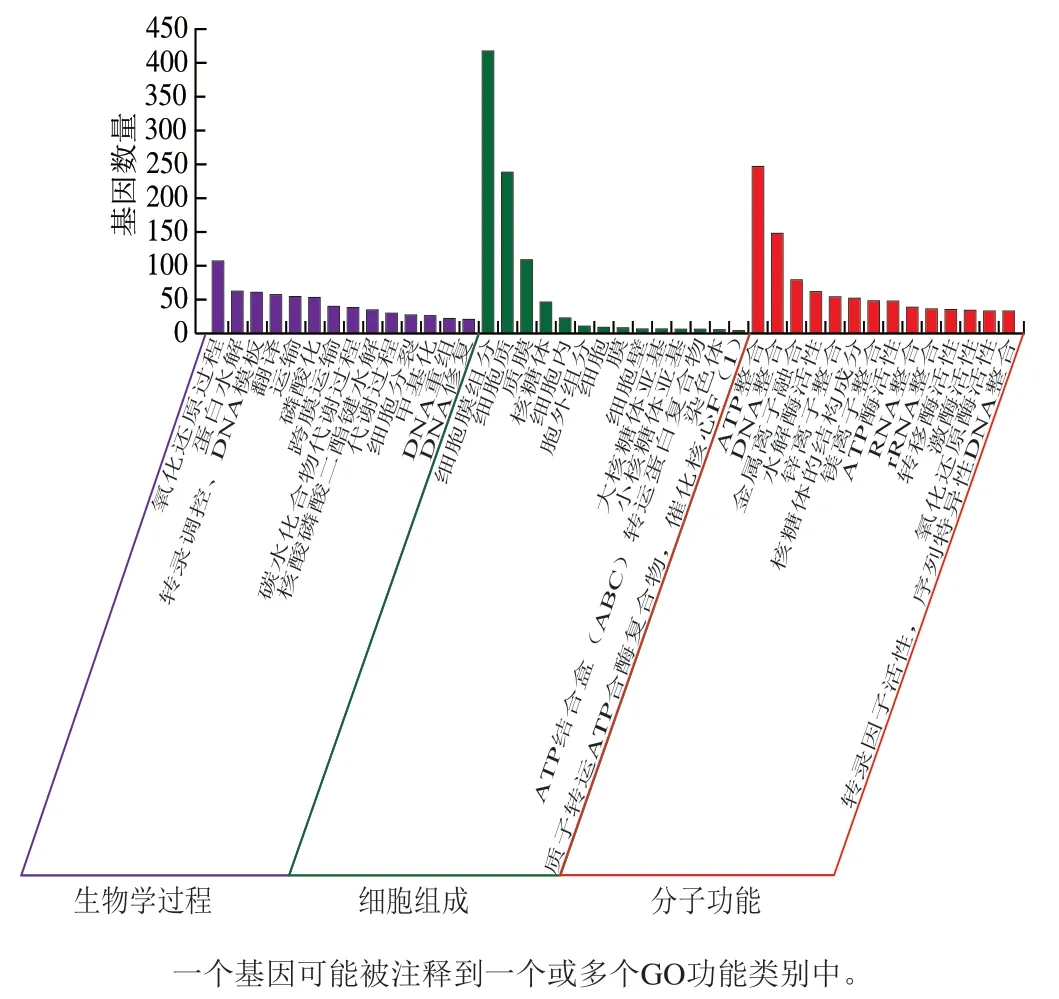
图3 S. thermophilus IMAU20551基因组GO功能注释结果Fig. 3 GO function analysis of genomic DNA from S. thermophilus IMAU20551
将S. thermophilusIMAU20551基因组数据与GO数据库比对,结果如图3所示。共有1 538个基因被分为3 类本体功能,分别为生物学过程、细胞组成和分子功能,进一步可细分为42个二级功能类别。其中,在生物学过程中的14个二级类别中,有较大一部分基因被注释到氧化还原过程中,另外被注释到蛋白水解、转录调控、翻译、转运和磷酸化中的编码基因也数量较大;在细胞组成类别中又分为14个二级类别,有418个基因被注释到细胞膜组分类别中,占GO总编码基因的22.19%,其次为细胞质和质膜,分别注释到239个基因和110个基因,有极少数的基因被注释到胞外基质、质膜外在组分、信号识别粒子等类别;在分子功能下的14个类别中,有绝大部分的基因被注释到ATP整合和DNA整合类别中,共397个编码基因,占GO总编码基因的21.07%,另外在水解酶、ATP酶和转移酶等酶活性类别中也注释到较多编码基因。
2.4 S. thermophilus IMAU2055基因组KEGG分析
将S. thermophilusIMAU20551基因组与KEGG数据库比对,结果如图4所示。S. thermophilusIMAU20551基因组中共有1 170个基因在KEGG代谢通路中得到注释,占到全部编码基因的62.10%。其中大部分的基因被注释到新陈代谢、遗传信息传递和生物体系统这3个一级类别中,被注释到基因数量分别为719、153个和143个,占到所有被注释到KEGG通路中的基因数量的61.45%、13.08%和12.22%。细胞过程、人类疾病以及环境信息加工这3个类别中注释到的基因较少,其中,被注释到有机系统的基因丰度最低,仅有16个。将6个一级分类细分为34个二级类别后发现,被注释到碳水化合物代谢、氨基酸代谢、膜转运以及翻译中的基因占大多数,仅有极少部分的基因被注释到消化系统、免疫系统、神经系统中。

图4 S. thermophilus IMAU20551基因组KEGG pathway分类统计Fig. 4 KEGG pathway classification of genomic DNA from S. thermophilus IMAU20551
2.5 S. thermophilus IMAU20551合成糖核苷酸
EPS的合成离不开前体物质糖核苷酸的供应。本实验也在KEGG通路中注释到一些可以将游离糖利用并合成糖核苷酸的酶的相关基因,如表2所示。这些酶可以将不同的碳源物质作为底物,经过一系列反应最终合成UDP-葡萄糖、dTDP-葡萄糖、UDP-半乳糖、dTDP-鼠李糖、UDP-N-乙酰葡萄糖胺以及UDP-N-乙酰半乳糖胺等前体糖核苷酸。由这些酶可以推测出S. thermophilusIMAU20551所产EPS可能是由葡萄糖、半乳糖、葡萄糖醛酸等多种单糖组分组成,为典型的HePS,这与前期测得的S. thermophilusIMAU20551 EPS单糖组成结果一致(数据暂未发表)。
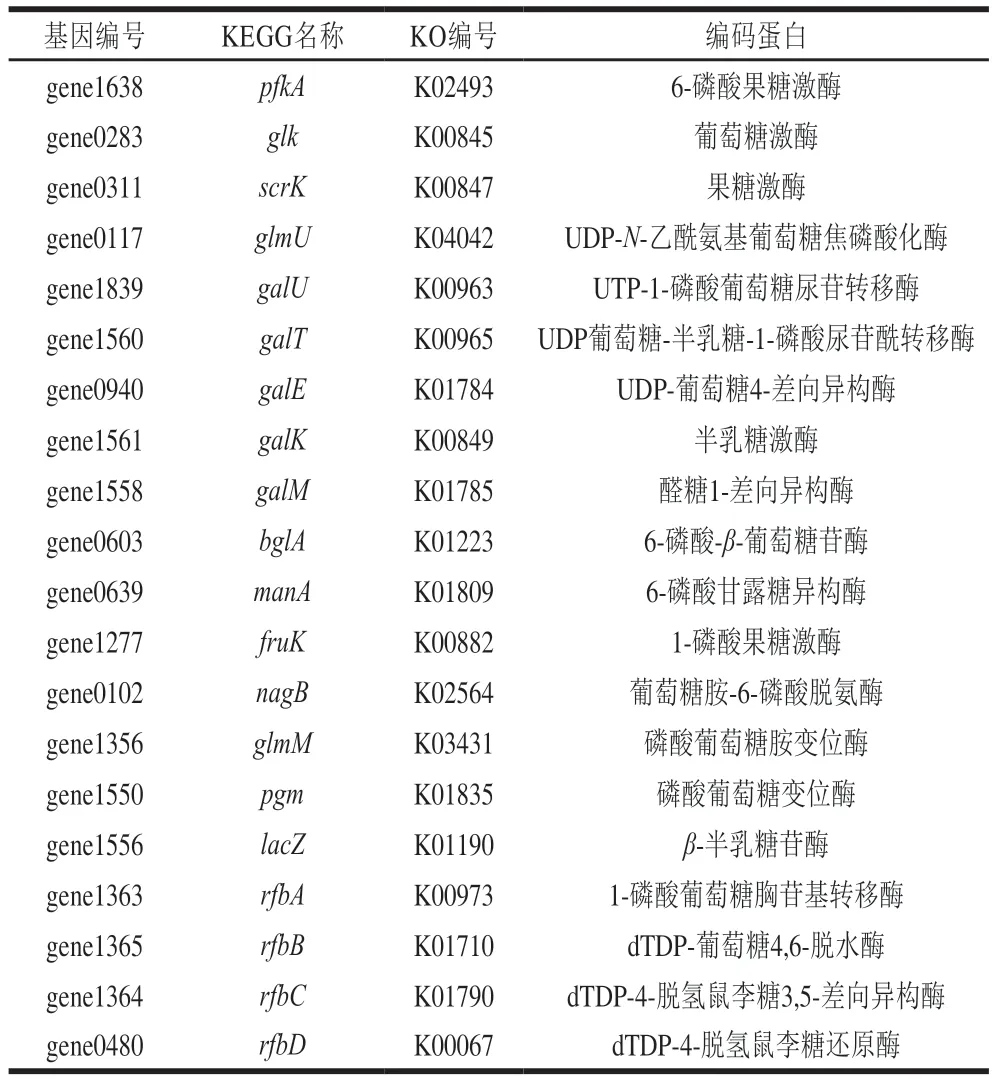
表2 KEGG注释S. thermophilus IMAU20551糖核苷酸合成部分过程中关键酶Table 2 Key enzymes in the synthesis of sugar nucleotides in S. thermophilus IMAU20551 annotated to the KEGG pathway
2.6 S. thermophilus IMAU20551 eps基因簇分析
注释基因组后发现,S. thermophilusIMAU20551拥有较为完整的eps基因簇,长度为19 376 bp。从图5可以看出,S. thermophilusIMAU20551的eps基因簇中,除假定蛋白外,与EPS有关的基因共有18个,其中已确定的糖基转移酶基因4个,分别为epsE、eps1F、eps2F以及epsJ;同时还注释得到负责调节EPS转录产生的基因epsA、epsB,控制EPS链长的基因epsC、epsD,控制磷酸化的epsP以及负责控制EPS输出的基因wzx和epsX。另外还注释到荚膜多糖生物合成蛋白、转座酶、磷酸甘油酸变位酶以及磷酸酶等与EPS合成相关的功能基因。
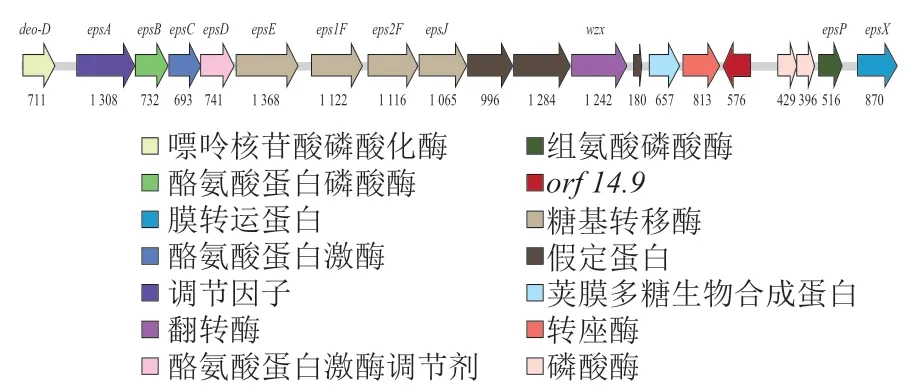
图5 S. thermophilus IMAU20551 eps基因簇Fig. 5 eps gene cluster of S. thermophilus IMAU20551
另外基因簇中还注释出位于5'端的deo-D和位于3'端的orf 14.9,其中,orf 14.9与eps基因簇中其他基因转录方向相反。eps基因簇中大部分的功能基因都位于deo-D和orf 14.9之间,只有负责磷酸化的epsP和控制EPS输出的epsX基因位于orf 14.9下游。多数S. thermophilus的eps基因簇均以orf 14.9结尾,但也有少部分菌株的eps基因簇结构与本研究类似,例如S. thermophilusMR-1C的eps基因簇中,epsU、epsV以及epsX均位于orf 14.9之后[23]。
以S. thermophilusND 03、ASCC 1275以及IMAU20561作为对照,比较实验菌株与这3 株菌株eps基因簇的差异(图6)。发现4 株S. thermophilus中的epsA、epsB、epsC、epsD以及epsE的位置和顺序几乎没有改变,说明这几个基因高度保守,稳定出现在所有产eps的S. thermophilus基因组中。而注释糖基转移酶的基因在不同的菌株eps基因簇中分布的种类、数量、位置以及顺序具有特异性。另外,与其他3 株S. thermophilus不同的是,ASCC 1275eps基因簇中出现了两对epsC和epsD,这可能与该菌株同时产生黏液多糖和荚膜多糖有关[24]。
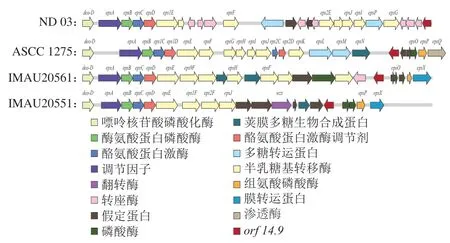
图6 4 株S. thermophilus eps基因簇对比Fig. 6 Comparison of eps gene clusters from four S. thermophilus strains
通过NCBI BLAST比对S. thermophilusIMAU20551eps基因簇中功能性基因的同源性,结果如表3所示。
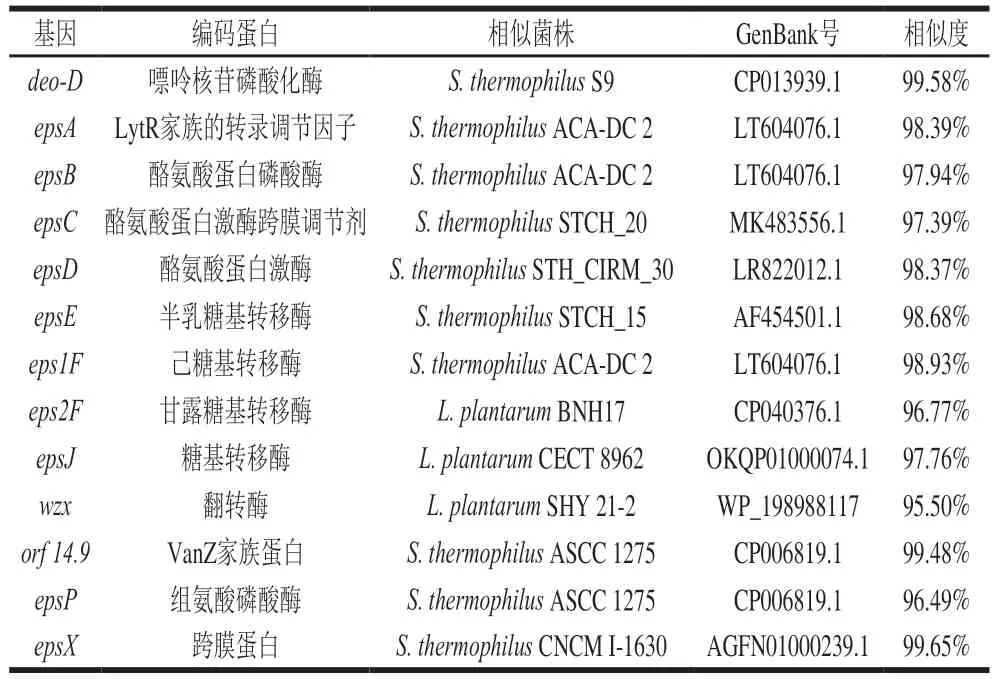
表3 S. thermophilus IMAU20551 eps基因簇功能Table 3 Functions of eps gene cluster from S. thermophilus IMAU20551
位于5'端的deo-D基因编码嘌呤核苷酸化酶,参与糖核苷酸的合成与分解反应[25],与S. thermophilusS9中的deo-D基因相似性极高,达99.58%。
epsA与epsB为eps基因簇中的保守基因,epsA编码为LytR家族的转录调节因子,调控EPS产生,Dertli等[26]通过敲除L. johnsoniiFI9785中的epsA基因后发现细胞表面EPS-1和EPS-2完全缺失,表明epsA是EPS产生的积极调节因子。epsB编码为酪氨酸蛋白磷酸酶,有研究发现乳酸乳球菌EPS合成受磷酸调节系统的控制,由epsB的非磷酸化形式驱动[27]。本实验注释得到的epsA和epsB与S. thermophilusACA-DC 2(GenBank:LT604076.1)的epsA和epsB的相似性较高,分别为98.39%和97.94%。
epsC和epsD负责控制EPS链长。epsC基因编码酪氨酸蛋白激酶跨膜调节剂,有报告称epsC可以与磷酸化激酶相互作用以控制EPS聚合物延伸链的长度[28]。本研究得到的epsC与S. thermophilusSTCH_20中的epsC具有较高的同源性;epsD编码酪氨酸蛋白激酶,与S. thermophilusSTH_CIRM_30中epsD基因序列最为相似。有研究表明,epsD可以控制epsE的活性,从而影响EPS合成过程中一系列糖基转移酶的表达[29]。
epsE、eps1F、eps2F以及epsJ均编码糖基转移酶,负责转运不同的糖核苷酸到脂质携带体,在EPS合成过程中发挥重要功能。其中epsE编码半乳糖基转移酶,是S. thermophilusIMAU20551eps基因簇中第一个糖基转移酶,称为引发糖基转移酶[30]。本实验中注释出的epsE与S. thermophilusSTCH_15中引发糖基转移酶的同源性最高,达到98.68%;eps1F编码己糖基转移酶,与S. thermophilusACA-DC 2中的epsF具有较高同源性(相似性98.93%);eps2F注释得到编码甘露糖基转移酶,和基因epsJ均与L. plantarum中编码糖基转移酶的基因最为相似。Pandey等[31]发现糖基转移酶可以具有广泛的底物选择范围。因此,本实验中引发糖基转移酶EpsE将UDP-半乳糖转运到与细胞膜相连的脂载体上,其他糖基转移酶Eps1F、Eps2F、EpsJ负责将UDP-葡萄糖、dTDP-葡萄糖、dTDP-鼠李糖、UDP-N-乙酰葡萄糖胺和UDP-N-乙酰半乳糖胺等糖核苷酸转按照一定的顺序和规律运到与脂载体相连的糖核苷酸上,不断延伸形成脂载体焦磷酸化重复单元(Und-PP-糖重复单元)。
wzx编码翻转酶,负责将EPS的重复单元进行易位,本实验中S. thermophilusIMAU20551基因组中注释出的wzx基因序列与L. plantarumSHY 21-2中的寡糖翻转酶家族所编码的基因相似性较高(95.5%)。目前,已知的EPS生物合成模式有4种,分别为Wzx/Wzy-dependent途径、ABC transporter-dependent途径、Synthase-dependent途径和使用单一蛋白酶进行胞外合成途径[32],除第四种途径外,其他3种途径均为HePS合成途径。在IMAU20551的eps基因簇中注释出Wzx翻转酶,推测S. thermophilusIMAU20551合成EPS的模式为Wzx/Wzydependent途径,与大多数乳酸菌HePS的合成模式相同。Wzx翻转酶是O-抗原生物合成途径的重要组成部分,负责寡糖O单元在革兰氏阴性菌中跨膜转运[33]。Wzx/Wzydependent途径是O-抗原生物合成途径,同时也作为HePS合成模型。
orf 14.9注释得到编码VanZ家族蛋白,存在于大部分S. thermophilus eps基因簇的3'端,orf 14.9与富产EPS的S. thermophilusASCC 1275菌株中的orf 14.9基因同源性最高,为99.48%。Tyvaert等[34]对不含orf 14.9的S. thermophilusNST2280和A054进行基因突变使之具有orf 14.9,对比后发现具有orf 14.9基因的突变体与原菌株的EPS产量差异不明显,但生长参数有明显区别,推测orf 14.9不影响S. thermophilusEPS的产量但与细胞生长有关。
epsP编码组氨酸磷酸酶,负责EPS磷酸化,目前对epsP的相关研究较少,本实验中epsP与S. thermophilusASCC 1275中的epsP基因相似性较高,为96.49%。
epsX编码蛋白为跨膜蛋白,负责将EPS转运至胞外。与S. thermophilusIMAU20551中epsX的基因序列最为接近的是S. thermophilusCNCM I-1630基因组中的epsX基因,相似性为99.65%。
2.7 S. thermophilus IMAU20551 EPS合成途径
通过实验所得结果,可以推测S. thermophilusIMAU20551 EPS的合成途径(图7)。果糖、蔗糖、纤维二糖以及甘露糖利用磷酸烯醇式丙酮酸-磷酸转移酶系统转变为磷酸化状态并进入细胞,其中纤维二糖会转化为6-磷酸纤维二糖,随后在6-磷酸-β-葡萄糖苷酶的作用下转换为6-磷酸葡萄糖,6-磷酸葡萄糖和6-磷酸果糖可以相互转化,而6-磷酸蔗糖和6-磷酸甘露糖则可以单向转化为6-磷酸果糖,6-磷酸果糖最终可以转化为UDP-N-乙酰葡萄糖胺和UDP-N-乙酰半乳糖胺,6-磷酸葡萄糖则最终转化为UDP-葡萄糖糖和dTDP-鼠李糖;乳糖、葡萄糖利用渗透酶进入细胞内,乳糖被β-半乳糖苷酶分解为葡萄糖和半乳糖,一部分半乳糖会通过渗透酶的作用被排出细胞外,另一部分被醛糖1-差向异构酶转化为a-D-半乳糖,随即进行磷酸化成为1-磷酸半乳糖,1-磷酸半乳糖会通过Leloir途径转化为u1-磷酸-葡萄糖,也可以在UDP葡萄糖-半乳糖-1-磷酸尿苷酰转移酶的作用下转化为UDP-半乳糖,另外,UDP-半乳糖和UDP-葡萄糖也是可以相互转化的。经过转化后的UDP-N-乙酰葡萄糖胺、UDP-N-乙酰半乳糖胺、dTDP-鼠李糖、dTDP-葡萄糖、UDP-葡萄糖以及UDP-半乳糖经过糖基转移酶的作用转移到脂质载体上成为Und-PP-糖重复单元,形成利用聚合酶将其聚合并形成特定的结构,再由翻转酶和跨膜蛋白共同作用将EPS转运到细胞外。另外,根据图7可以推测得出磷酸化作用是将糖分子转化为核苷酸的基础。
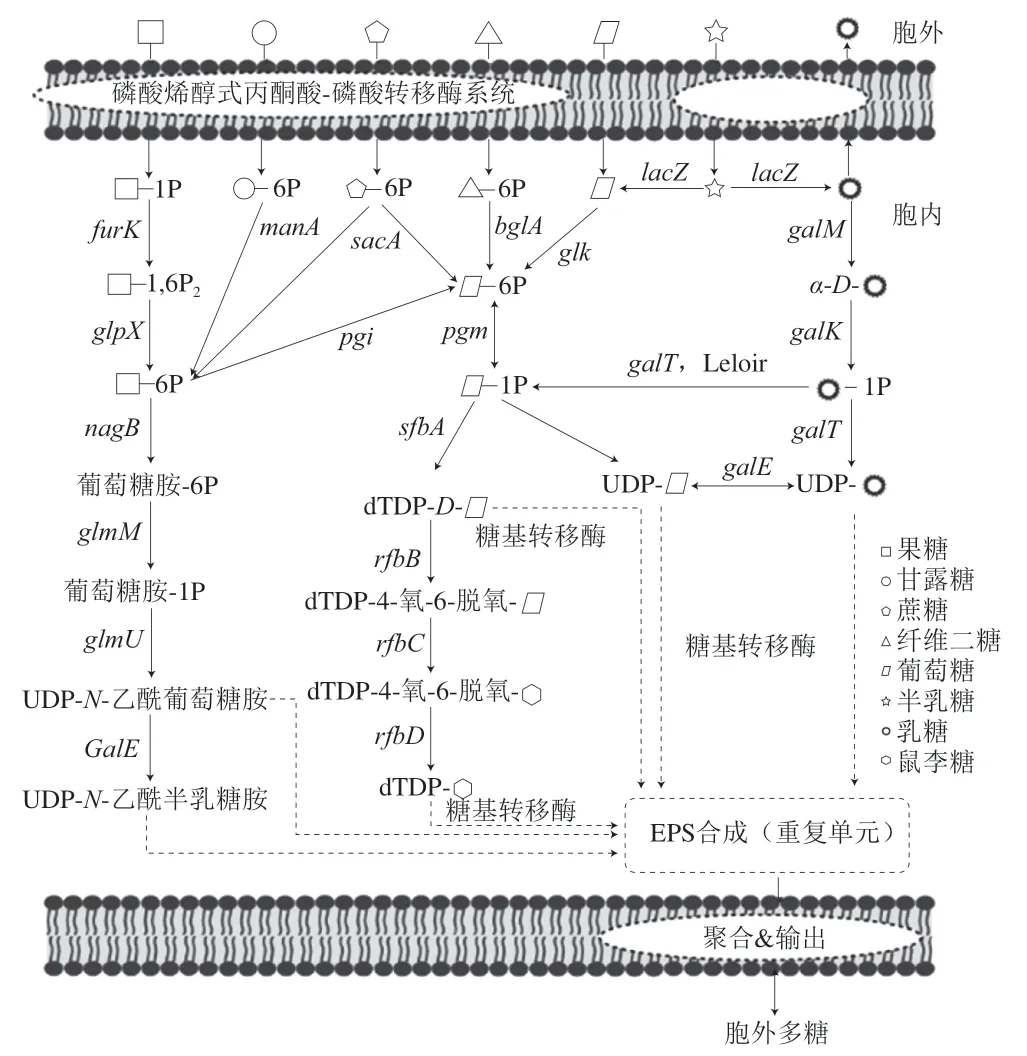
图7 S. thermophilus IMAU20551 EPS合成途径Fig. 7 Exopolysaccharide synthesis pathway of S. thermophilus IMAU20551
2.8 real-time PCR分析
2.8.1 RNA提取结果
real-time PCR技术可以对基因的表达量进行较为准确的描述,已成为常用的基因表达检测方法。S. thermophilusIMAU20551 RNA提取后质检结果如表4、图8所示。所有样品RNA条带清晰明亮,无色素、蛋白、糖类等杂质污染,无DNA污染,28/23S亮度大于18/16S,OD260/280≥1.8,OD260/230≥1.0,质量浓度≥400 ng/μL,总量≥4 μg,可以进行后续实验。
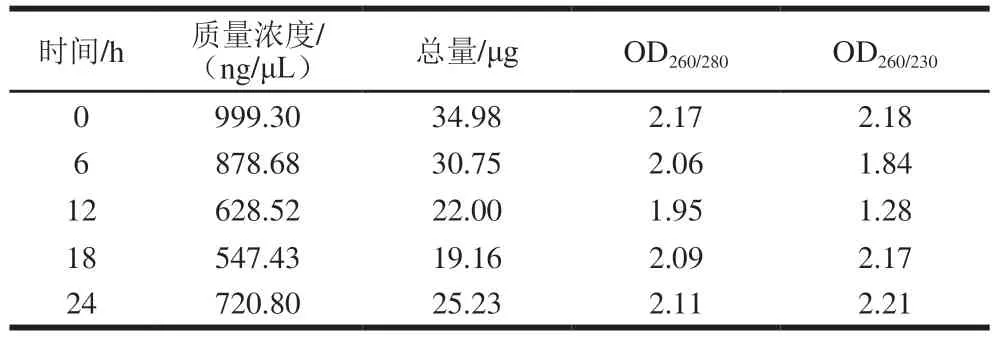
表4 S. thermophilus IMAU20551总RNA提取结果Table 4 Amount of total RNA extracted from S. thermophilus IMAU20551
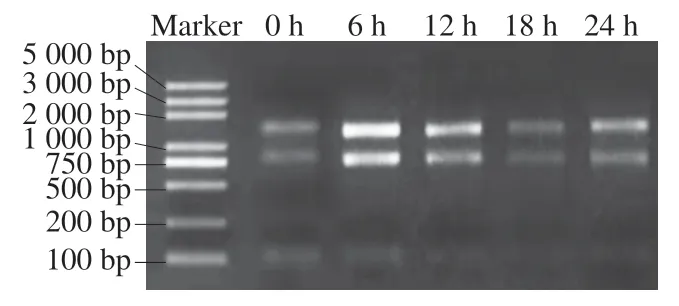
图8 S. thermophilus IMAU20551 RNA琼脂糖凝胶电泳结果Fig. 8 Electrophoretogram of RNA from S. thermophilus IMAU20551
2.8.2 real-time PCR分析结果
对S. thermophilusIMAU20551eps基因簇中的epsA、epsB、epsE、eps1F、eps2F以及epsX的相对表达量进行测定,结果如图9所示。可以看出,除eps2F、epsJ外,其他被测基因均在6 h达到最大表达量,说明在菌株生长到6 h是菌株代谢EPS的关键阶段;12 h时,eps2F、epsJ的表达量有一定增长,eps2F的表达量达到最大(1.022),但其余基因的表达量均有不同程度的下降,说明这段时间内菌株仍继续生产EPS,但合成速率减缓;菌株培养18 h,epsE、eps1F和负责转运EPS的epsX相对表达量出现回升,基因epsJ表达量继续增长,达到峰值(0.576),推测此时S. thermophilusIMAU20551正处于平台期,在不间断产出EPS的同时也产出其他丰富的次级代谢物;除epsA外,其他被测基因在菌株生长到24 h相对表达量继续下降,说明24 hS. thermophilusIMAU20551已经处于衰亡期,基因的表达能力下降,无法产生更多的EPS或其他代谢产物。
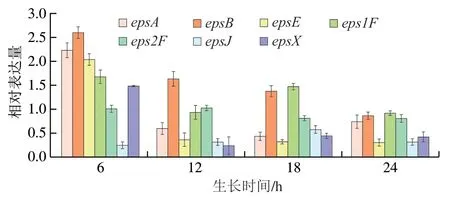
图9 eps基因簇部分基因的相对表达量Fig. 9 Relative expression of key genes in the eps gene cluster
3 讨 论
大部分S. thermophilus的基因组大小在1.85 Mbp左右,编码大约2 000个基因,涉及菌株生长代谢等各个方面[4]。S. thermophilusEPS的生物合成是由eps基因簇调控的。王俊沪等[35]认为S. thermophiluseps基因簇几乎全部位于染色体DNA上,在传代过程中eps基因簇丢失的概率较低,保证了菌株产EPS的稳定性。而其他乳酸菌eps基因簇大多位于质粒上,在传代或进化过程中容易出现质粒丢失现象从而失去产EPS能力。通过基因组重测序和生物信息学分析,本实验在S. thermophilusIMAU20551染色体DNA上发现了完整的eps基因簇,该基因簇共有18个基因,全长19 376 bp,负责调控EPS生物合成、输出以及聚合等过程。
本实验利用real-time PCR对S. thermophilusIMAU20551eps基因簇中关键基因的相对表达量进行了检测分析,之后将继续利用基因敲除及回补实验验证eps基因簇表达能力的准确性,以及基因之间的互作关系,另外,有部分研究认为eps基因簇中编码糖基转移酶的基因数量会影响菌株的EPS产量,后续实验也将利用基因克隆或基因过表达实验等验证eps基因簇中糖基转移酶数量以及与糖核苷酸代谢相关酶类与EPS产量之间的联系。
4 结 论
利用Ilummina HiSeq、SOPAdenovo、prokka等对产EPS的S. thermophilusIMAU20551基因组进行测序、组装及注释后发现,S. thermophilusIMAU20551基因组中各代谢通路基因丰度较高,且拥有一条完整的eps基因簇,涵盖负责调控、组装、转运以及输出EPS。real-time PCR结果显示,S. thermophilusIMAU20551eps基因簇中的关键基因在菌株各个生长时段均可顺利表达,且在菌株培养6 h时eps基因簇中的部分基因表达量达到最高,是菌株代谢产生EPS的关键性阶段。

