春尺蠖几丁质酶AcinCht10基因的克隆及在逆境胁迫下的表达
陈 龙, 董飞龙,寇 峰,查 干,崔阔澍,马杰茹
(1. 河套学院农学系,内蒙古 巴彦淖尔 015000; 2. 磴口县林业和草原有害生物防治检疫站,内蒙古 巴彦高勒 015299; 3. 磴口县防沙治沙局,内蒙古 巴彦高勒 015299; 4. 巴彦淖尔市草原工作站,内蒙古 巴彦淖尔 015000; 5. 四川省农业技术推广总站,成都 610041)
【研究意义】春尺蠖(Apocheimacinerarius)是鳞翅目、尺蛾科杂食性昆虫。该昆虫在我国多个省(市、自治区)危害牧草、林木和经济树种[1-2],在内蒙古地区主要危害柠条。近年来,春尺蠖的危害对象逐渐由杨、桑、柳、柠条以及经济类果树等林木扩散至玉米和小麦等大田作物,区域也由北部地区的内蒙古及新疆向四川、云南等南部地区扩散。幼虫最早于4月上旬开始发生危害,此时当地昼夜温差较大,最低可达-10 ℃,同时在荒漠化草原中生存需要很强的耐饥饿能力,春尺蠖3龄和4龄幼虫能在如此环境中生存下来且发生危害,与其极强的抗寒性和耐饥性密切相关。【前人研究进展】几丁质在昆虫的角质层、内表皮、围食膜和体壁均有分布,同时还分布于呼吸系统、腔肠结构等组织,在昆虫中起到维持体型、限制昆虫生长及抵御外界不利环境对机体的伤害等作用[3-5]。在昆虫生长发育及蜕皮的发育过程中,部分旧角质层中的几丁质需要降解,用新生成的角质层来替代[6-7]。几丁质酶( Chitinase)和β-N-乙酰葡糖胺糖苷酶(β-N-acetyglucosaminidase)在昆虫蜕皮中具有重要作用,只有二者共同作用才能完成该生理过程[8]。几丁质酶在昆虫的蜕皮、围食膜的降解和昆虫消化等生理过程[9-13]均发挥重要的作用,昆虫生长发育的各个阶段与几丁质酶有紧密的联系。因此,只要几丁质酶在昆虫不同组织和不同生长发育阶段维持一定的表达量,昆虫的正常生长发育过程才能得到保证[14]。根据几丁质酶基因所编码氨基酸的序列同源性,昆虫几丁质酶隶属于18家族几丁质糖基水解酶,该类基因的特点为:大多数均由信号肽,催化区、连接区和几丁质结合区构成。通过生物信息学分析与构建系统进化树等方式,可将几丁质酶及其类似蛋白划分为I~VIII型[11],在昆虫不同的生长发育阶段和不同组织中均体现了这些类型几丁质酶基因的功能差异[15]。赤拟谷盗(Triboliumcastaneum)的III型几丁质酶由2个催化区和1个几丁质结合域构成,在其N端还发现1个跨膜结构域,属于膜结合蛋白,其在两个完全不同的方面(蛹腹部收缩和翅伸展)均发挥重要作用[11]。相关研究表明,几丁质酶还在植物抗菌、抗逆境、生长发育以及共生固氮等方面扮演重要角色[16-22]。拟南芥(Arabidopsisthaliana)几丁质酶基因的表达与逆境胁迫相关[23]。目前,关于昆虫几丁质酶在逆境胁迫功能探究报道较少。在小胸鳖甲(Microderapunctipennis)低温胁迫的转录组数据中发现大量几丁质酶基因与低温胁迫的响应密切相关[24],同时还发现MpCht19和MpCht8c均受低温诱导表达,且MpCht19基因具有组织特异性[25-26]。在经过饥饿处理的3龄桔小实蝇(Orientalfruit)幼虫体内发现BdCht2上调表达,当恢复饲喂后BdCht2基因的表达则被抑制,表明在桔小实蝇蜕皮过程中BdCht2起着重要作用[27]。已有研究证实,赤拟谷盗中(Triboliumcastaneum)TcCht10参与在卵至成虫的各个阶段[28],同时发现第 III 组昆虫几丁质酶 Chitinase 7还在生长发育过程中的几丁质的排布[29]。黑腹果蝇(Drosophilamelanogaster)X组几丁质酶Cht2有助于提高表皮的沉积以及抵御细菌渗透的稳定性[30]。【本研究切入点】为深入了解几丁质酶基因在春尺蠖抵御外界不良环境中的作用,本研究利用本实验室已测春尺蠖3龄和4龄转录组数据,基于测序所得基因序列信息,结合NCBI blastp和RT-PCR技术克隆获取1个几丁质酶基因,分析其序列分子特征及氨基酸理化性质,对其序列进行比对并构建系统进化树,同时分析其在不同温度和饥饿胁迫下的表达谱。【拟解决的关键问题】明确几丁质酶基因在春尺蠖逆境胁迫中作用,为进一步揭示春尺蠖响应逆境胁迫的分子机理奠定基础。
1 材料与方法
1.1 供试昆虫及样品处理
将2021年春季采集于内蒙古巴彦淖尔市乌拉特前旗柠条草场(108°45′23.63″E, 40°46′4.19″N)的春尺蠖雌雄成虫置于实验室内在室温配对饲养(饲喂柠条锦鸡儿),待其产卵后放置于(22±1) ℃,光周期L∶D=18∶6,相对湿度为55%~59% 的ZXQP-R1700上海智城人工气候箱中孵化,孵化后的1龄幼虫以柠条锦鸡儿连续饲养,待幼虫分别发育至3龄和4龄时,进行以下处理:①低温处理(3龄幼虫):选取3龄幼虫(蜕皮后2 d)分别在-10、-5、0、5和25 ℃(对照)下各处理1 h,每个温度处理5头;②饥饿胁迫(4龄幼虫):选取4龄幼虫(蜕皮后2 d)分别饥饿处理 0(CK)、6、24和72 h,每组处理3头。上述处理的昆虫用液氮速冻后冻存于-80 ℃冰箱备用。
1.2 主要试剂
RNA提取、反转录、pMD19-T载体、2×PCR Master Mix等均购自大连宝生物有限公司;大肠杆菌感受态细胞DH5α购自于北京天根生化科技有限公司。
1.3 RNA的提取及cDNA第1链的合成
将春尺蠖各供试样品从-80 ℃冰箱中取出,放置于灭菌后且已用液氮预冷的研钵中研磨,RNA提取过程参照TaKaRa RNA提取试剂盒说明书,待提取完成后分别利用Nano PhotometerTMP-Class超微量分光光度计和1.0%的琼脂糖凝胶电泳检测提取RNA的浓度和质量,待浓度和质量检测合格后反转录合成第1链。
1.4 春尺蠖AcinCht10基因的克隆
本实验通过分别分析春尺蠖3龄不同温度处理下及4龄幼虫不同时间饥饿胁迫下的转录组差异表达情况,结合NCBI blastp与基因注释结果,筛选获取一条在上述两种胁迫下均差异表达且具有完整CDS序列的几丁质酶基因。基于转录组基因序列,利用Primer Premier 5.0软件设计引物扩增春尺蠖几丁质酶基因。以在上述两种处理条件下PCR扩增产物最亮条带样品的cDNA为模板。采用两段式扩增目的基因,第一段扩增引物(250~2166 bp)为F:5′-TTGTGTGGTCAAGGATATCTTGTAAAA-3′和R:5′-CTTCATAGACGAACTGGTTCATGCG-3′;第二段扩增引物(1939~3282 bp)为F: 5′-ACCCTAATCTCTGCACTCACATGAT-3′和R: 5′-GTTTTTGTTTAAGGGTATCTGCTAAT-3′。
PCR采用25 μL反应体系,其中PCR扩增cDNA模板1 μL,正反引物各取1 μL,Master Mix 12.5 μL,剩余体积以RNase-free Water补齐。设置PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,62 ℃/60 ℃退火30 s,72 ℃延伸运行1 min,循环运行30次;72 ℃补齐延伸10 min,而后4 ℃保存。利用1%的琼脂糖凝胶电泳将两段PCR扩增产物电泳检测片段,而后经胶回收和亚克隆送上海生物工程有限公司测序。
1.5 春尺蠖AcinCht10基因的生物信息学分析
利用NCBI在线网站ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测春尺蠖几丁质酶基因的编码蛋白区。使用在线软件 ExPASy-ProtParam tool(http://web.expasy.org/protparam/)对春尺蠖几丁质酶编码氨基酸的基本理化性质进行预测;使用DNAMAN 6.0(Lynnon Biosoft, Canada)软件对选取的其他昆虫几丁质酶基因与春尺蠖几丁质酶基因的序列一致性进行分析;利用SignalIP 5.0(http://www.cbs.dtu.dk/services/SignalP/)在线预测工具对春尺蠖几丁质酶N端信号肽进行预测;蛋白跨膜区则利用TMHMM在线网站(http://www.cbs.dtu.dk/services/TMHMM/)预测;利用NetPhos 2.0 Server在线工具 (http://www.cbs.dtu.dk/services/NetPhos/)预测其磷酸化位点,同时利用DictyOGlyc 1.1在线工具(http://www.cbs.dtu.dk/services/DictyOGlyc/)对春尺蠖几丁质酶进行糖基化位点预测。利用MEGA 6.0软件构建系统进化树,采用邻接法(Neighbor-Joining, NJ)、p距离(P-distance),重复运算1000次。
1.6 春尺蠖AcinCht10基因逆境条件下的表达
为明确春尺蠖幼虫在逆境胁迫下(低温与饥饿)的表达谱,本研究基于前期实验室已测转录组数据(不同低温及不同时间饥饿处理),基于FKPM值,对春尺蠖AcinCht10基因的表达情况进行分析。
1.7 数据统计与分析
利用SPSS 17.0软件中的单因素中Duncan多重比较分析AcinCht10基因表达结果,同时应用GraphPad Prism 7.0软件绘制柱形图,方差分析结果采用平均值±标准误的形式展现,显著水平P<0.05。
2 结果与分析
2.1 春尺蠖AcinCht10基因的克隆
基于转录组数据中的FASTA文件的几丁质酶基因的序列信息,利用两对引物分段扩增,经1.0%浓度的琼脂糖凝胶电泳检测发现,分别在1917 bp(图1-A)和1344 bp(图1-B)处有特异性单一条带,经测序后将2段序列拼接后共获取2967 bp,与转录组数据比对后,序列信息一致,命名为AcinCht10(基因登录号:OK504624)。
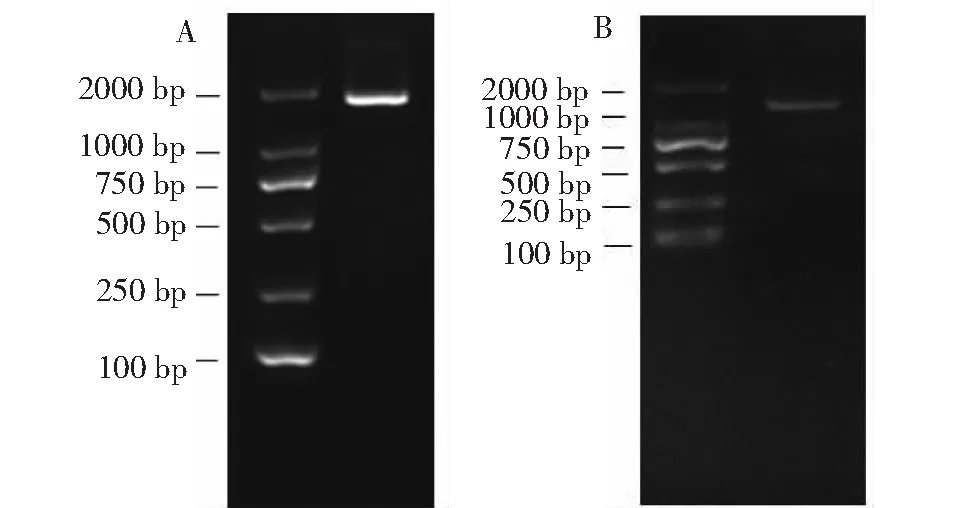
图1 AcinCht10基因片段PCR产物检测Fig.1 PCR production detection of AcinCht10 from Apocheima cinerarius
2.2 AcinCht10基因蛋白的生物信息学分析
春尺蠖AcinCht10基因编码蛋白理化性质预测结果表明,春尺蠖AcinCht10基因序列CDS全长为2967 bp,共编码988个氨基酸,预测分子式为C5017H7689N1349O1490S39,蛋白的预测分子量111.99 kDa,理论预测等电点为6.25;半衰期和脂肪指数分别为30 h和69.62,负电荷残基(Asp+Glu)总电荷为136,正电荷残基(Arg+Lys)总电荷为127;该蛋白的亲水性系数为-0.547,为亲水性蛋白。N端氨基酸为蛋氨酸(Met, M),预测不稳定系数值为37.19,因其小于40,所以该蛋白为稳定蛋白。AcinCht10在N端含1条含26个氨基酸的(MWPPRLLRWTL TILLILAVIAPTSNS)信号肽(图2-A),无跨膜区(图2-B)。此外,该蛋白具有2个催化区,分别位于第95~439位和第531~870位氨基酸,同时具有1个几丁质结合域,位于第923~977位氨基酸(图3),磷酸位点检测(图2-C)发现丝氨酸48个,苏氨酸43个,酪氨酸14个;未检测到糖基化位点(图2-D)。
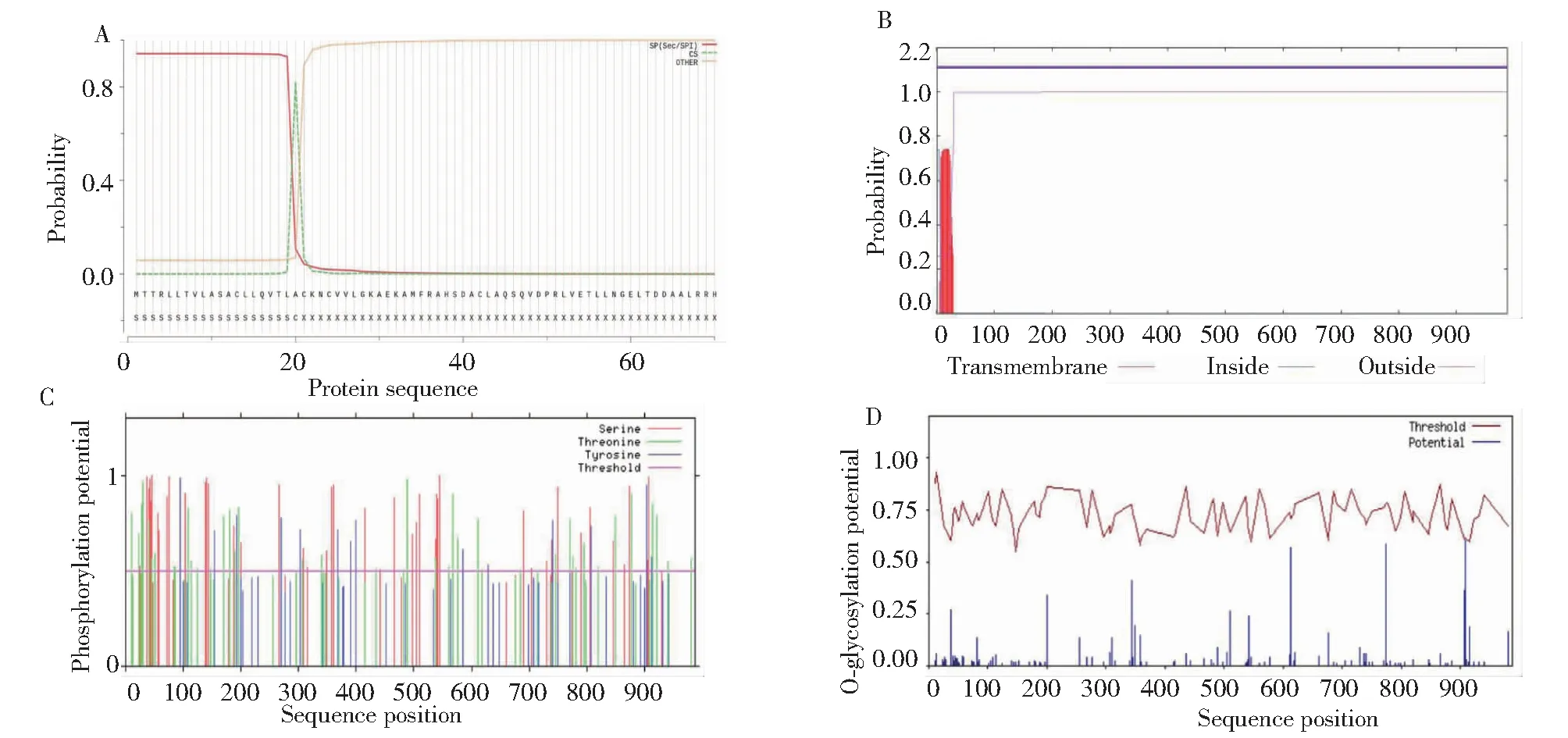
Transmembrane. 跨膜区;Inside. 蛋白质在膜内的概率;Outside. 蛋白质在膜外的概率Transmembrane. Transmembrane region; Inside. Probability of protein inside the membrane; Outside. Probability of protein outside the membrane图2 春尺蠖几丁质酶AcinCht10的信号肽、跨膜结构、磷酸化位点和糖基化位点预测Fig.2 Prediction of the signal peptide, transmembrane structure, phosphorylation sites and glycosylation sites of the AcinCht10 from Apocheima cinerarius

起始密码子与终止密码子用下划线标注;催化结构域(CAD)用阴影标注;几丁质结合域(CBD)用下划线标注Start codon (ATG) and stop codon (TGA) are underlined; Catalytic domain(CAD)is shaded; Chitin binding domain (CBD)are underlined图3 春尺蠖AcinCht10基因的核苷酸及推导的编码氨基酸序列Fig.3 Nucleotide and encoded deduced amino acid sequence of AcinCht10 in Apocheima cinerarius
2.3 春尺蠖几丁质酶基因的同源性比对及系统进化关系分析
在NCBI中搜索其它鳞翅目昆虫几丁质酶基因的氨基酸序列,将搜索获取的序列与春尺蠖AcinCht10进行序列一致性分析,结果(图4)表明,AcinCht10与同鳞翅目昆虫之间的一致性均≥91%,其中,春尺蠖AcinCht10与烟草天蛾(Manducasexta)的一致性最高,为93.12%,其次为粉纹夜蛾(Trichoplusiani)和亚洲玉米螟(Ostriniafurnacalis),分别为92.53%和92.32%,其与甜菜夜蛾(Spodopteraexigua)、桃蛀螟(Dichocrocispunctiferalis)和粘虫(Mythimnaseparata)也均保持较高的一致性,分别为 91.90%、91.60%和91.30%。

Cht来源物种及其 GenBank 登录号: 春尺蠖(AcinCht10,KO504624);烟草天蛾(MsexCht10,XP_030022210.1);粉纹夜蛾(TniCht10,XP_026728882.1);亚洲玉米螟(OfurCht10,AGX32025.1);甜菜夜蛾(SexiCht7,AFM38213.1);桃蛀螟(CpunCht7,ASM94207.1);粘虫(MsepCht,AUF40393.1);共有序列Source species of Cht proteins and their GenBank accession numbers:Apocheima cinerarius(AcinCht10,KO504624); Manduca sexta(MsexCht10,XP_030022210.1); Trichoplusia ni(TniCht10,XP_026728882.1); Ostrinia furnacalis (OfurCht10,AGX32025.1); Spodoptera exigua(SexiCht7,AFM38213.1); Conogethes punctiferalis(CpunCht7,ASM94207.1);Mythimna separata(MsepCht,AUF40393.1); Consensus 图4 春尺蠖与其它昆虫Cht基因的氨基酸序列比对Fig.4 Multiple amino acid sequence alignment of Cht from Apocheima cinerarius and other insects

使用MEGA 6软件中的邻接法构建系统进化树, 春尺蠖Cht蛋白用三角标记The tree was constructed by MEGA 6.0 using the Neighbor-Joining (NJ) method, the Cht protein of Apocheima cinerarium was marked with filled triangle图5 春尺蠖与其它昆虫Cht氨基酸序列构建的系统进化树Fig.5 Phylogenetic tree based on amino acid sequence of Cht from Apocheima cinerarium and other insects
在NCBI中以Chitinase为关键字搜索其它昆虫的几丁质酶基因的氨基酸序列,结合春尺蠖AcinCht10基因的氨基酸序列构建系统进化树。系统进化结果(图7)显示,各类昆虫的8种几丁质酶分别聚为一支上,同时春尺蠖AcinCht10与同为鳞翅目昆虫的烟草天蛾MsexCht10、粉纹夜蛾TniCht10和亚洲玉米螟OfurCht10聚为一类。
2.4 春尺蠖AcinCht10基因在逆境胁迫下的表达分析
从图6-A可以看出,AcinCht10基因随着饥饿处理时间的增加,表达量依次降低且相邻处理间的表达量差异不显著(P>0.05)。AcinCht10基因在25 ℃处理下表达量最高,而在其余温度胁迫下表达量均处于较低水平且差异不显著(P>0.05,图6-B)。
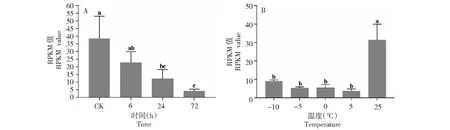
图6 春尺蠖AcinCht10基因在不同时间饥饿处理和不同温度胁迫下的表达Fig.6 Expression profile of AcinCht10 different starvation and at different temperature stress of Apocheima cinerarius
3 讨 论
本研究基于前期已获取的春尺蠖3龄幼虫不同低温处理和4龄幼虫不同饥饿时间处理的转录组数据,筛选出在两数据库中共有差异表达的几丁质酶基因,在NCBI中的blastp中验证后,结合RT-PCR技术克隆获取该基因的编码蛋白区。经序列分析后发现,春尺蠖几丁质酶基因具有两个催化区域,一个几丁质结合区域,符合几丁质酶III型基因的特性[31],而后经序列比对和系统进化分析,发现春尺蠖AcinCht10分别与烟草天蛾MsexCht10、粉纹夜蛾TniCht10和亚洲玉米螟OfurCht10等几丁质酶III型基因序列一致性均在92%以上,且聚为一类,故将春尺蠖几丁质酶基因鉴定为几丁质酶III型基因,并命名为AcinCht10。
通过对AcinCht10基因进行生物信息学分析,AcinCht10基因的CDS全长2967 bp,共编码988个氨基酸,预测分子量为111.99 kDa,与大多数昆虫的几丁质酶III型基因相似。该蛋白的理论预测等电点为6.25,亲水性系数为-0.547,说明属于亲水性的酸性蛋白。AcinCht10基因蛋白第1~26位氨基酸位信号肽,属于分泌蛋白,符合大多数几丁质酶需要分泌道胞外特点。AcinCht10基因蛋白未发现跨膜区,说明其可能是非膜相关蛋白[32]。磷酸位点检测发现丝氨酸48个,苏氨酸43个,酪氨酸14个可能成为磷酸位点,推测其可能在信号转导中扮演重要角色[33]。从AcinCht10与其它目昆虫的几丁质酶基因构建的系统进化树中可以看出,8类几丁质酶分别聚在一支上,而同一昆虫的不同类型几丁质酶基因则分布在不同分支上,在棉铃虫(Helicoverpaarmigera)和荒漠甲虫小胸鳖甲(Microderapunctipennis)的研究中也有相同结果[19,34],说明几丁质酶基因在进化过程中分化程度较大,分化而来的8种不同类型的几丁质酶基因具有不同的生理功能[17]。
昆虫几丁质酶在昆虫生长发育的各个时期均扮演着重要角色,参与诸如昆虫的蜕皮、围食膜降解等过程,同时还在细胞增殖和应对外界不利环境的免疫等过程发挥作用[35-36]。近年来,相关研究发现几丁质酶基因在应对低温胁迫有作用。本研究中,在低温处理下AcinCht10表达量并无明显变化,但小胸鳖甲Mpcht19和MpCht8c在4 ℃条件下7 h后表达量显著上调[18-19],可能是由于AcinCht10处理时间为1 h,而小胸鳖甲的处理时间为7 h。有研究表明,长时冷驯化会伴随着基因表达的较大变化[37-38],而短时、快速驯化则很少有证据表明需要合成新的基因产物。在红尾肉蝇的研究中也得到证实,该虫0 ℃处理2 h后虽然使得抗寒性增强,但并未引起基因表达的变化[39]。本研究表明,春尺蠖AcinCht10基因在低温胁迫下基因表达未发生显著变化,可能由于处理时间过短,虫体内不需要合成新的产物来抵抗外界低温,但在25 ℃样品处理的各环节中未发生异常,为何AcinCht10高表达,有待于今后进一步探究。
几丁质酶基因家族中的几丁质酶类型具有多样化,每种类型的昆虫体内具有的功能也不尽相同,在作用效应和呈现出的表型也有差异[31]。在桔小实蝇的研究中,饥饿增加了3龄幼虫体内BdCht2的表达[20],与本研究的结果相反,可能是由于二者隶属于几丁质酶家族的不同成员,BdCht2是VII型,而AcinCht 10则是III型几丁质酶基因。鳞翅目昆虫属于全变态昆虫,幼虫生长到一定阶段就会退掉旧皮,形成新的表皮,这个过程中则需要几丁质合成通路及几丁质降解途径配合才能实现[40-41]。近年来,在对赤拟谷盗、甜菜夜蛾(Spodopteraexigua)、褐飞虱(Nilaparvatalugens)等昆虫海藻糖酶—几丁质调控通路的研究中发现,几丁质的合成与分解在一定程度上受海藻糖酶(TRE)的调控[42-45]。如在褐飞虱虫体内注入海藻糖酶抑制剂后,10个几丁质酶基因表达都显著或者极显著下降[46]。以上研究表明,几丁质酶基因和海藻糖酶基因的表达是同步的。在本研究中,伴随着饥饿处理时间的增加,几丁质酶基因的表达量呈现下降趋势,可能是由于随着饥饿时间的增加,体内积累的海藻糖逐渐被分解,没有多余的海藻糖可以分解(饥饿72 h后春尺蠖表现为活动减弱,呈现虚弱状态),导致海藻糖酶基因表达降低(未发表),促使几丁质酶基因下调表达。
虽然本研究已完成AcinCht10的克隆,分析了其分子特征,同时明确了该基因与其它昆虫的系统进化关系,以及明确了其在低温和饥饿胁迫下的表达情况,但是还有一些问题需要今后进一步明确。首先,本研究虽然分析了春尺蠖几丁质酶基因在低温和饥饿胁迫下的表达谱,但是还缺乏在上述两种胁迫下的组织表达谱,基因的表达进一步精确到特定组织。其次,还未明确AcinCht10在同一温度不同处理时间的表达情况,同时低温胁迫还缺乏递进式降温处理,以此来模拟真实的自然环境。最后,春尺蠖体内还存在其它类型的几丁质酶基因,还需今后进一步去挖掘,才能够系统全面的明确春尺蠖在逆境胁迫下几丁质酶基因调控的分子机理。
4 结 论
本研究基于转录组数据克隆获取了春尺蠖几丁质酶基因AcinCht10,并对其序列特征、理化性质及系统进化关系进行了分析,同时分析了该基因在低温及饥饿胁迫下的表达情况。研究结果证明,春尺蠖几丁质酶基因AcinCht10属于III型,饥饿胁迫均可诱导下调表达,而在短时低温胁迫下,表达量有变化但差异不显著,以上结果揭示AcinCht10为春尺蠖在逆境条件下可被诱导表达,本研究结果有助于为今后开展春尺蠖抵御逆境胁迫分子机理的研究奠定基础。

