基于加权基因表达网络和LASSO回归筛选骨肉瘤预后和转移相关基因
张 毅,姚士军,刘朝旭
骨肉瘤(osteosarcoma,OS)是一种在青少年中常见的恶性骨肿瘤,是导致青少年肿瘤死亡及截肢率的主要疾病之一[1]。近年来随着临床治疗的方法不断获得更新、优化、改进,OS患者的预后有了明显的改善并同时提高了其生存质量。但对于存在同时或异时性远处转移的OS患者其5年生存率极差[2~4]。而目前OS发生转移的潜在分子机制仍尚不明确,同时其发生、发展及转移的相关标志物也未获得较好的验证。因此,寻找OS新型有效的分子标志物及基于该标志物构建治疗的新靶点,将对判断OS预后和治疗提供一种全新的策略。
1 材料与方法
1.1 实验材料
利用UCSC Xena(https://xena.ucsc.edu/)网站下载癌症基因组图谱-产生有效治疗方法研究项目(The Cancer Genome Atlas,Therapeutically Applicable Research To Generate Effective Treatments,TCGA-TARGET)OS患者转录组基因表达数据谱和临床相关信息(检索起止:建库至2021年5月31日)。剔除数据库中生存情况及病理分期不明患者,最终纳入84例OS患者,其中男性37例,女性47例;平均年龄14.99岁(标准差7.80岁)。原发肿瘤位置:脚/足76例;手/手臂6例;骨盆2例。初始诊断即存在远处转移患者21例。从ONCOMINE数据库(https://www.oncomine.org)选出4个数据集,用于外部验证。
1.2 方法
1.2.1 基于加权共表达基因网络筛选核心模块
加权共表达基因网络(weighted gene co-expression network analysis,WGCNA)是一种对数据集中每个样本的全部数据,包括基因表达数据、临床信息数据等,进行整合分析的一种生物信息学分析方法。其主要是通过将每个样本的数据进行均值归一化后构建无序网络并进行关联分析,同时当其应用于临床样本的测序数据中,可以将临床信息与基因表达进行关联,目前已被广泛应用于生物体内识别和筛选复杂疾病的标志物[5]。简单的概述该方法:首先利用R软件(4.0.1版本)中的“WGCNA”分析包构建该模型[6,7];构建模型中的相关矩阵,计算所有基因的邻接关系,确定软阈值大小;基于软阈值的大小,对基因之间的无序邻接关系进行截断,并最终将其转化为拓扑重叠矩阵(topological overlap matrix,TOM),用以度量基因之间的网络连通性[8,9]。并基于TOM的差异程度进行层次聚类,从而将基因表达差异程度相似的基因纳入同一个基因模块(module eigengenes,ME)中[6,7]。
1.2.2 特征模块和核心基因筛选
为明确实验研究所构建的WGCNA模型哪个模块为对整体样本贡献度最大,通过Pearson相关分析计算每个模块与每个样本之间的关联系数,并通过取绝对值后累加获得关联系数最大模块,深蓝(midnight blue)模块。利用STRING网站(https://string-db.org/)对深蓝模块中所有基因进行蛋白质-蛋白质相互作用(protein-protein interaction,PPI)分析,并利用Cytoscape软件中的复杂分子关联分析(molecular complex detection,MCODE)插件进一步分析。同时利用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)回归分析筛选深蓝模块中核心基因。基于上述3种方法最终获得了3个基因核糖体RNA加工8基因(ribosomal RNA processing 8 gene,RRP8)、核糖体RNA加工9基因(ribosomal RNA processing 9 gene,RRP9)、线粒体核糖体蛋白48基因(mitochondrial ribosomal protein L48 gene,MRPL48),用以后续验证。
1.2.3 GO功能学、KEGG通路分析和GSEA分析
为了解深蓝模块涉及的功能学和通路,对深蓝模块中所有基因进行基因本体论(gene ontology,GO)和京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析,用以筛选与其相关的功能和通路。为了解核心基因在OS患者中的潜在功能,利用TCGA-TARGET OS患者数据集进行基因组富集分析(gene set enrichment analysis,GSEA)。将P<0.05和富集评分(enrichment score,ES)>0.3作为截止标准。
1.3 统计学方法
t检验用来分析两组样本之间是否存在差异。利用X-tile软件,基于最小P值和最大Log-rankχ2值进行分析计算基因的最佳取值点[10]。受试者工作特性(receiver operator characteristic,ROC)曲线被用于评估核心靶基因表达高低预测OS转移能力。利用卡普兰-迈耶曲线(Kaplan-Meier,K-M)分析基因高低表达对OS患者预后的影响。P<0.05为差异有统计学意义。
2 结果
2.1 构建共表达模块
基于WGCNA方法,对TCGA-TARGET数据集中所包含的转录组基因基于其每个基因的相对表达量进行模块富集分析(图1)。最终共获得了67个相应的ME(图2),依据各模块在各个样本上皮尔森系数绝对值相加为最高者认定为响应系数最高模块,最后筛选获取了深蓝模块,深蓝模块共含有214个基因。

图1 84例OS患者的转录基因WGCNA模式图Fig.1 Transcriptional WGCNA pattern of 84 OS patients
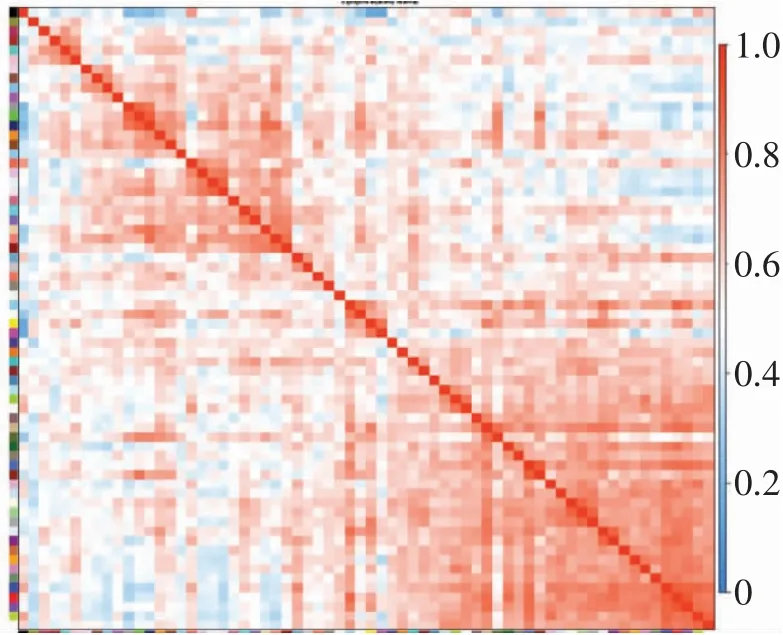
图2 67个ME相关分析图Fig.2 Correlation analysis diagram of 67 ME
2.2 深蓝模块相关功能分析及核心分子的筛选
依据深蓝模块中相关基因筛选表达响应基因参与的信号通路关联(图3A、B)。研究结果提示,主要与凋亡进程、mRNA修饰、胰岛素释放等相关。利用PPI分析和MOCDE插件筛选位于调控核心位置的基因(图4),并进一步采用了LASSO分析(图5),筛选出12个与预后相关的靶基因:蛋白酶体20S亚基7基因(proteasome 20S subunit alpha 7 gene,PSMA7)、RRP9、1型路障动力轻链蛋白基因(dynein light chain roadblock-type 1 gene,DYNLRB1)、自噬相关4D半胱氨酸肽酶基因 (autophagy related 4D cysteine peptidase gene,ATG4D)、RRP8、蘑 菇 家 族 成 员2基 因(shroom family member 2 gene,SHROOM2)、序列相似家族86个成员C1基因(family with sequence similarity 86 member C1 gene,FAM86C1)、RAS致癌基因家族成员24基因(member RAS oncogene family 24 gene,RAB24)、雌激素相关受体α基因(estrogen related receptor alpha gene,ESRRA)、MRPL48、假尿苷合酶1基因(pseudouridine synthase 1 gene,PUS1)和脂肪非典型钙黏蛋白4基因 (FAT atypical cadherin 4 gene,FAT4)。将上述3种方法所筛选结果综合分析,获得RRP9、RRP8和MRPL48。

图3 深蓝模块GO分析(A)和KEGG分析(B)Fig.3 GO analysis(A)and KEGG(B)analysis of midnight blue module
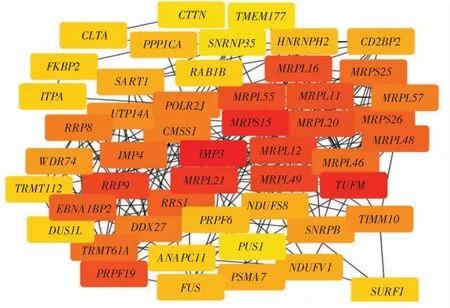
图4 深蓝模块中相关基因PPI分析Fig.4 PPI analysis of related genes in midnight blue module
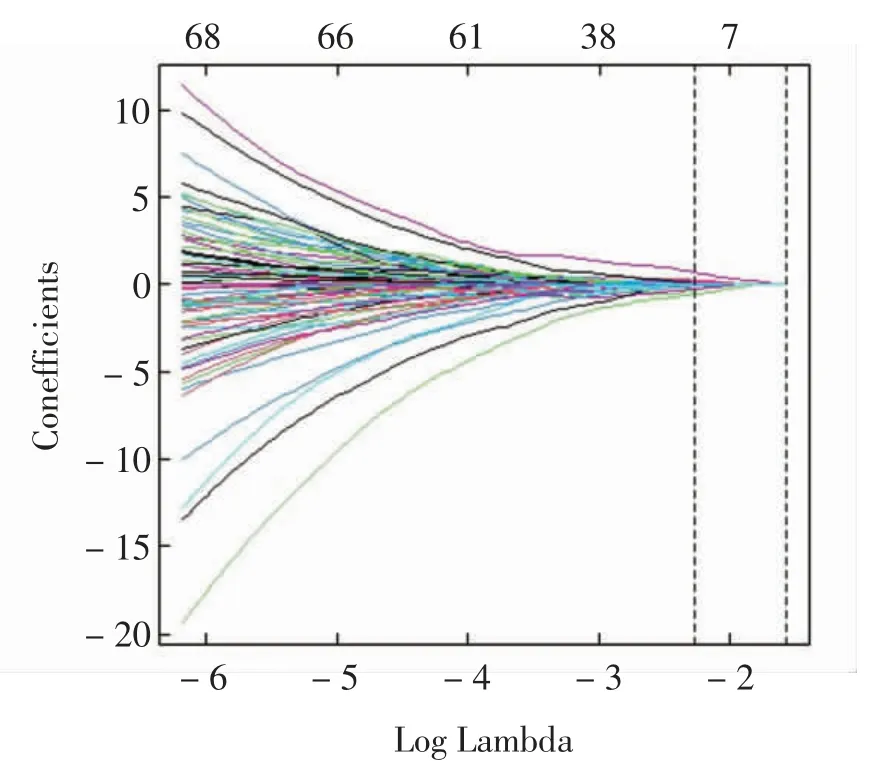
图5 深蓝模块中相关基因LASSO筛选分析图Fig.5 LASSO screening analysis diagram of related genes in midnight blue module
2.3 RRP8、RRP9和MRPL48验证预后及转移相关表达
为验证RRP8、RRP9和MRPL48预测患者预后,应用X-tile软件选取RRP8、RRP9和MRPL48最佳截点。以RRP8表达量为1.55、RRP9表达量为3.63和MRPL48表达量为2.29为截点将获得最佳结果,高表达RRP8和RRP9的患者有较好的无病生存期(P=0.085、0.002;图6A、B);低表达MRPL48的患者有较好的无病生存期(P<0.001;图6C)。高表达RRP8和RRP9的患者有较好的总生存期(P=0.071、0.017;图6D、E);低表达MRPL48的患者有较好的总生存期(P=0.001;图6F)。

图6 RRP8、RRP9和MRPL48在TCGA-TARGET数据集中OS患者K-M曲线分析Fig.6 K-M analysis curves of RRP8,RRP9 and MRPL48 of OS patient in TCGA-TARGET datasets
实验利用ONCOMINE数据库对RRP8、RRP9和MRPL48基因进行Meta分析(图7)。在纳入的4个数据集中,RRP8和RRP9在癌组织中表达较癌旁组织低(P=0.060和P=0.172);MRPL48在癌组织中较癌旁组织高表达(P=0.035)。进一步分析RRP8、RRP9和MRPL48在转移病人中表达情况(图8),结果提示在转移组中RRP8、RRP9和MRPL48均呈现高表达(RRP8:转移组1.66±0.33vs非转移组1.62±0.26;P=0.512。RRP9:转移组3.88±0.99vs非转移组3.84±0.52;P=0.811。MRPL48:转移组2.56±0.52vs非转移组2.31±0.43;P=0.029)。

图7 RRP8、RRP9和MRPL48在ONCOMINE数据集中OS患者癌组织和癌旁组织中表达Fig.7 Expression analysis of RRP8,RRP9 and MRPL48 betweeen cancer tissues and adjuvant cancer tissues in OS patients of ONCOMINE datasets

图8 RRP8、RRP9和MRPL48在OS转移和非转移患者中表达比较柱状图Fig.8 Comparison of expression analysis of RRP8,RRP9 and MRPL48 between metastasis and non-metastasis OS patient
为进一步了解实验所筛选的3个基因对于预测OS转移能力,进一步利用ROC曲线分析(图9)。结果提示,RRP8、RRP9和MRPL48三者对于预测OS转移其曲线下面积(area under curve,AUC)分别为:RRP8AUC=0.504,P=0.955;RRP9AUC=0.509,P=0.913;MRPL48AUC=0.663,P=0.025。上述结果进一步提示了MRPL48可以较好地预测OS患者是否发生转移。

图9 ROC曲线检测RRP8、RRP9和MRPL48预测OS转移能力Fig.9 Curves of RRP8,RRP9 and MRPL48 predicted metastasis in OS patients by ROC
2.4 GSEA功能学分析
如前述,在实验筛选所获得的基因中,MRPL48在预测OS患者预后和转移均表达了较强的作用。为了解MRPL48涉及的功能调控,利用TCGA-TARGET数据库并采用GSEA分析其所涉及的下游通路。MRPL48主要涉及了细胞黏附机制和碱基切除等(图10)。

图10 MRPL48在预测OS患者预后和转移的GSEAFig.10 GSEA diagrams of MRPL48 predicted prognosis and Metastasis in OS patients
3 讨论
尽管目前手术、化学治疗和放射治疗等多种治疗方案已被证实对OS有较好的疗效且已广泛应用于OS的治疗当中,但由于部分患者出现同时性或异时性远处转移,其5年生存率极低。因此,如何更为精准地识别初诊患者是否具有高危转移风险是临床中亟待解决的问题。笔者利用TCGA-TARGET数据库中84例OS患者的转录组测序数据和对应的临床样本的临床数据,利用基于无序网络的WGCNA算法并结合LASSO、PPI分析、MOCDE分析筛选获得了MRPL48,并通过内部验证MRPL48能够较好地预测OS患者的预后及转移情况。同时笔者利用了外部的ONCOMINE数据库验证了MRPL48在OS癌组织和癌旁组织中的表达。结果提示MRPL48在OS的患者中可能扮演癌基因的角色。
近年来,随着基因芯片、测序等多种高通量检测手段的高速发展给人们探索不同疾病的发病机制、分子标志物等做出了巨大的贡献。基于不断发展的高通量检测手段,目前在多种肿瘤中,已发现了较多应用前景较为广阔的分子标志物;同时基于对疾病的发病机制探索,也有针对个体突变分子的成熟靶向药物上市,并获得了较好的临床结果。因此基于高通量检测手段将对人们探索OS患者有效分子标志物及其潜在治疗靶点产生重要的影响。同时近年来随着针对基因表达情况的算法不断改进及优化,进一步促进人们更好地探索疾病的机制及分子标志物。笔者研究基于公共数据库中所含有的OS基因芯片数据并结合其所对应的临床数据,利用了目前被广泛使用的WGCNA算法[11,12],该算法通过系统的绘制个体生物网络互作图[7,9],可以有效地避免传统筛选方法:差异基因筛选算法所导致的遗漏调控过程中的核心分子。同时该算法有效地将患者临床资料和基因表达数据进行了有机的关联和结合,使得人们的研究有效地从基础研究进入了临床前应用中[13,14]。笔者研究利用WGCNA算法对TCGA-TARGET数据库中OS患者转录组数据深入分析,筛选获得3个核心基因:RRP8,RRP9,MRPL48。进一步通过生存分析验证笔者所筛选的核心基因是否能准确地预测OS患者的预后情况,结果提示:RRP9和MRPL48能较好地预测OS患者的无病生存和总生存率,但RRP8不能获得较好的结果。对于OS患者,当发生了远处转移后,其预后极差。因此,笔者进一步分析上述3个基因在OS转移和非转移患者中的表达情况,其中只有MRPL48在转移患者和非转移患者中呈现表达差异。同时在预测患者是否发生转移时,MRPL48基因表现出较好的结果。而RRP8和RRP9均未表现出较好的预测能力。同时,笔者在外部的ONCOMINE数据库中检测RRP8、RRP9和MRPL48三者在OS癌组织和癌旁组织中表达情况,结果提示在OS患者中,MRPL48在癌组织的表达量高于癌旁组织。上述结果均进一步证明了MRPL48可以作为预测OS预后和转移的有效分子标志物。
MRPL48属于线粒体核糖体蛋白家族,目前针对MRPL48在肿瘤中所发挥作用的报道较少。Hu TT等[15]报道显示,通过CRISPR-Cas9技术敲减MRPL48可以显著增加结直肠癌细胞对西妥昔单抗的敏感性。Fernandez-Ranvier GG等[16]报 道 显 示,MRPL48可 以做为肾上腺皮质癌的抑癌基因。如上述,笔者发现MRPL48在肿瘤研究中存在着分歧,在部分肿瘤中其发挥了癌基因的作用,在部分肿瘤中发挥了抑癌基因的作用。因此进一步在不同肿瘤中探索其所产生的作用将对研究其在肿瘤中发挥不同作用的机制产生较大的帮助。笔者研究结果发现,MRPL48在OS患者中,高表达MRPL48与更差的预后相关,同时在癌组织中MRPL48高表达,上述结果提示MRPL48在OS中扮演癌基因的角色,并涉及OS患者预后及转移。同时笔者研究进一步通过GSEA分析探索了MRPL48可能涉及的下游功能,结果提示MRPL48可能通过调控细胞黏附调控了OS的发生、发展及转移。
总之,笔者研究通过TCGA-TARGET数据库中OS转录组数据及其相对应的临床资料,利用WGCNA算 法 和LASSO、PPI、MOCDE方 法 筛 选 并 验 证MRPL48高表达与OS患者较差预后相关。MRPL48在OS患者中的癌组织较癌旁组织中表达高,其在转移患者中较非转移患者表达也更高。MRPL48有可能成为OS新的预后和转移指标,有助于OS患者个性化治疗及临床预后判断。

