番茄红素合成调控基因SISGR1的分子特征及sgRNA分析
刘江娜,张西英,李荣霞,张小伟,白云凤,张爱萍
(新疆生产建设兵团第六师农业科学研究所,新疆五家渠 831300)
番茄红素(Lycopene)含量与番茄果色相关,也是衡量番茄营养品质的重要指标。番茄红素是一种功能性天然色素,在各种类胡萝卜素中抗氧化能力最强,能助于人体细胞免受氧自由基的伤害,保护生物膜,对于提高人体免疫力和延迟机体细胞衰老及预防癌症具有重要作用[1]。人体不能自主合成番茄红素,只能通过膳食摄取。番茄(Solanum lycopersicum)则是人们通过日常饮食补充番茄红素的重要来源。
滞绿蛋白STAY-GREEN(SGR)最初是在草甸羊茅(Festuca pratensisi)中发现的[2],随后也在多种植物中被发现,该蛋白位于叶绿体中,主要参与调控叶绿素的降解代谢[3]。1999年有学者发现成熟过程中仍保留叶绿素的gf突变体番茄,该突变体叶片完全滞绿,果实在成熟过程中由于仍含有相当数量的叶绿素,导致果实出现褐色或铁锈色的表型[4]。直至2008年番茄中的SGR1基因首次由康奈尔大学从gf突变体中克隆出来,并且将其命名为SlSGR1[5]。目前关于番茄滞绿基因SGR1的研宄主要集中在叶片及叶绿素降解途径中,有研究表明SGR1通过影响叶绿素合成途径中关键基因PAO酶的活性来发挥其作用[6]。Sakuraba等[7]发现SGR1在叶绿素降解过程中具有招募作用,可能招募叶绿素分解代谢酶,同时与捕光复合体结合形成SGR-CCEs-LHCII复合体,从而调节叶绿素的降解;在非生物胁迫下,过表达SGRL的呢喃届出现早期叶片变黄,SGRL-1突变体在盐胁迫下出现滞绿表型[8];然而针对滞绿突变体在番茄果实色素积累方面的研究较少,有研究发现八氢番茄红素是合成番茄红素的前体,其合成量直接影响番茄红素的形成[5]。八氢番茄红素合成酶(phytoene synthase,PSY)催化两个GGPP(geranylgeranyl pyrophosphate,牻牛儿基牻牛儿基焦磷酸)分子聚合生成八氢番茄红素,是番茄红素合成途径中第一个也是最为关键的限速酶。番茄中过表达SlPSY1基因,可显著提高果实中番茄红素含量[9]。番茄SlSGR1蛋白能与八氢番茄红素合成酶SlPSY1互作,抑制SlPSY1在番茄果实成熟期的活性,RNAi沉默SlSGR1基因,可提高SlPSY1的活性,增加番茄果实的番茄红素含量[10-12]。
近年来以多种新型高效的DNA靶向内切酶为基础建立的基因编辑技术[13],可以对植物靶基因进行定点突变或替换,不必再通过外来转基因调控靶基因功能[14]。在各种基因编辑技术中,CRISPR/Cas9技术操作较为简单、效率高,应用更为广泛,它通过特异性RNA(Guide RNA,gRNA)与靶基因碱基互补配对,将Cas9核酸酶引导至靶基因特定位点,将DNA双链切断。断裂后的DNA修复主要有两种机制,一是非同源末端连接(Nonhomologous end joining,NHEJ),在连接过程中会在断裂位置产生少量核苷酸的插入或删除,造成移码,使基因功能失活;二是同源重组(Homologous recombination,HR),会产生精确的定点替换或者插入突变[15]。利用CRISPR/Cas9技术已对番茄的多个基因,如转录因子RIN、RNA编辑因子SlORRM4[16]及生长相关的DELLA基因[17]进行了编辑,也为提高番茄红素含量提供了新思路[18]。
本文分析番茄SISGR1的特征,设计和筛选该基因编码区及其上游启动子区的sg RNAs,以期为利用CRISPR/Cas9技术调控SISGR1表达及提高番茄果实中的番茄红素含量提供参考。
1 材料与方法
1.1 SlSGR1保守结构域分析
利用NCBI搜索番茄SlSGR1蛋白质序列及其保守结构域,BlastP比较SGR1家族的多态性。
1.2 染色体定位和基因组结构的确定
利用NCBI核酸数据库查询获得SlSGR1的cDNA,以此作query,搜索番茄基因组数据库得到相应的gDNA序列和所在染色体信息,序列比对确定SlSGR1的基因组结构以及外显子所在染色体位置。
1.3 数字表达谱的建立
以番茄功能基因组数据库网站(http://ted.bti.cornell.edu/cgi-bin/)中RNA-seq data的数据作基础,建立SlSGR1基于RNA-seq的数字表达谱。
1.4 sgRNA的设计和选择
根据CRISPR-Cas9靶点设计原则,利用CRISPRdirect(https://crispr.dbcls.jp/)[19]筛选SlSGR1及其启动子区域的sg RNA序列,即3′端有PAM元件的20个连续的碱基序列,PAM元件设定为NGG,sgRNA的结构为5′-(N)20NGG-3′,N为任意核苷酸。
1.5 SlSGR1启动子顺式作用元件分析
根据SlSGR1gDNA所在染色体信息,获取SlSGR1基因上游1 500 bp的启动子序列。利用PlantCare服务器(http://bioinformatics.psb.ugent/webtools/plantcare/html/search_CARE.html)[20]对SlSGR1启动子序列进行顺式作用元件分析。
2 结果与分析
2.1 番茄SlSGR1的分子特征
从GenBank搜 索 到番茄SlSGR1的蛋白质序列(Gen Bank Accession:AAY98500.1),该蛋白由272个氨基酸组成,平均分子质量30 532.49 u,摩尔消光系数40 540 m L/(mg·cm),理论等电点pI为8.65,GRAVY(疏水性算术平均值)为-0.34,属于亲水蛋白,没有跨膜结构域、信号肽和核定位序列。该蛋白的48~200 aa具备典型的staygreen superfamily保守结构域。序列比对表明,不同物种氨基酸序列多态性主要发生在staygreen结构域的下游(图1)。

图1 番茄SlSGR1的保守结构域及与其他物种序列比对Fig.1 Conserved domain and multiple alighnment analysis of SlSGR1 in tomato
2.2 SlSGR1的基因组定位和结构
以SlSGR1序 列(GenBank Accession:AAY98500.1)作querry在番茄的基因组数据库进行Blast,结果表明SlSGR1位于番茄的第8号染色体(CP023764.1),起始密码子至终止密码子的位置在63 418 923~63 416 720 nt,长2 204 nt,由4个外显子、3个内含子组成,外显子总长819 nt,内含子总长1 385 nt,外显子的长度在117~360 nt,内含子的长度变化较大,在100~1 118 nt(图2)。
2.3 基于RNA-seq的SlSGR1数字表达谱
以番茄功能基因组RNA-seq数据库为基础,分析SlSGR1基因(ID TC119167)在番茄不同组织以及不同生长时期的的表达谱(图3)。结果显示,SlSGR1基因在成熟的红果中大量表达,在破色期的果实和开放的花中有少量表达,而在种子、胚根、顶端分生组织、不同时期的幼苗、根系、叶片、花蕾、绿果中均无表达,验证SlSGR1基因是与果实中番茄红素合成和代谢相关的基因。
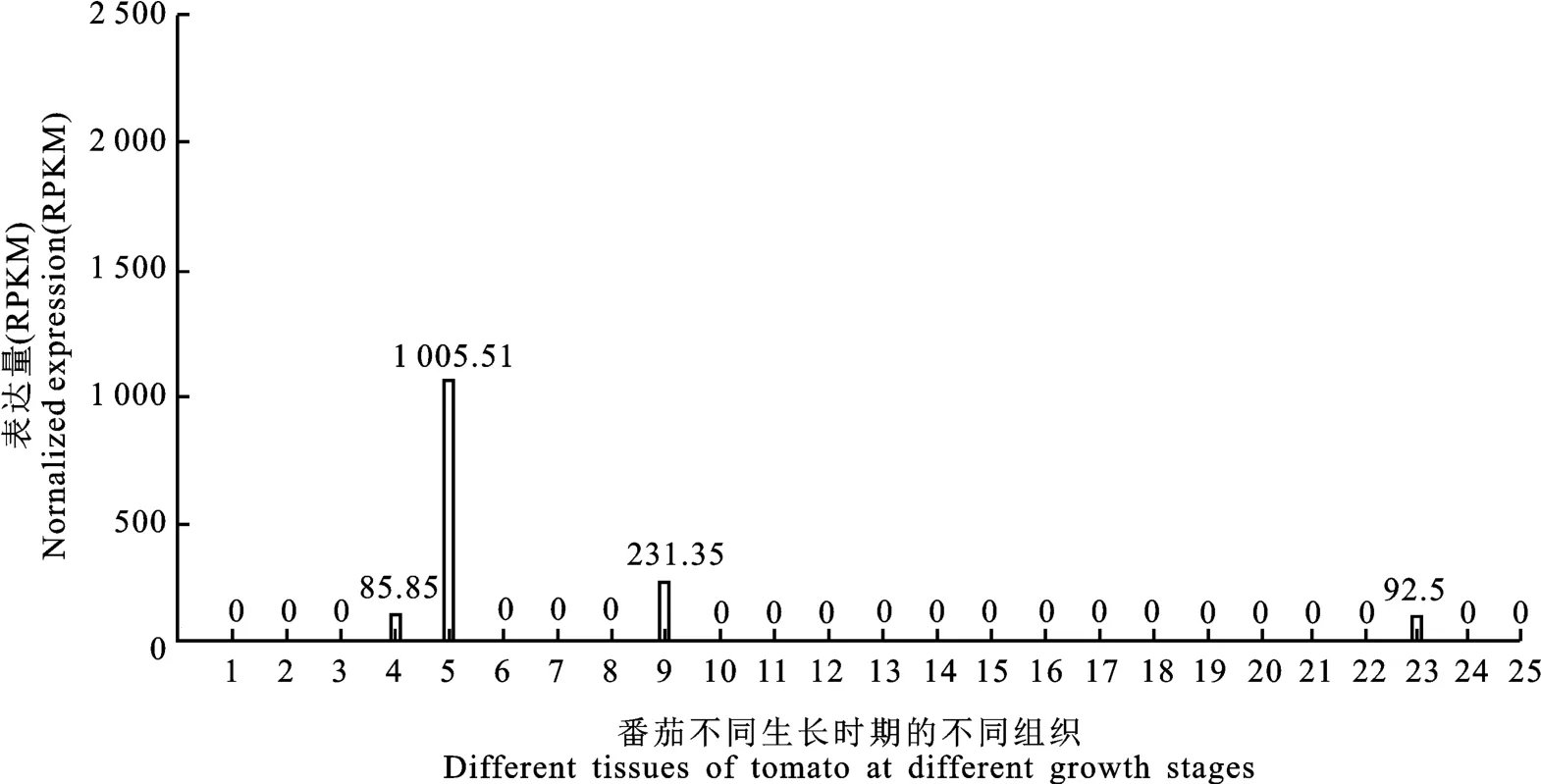
图3 基于RNA-seq的SlSGR1数字表达谱Fig.3 Digital expression profile of SlSGR1 based on RNA-sequence
2.4 sgRNA的设计和评价
设定PAM为NGG,CRISPRdirect在线分析表明SlSGR1序列分布76条sgRNA(图4),其中正链分布46条,负链分布30条,其中12条横跨两个外显子邻接处,不适合进行编辑,另有16条含有TTTTs序列,使用pol III启动子时应尽量避免选用。在其余的58条sg RNA中,有1条其邻近PAM 12 nt的种子序列在番茄全基因组上是唯一序列,特异性最好,位于681~703 nt,另有5条sgRNA的邻近PAM 12 nt的种子序列除了靶位点外,在基因组的其他位置只有1条与其完全匹配的序列,可作为候选序列。
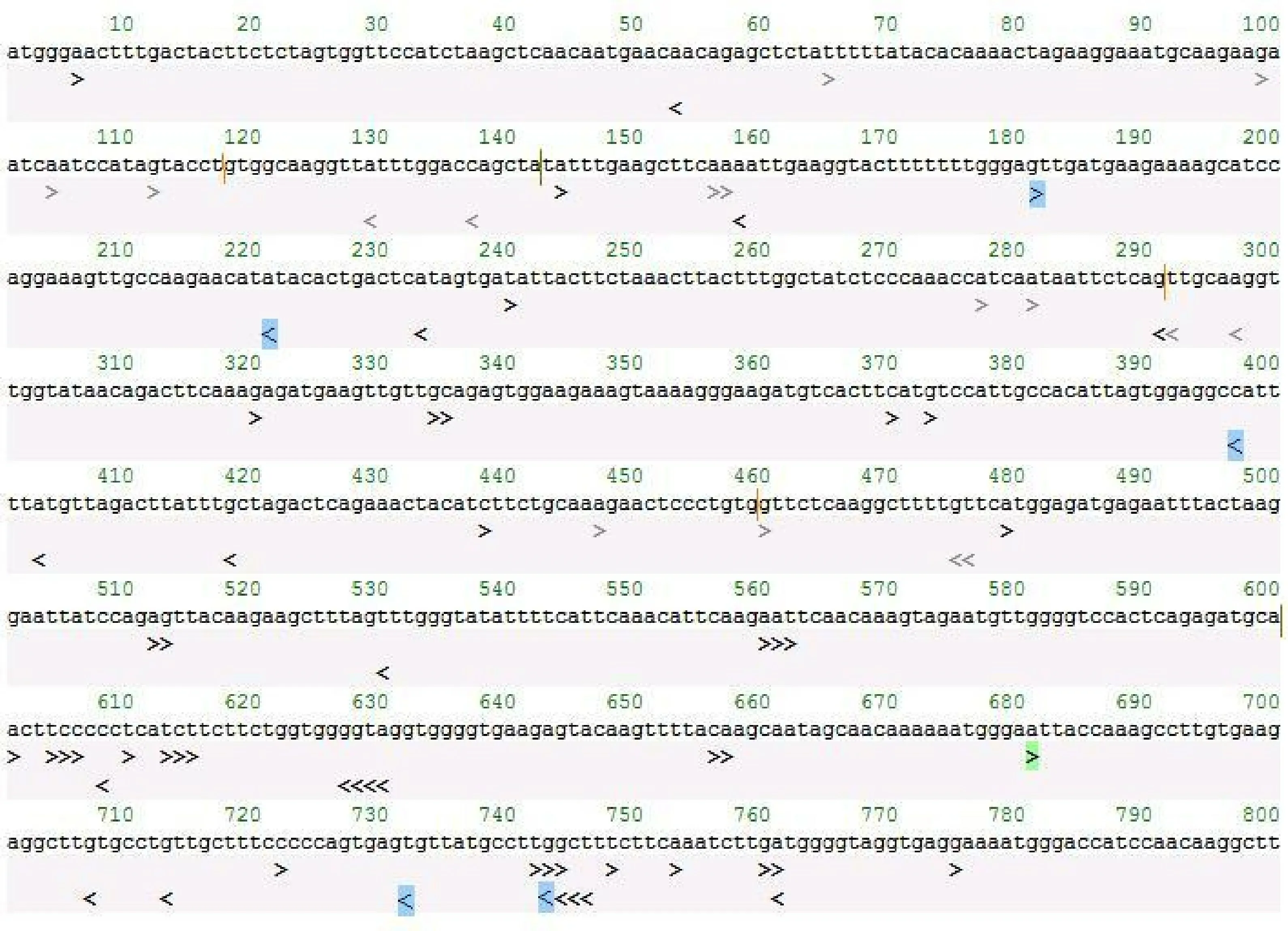
图4 sgRNAs在SlSGR1的分布Fig.4 Position of sgRNAs in SlSGR1
2.5 SlSGR1启动子顺式作用元件和sgRNA的分布
基因表达主要受其上游的启动子上分布的顺式作用元件调控。利用在线工具PlantCare分析SlSGR1上游1 500 bp的启动子序列。结果显示,该序列除了含有典型的TATA-box和CAAT-box核心启动子元件外,还分布有ABRE3a、ABRE4、AE-box、AP-1、ARE、AT~TATA-box、CAAT-box、ERE、G-Box、MYC、Myb、STRE、TATA-box、TC-rich repeats等众多顺式作用元件,其中一些诱导型元件的分布见表1。

表1 SlSGR1启动子顺式作用元件分析Table 1 Predicted cis-regulatory elements in SlSGR1 promoter
搜寻SlSGR1启动子序列分布的sg RNA,发现含有86条(图5),其中正链分布44条,负链分布42条,剔除15条含有TTTTs的序列,尚有71条可供选择,其中在启动子上游有2条特异性较高,分别位于107~85(负链)和215~193 nt(负链),其邻近PAM 12 nt的种子序列在番茄全基因组上是唯一序列,特异性较好。有2条sg RNA(分别位于305~327nt和306~308)包含了AEbox元件,有5条sgRNA(分别位于625~647 nt、634~656 nt、635~657 nt、1 032~1 054 nt和1 033~1 055 nt)包含ABRE和G-box元件,位于1 119~1 231 nt的sgRNA则含TAC-element元件,意味着选用这些sgRNA进行基因编辑,有可能使所含的启动子元件序列发生突变致使功能变化。

图5 SlSGR1启动子包含的诱导型顺式作用元件和sgRNAsFig.5 Position of cis-acting elements and sgRNAs in promoter of SlSGR1
3 讨论
番茄果实成熟时,随着叶绿素逐步降解和有色体合成,呈现不同果色[21]。番茄滞绿基因SlSGR1突变[5],使得果实中叶绿素降解不完全而“滞绿”。番茄滞绿突变多为点突变,有不同的突变体[22],表现不同的滞绿程度,使果色深浅不一。番茄滞绿突变是非功能型突变,叶绿素降解过程受到抑制,但不影响植株衰老进程中的其他环节[23]。SlSGR1蛋白可与PlPSY互作并影响其活性,调控果实中番茄红素的含量[10]。近年来以多种新型高效的DNA靶向内切酶为基础建立的基因组编辑技术[13],因具有高效准确、制作简单的特点,已被广泛应用到植物基因功能研究和定向改良植物性状方面[24],该技术可以在不同物种中对目标基因进行定点敲除、单核苷酸或多核苷酸片段置换、添加等靶向修饰,不必通过导入反义基因或RNAi基因抑制靶基因功能。本研究中CRISPRdirect在线分析表明SlSGR1序列分布76条sg RNA序列,其中6条sgRNA可作为后续种子序列,为利用CRISPR/Cas9基因编辑获得不同位点、不同类型的突变、提高PlPSY活性提供了可能。另外,其中的一些sg RNA序列具有较高的序列特异性,基因编辑时有可能规避脱靶效应,可作为优先选用序列。
基因表达产物取决于编码序列,表达的时空量则由启动子调控。利用CRISPR/Cas9突变相关基因的启动子而不是这些基因本身,能够实现对数量性状的精细调节,微调基因表达而不是剔除或灭活它们编码的蛋白[25]。Rodriguez等[26]利用CRISPR/Cas9技术对启动子区域进行基因组编辑,实现对番茄产量性状(果实大小、花序分枝和株型)的精细调控。对水稻Wx基因启动子上的关键顺式作用元件进行基因编辑,创制了多个可微调直链淀粉含量和蒸煮食味品质的新Wx等位基因[27],这些研究表明对启动子进行基因编辑可以适度调节目的基因的表达。
利用在线工具PlantCare分析番茄SlSGR1上游1 500 bp的启动子序列,发现SlSGR1启动子区分布多条顺式作用元件,如脱落酸应答元件ABRE、水杨酸应答原件TCA-element,光应答原件AE-box、G-Box、逆境胁迫响应元件TC-rich repeats等,可能预示SlSGR1的表达受光照、激素及逆境胁迫的调控,为通过调节日长、使用激素或胁迫刺激等途径调节SlSGR1的表达特性提供了可能。利用CRISPR/Cas9基因编辑可使番茄SLCLV3基因的启动子区产生多组突变,诱导番茄的花序、果实、株型发生广泛变异[26],实现对基因表达的微调和数量性状的调控。另外,选用2条sg RNA进行双编辑,将有可能定点删除2条sg RNA之间的启动子片段及顺式元件序列,对番茄SlSGR1启动子序列进行分析,发现该序列含有86条sgRNA,其中2条特异性较高且位于序列上游,其邻近PAM 12 nt的种子序列在番茄全基因组上是唯一序列,可有效降低脱靶效益,二者的间隔距离为108 nt,可作为后续基因编辑载体构建的首选sg RNA。其中7条sg RNA还包含顺式作用元件序列,预示如选用这些sgRNA进行基因编辑将使所含的顺式元件序列发生突变,为原位探明这些元件的功能,进而调节启动子表达特性、提高番茄果实的番茄红素含量探索一条新途径。
在进行基因编辑时,核酸内切酶切割导致产生DNA双链断裂,然后细胞通过非同源末端连接或同源重组修复断裂。同源重组可产生精确的定点替换或者插入突变,但发生频率较低,也有研究报道,借助双生病毒的复制元件能够提高番茄细胞中的同源重组模板DNA的表达量,提高重组效率[28]。本研究分析表明,番茄SlSGR1基因及其启动子区分布诸多g RNA,为通过同源重组修复在SlSGR1基因及其启动子区插入或替换特定序列、创造新的等位基因提供了基础。

