基于甲基化位点的筛选建模诊断结直肠癌
薛春萌,高洁,李嘉乐,李荣佳,刘畅,梁建伟
1.山东第一医科大学第一附属医院(山东省千佛山医院)健康管理学,山东省健康体检工程实验室,山东济南 250000;2.山东第一医科大学基础医学院,山东济南 250000;3.山东省泰安市中心医院普外科,山东泰安 271000
结直肠癌(colorectal cancer,CRC)作为消化系统好发癌症之一,近年来在全世界范围内发病率呈显著上升的趋势[1]。在我国,CRC的疾病负担也较重,其呈现男性高发、高龄高发的总体趋势[2]。临床早期CRC患者常无明显症状,易被忽视,发现时往往已进展至中晚期。结肠镜下取组织活检是CRC诊断的金标准。但该操作对患者造成损伤较大,患者配合度低,加之对医疗人员技术水平要求较高[3]。而目前已应用的CRC诊断的标志物在实践过程中有一定局限性。糖类蛋白肿瘤标志物CA19-9缺乏器官特异性,对早期患者的敏感度仅为30%[4-5]。癌胚抗原(carcino-embryonic antigen,CEA)在CRC早期时阳性率较低,不易被检测出[6-7]。因此,开发出特异性强灵敏度高、且创伤性小的诊断方式系当务之急。CRC的遗传分析显示CRC的发生源于基因突变和表观遗传变化的积累[8],特定基因甲基化水平的变化与CRC的发病阶段以及患者预后相关[9]。甲基化标志物可在患者的血液、粪便和手术标本中检测到[10],并且稳定性强、检测简便,在CRC诊断方面具有较大潜力。
由于近年来科研人员已经依据癌症基因组图谱数据库(The Cancer Genome Atlas,TCGA)在多种肿瘤中发现了潜在的临床标志物和治疗靶点[11-13],2020年12月-2021年9月本研究应用机器学习(Machine Learning)的方法,利用TCGA数据库中的结直肠癌27 K甲基化数据和临床信息,运用SPSS分析与CRC不良预后有关的因素。应用Weka3-9-4建立起基于数个DNA甲基化位点的诊断模型,并在GEO数据库(Gene Expression Omnibus)中获取独立数据集(GSE131013)来验证模型,以期对临床上CRC的早期诊断和预测提供帮助。现报道如下。
1 资料与方法
1.1 一般资料
在TCGA网站下载207例结直肠癌27 K甲基化测序数据和相关临床资料。27 K甲基化数据包括169例结直肠癌组织(其中有5例资料缺失)和38例癌旁正常组织的CpG岛基因位点的甲基化程度值。临床随访数据包括169例CRC患者的社会人口学特征、临床病理信息和随访时间。其中社会人口学特征包括年龄、性别、种族等信息,临床病理信息包括肿瘤切除或活检部位、肿瘤分期。本研究以年龄中位值72岁为阈值,将CRC患者分为高龄组和低龄组,各82例;以甲基化位点(cg24446548)Beta值的中位值0.783为阈值,将CRC患者分为高甲基化组(n=81)和低甲基化组(n=83)。stageⅠ,stageⅡ,stageⅡa,stageⅡb为CRC早中期,stageⅢ,stageⅢa,stageⅢb,stageⅢc,stageⅣ,stageⅣa为CRC晚期。见表1。
1.2 差异甲基化位点的筛选
使 用R3.1.0(http://www.cran.r-project.org/)中edger软件包筛选差异甲基化位点。差异甲基化位点筛选的设定条件为:差异倍数(fold change,FC)>4或<0.4,P<0.05。FC即为两组样品间位点甲基化程度的比值,是表示差异倍数的变量。对筛选出的位点进行受试者操作特征(receiver operating characteristic,ROC)分析,使用SPSS软件绘制受试者操作特征曲线(receiver operator characteristic curve,ROC曲线),计算曲线下面积(area under the curve,AUC),保留曲线下面积较大的12个位点,从而筛选出检验效能较大的位点;在筛选出的12个位点中选取检验效能最大的位点(cg24446548)和检验效能最小的位点(cg05345286)作ROC曲线图。使用SPSS逐步回归分析进一步筛选,筛选标准是P≤0.05进入模型,P≥0.1自动排除。
1.3 方法
结直肠癌诊断模型的构建方法。使用人工神经网络(artificial neural network,ANN)、逻辑回归(Logistic回归)、支持向量机(support vector machine,SVM)3种方法建立模型。将逐步回归筛选出来的位点导入Weka系统。为了提高计算效率,增强模型的稳定性和准确度,选用Discretize过滤器离散化的甲基化值,此时甲基化数据已被分为较均等的多个子空间[14],异常数据对模型的影响降低。接着使用ANN、Logistic回归及SVM3种方法分别建立模型,并进行10折交叉验证。10折交叉验证即将数据集随机分成10份,依次将其中9份作为训练数据,1份作为测试数据进行试验;取10次实验结果的平均值作为模型的精确度估计,来进一步优化模型。
1.4 使用独立数据集评估结直肠癌诊断模型的性能
为进一步验证模型的预测能力,于GEO数据库中下载GSE131013数据集。用数据集中的96例肿瘤组织样本和144例正常组织样本作为验证集对ANN模型、Logistic模型、SVM模型分别进行测试。利用已建立的混淆矩阵和相关指标,选出诊断性能较优的模型。模型的分类预测效果用平均准确率、漏诊率来评价。平均准确率较高且漏诊率较低的模型诊断性能较好。同时使用特异度、灵敏度、AUC、Kappa值等指标来辅助评价模型。较大的AUC代表了较好的预测性能;Kappa>0.75,说明两种诊断方法结果一致性较好。
1.5 统计方法
采用SPSS统计学软件对CRC患者临床随访数据进行统计分析。首先进行Kaplan-Meier(KM)生存分析,采用对数秩检验(Log-Rank法)比较不同组别患者生存率。然后采用多因素Cox回归模型分析性别、年龄、肿瘤分期和位点甲基化程度对CRC患者预后的影响。同时,用相关性分析探究甲基化位点与年龄、肿瘤分期和基因表达量之间的相关性。相关系数反映其相关性,>0表示正相关,<0表示负相关。相关系数的绝对值,0~0.1为没有相关性,0.1~0.3为弱相关,0.3~0.5为中等相关,0.5~1.0为强相关。P<0.05为差异有统计学意义。
1.6 分析结果的核实与补充
采用甲基化分析资源工具(shiny methylation analysis resource tool,SMART)分析位点的甲基化程度在结肠癌组织与癌旁组织之间有无明显差异以及甲基化程度与基因表达量之间有无相关性。
2 结果
2.1 差异甲基化位点的筛选
采用R语言“edger”软件包筛选出63个位点,其中logFC最大的18个位点和logFC最小的18个位点。见图1。ROC分析筛选出了12个检验效能较大 的 位 点(cg00240432、cg05345286、cg06151165、
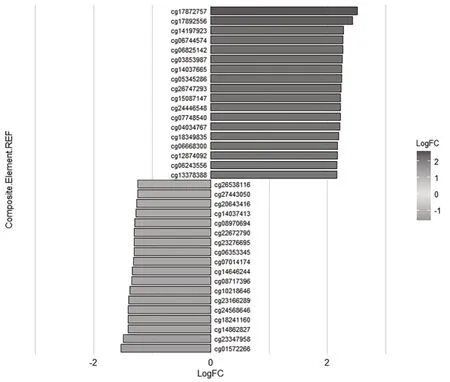
图1 差异分析中logFC最大的18个位点和logFC最小的18个位点
cg08090772、cg13577076、cg14197923、cg15087147、cg17872757、cg18349835、cg22879515、cg24446548
和cg06744574)。见图2。将筛选出的12个位点纳入逐步回归分析,筛选出6个位点(cg00240432、
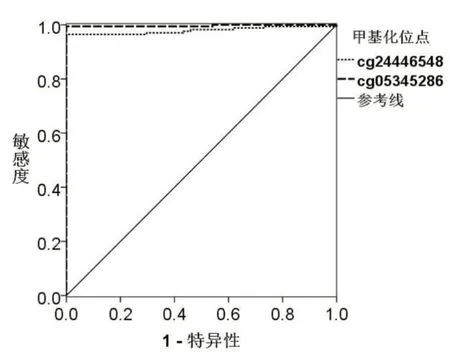
图2 甲基化位点cg24446548和cg05345286的ROC曲 线
cg06744574、cg08090772、cg13577076、cg17872757、cg24446548),这6个位点的甲基化程度在结肠癌组织与癌旁组织之间差异有统计学意义(P<0.05)。见图3。
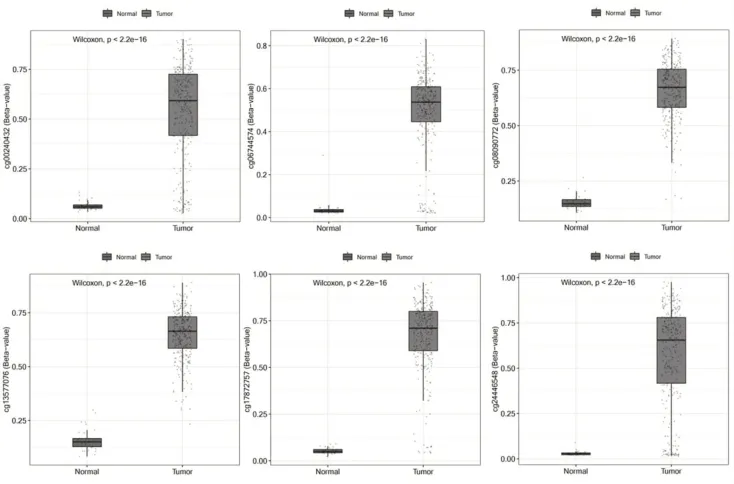
图3 结肠癌的癌旁组织与癌症组织的6个差异甲基化位点的Beta值分布
2.2 诊断模型的构建与评价
基于6个差异甲基化位点建立模型,结果见表1。SVM模型、ANN模型和Logistic回归模型的平均准确率分别为99.5%、99.0%和98.0%。ANN模型(图4)、Logistic回归模型和SVM模型的漏诊率分别为1.0%、2.0%和0.5%。ANN模型、Logistic回归模型和SVM模型AUC值分别为0.999、0.994和0.997。独立数据集验证结果见表2。ANN模型、Logistic回归模型、SVM模型的准确率分别为92.9%、85.8%和91.2%,漏诊率分别为7.1%、14.2%和8.8%。

表2 独立数据集评估3种模型性能
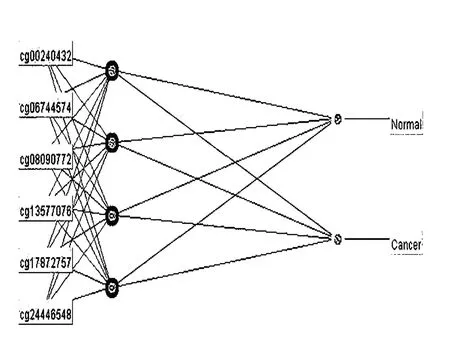
图4 基于6个甲基化位点的神经网络模型

表1 10折交叉验证评估3种模型的性能
2.3 影响CRC患者预后因素的生存分析
Log-Rank检验结果显示男女两性别之间的生存分析差异无统计学意义(P>0.05);以72岁(中位数)为界限数值分类时,高龄组(≥72岁)和低龄组(<72岁)生存分析差异无统计学意义(P>0.05)。高甲基化组与低甲基化组生存分析比较,CRC早中期与晚期的生存分析比较,差异有统计学意义(P<0.05)。将P<0.20的性别、年龄、肿瘤分期、和甲基化位点(cg24446548)纳入Cox多因素分析,性别、年龄和甲基化位点对生存时间的影响无统计学意义(P>0.05);肿瘤分期对生存时间的影响有统计学意义(HR=4.423,P<0.05)。见图5、表3。
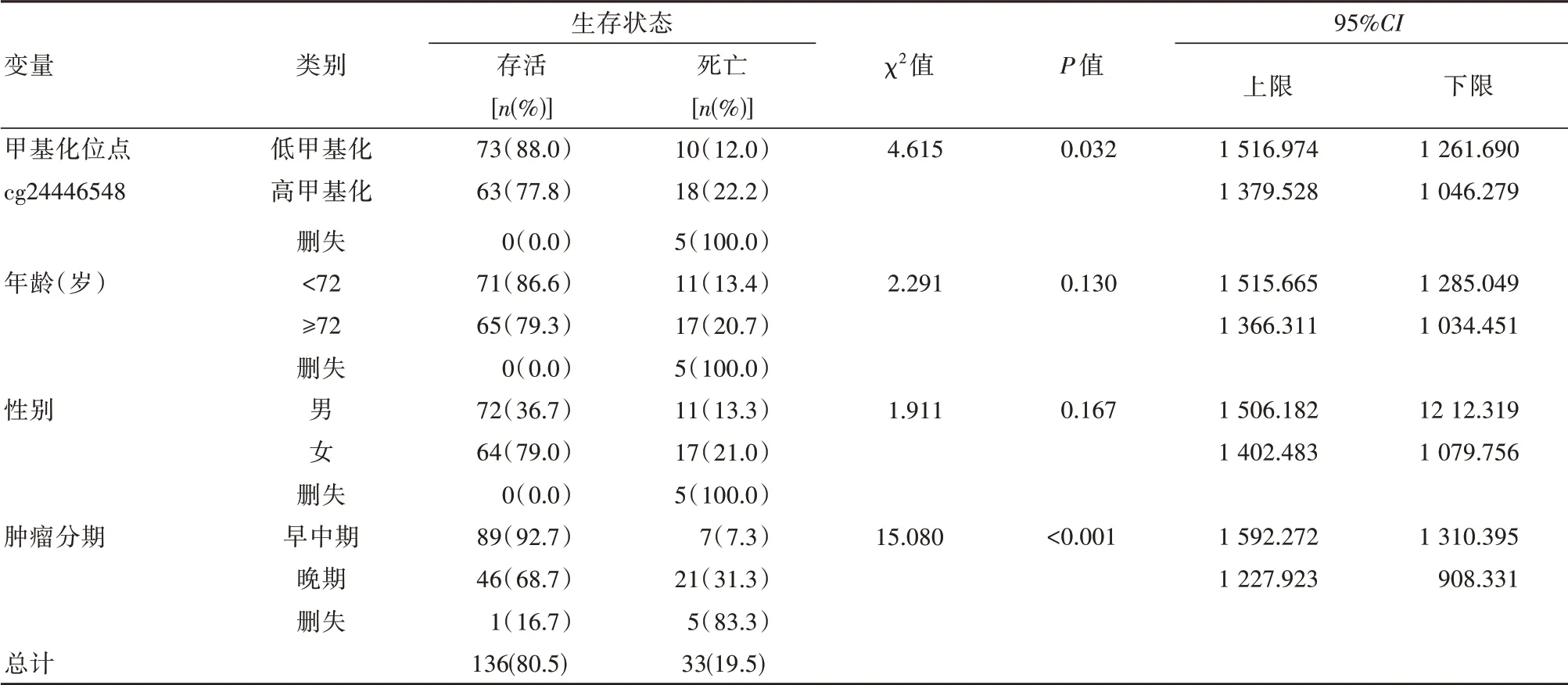
表3 KM生存分析中的参数估计及Log-Rank检验结果
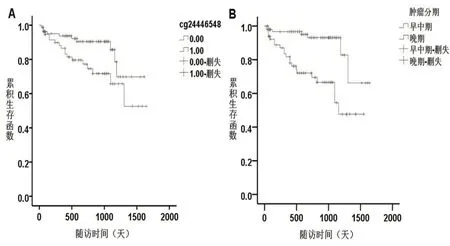
图5 169例CRC患者临床随访数据生存分析
2.4 相关性分析
相关性分析结果显示,cg17872757与年龄呈现正相关(r=0.227,P<0.05)。甲基化水平与肿瘤分期无相关性(P>0.05)。为了探究位点甲基化程度与所在基因表达量的相关性,采用SMART对筛选出的6个差异甲基化位点进行相关性分析。甲基化程度与基因表达量具有一定相关性,其中,cg08090772甲基化程度与ADHFE1基因表达呈现负相关(r=-0.700,P<0.05);cg17872757甲基化程度与FLI1基 因 表 达 呈负 相 关(r=-0.410,P<0.05);cg06744574甲基化程度与BEND5表达呈负相关(r=-0.350,P<0.05)。
3 讨论
DNA甲基化是表观遗传学的重要表现之一,是指在DNA甲基化转移酶作用下,基因组CpG二核苷酸胞嘧啶的5’碳位与甲基基团共价结合[15]。CpG二核苷酸序列通常成串出现在DNA上,称为CpG岛,常出现在真核生物编码基因的调控区。正常状态下,启动子中CpG岛处于未甲基化状态,CpG序列中出现C甲基化则可能会导致正常基因转录被抑制。Hu YH等[15]的研究显示,ADHFE1在CRC组织中下调和高甲基化,ADHFE1的下调与CRC患者的分化差和晚期TNM分期相关。相关性分析结果表明:cg08090772高甲基化与其所在的ADHFE1基因低表达高度相关(r=-0.700,P<0.05)。ADHFE1基因的高甲基化可能会抑制基因的表达。
DNA甲基化异常已被发现是肿瘤发生过程中出现的最早分子事件之一,且在正常细胞向肿瘤细胞转化的过程中就能被检测到[16]。因此,基因异常甲基化的检测在恶性肿瘤的早期筛查中具有潜在的应用价值。本研究利用TCGA数据库中结直肠癌27 K甲基化数据,采用生物信息学数据的处理方法,筛选出了6个具有诊断CRC潜力的位点,其中cg24446548的高甲基化与CRC患者不良生存显著相关(P<0.05)。然后通过机器学习的方法初步建立了基于6个甲基化位点的ANN、Logistic和SVM诊断模型。ANN模型、Logistic回归模型和SVM模型的漏诊率分别为1.0%、2.0%和0.5%,AUC值分别为0.999、0.994和0.997,模型分类能力较好。3种模型均能够根据现有数据建立起预测性能良好的模型。其中,ANN和SVM的分类和预测性能较佳。SVM模型的Kappa系数为0.984,MAE值为0.005,因而SVM模型诊断CRC的诊断一致性很高。而后通过GEO数据库的独立数据集进行验证,进一步比较3种模型的诊断效果,结果同样显示ANN模型与SVM模型的分类和预测性能较优。Log-Rank检验结果表明:高vs低甲基化组和CRC早中期vs晚期的生存分析显示差异有统计学意义(P<0.05)。因此,cg24446548位点的高甲基以及CRC晚期(Ⅲ、Ⅳ期)预示着患者的不良生存预后。Cox多因素分析结果表明,CRC晚期(Ⅲ、Ⅳ期)预示着CRC患者不良预后(HR=4.423,P<0.05),且晚期(Ⅲ、Ⅳ期)癌症患者的死亡风险为早期(Ⅰ、Ⅱ期)患者死亡风险的4.423倍。
本研究的创新之处在于建立了准确率较高的CRC诊断模型。Hou PZ等[17]分别采用12个指标、4个指标和7个指标建立了3个SVM模型,分类准确率分别为76.7%、83.3%和90.0%,对CRC的诊断具有较高价值。Zhang B等[18]基于5个血清标志物CEA、CA199、CA242、CA125、CA153建立SVM和BP神经网络模型,准确率分别为82.5%和75.0%。本研究基于6个甲基化位点建立的SVM模型分类准确率为91.2%,取得了较好的分类效果,同时具有较低的漏诊率。
局限性:本研究筛选的甲基化位点建立的模型可以有效地诊断结直肠癌。位点所在的基因甲基化程度有待大样本的实验验证或测序检验。由于目前测序成本较高,所以对该方法的推广有一定限制。
综上所述,流程筛选的6个甲基化位点,具有诊断结直肠癌的潜能;建立的ANN和SVM模型可以有效区分肿瘤组和癌旁正常组。cg24446548位点的高甲基化以及肿瘤晚期预示着不良生存预后。

