油莎豆SRAP-PCR体系优化及遗传多样性分析
赵琦琦郭玉静于梦斐王颖高文伟张斌
(1.新疆农业大学农学院,新疆 乌鲁木齐 830000;2.山东省农业科学院农作物种质资源研究所,山东 济南 250100;3.湖南大学研究生院隆平分院,湖南 长沙 410125;4.曲阜师范大学生命科学学院,山东 曲阜 273165)
油莎豆(Cyperus esculentus)是莎草科莎草属的禾本科作物,原产于非洲及地中海沿岸的干旱和半干旱地区[1,2],现广泛分布于我国的东北、华北及长江流域等中、低纬度地区,性喜温暖湿润气候,具有生长速度快、生物量大、抗逆性强等优点[3],耐旱、耐涝、耐贫瘠、耐盐碱[4]。油莎豆块茎含油量达27%左右[5],且含有相当丰富的膳食纤维、矿物质(如钾、磷、钙、镁、锌、铜)、维生素C和E以及人体必需脂肪酸(如肉豆蔻酸、油酸和亚油酸)等[6]。目前,大多数油莎豆研究主要集中在种植技术[7]、油脂提取工艺[8,9]、营养成分[10]及药用价值分析[11]等方面,对于其种质资源遗传多样性的研究相对较少。
SRAP又称相关序列扩增多态性(sequencerelated amplified polymorphism,SRAP),由美国加州大学作物系Li和Quiros于2001年提出[12],是一种无需任何序列信息即可直接PCR扩增的新型分子标记技术,具有简便、高共显性、易分离条带及测序等优点,已被广泛应用于生物遗传多样性分析、遗传图谱构建、比较基因组及品种鉴定等研究[13,14]。目前,SRAP分子标记技术在紫花苜蓿[15]、马铃薯[16]、紫锥菊[17]、草果菊[18]、石榴[19]等多种生物的遗传多样性及亲缘关系分析中取得重大进展,但在探究油莎豆种质资源遗传多样性和亲缘关系上的应用却很少。
本研究利用SRAP分子标记技术,采用单因素试验与正交试验相结合的方法,基于引物浓度、混合酶体积、模板DNA浓度三因素,探究了SRAPPCR的最佳扩增体系,并对收集到的16份油莎豆种质资源进行遗传多样性分析,以探明不同地区油莎豆种质资源的遗传距离和亲缘关系,以期为油莎豆的种质资源鉴定和遗传育种提供依据。
1 材料与方法
1.1 试验材料
供试16份油莎豆种质资源材料的来源及特性见表1。于2021年4月收集种子,5月底浸种3~5 d,6月初种植于山东省农业科学院核心区温室大棚内底径26 cm、高23 cm、口径34 cm的花盆中,盆土为沙质土。每盆播种3穴,每穴1粒种子,穴深4~5 cm。播种后及时浇水,保持土壤湿润,便于种子萌发。播种后10 d左右出芽,20 d左右采集油莎豆幼嫩叶片,保存于-80℃超低温冰箱,用于提取DNA。7~12月进行油莎豆SRAPPCR体系优化试验及相关遗传多样性分析。所用引物见表2。

表1 供试16份油莎豆材料的特性及来源
1.2 油莎豆基因组DNA提取与检测
采用改良的CTAB法提取油莎豆幼嫩叶片基因组DNA,通过微量核酸蛋白浓度测定仪测定DNA浓度。将5μL DNA样品与1μL 6×Loading buffer混合均匀后点入1%琼脂糖凝胶,置于100 V电压下电泳20 min,检测DNA质量。
1.3 SRAP-PCR扩增体系建立与优化
选用条带清晰且多态性高的Me2+Em2引物对,以2号油莎豆种质材料DNA为模板,采用单因素试验与正交试验相结合的方法确定SRAPPCR最佳扩增体系。
SRAP-PCR扩增初始体系(15μL):模板DNA 60 ng,2×PCR Master Mix 7.5μL,正反向引物各0.3μL,ddH2O补足至15μL。SRAP-PCR程序:94℃预变性5 min;94℃1 min,35℃1 min,72℃1.5 min,10个循环;94℃1 min,50℃1 min,72℃1.5 min,35个循环;72℃延伸10 min。用2%琼脂糖凝胶检测PCR产物。
1.3.1 单因素试验设计 影响SRAP-PCR扩增体系的主要因素有引物浓度、混合酶体积、模板DNA含量,所以,基于初始扩增体系,对这3个因素分别设计6个浓度梯度(表2)进行单因素试验,以确定各因素的基础浓度范围。
1.3.2 正交试验设计 在单因素所选浓度范围基础上设计L9(33)正交试验,共9个处理组合,如表3所示。利用陈璨等[20]的方法,对正交试验结果进行直观分析,即:依据扩增条带的清晰度和稳定性对9个组合的PCR扩增结果进行评分,条带数量最多、清晰度最高的产物记16分,最差的产物记1分;根据各处理组合评分结果,计算各因素各水平的得分均值ki,进而计算该因素的极差R=kimax-kimin。
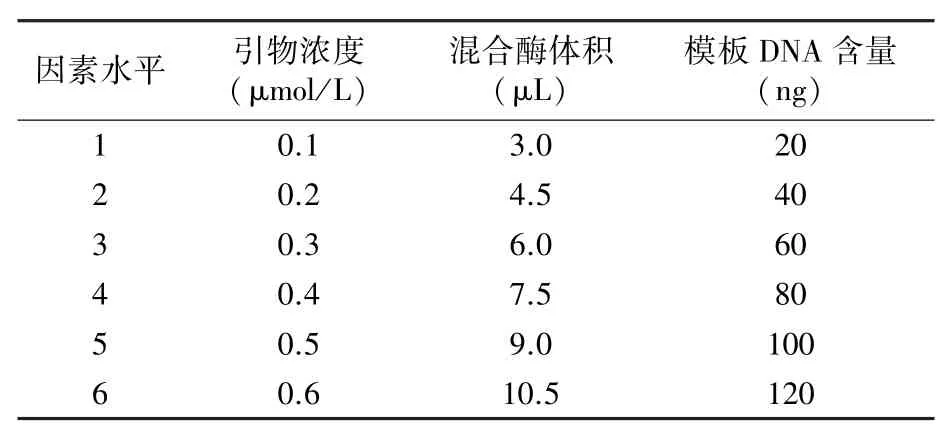
表3 油莎豆SRAP-PCR扩增体系的单因素试验设计

表4 油莎豆SRAP-PCR扩增体系的正交试验设计
1.4 供试16个油莎豆种质材料的SRAP-PCR遗传多样性分析
遗传多样性分析所用SRAP引物由上海生工生物工程有限公司合成,设计正向引物6条、反向引物17条(表2),组合成102对引物。利用上述试验确定的最佳扩增体系对16份油莎豆材料进行扩增,采用6%非变性聚丙烯酰胺凝胶对PCR产物进行电泳,恒压200 V,电泳1.0~1.5 h,用银染法显影,拍照记录。
利用人工读带的方法统计PCR产物中相同位置清晰的条带数,有条带的记为“1”,无条带的记为“0”,转换为“0/1”数字矩阵。利用NTSYS 2.1计算遗传一致度和遗传距离,采用UPGMA法绘制聚类图,进而分析16份油莎豆材料的亲缘关系。
2 结果与分析
2.1 DNA提取质量检测结果
经过检测,提取的DNA浓度范围在141.5~269.7 ng/μL,OD260/OD280值 在1.8以 上,OD260/OD230值在1.96~2.18之间;DNA条带清晰完整,无降解和拖尾现象(图1)。综上,利用改良CTAB法提取的油莎豆DNA质量较好,浓度较高,可用于SRAP-PCR反应。

图1 16份油莎豆材料的DNA电泳检测图谱
2.2 SRAP-PCR扩增体系的优化
2.2.1 单因素试验结果分析 由图2可知,随着引物浓度提升,条带逐渐清晰且多态性增加,最终根据条带的扩增情况选择0.4、0.5、0.6μmol/L三个引物浓度用于正交试验。混合酶体积在3.0μL时无扩增条带,在4.5、6.0μL时扩增条带较模糊,在7.5、9.0、10.5μL时条带扩增情况最好,所以选择7.5、9.0、10.5μL用于正交试验。DNA模板用量为20 ng时扩增条带较模糊,其他浓度时扩增情况均较好,选择60、80、100 ng用于正交试验。
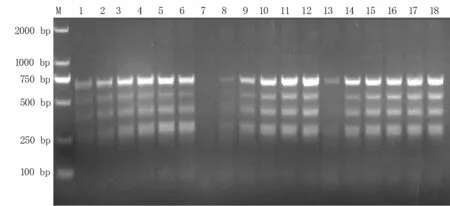
图2 SRAP-PCR扩增体系单因素试验结果
2.2.2 油莎豆SRAP-PCR最佳扩增体系的确立
从图3可看出,处理组合1、7没有扩增出条带,处理组合4、8扩增条带多态性较少,其余组合扩增条带较好但清晰度存在差异。正交试验直观分析结果(表5)显示,3个因素对SRAP-PCR反应体系的影响为混合酶体积>模板DNA含量>引物浓度,引物浓度为0.5μmol/L、混合酶体积为10.5 μmol/L、模板DNA含量为80 ng时扩增效果最佳。以此组合作为最佳反应体系,选用不同引物组合,用4号、5号油莎豆材料进行验证试验,结果(图4)表明,不同引物组合在两种质中均能稳定扩增,说明所选体系稳定可靠,适用于油莎豆SRAP分子标记研究。
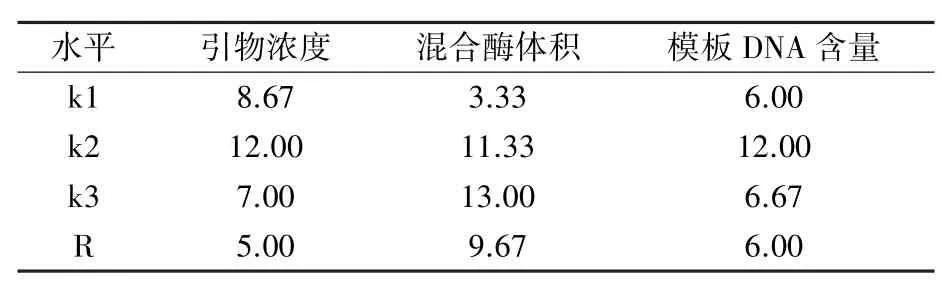
表5 正交试验直观分析结果

图3 SRAP-PCR扩增体系的正交试验结果
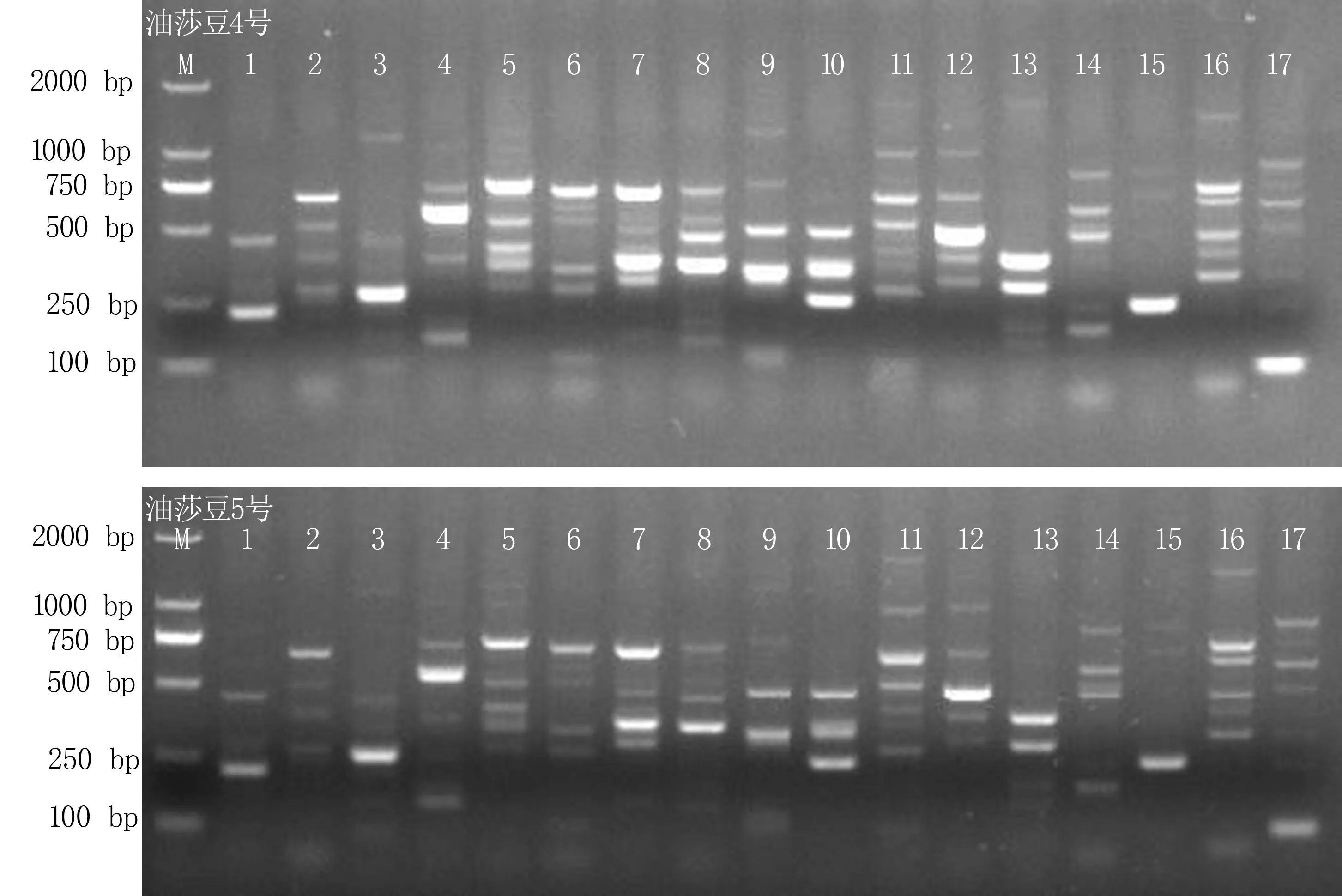
图4 SRAP-PCR最佳扩增体系在不同品种不同引物稳定扩增结果
2.3 16份油莎豆种质材料的遗传多样性分析
2.3.1 SRAP-PCR多态性分析 利用6%非变性聚丙烯酰胺凝胶电泳从102对引物组合中筛选出30对条带清晰且多态性较好的引物组合(表6),用这30对引物组合共扩增出312个位点,其中多态性位点289个,平均多态性比率达92.82%;每对引物扩增出的总位点数在6~17个之间,平均10.40个位点;多态性位点有5~16个,平均9.63个位点。表明筛选出的30对SRAP分子标记在16份油莎豆材料中所扩增出的条带都具有较好的多态性,可用于遗传多样性分析。图5是引物Me1+Em7和Me2+Em1对16份油莎豆材料的SRAP扩增结果。
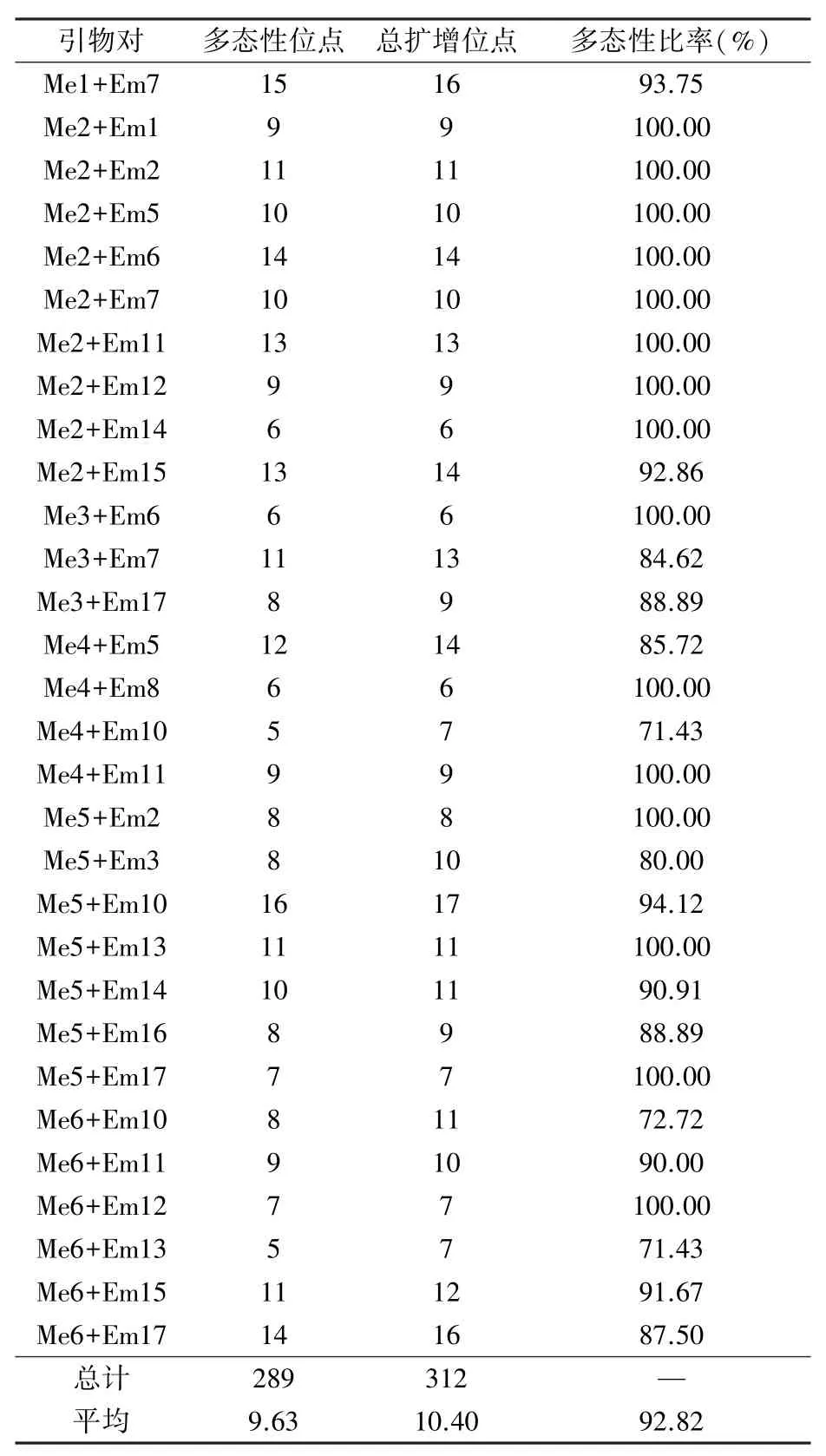
表6 多态性引物组合扩增结果

图5 Mel+Em7和Me2+Eml对16份油莎豆种质材料的扩增结果
2.3.2 16份油莎豆种质材料的SRAP-PCR遗传距离与遗传一致度分析 由表7可知,遗传一致度范围为0.60~0.93,遗传距离范围为0.07~0.40。8号与11号种质的遗传一致度最低,亲缘关系最远,遗传距离最大;来自新疆维吾尔自治区的14号与来自河北省的15号材料遗传一致度最高,亲缘关系最近,遗传距离最小。由于14、15号材料的品种特性都为黄色圆粒,结合遗传一致度结果,推测它们可能为同一品种。综合分析油莎豆材料的遗传距离和遗传一致度,16份油莎豆种质材料间遗传一致度较高,遗传背景较狭窄,遗传差异较小,这可能是因为我国的油莎豆种质是由国外引种而来,引种后地域间种质交流较频繁,从而造成了油莎豆种质资源具有较高的遗传一致度。

表7 16份油莎豆材料遗传距离与遗传一致度
2.3.3 16份油莎豆种质材料的SRAP-PCR聚类分析 利用UPGMA法对16份油莎豆种质材料进行聚类分析,得到树状聚类图见图6,可见聚类结果与遗传一致度和遗传距离的结果一致。在遗传相似系数0.824处,可将16份油莎豆种质材料划分为4个类群,第一、第二、第四类群均包含1份油莎豆种质材料,第三类群包含13份种质材料。在遗传相似系数0.872处,可进一步将第三类群划分为5个亚类,其中第1亚类包含河南省的3份油莎豆材料,大部分为圆粒;第2亚类包含吉林省的3份油莎豆材料,均为长粒;第5亚类包含其他4个地区的5份油莎豆材料,绝大部分为圆粒;来自吉林省的长粒油莎豆7号和来自云南省的长粒油莎豆11号材料单独归于一个亚类。表明,油莎豆种质资源的地域来源和品种特性均对聚类结果产生影响。

图6 16份油莎豆种质材料的UPGMA聚类结果
3 讨论
3.1 油莎豆SRAP-PCR体系的优化
利用PCR扩增体系获得条带清晰、多态性高的引物组合是研究SRAP分子标记的基础。本研究利用单因素试验和正交试验确定了适合油莎豆的SRAP-PCR最佳扩增体系,即引物为0.5μmol/L,混合酶体积为10.5μL,模板DNA含量为80ng,用ddH2O补足15μL。这与赵永国等[21]确定的油莎豆SRAP优化体系(15μL扩增体系包括DNA模板25 ng、Mg2+1.5 mmol/L、引物浓度1 μmol/L、dNTPs 0.3 mmol/L和Taq酶1 U)不一致,可能与DNA提取方法不同有关,另外,不同厂家的试剂、不同的实验操作方法也会对SRAPPCR的扩增结果产生不同的影响。
本研究确定了3个因素对油莎豆SRAP-PCR扩增体系的影响大小为混合酶体积>模板DNA含量>引物浓度。但不同植物中SRAP-PCR扩增体系各因素产生的影响不同:戚华沙等[22]在海南油茶SRAP-PCR扩增体系优化中发现,各因素影响大小为引物浓度>dNTPs浓度>TaqDNA聚合酶用量>模板DNA含量;严莉等[23]对山桐子SRAP-PCR扩增体系的优化结果表明各因素的影响大小为TaqDNA聚合酶用量>Mg2+浓度>引物浓度>模板DNA浓度>dNTPs浓度。
3.2 SRAP分子标记探究油莎豆遗传多样性
SRAP分子标记使用通用引物进行扩增,常被用于分析遗传多样性和亲缘关系,如沈秀芬等[24]利用SRAP标记分析草菇遗传多样性,发现草菇种质资源在一定程度上出现了地域基因分化;Deng等[25]利用SRAP标记首次对中国特有的10种莪术药材进行了亲缘关系分析。这些研究表明,SRAP分子标记技术可有效区分种质资源间的遗传差异,明确其亲缘关系。本研究对收集到的16份种质资源进行遗传多样性分析,筛选到30对多态性较好的引物组合,并用其扩增出312个位点,平均多态性比率为92.82%,说明16份油莎豆种质材料具有丰富的多态性。聚类分析结果表明,16份油莎豆种质材料的遗传差异与品种特性及来源均有关。
4 结论
本研究基于引物浓度、混合酶体积和DNA模板含量3个因素,通过单因素试验和正交试验,确立了油莎豆SRAP-PCR的最佳扩增体系为:引物0.5μmol/L,混合酶体积10.5μL,模板DNA含量80 ng,用ddH2O补足15μL。利用该体系从102对引物组合中筛选出30对多态性引物,并用于对16份油莎豆种质材料的遗传多样性进行分析,发现油莎豆种质材料间的遗传多样性受品种特性和地域来源的双重影响。本研究结果可为油莎豆种质资源的品种鉴定和选育等提供一定的参考依据。

