基于近红外高光谱成像技术识别紫斑和霉变大豆的方法
李凯楠,沈广辉,叶文武*,郑小波,王源超
(1.南京农业大学植物保护学院,江苏 南京 210095;2.江苏省农业科学院农产品质量安全与营养研究所,江苏 南京 210014)
真菌病害不仅在大豆生产中造成产量损失,也对种子质量和食品安全造成许多负面影响。大豆种子受病原真菌侵染后,经常出现色斑或霉变,不仅影响外观,而且影响其营养品质,并携带对人畜有害的真菌毒素。例如,引起大豆紫斑病的菊池尾孢菌(Cercosporakikuchii)可产生尾孢毒素,致使种皮甚至整颗豆粒呈紫色,严重影响大豆外观品质,并导致发芽率下降[1];大豆拟茎点种腐病菌(Phomopsislongicolla)[2]能够侵染种皮、子叶及胚,消耗种子的蛋白质和脂肪等营养物质,种子常出现皱缩、延长或开裂,受潮后易霉变和腐烂[3-5];黄曲霉(Aspergillusflavus)和镰孢菌(Fusariumspp.)分别能够产生赭曲霉毒素A(ochratoxin A,OTA)和呕吐毒素(deoxynivalenol,DON)及伏马菌素(Fumonisin FB)等真菌毒素,对人畜具有重大安全隐患[6-8]。带菌种子不仅萌发率低,而且在播种后容易造成新一轮的病害和产量损失,调运播种未筛查的带菌种子是病害传播蔓延的重要途径之一。因此,快速识别和筛查带菌特别是病变的种子对大豆生产与品质安全均具有重要意义。
传统的农作物质量检测手段包括感官评价与理化分析等,存在处理时间长、对鉴定人员的经验要求较高、鉴定的准确性易受到主观因素影响等问题。近年来,基于近红外光谱技术(near infrared reflectance spectroscopy,NIRS)的植物化学组分检测分析方法得到越来越普遍的应用[9]。例如,Lim等[10]使用NIRS实现了对小麦、脱壳大麦和裸大麦中镰刀菌污染麦粒的快速无损鉴别;齐星炜[11]使用傅里叶变换近红外光谱仪,探索基于NIRS结合主成分分析对不同活力水平大豆种子进行分类的可行性;耿立格等[12]和Al-Amery等[13]分别利用NIRS结合偏最小二乘法模型(PLS)的方法,对大豆种子的活力进行了预测与鉴别。此外,近红外高光谱成像技术(near infrared hyperspectral imaging,NIR-HSI)把近红外光谱与成像技术相结合,不但可以同时获取样品内部和外部的信息,还可以分析不同组分在样品中的空间分布,被广泛应用到各个领域[14-20]。在大豆研究中,王海龙等[21]研究了使用NIR-HSI结合化学计量学对转基因大豆的鉴别方法,其构建的偏最小二乘判别分析模型(PLS-DA)对验证集的识别准确率在80%以上;Zhu等[22]基于NIR-HSI对3个大豆品种进行分类识别,建立了卷积神经网络(convolutional neural networks,CNN)模型,识别准确率均达90%;李亚婷[23]将NIR-HSI与多种数据降维方法和建模算法相结合,对大豆种子活力进行了准确鉴别和高精度的定量检测;柴玉华等[24]利用高光谱图像技术,采取1 000~2 500 nm的波长建立了对大豆5个分类等级的识别模型,预测准确率达92%。相较于传统的检测识别技术,NIR-HSI在检测分析大豆种子的化学组分方面具有快速、灵敏和准确等技术优势,适用于种子活力分析和品种鉴别。然而,目前尚未见对大豆病变种子进行鉴别的相关报道。
本研究针对大豆种子常见的紫斑和霉变2种病变类型,采用主成分分析(principal component analysis,PCA)结合最大类间方差法(Otsu)对采集的种子高光谱图像进行背景分割,通过提取指纹图谱,结合化学计量学算法构建判别分析模型,探究基于NIR-HSI对大豆正常粒、紫斑粒和霉变粒进行鉴别的可行性,旨在为后续开发大豆病变种子的自动识别与分选设备提供技术和理论支撑。
1 材料与方法
1.1 供试大豆种子采集
供试大豆种子(品种‘南农50’)保存于南京农业大学真菌与卵菌分子实验室。共采集了983粒种子,其中正常粒365粒、霉变粒381粒和紫斑粒237粒,均经过经验丰富的实验员视觉区分确认。通过随机采样的方法将样品分为校正集(用于模型构建)和验证集(用于模型评估),2个分集中正常粒、霉变粒和紫斑粒的粒数分别为279和86、297和84、163和74。
1.2 高光谱图像采集
所用设备为近红外高光谱成像系统(Gaiafield-N17E,四川双利合谱有限公司),由光源、光谱相机、电控移动平台、计算机及控制软件等组成。光源为2个卤素灯穹顶光源,光谱采集范围为900~1 700 nm,光谱和相机分辨率分别为5 nm和320×256像素,镜头高度为30 cm,样品台移动速度设为0.6 cm·s-1,曝光时间是20.6 ms。图像采集时将大豆种子随机单层平铺到样品台上,获取其图像。为消除暗电流和光源强度不均匀的影响,需要对原始图像进行黑白校正。校正公式:I=(I0-B)/(W-B)。式中:I为经校正后的图像;I0为原始的高光谱图像;B为黑色标定背景的信息;W为白板(聚四氟乙烯材料)标定图像信息[25]。
1.3 高光谱图像分割及单粒种子的光谱提取
使用ENVI 5.3软件裁剪黑白校正后的图像,在去除969~1 620 nm外的非必要波段后,使用基于PCA得分的Otsu算法进行图像分割和背景去除,提取只含有种子信息的图像[25]。使用MATLAB 2017a软件中的Bwlabel函数对图像中的种子进行编号,实现单粒种子指纹图谱的自动提取,并计算每粒种子的平均近红外光谱,用于后续判别分析。
1.4 光谱数据预处理
为了消除系统噪声并增强化学成分对光谱信号的贡献,分别比较了Autoscale、Savitzky-Golay多项式卷积平滑、一阶和二阶导数处理、多元散射校正(multiplicative scatter correction,MSC)、基线法(Baseline)和去趋势的标准正态方差(standard normal variance with de-trending,SNV-D)等算法,从中选择最优的算法进行光谱预处理。
1.5 基于多元数据的判别分析
为实现对正常粒、紫斑粒和霉变粒的快速区分,首先通过二阶导数谱解析了3类种子之间的光谱差异,并采用PCA探究其聚类趋势;然后使用判别分析结合特征波长筛选方法,探究基于NIR-HSI对每类种子进行区分的可行性;最后探讨同步快速识别3类种子的可行性。
利用PCA[26]从多变量数据集中提取主要信息,对3类种子的数据进行聚类及初步检验。使用PLS-DA和SVM-DA 2种判别分析方法,前者是一种双线性建模方法,用于分析一组独立的光谱变量和单个因变量之间的关系[27],后者是一种化学计量学技术,对每个分类模型创建一个超平面,当低维输入空间中的线性边界不能实现适当分类时,可在高维度特征空间中进行线性分离[28]。核函数的选择对SVM-DA算法的性能至关重要,本研究选择径向基核函数(radial basis function,RBF)[29]。在全谱判别分析的基础上选择合适的特征选取方法,包括连续投影法(SPA)[30]和竞争性自适应权重取样法(CARS)[31]。
将279粒正常粒和460粒病变粒(含紫斑粒与霉变粒)2组样品设为校正集,分别基于全谱段和CARS选取的特征波长,采用留一交互验证(leave-one-out cross validation)的方式构建PLS-DA和SVM-DA 2种模型,并对外部验证集(86粒正常粒和158粒病变粒)进行预测,判断模型的识别正确率。再将279粒正常粒、297粒霉变粒和163粒紫斑粒3组样品设为校正集,基于全谱段和SPA特征波长分别构建PLS-DA和SVM-DA 2种模型,并对外部验证集(86粒正常粒、84粒霉变粒和74粒紫斑粒)进行预测,判断模型的识别正确率。上述模型的构建均在MATLAB 2017a软件中实现。
2 结果与分析
2.1 3类种子的图像获取与分割
正常粒、霉变粒和紫斑粒3类种子样品的原始图像如图1-A所示。为了自动扣除背景和提取种子光谱,以经过裁剪后的高光谱图像为对象进行PCA,提取第1主成分的得分并结合Otsu算法,对图像进行双阈值(two-threshold)分割。处理后的二值化图像如图1-B所示。

图1 3类种子样品的原始图(A)和阈值分割后的二值化图(B)Fig.1 Original image(A)and two-threshold segmentation binarization image(B)of the three types of seed samples
2.2 3类种子的光谱特性比较
使用MATLAB 2017a软件的Bwlabel函数对983粒种子的光谱信息分别进行了提取(图2-A)。比较3类种子的平均光谱,发现霉变粒的光谱反射率普遍高于正常粒,紫斑粒与正常粒的光谱重叠程度高,不易区分(图2-B)。对样品平均光谱进行二阶导数处理,发现3类种子在1 004、1 085、1 170、1 246、1 362和1 416 nm波长附近存在较大差异(图2-C)。经比对,发现这些波长的吸收峰主要与种子的蛋白质和脂肪含量相关[32],这可能与病原菌侵染过程中消耗了种子中的一些营养物质有关。

图2 3类种子的光谱特性比较Fig.2 Comparison of the spectra data among the three types of seed samplesA. 全部光谱All spectra;B. 平均光谱Average spectra;C. 二阶导数处理后的结果 Result after second derivative processing.
2.3 3类种子光谱的PCA
对3类种子的光谱进行PCA,前3个主成分(PC1、PC2和PC3)的三维聚类结果如图3所示,霉变粒和其他2类种子间具有明显的区分度,而紫斑粒与正常粒的重合度较高,区分不明显,这可能是由于霉变粒(与紫斑粒相比)受真菌侵染程度较重,内部品质变化较为明显。

图3 3类种子光谱的主成分分析结果Fig.3 PCA result of the three types of seed samples
2.4 正常和病变种子的判别分析
2.4.1 基于全谱段的判别分析基于全谱构建了PLS-DA和SVM-DA 2种模型。PLS-DA模型中,根据交互验证均方根误差确定最佳潜变量数为15,结果如表1所示。校正集中正常粒和病变粒的识别正确率均大于94.00%,总识别正确率为95.81%;验证集的总识别正确率为96.31%,仅有7粒正常粒和2粒病变粒被误判。SVM-DA 模型中,惩罚系数(c)为100,核函数参数(g)为3.16×10-2,校正集中正常粒的识别正确率为 94.72%,而验证集中对正常粒存在较多误判,导致总识别正确率仅为85.25%。与PLS-DA模型的判别结果相比,SVM-DA模型对2类种子的识别正确率相对偏低。
2.4.2 基于CARS特征波长的判别分析使用CARS算法对全波段光谱数据提取特征波长,以减少冗余信息。经多次试验后,设置蒙特卡罗采样次数为50、交互验证的组数为5,按照交叉验证误差(cross-validated error)最小的原则筛选最优的特征变量,选择了15个最优特征波长(985、1 004、1 010、1 070、1 073、1 085、1 114、1 167、1 221、1 340、1 350、1 378、1 381、1 453和1 479 nm)作为变量集(图4)。
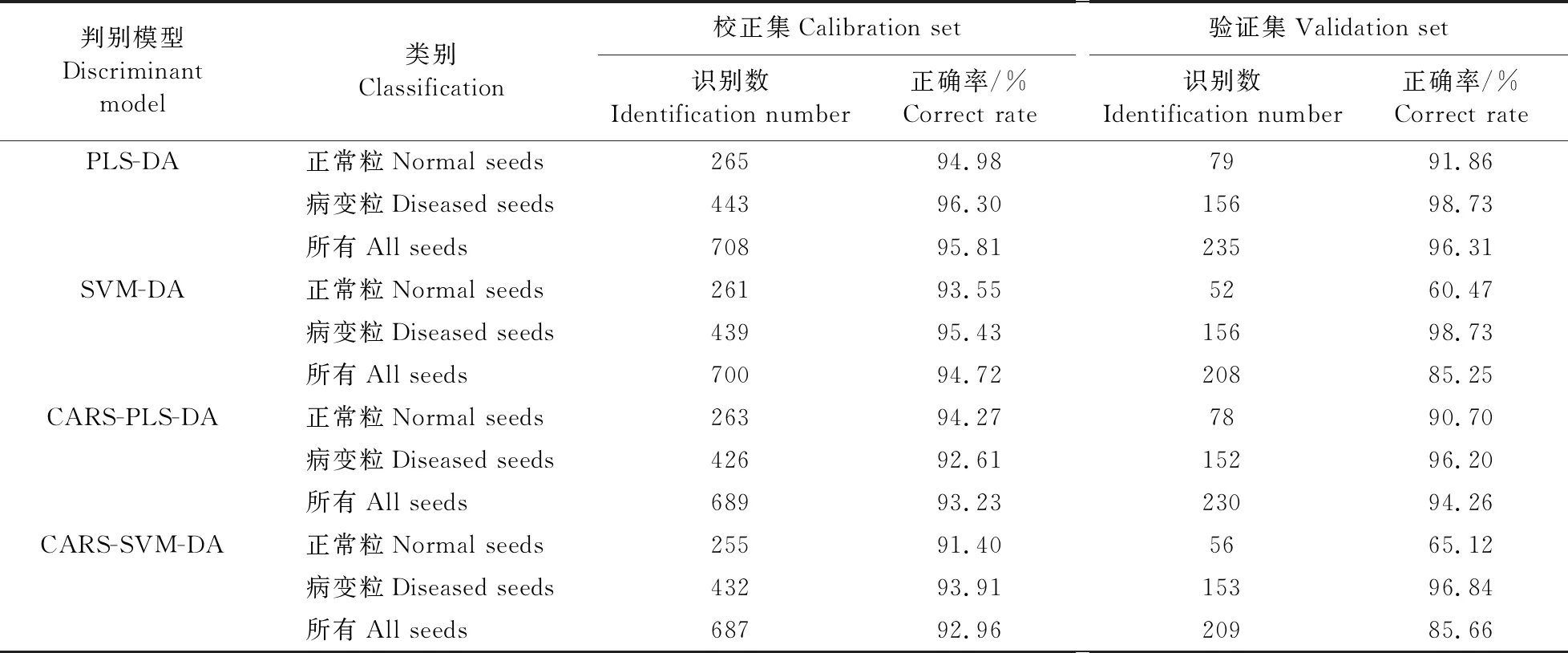
表1 基于全谱和CARS特征波长对正常和病变种子的判别分析结果Table 1 Discriminant analysis results for the normal and diseased seeds based on full spectrum and CARS characteristic wavelength

图4 使用CARS算法对全波段光谱数据的分析结果Fig.4 Analysis results of full band spectral data using CARS algorithm A. 每次抽样的特征变量数Number of characteristic variables per sampling;B. 每次抽样的交互验证误差Cross-validated error per sampling;C. 筛选出的特征波长Filtered characteristic wavelength.
基于CARS所筛选的特征波长构建了CARS-PLS-DA和CARS-SVM-DA两种模型(表1)。CARS-PLS-DA模型与基于全谱的PLS-DA模型结果相似,校正集和验证集的总识别正确率均在93.00%以上;CARS-SVM-DA模型与基于全谱的SVM-DA模型结果相似,对正常粒的识别正确率略有提升,在验证集中达到65.12%,但仍远低于使用PLS方法构建的模型。上述结果表明,基于全谱和CARS特征波长分别构建的PLS-DA和CARS-PLS-DA模型可有效识别病变粒,其中基于全谱的PLS-DA模型具有更好的识别效果,但是基于特征波长的CARS-PLS-DA模型,去除了无用信息变量,为便携式多光谱设备的开发提供了基础。
2.5 3类种子的判别分析
2.5.1 基于全谱段的判别分析基于全谱构建了PLS-DA和SVM-DA两种模型。PLS-DA模型中,潜变量数设为12,对校正集中3类种子的识别正确率均大于91.00%,其中霉变粒最高(96.97%),总识别正确率为93.50%;外部验证集的总识别正确率为90.16%。SVM-DA模型中,对校正集中正常粒、霉变粒和紫斑粒的识别正确率分别为93.19%、95.96%和87.12%,与PLS-DA模型的判别结果相比略有下降;外部验证集的总识别正确率也均低于PLS-DA模型的相应结果(表2)。

表2 基于全谱和SPA特征波长3类大豆的判别分析结果Table 2 Discriminant analysis results for the three types of seeds based on full spectrum and SPA characteristic wavelength
2.5.2 基于SPA特征波段的判别分析使用SPA算法挑选特征波长,最大选定波长数设为30,共筛选到17个特征波长(985、988、994、1 010、1 013、1 029、1 032、1 067、1 095、1 164、1 202、1 315、1 353、1 378、1 419、1 479和1 538 nm)作为变量集(图5)。

图5 使用SPA算法对全波段光谱数据的分析结果Fig.5 Analysis results of full band spectral data using SPA algorithm
基于SPA筛选的特征波段构建了SPA-PLS-DA和SPA-SVM-DA两种模型。SPA-PLS-DA模型下,校正集的总识别正确率为93.91%,与PLS-DA结果相比略有提高;验证集的总识别正确率也与PLS-DA结果相近。SPA-SVM-DA模型的结果与SVM-DA模型相似,对正常粒的识别正确率均低于PLS-DA模型,在验证集中仅为61.63%。上述结果表明,基于SPA特征波长构建的SPA-PLS-DA模型可有效实现对3类种子的识别,而SPA-SVM-DA对正常粒的误判现象较多(表2)。
综上所述,4种模型均可实现对3类种子的识别,其中基于全谱段的PLS-DA模型和基于SPA特征波长的SPA-PLS-DA模型对霉变粒和紫斑粒的识别正确率更高,SPA-PLS-DA可在保证模型识别正确率基本不变的情况下大幅降低变量数,更有利于便携式多光谱设备的开发。
3 讨论
本研究探索了基于近红外高光谱成像技术对大豆紫斑粒和霉变粒进行快速识别的方法。通过分析比较正常粒、霉变粒和紫斑粒3类种子的原始光谱可知,正常粒和紫斑粒的原始光谱相似,霉变粒光谱反射率明显高于前两者,其主要原因可能是霉变种子(受真菌侵染后)出现皱缩和颜色发白;进一步对3类种子的原始光谱进行二阶导数处理,发现6个存在较大差异的波长。大豆种子霉变后蛋白质和脂肪等营养成分的含量显著下降,而大豆紫斑病只影响种皮结构,对内部物质的影响较小。近红外光谱主要是反映含氢基团(C—H、N—H、O—H)的吸收,这些特征波长可能与种子中营养成分的差异有关[33]。
本研究采用CARS和SPA两种算法进行光谱的变量筛选,前者适用于正常粒和病变粒2类种子的判别,后者被用于正常粒、霉变粒和紫斑粒3类种子的判别。CARS法筛选的特征波长与二阶导数处理后发现的差异波长以及SPA法筛选的特征波长基本一致,其主要原因是近红外区域的吸收多为宽峰且重叠严重,无法直接分辨是哪一种物质的吸收峰,需要借助化学计量学对光谱信息进行解析。
为了准确区分正常粒与病变粒,本研究分别基于全谱和CARS法筛选的特征波长构建了4种模型,发现SVM-DA和CARS-SVM-DA模型对正常粒的误判率较高,而PLS-DA和CARS-PLS-DA模型对校正集和验证集的识别正确率都在93%以上,其中PLS-DA模型达到95%,说明所建立的PLS-DA和CARS-PLS-DA模型能较好地识别病变种子。
为了进一步探究区分正常粒、紫斑粒和霉变粒3类种子的可行性,本研究基于全谱和SPA法筛选的特征波长构建了4种模型,发现所有模型对校正集和验证集的识别正确率都在80%以上,其中正常粒与霉变粒均可以有效区分,而紫斑粒存在误判为正常粒的问题,尤其是在基于特征波长的判别模型中。这可能是因为紫斑粒上斑点的大小与位置不均一,当采集的光谱信息来自无斑点一侧时容易将之误判为正常粒。此外,实际材料中的紫斑粒和正常粒也具有霉变的情况,导致这些种子被判定为霉变粒。因此,需要进一步对不同病变程度的大豆进行精准分析。赵丹婷[34]基于图像处理技术构建的BP神经网络模型对正常、病斑、霉变、虫蚀和破碎的大豆进行识别,平均正确率为90.33%;侯升飞[35]基于高光谱成像技术构建的人工神经网络对不同质量等级的大豆进行识别,准确率为92%。这说明高光谱成像技术相较于图像处理技术可能在大豆判别分析中更优,而本研究所构建的模型,在准确识别大豆的前提下,对高光谱数据进行了降维,筛选到与大豆病变相关的特征波长,因此更利于光谱设备的开发。
本研究只使用单一的大豆品种进行研究,尚不清楚在不同品种之间判别情况的差异。下一步研究应使用不同遗传背景和地域来源的大豆材料探索不同病变程度大豆之间的广谱特征差异,以验证所建模型的稳定性和方法的可行性,最终构建一套行之有效的、高通量快速无损识别病变大豆的理论和技术体系。

