基于深度级联森林的乳腺癌基因数据分类研究
秦喜文 王 芮 张斯琪
1(长春工业大学大数据科学研究院,长春 130012)
2(长春工业大学研究生院 长春 130012)
3(长春工业大学数学与统计学院 长春 130012)
引言
在数据挖掘方法出现之前,疾病都是通过生物学以及临床诊断进行区分的,由于不同临床反映的疾病可能属于同类型,判定误差较大。 随着生物和医学技术的不断发展和深入,可以从正常组织以及病变组织中获得基因表达数据,利用数据挖掘技术分析基因数据,可以有效加深对病理的了解。
在对基因表达数据挖掘过程中,可以通过学习器对已知类别样本进行训练,使得该学习器对未知类别样本进行分类预测,以提高疾病诊断的有效性和准确性。 然而基因表达数据具有高维小样本的特性,往往存在冗余和噪声信息,使得学习器在建模过程中出现“维数灾难”或分类准确度不高等问题。 因此在建模之前需要对高维基因表达数据进行降维处理。
刘旭东[1]提出使用信息增益进行特征选择,排除无关特征,再使用互信息去除冗余特征,结果表明,所提出的方法优于信息增益特征选择的方法。谢东迅[2]将邻域互信息理论应用到特征基因选择中,并将基于优化特征的邻域互信息作为相关度的度量标准,所提出的方法在特征基因的识别准确率和数量等方面效果显著。 陈俊颖[3]利用互信息最大化找出同类中相关性最强的基因,结合自适应遗传算法增强变异能力,该方法能够显著降低基因表达数据的维度,减少分类冗余。 郭园园[4]将原最大相关最小冗余(max-relevance and min-redundancy,mRMR)算法中度量相关性的互信息使用最大信息系数(maximal information coefficient, MIC)来代替,并使用近似计算最大信息系数 (chi-square maximum information coefficient, ChiMIC)算法来近似估计MIC 值,公用数据集验证结果表明,所提出的mRMR-ChiMIC 算法较原mRMR 算法提取的特征具有更高的分类准确率,有效降低了计算复杂度。
在基因表达数据分类研究方面,Kong[5]提出了一种新的分类器森林深度神经网络(forest-deep neural network, fDNN),将深度神经网络结构与有监督的森林特征检测器相结合,以缓解过拟合问题。梁壮[6]提出一种基于堆栈自动编码器与Boosting 相结合的方法,使用主成分分析对基因表达数据降维,将堆栈自动编码器作为Boosting 的基学习器进行学习训练,最后组合多个堆栈自动编码器进行决策,显著提高分类准确率。 高振斌[7]使用PCA 对基因数据进行降维处理,采用LS-SVM 对信息基因集进行分类,在两个基因表达数据集上具有较高的测试准确率。 范怡敏等[8]提出一种改进深度森林模型(two boosting deep forest,TBDForest),采用均等式特征利用方法对原始特征进行变换,并将上层最重要的部分判别特征输入到下一级联层,在原级联层采用子层级联的结构,避免模型对参数的依赖,验证结果表明,改进的算法达到了更好的分类效果。颜建军等[9]针对中医问诊复杂性和非线性的特点,采用深度级联森林算法(multi-grained cascade forest, gcForest)构建慢性胃炎中医问诊证候分类模型并与多种模型进行比较。 实验结果表明,基于gcForest 的方法在分类准确率上都优于其他算法,能有效地解决慢性胃炎中医问诊证候分类问题,为gcForest 方法在疾病诊断方面提供参考。
本研究针对基因表达数据降维问题,使用改进的最大相关最小冗余变量选择方法,利用特征与响应变量之间的相关性选择最大相关特征,并计算条件互信息下的冗余特征,以到达选择较少变量并提高分类器准确率的目的。 在分类问题上,提出使用深度级联森林方法作为分类器,对比试验表明,深度级联森林在乳腺癌基因数据上的分类效果优于其他传统分类器。 最后将所提出的变量选择方法与深度级联森林相结合,显著提高分类准确率。
1 方法
1.1 实验过程
对乳腺癌基因表达数据分类主要由3 个阶段组成:一是预处理阶段,为克服不同特征不同量纲对结果的影响,首先对数据进行标准化处理,使数据分布在[0,1]区间;二是特征提取阶段,使用改进的最大相关最小冗余方法对特征进行重要性排序,在[0,600]区间,每50 个特征作为一个特征子集选择节点,每次向特征集中增加50 个特征;三是分类阶段,使用深度级联森林对提取特征后的数据进行分类,十折交叉验证后的分类准确率作为模型的评价指标。
1.2 数据来源及预处理
乳腺癌数据取自博德基因研究所的公开实验数据 ( http:/ /portals.broadinstitute.org/cgi-bin/cancer/datasets.cgi)。 该数据集含有98 个乳腺癌受试者的观测数据,每位受试者拥有1 213 个特征基因,98 位受试者分别被诊断为A1,A2,A3 共3 类乳腺癌疾病状态。 具体工作是需要通过这1 213 个基因在不同个体上的表达值诊断该受试者的疾病状态。 数据是不平衡的,其中A1 类数据的数量为11,A2 类数据的数量为51,A3 类数据的数量为36。
获得数据后,为克服量纲对结果的影响,保证分析结果的有效性,对数据进行最大最小标准化处理,使数据分布在[0,1]区间,其表达式为

式中xi是实际观测值,xmin是所有观测中的最小值,xmax是所有观测中的最大值。
1.3 最大相关最小条件冗余变量选择
癌症可能发生在人体的任何部位,值得注意的是,癌症的初期治疗要比晚期容易的多,基于基因数据的分析已经成为早期癌症鉴定的有效方法,在基因表达数据的收集过程中,由于临床受试者数目有限以及受试群体之间的异质性,导致样本数量远小于变量(基因)数目,为了进行分类,识别出一小部分导致疾病发生的主要基因是十分重要的,这可有助于去除不适当的和无效的基因,提高对分类模型的理解。
假定Xi是候选变量,Y是响应变量,Xj∈S是一个被选变量,S是被选变量子集。 定义候选变量Xi与响应变量Y之间的互信息为相关项,候选变量Xi与被选变量Xj之间的互信息称为冗余项;任何变量选择问题的目标都是选择相关项,排除无关项。对于冗余项,可以看作是存在相依性的有用变量。例如,在测量相关变量时出现一些错误,则预测器工作性能很差,但如果预测器选择了一些相关变量的冗余项,这些错误即可纠正。 所以,预测器可选择某些冗余变量,以提高预测的鲁棒性。
因此,在基因数据的变量选择过程中,将基因视为自变量,受试者状态标签(A1/A2/A3)视为响应变量,其目的是在变量集中选取对标签变量起作用的相关基因,排除无关基因,选择冗余基因。
从候选变量的角度看, 互信息( mutual information,MI)[10]是变量选择问题中变量相互依赖性的一种度量。 对于两个离散的变量X和Y,其互信息定义如下:

式中,p(x,y) 是联合分布,p(x) 和p(y) 是边际分布。
互信息也可以被条件化,条件互信息[11-14]的定义如下:

变量相关性分析和冗余性分析一直是变量选择领域的两个挑战性问题。 近年来,针对分类问题,学者们提出了多种基于最大相关最小冗余准则的MI 变量子集选择方法,首先计算待选择的变量相关性,其次计算关于先前已选择变量子集的变量冗余性。 Battiti[15]首次利用最大相关最小冗余的启发式 MI 逼近(mutual information based feature selection, MIFS)来选择变量子集:
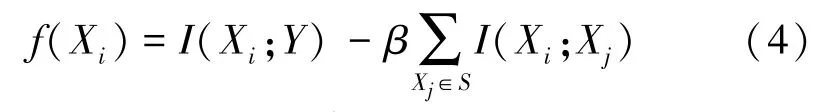
式中,β调整相关项和冗余项的减法可比性,f用于估计变量Xi的优良性。 但是参数β的选择是困难的,因此,Peng 等[16]提出的最大相关最小冗余(max-relevance and min-redundancy, mRMR)准则如下:

式中,Xi为候选变量,Y为响应变量,S为已选变量子集。
然而大多数基于MI 的方法仅计算I(Xi;Xj) ,而没有计算候选变量、S中的已选变量、以及响应变量Y之间的冗余信息。 作为将被选择的变量,应该不能由S中的已选变量来预测、且对于响应变量Y必须是有信息的[17]。 因此,考虑条件互信息的作用,将mRMR 方法的后半部分改进为计算候选变量Xi与已选变量Xj在目标变量条件下的条件互信息

因此,改进的最大相关最小条件冗余(maxrelevance and min-conditional redundancy, mRMCR)可以表示为

该方法流程如图1 所示。

图1 最大相关最小条件冗余算法流程Fig.1 Flow chart of mRMCR algorithm
1.4 深度级联森林
深度级联森林是一种基于树模型的集成学习算法,包括多粒度扫描和级联森林两部分[18]。 多粒度扫描解决了高维输入问题,增强输入特征的差异性。 级联森林通过模拟深度神经网络的结构进行表征学习,提高输入特征分类能力。 图2 为深度级联森林的总体过程。

图2 深度级联森林的总体过程(左侧虚线框表示多粒度扫描过程,右侧虚线框表示级联森林过程)Fig.2 The general process of gcForest (The left dashed box represent the multi-grained scanning process, the right dashed box represent the cascade forest process)
将多粒度扫描展开得到图3 所示过程图,多粒度扫描结构通过滑动窗口对原始输入进行局部采样,得到多个不同维度的特征实例,然后经过随机森林和完全随机森林分类器产生类概率向量,最后通过类向量的连接转换为级联的输入特征。 以1 213维特征数据为例,滑动窗口大小为100 维,滑动步长为1,经过滑动后,产生(1 213-100)/1+1 =1 114个特征子样本,针对三分类问题,每个子样本经过随机森林和完全随机森林训练后,分别生成一个3 维的类概率向量,最后得到一个1 114×3×2 =6 684维的特征向量。 使用不同维度的滑动窗口可以生成不同粒度大小的特征向量,类似地,对于每个原始的训练样本,若使用200 维和300 维的滑动窗口将分别生成6 084 和5 484 维的特征向量。

图3 多粒度扫描结构(虚线框代表滑动窗口)Fig.3 Multi-grained scanning (The dashed box represent sliding window)
级联森林阶段通过对特征进行逐层表征学习,体现其深度学习的过程。 级联森林的第一级将多粒度扫描后的特征信息作为输入,之后的每一级都接收原始输入和从上一级输出的特征向量连接作为该级的输入,使特征信息得到了增强。 级联的每一层包含两个随机森林和两个完全随机森林,通过使用交叉验证产生每个森林的类向量,并将输出结果和原始输入拼接到一起输入到下一级中。
以100 维的滑动窗口扫描为例,对于三分类问题,经过两个随机森林和两个完全随机森林后,每个级别生成4×3 =12 维增强特征向量,与扫描变换后得到的6 684维特征向量拼接后得到6 696维特征向量作为第2 级的输入,以此类推,第2 级经训练后生成1 个12 维的特征向量,与200 维滑动窗口扫描后变换得到的6 084 维特征向量拼接,所得到的6 096 维类向量作为第3 级训练的输入,第3 级经训练后生成一个12 维的特征向量,与300 维滑动窗口扫描变换后得到的5 484 维特征向量拼接,得到5 496 维类向量,再作为下一级的输入,重复上述过程,直到没有显著的性能增益,训练过程停止。
针对深度级联森林方法,使用Python 软件实现,代码来源于GitHub 网站,由算法的提出者共享。下附代码来源网址: https:/ /github.com/kingfengji/gcForest
1.5 评价指标
由于数据存在不平衡问题,因此采用十折交叉验证,从每类数据中按比例抽样,保证每个交叉验证的数据集含有所有的分类标签。 采用分类平均准确率作为评判标准,计算公式为

式中,i表示第i折交叉验证,Ni(TRUE) 表示第i次交叉验证中分类正确的数目,N(ALL) 表示所有分类标签数目,准确率用10 折交叉验证的平均值表示。
2 结果
从基因数据中的许多基因中选择一小部分合适的数据对精确的癌症分类至关重要,传统的方法通常根据基因的差异表达对基因进行排序,并选择排名靠前的基因进行分类任务。 因此,在变量选择方面,选择使用随机森林特征重要性和最大相关最小冗余作为对比方法。 在对基因表达数据进行特征选择和分类时,根据数据集的大小选择不同规格的特征间隔是一种常用方法,其目的是快速验证该方法的有效性和鲁棒性。 因此,为验证变量选择方法的有效性,设定600 为变量选择中心阈值,使用随机森林(random forest, RF)[19],支持向量机(support vector machine, SVM)[20], BP神经网络(back propagation neural network, BPNN)[21]作为深度级联森林的对比分类器,在[0,600]区间,每次向特征集中增加50 个特征[22-23],并结合分类器分析特征选择方法的效果。
将3 种变量选择方法与4 种分类器相结合应用于乳腺癌基因数据分类,得到如表1 及图4 所示信息。
表1 中加粗部分表示相同变量数目下的最佳分类准确率。 由表1 可知,所使用的最大相关最小条件冗余方法与随机森林方法相结合时,仅在数据量为400 时,准确度不敌最大相关最小冗余方法,在变量数为50 时,最大相关最小条件冗余方法结合随机森林分类效果达到92.83%,远高于不进行变量选择时的分类准确率88.61%。

表1 变量选择方法在乳腺癌基因数据集上的分类准确率(%)Tab.1 Classification accuracy (%) of feature selection method on breast cancer gene dataset
在与支持向量机相结合时,变量数目小于200时,其分类效果优于最大相关最小冗余方法,但稍逊色于基于随机森林特征重要性方法,变量数大于等于200 时,分类效果优于其他两种方法,尤其在变量数为550 时,改进的方法结合支持向量机分类效果达到89.89%,远高于不进行变量选择时的分类准确率86.78%。
在与BP 神经网络相结合时,变量数目分别为50,100,200 时,其分类效果优于最大相关最小冗余方法但稍逊色于基于随机森林特征重要性方法,变量数大于200 时,分类效果优于其他两种方法,尤其在变量数为450 时,改进的方法结合BP 神经网络分类效果达到87.56%,高于不进行变量选择时的分类准确率84.56%。
在与深度级联森林相结合时,分类效果明显优于基于随机森林特征重要性方法,在变量数为100时,达到最佳分类准确率93.78%,远高于不进行变量选择时的分类准确率90.67%。 这也是所有方法中,表现最佳的一组。
图4 表示变量选择方法在不同分类器上随变量数目变化的表现。 由图可见,最大相关最小条件冗余方法效果明显优于其他两种变量选择方法。

图4 变量选择方法在不同分类器上的表现。 (a)随机森林;(b)支持向量机;(c)BP 神经网络;(d)深度级联森林Fig.4 Performance of variable selection method on different classifiers.(a) Random forest; (b) Support vector machine; (c) BP neural network; (b) Multi-grained cascade forest
表2 和表3 分别显示了所使用的变量选择方法相对于其他两种方法在分类准确率上的改进效果及其平均值。 mRMCR 相对于随机森林重要性方法在随机森林,支持向量机,BP 神经网络和深度级联森林分类器上的平均准确率分别提高了2.19%,1.63%,1.92%和3.24%。 mRMCR 相对于mRMR方法在随机森林,支持向量机,BP 神经网络和深度级联森林分类器上的平均分类准确率分别提高了1.03%,1.54%,3.81%和0.54%。
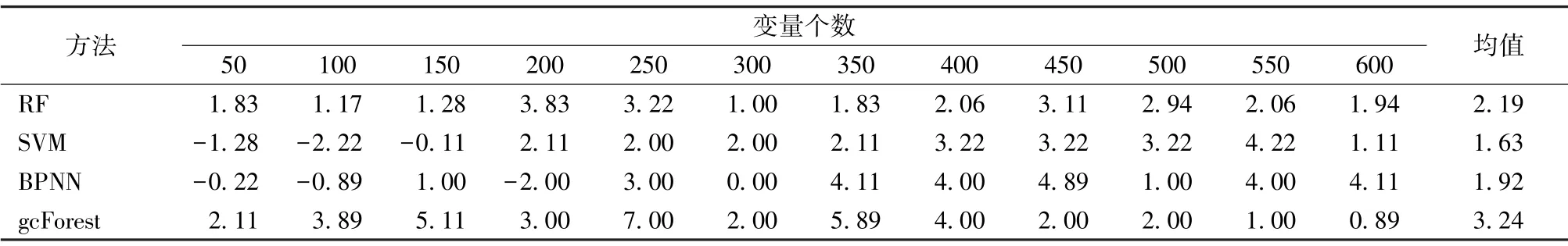
表2 mRMCR 相对于随机森林重要性方法提高的分类准确率(%)Tab.2 The improved classification accuracy (%) of the mRMCR over the random forest feature importance method
为了对比变量选择方法的稳定性,计算从50~600 个变量的平均分类准确率,绘制如图5 所示的柱状图。 图5 清楚的显示出最大相关最小条件冗余方法表现出比基于随机森林特征重要性和最大相关最小冗余方法更高的分类准确率和在分类器上的稳定性。 由图5 可知,最大相关最小条件冗余方法在各个分类器上都有良好的效果,为了更加直观的对比最佳分类器,将最大相关最小条件冗余结合四种分类器的效果绘制如图6 所示折线图,由图可见,最大相关最小条件冗余和深度级联森林结合方法效果明显优于其他3 种方法的分类效果,在变量数为100 时,即达到最佳分类准确率,为93.78%。
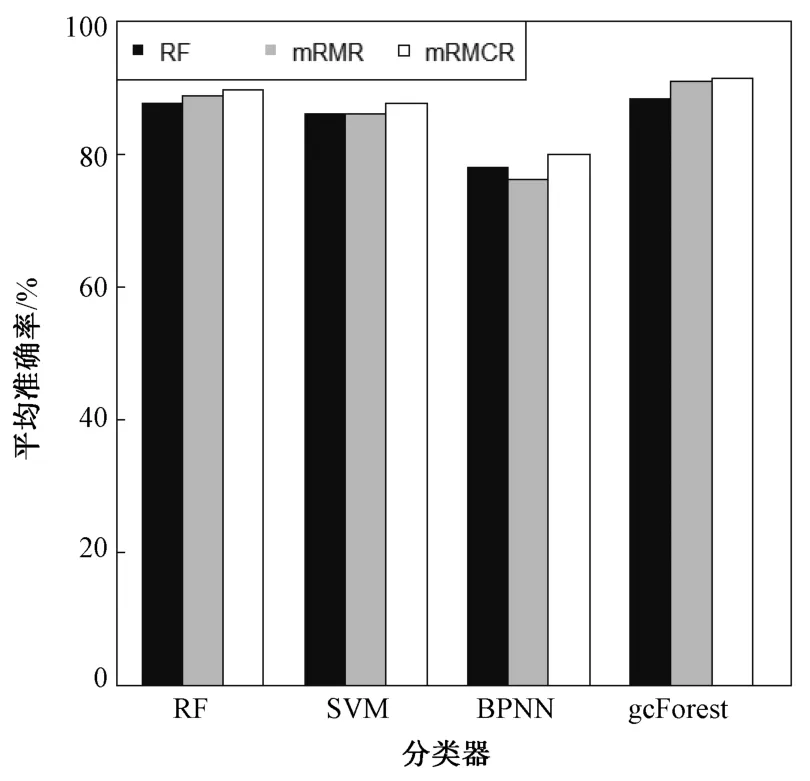
图5 从50 到600 个变量的平均分类准确率Fig.5 Average classification accuracy from 50 to 600 variables

图6 4 种分类器结合最大相关最小条件冗余方法准确率Fig.6 Accuracy of four classifiers combined with mRMCR
3 讨论
癌症的早期诊治非常关键,一种有效的基因数据降维分类方法在临床诊断与治疗中是至关重要的。
互信息是一种流行的相关性度量方法,也是在基因数据特征选择过程中常用的基本方法。 陈俊颖[2]利用互信息最大化找出同类中相关性最强的基因,结合自适应遗传算法显著降低了数据维度,减少了分类冗余。 陈昊楠等[23]在基因表达数据的癌症分类研究中提出了同时考虑相关性、冗余度以及增益性等三大特性的多特征交互的特征选择方法,解决了癌症表达谱数据的高维、高冗余问题。
Zhou 等[18]提出的深度级联森林算法,从手写识别、人脸识别、音乐片段识别等方面的数据集中被证明是一个可以媲美DNN 而且比传统机器学习模型有明显优势的模型,具有参数少,模型训练简单,可扩展等优点。 此外作为一种基于树的方法,gcForest 在理论分析方面也比深度神经网络更加容易。 范怡敏等[8]在此基础上对原始特征进行变换,将上层最重要的部分作为下一层级联的输入,采用子层级联的结构,提出一种改进深度森林模型TBDForest,获得了良好的分类性能。 刘超等[25]在DNA 甲基化的癌症分类研究中使用了gcForest 方法,也证实了gcForest 分类模型在小规模不平衡数据集中性能优势。
通过对博德研究所基因组数据分析中心的实验数据进行实证分析,考虑响应变量存在条件下,候选变量与已选变量之间的条件互信息,并将其视为冗余,将mRMCR 变量选择算法与mRMR,基于随机森林特征重要性方法进行对比,分别结合随机森林(RF),支持向量机(SVM),BP 神经网络(BPNN)和深度级联森林(gcForest)验证效果,结果表明,在4 个分类器中,所使用的变量选择方法在相比之下提高了1.03%~3.81%的平均分类准确率,mRMCR相对于随机森林重要性方法在深度级联森林提高3.24%的平均分类准确率;相对于mRMR 方法在BPNN 上提高了3.81%的平均分类准确率。 分类结果表明,最大相关最小条件冗余和深度级联森林结合方法对乳腺癌基因数据的分类效果显著,明显优于其他组合方法,在选择100 个变量的情况下达到93.78%的最佳分类准确率。 该方法不仅可以应用在基因表达数据分类方面,还可以应用于金融数据、机械故障诊断等其他众多高维小样本数据领域,为在不同领域研究变量选择与分类等问题提供了可行的思路和模式。
4 结论
基因表达是一个高维的数据,但是只有少部分基因能够直接导致癌症的发生。 因此,基于基因表达数据子集的分类(通过合适的变量选择方法进行选择)是一种常见的方法。 在研究中,使用一种新的冗余措施来改进mRMR 的变量选择,然后用不同的分类器进行分类。 与对比方法相比,使用的最大相关最小条件冗余方法考虑响应变量存在条件下,候选变量与已选变量之间的条件互信息,并将其视为冗余,有效压缩数据维度,达到变量选择的目的。针对乳腺癌基因表达数据集,对数据进行标准化处理,结合不同的分类器研究结果表明,该方法能够在选择较少变量的同时明显提高分类准确率,拥有较高的准确性和稳定性。 基于基因数据的癌症分类是一项敏感的任务,需要拥有高度的准确性。 使用最大相关最小条件冗余和深度级联森林结合方法优于其他方法效果,可明显提高乳腺癌的分类准确度。 所提出的方法对基于基因数据的乳腺癌分类诊断具有重要的理论意义与实用价值,可以为病人及时提示和预警,为医护人员提供科学的决策支持。

