版纳微型猪近交系AURKC 的分子特征及其在睾丸中的转录调控研究
代红梅,刘志朋,张 霞,霍海龙,王 配,赵 筱,霍金龙
(1. 云南农业大学 动物科学技术学院,云南 昆明 650201;2. 吕梁学院 生命科学系,山西 吕梁 033001;3. 云南农业职业技术学院,云南 昆明 650212)
极光激酶C(AURKC)属于丝氨酸/ 苏氨酸极光激酶(Aurora)家族成员,是雄性生殖细胞减数分裂过程中纺锤体形成的必需酶,该家族由控制染色体分离和浓缩、中心体复制、G2/M 期转换、着丝粒附着和胞质分裂的调节因子组成[1],包括AURKA、AURKB 和AURKC 3 个成员,属于细胞周期调节酶,能确保细胞分裂过程中染色体分离和纺锤体形成的正常进行,并且在减数分裂过程中调节联会复合体的动力[2]。该家族的3 个成员之间有60%以上的氨基酸序列同源性,但每种蛋白质在细胞中特定的定位模式和独特的功能又有所不同[3]。与在多组织中广泛表达的AURKA 和AURKB 不同,AURKC 主要在睾丸中高表达,在肺、骨骼、卵巢和血液白细胞中弱表达[4],AURKC 在减数分裂中期定位于着丝粒和染色体,在后期则定位于纺锤体中带和中间体[5],是减数分裂期间染色体正确排列和分离的必需因子,在分裂期间的纺锤体形成中起关键作用,并在减数分裂过程中纺锤体组装检验点中发挥作用,辅助染色质在减数分裂期间的浓缩和分离[6]。AURKC 也是染色体乘客复合体(Chromosomal passenger complex,CPC)的一个重要组成部分,它能结合染色体乘客复合体亚基并在丝氨酸残基上磷酸化组蛋白H3,从而调节染色体的浓缩和胞质分裂,以确保减数分裂中染色体和微管之间的正确结合[7]。在雄性小鼠中敲除AURKC,小鼠的睾丸重量和精子数量正常,但精子易发生头部变钝、顶体丢失、染色质凝聚异常等缺陷,小鼠生育力明显下降[8]。睾丸中的AURKC可调节精子细胞的分裂,确保每一个新的精子细胞的形态正常并包含每条染色体的一个拷贝,在精子发生中,AURKC基因突变会产生无活性或可快速降解的蛋白质,缺乏正常的AURKC蛋白会使精子细胞的分裂受阻,引起人类大头精子症,进而导致男性原发性不育[9]。AURKC的异常表达还能导致基因组不稳定并引发恶性细胞转化,其在人体组织中过表达会导致癌细胞的非典型有丝分裂,驱动许多癌细胞系的肿瘤发生[10]。
版纳微型猪近交系(Banna mini-pig inbred line,BMI),已利用全同胞和亲子交配的高度近交方式繁育41 年,是云南农业大学培育的大型近交系哺乳动物模型[11]。本研究以BMI 睾丸为研究材料,进行转录组测序,获得AURKC基因的表达水平;克隆BMIAURKC基因的编码区序列,分析其基因的分子特征,检测编码区序列是否存在SNP;获悉AURKC 蛋白质的基本特性,挖掘AURKC 蛋白的互作蛋白;并通过注释AURKC基因,构建其ceRNA 转录调控网络,为进一步研究AURKC基因在BMI 精子发生中的作用奠定基础。
1 材料与方法
1.1 试验动物
BMI 12月龄成年公猪4头,去势取睾丸组织样品。
1.2 转录组测序及AURKC 基因表达量分析
对4 个BMI 睾丸组织样品构建文库,利用Illumina Hiseq 4000 平 台 进 行RNA-seq 测 序,利用Illumina novaseq 6000 平 台 进 行small RNA 测序。利用fastp 软件对RNA-seq 原始数据质控并过滤低质量数据,使用bowtie2-2.1.0 比对工具将过滤好的数据比对到猪核糖体参考序列,并去除比对到核糖体的序列。从Ensembl 网站下载猪参考基因组(Sus scrofa 11.1)和注释文件(gtf 11.1),使用STAR-2.5.2a 软件构建参考基因组的索引,并将已去除核糖体序列的数据与猪参考基因组比对。用featureCounts-2.0.1 软件 和salmon-1.5.1 软件进行表达量计算,得到AURKC基因的原始表达量和TPM 值校正表达量。对small RNA 原始数据进行质控,利用bowtie 软件去除样本中的rRNA、tRNA、snRNA 和snoRNA,把剩余的clean reads 与miRBase 数据库中所有物种的前体miRNA 和成熟的miRNA 序列进行比对,获得样本中miRNA 的表达量值。
1.3 基因扩增及序列测定
根 据AURKC转 录 组 数 据 钓 取 到Ensembl 数 据 库 对 应 的 猪AURKC转 录 本ENSSSCT00000051895.2,利用该转录本序列设计特异性引物(F: CCCTTCTCAGGATCACCCA;R:AGACAGATGAGATACCAGAGC),以BMI 睾 丸cDNA 为模板扩增AURKC基因全长编码区。反应体系25 μL:Premix TaqTM version 2.0 12.5 μL,10 μmol/LAURKC上下游引物F/R 各1 μL,25 ng/μL cDNA 1 μL,H2O 9.5 μL;扩增程序:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 45 s,72 ℃ 2 min,30 个循环;72 ℃ 10 min。扩增产物送昆明擎科生物公司测序。
1.4 AURKC 功能分析
利用Lasergene7.1 校对测序的AURKC序列;利用NCBI 的ORFfinder 工具对AURKC的开放阅读框进行查找分析;用ProtParam 程序预测AURKC蛋白质的分子量、分子式、等电点、正负电荷残基数;AURKC 蛋白质的二级结构、三级结构、疏水结构和功能位点分别使用SOPMA、Swiss-model、ProtScale 和Prosite 预 测; 使 用TMHMM 2.0 和SignalP 5.0 分别预测AURKC 蛋白的跨膜结构和信号肽;使用MEGA7 构建AURKC 蛋白的系统发育树;使用Weblogo 工具对结构域区的氨基酸序列进行多物种保守性分析;用String v11.0b 进行蛋白互作网络分析;通过Ensembl 数据库下载其它猪种的AURKC基因编码区序列并与BMI 进行比对。
1.5 AURKC 的功能注释和ceRNA 调控网络的构建
利 用Uniprot 进 行GO(Gene Ontology) 注释;利用已获得的猪RNA-seq 数据进行miRNA 和lncRNA 表达分析;利用miRanda 3.3 和RNAhybrid 2.1.2 软 件 挖 掘 潜 在 调 控AURKC的miRNA 和lncRNA,用Cytoscape 3.8.2 绘制可视化网络图。
2 结果与分析
2.1 AURKC 基因及氨基酸结构信息
RNA-seq 测序分析结果显示,每个样品获得6 Gb clean data,平均测序深度为38.6 百万reads;比对率均在96%以上,共注释到17 040 个编码基因、63 023 个 转 录 本 和9 342 个lncRNA。Small RNAseq 测序分析结果显示,每个样品均获得了10 M 以上的clean data,平均测序深度为14.7 百万reads,注释到成熟的miRNA 共458 个。其中AURKC基因在BMI 睾丸中的原始平均表达量为721.25,其对应Ensembl 网站的转录本ENSSSCT00000051895.2 的平均表达量(TPM)值为18.37。利用AURKC-F/R 引物扩增BMIAURKC基因的完整编码区(CDS)及部分非编码区(UTR),获得1 063 bp 长的产物(图1A)。AURKC基因CDS 序列长894 bp,编码297个氨基酸(图1B)。该基因定位于猪(Sscrofa11.1)6 号染色体,全长5 464 bp,Ensembl 网站分析发现包含7 个外显子和6 个内含子,AURKC 蛋白含有STKc_Aurora-B_like 保守结构域(图1C)。
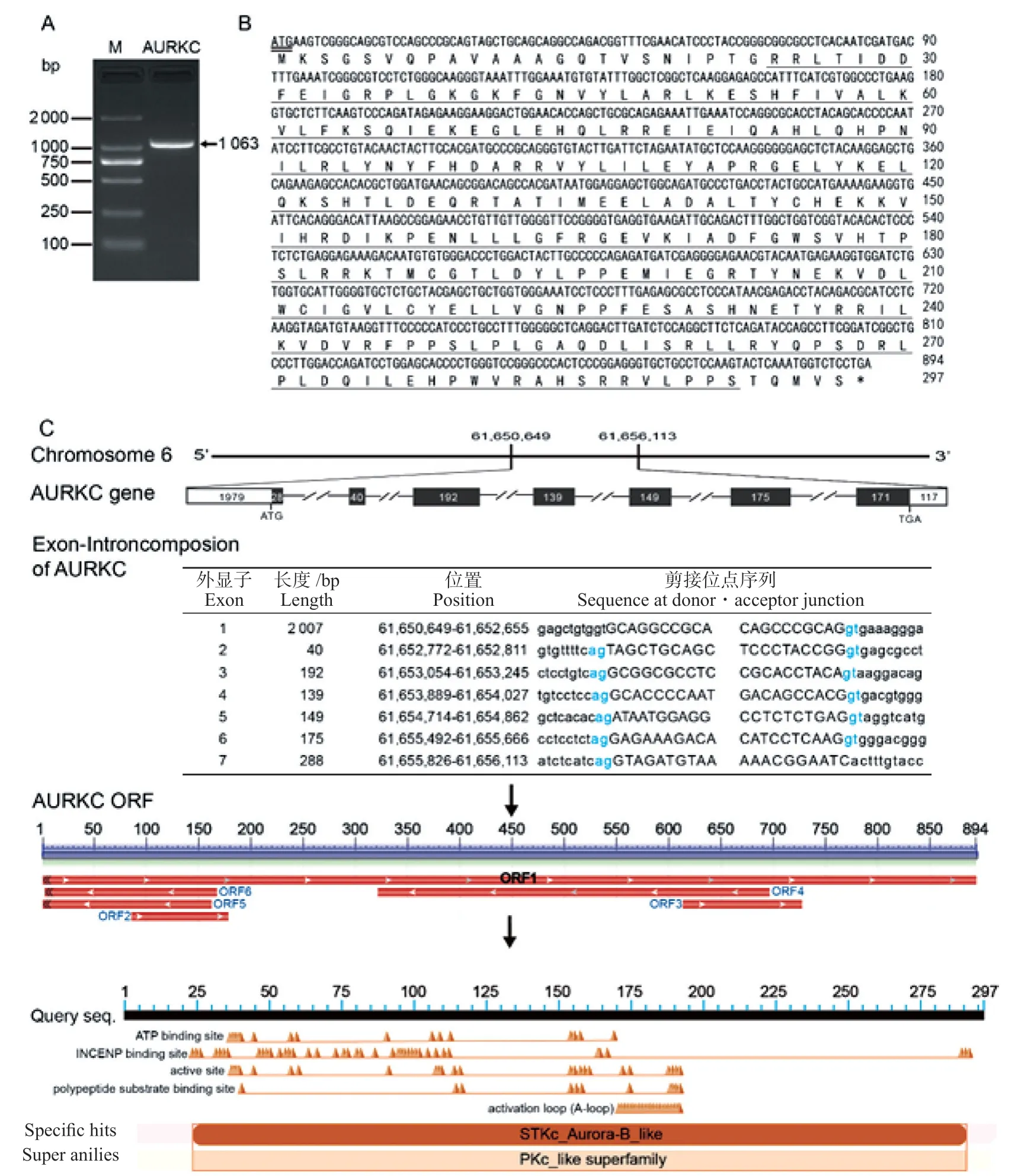
图1 AURKC 基因结构Fig. 1 Gene structure of AURKC
2.2 BMI AURKC 蛋白质序列及结构分析
猪AURKC 蛋白质分子量34.2 kD,分子式C1539H2445N435O431S9,等电点8.93,负电荷残基数为36,正电荷残基数为41,为碱性氨基酸。AURKC蛋白质297 个氨基酸的二级结构中无规则卷曲占比最大(38.05%),包含113 个氨基酸;α 螺旋次之(36.36%),包含108 个氨基酸;延伸链占18.52%,包含55 个氨基酸;β 转角最少,仅占7.07%,有21 个氨基酸(图2A)。利用SWISSMODEL 构建三级结构可知,AURKC 三级结构与二级结构相似,主要包含无规则卷曲、α 螺旋、延伸链和β 转角(图2B)。蛋白质的第59 位氨基酸具有最大疏水值2.667,第234 位氨基酸处具有最小疏水值-2.224,N 端亲水、C 端疏水。含1 个蛋白质功能域S_TKc,含有酶磷酸化活化位点,无亮氨酸富集的核输出信号、无跨膜结构域,无信号肽。
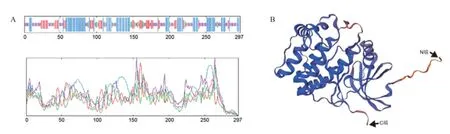
图2 AURKC 蛋白的空间结构Fig. 2 The spatial structure of AURKC protein
2.3 AURKC 基因的保守性及系统进化分析
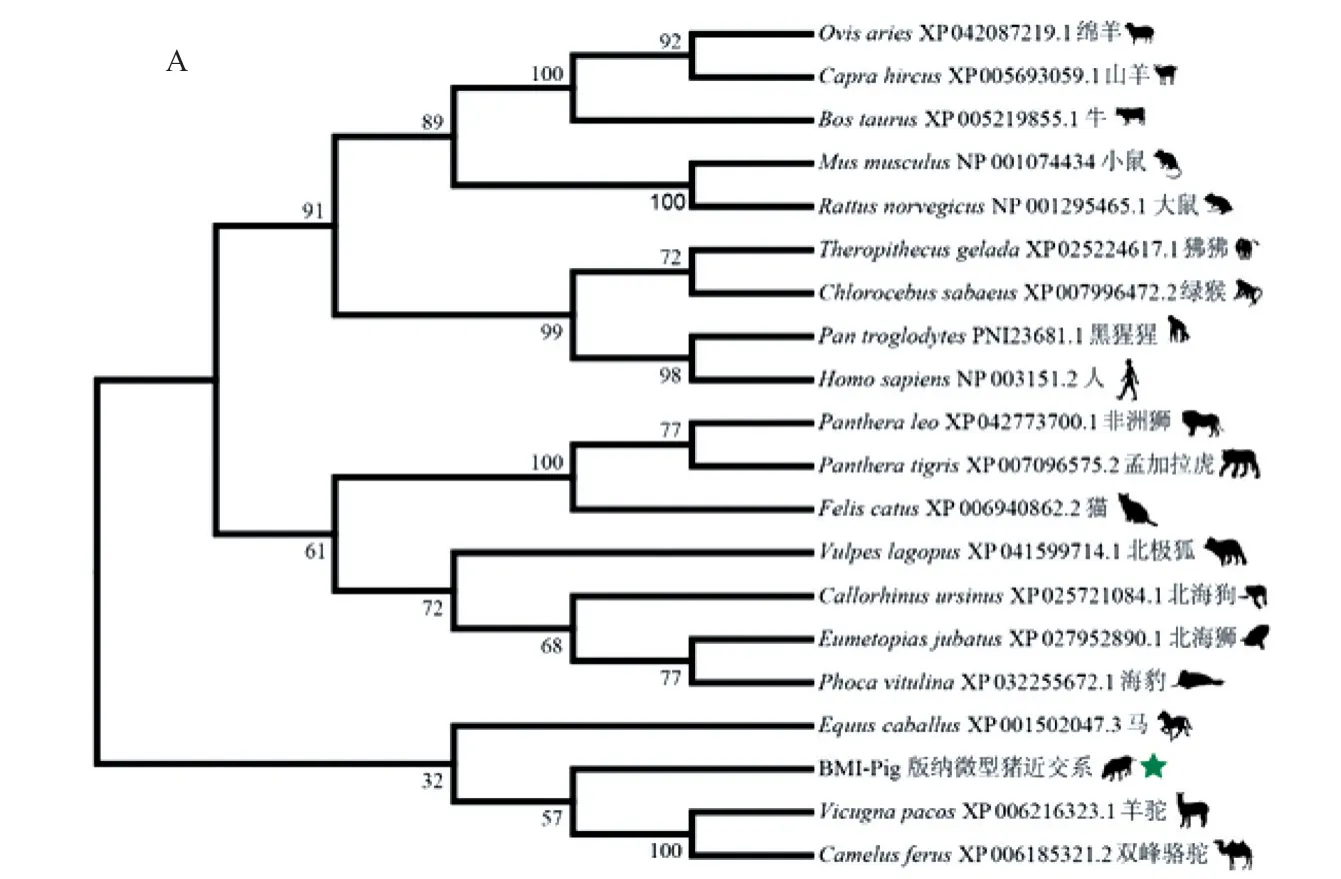
将BMIAURKC基因的CDS 序列与野猪(ENSSSCT00000051895.2)、大白猪(ENSSSCT 00025104349.1)、 长 白 猪(ENSSSCT000450640 29.1)、巴 克 夏 猪(ENSSSCT00065011958.1)、汉普夏猪(ENSSS CT00035013414.1)、皮特兰猪(ENSSSCT00055002061.1)、八眉猪(ENSSSCT000 50027876.1)、金华猪(ENSSSCT00060104185.1)、梅 山 猪(ENSSSCT00040097989.1)、 荣 昌 猪(ENSSSCT00030011616.1)、藏猪(ENSSSCT00 015108641.1)等11 个猪品种AURKC的CDS 序列进行比对,未发现AURKC基因CDS 序列在不同品种猪间存在差异位点。20 个哺乳动物AURKC 氨基酸序列比对发现,BMI 与马的相似度最高(91.9%),其次为双峰骆驼(91.6%)、羊驼(90.4%)、北海狮(90.4%)、北海狗(90.1%)、海豹(89.8%)、北极狐(89.8%)、黑猩猩(87.7%)、人(87.4%)、猫(87.4%)、孟加拉虎(87.1%)、非洲狮(87.1%)、狒狒(85.6%)、绿猴(85.3%)、山羊(84.1%)、牛(82.9%)、绵羊(77.2%)、大鼠(76.3%),与小鼠的相似性最低(69.5%)。系统进化分析表明,BMI 与马、双峰骆驼、羊驼聚为一支(图3A)。通过构建20 个哺乳动物AURKC 保守结构域S_TKc的Weblogo 图发现AURKC 在物种间较为保守(图3B),20 种哺乳动物间共有86 个氨基酸差异位点;除大鼠、小鼠外,其他18 种哺乳动物间有57 个氨基酸差异位点(图3C)。
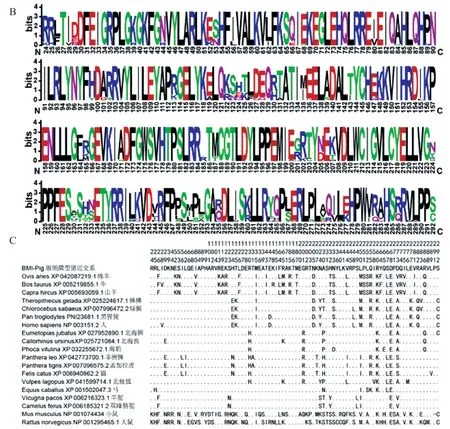
图3 20 种哺乳动物AURKC 的氨基酸序列分析Fig. 3 Amino acid sequences analysis of AURKC from 20 mammals
2.4 AURKC 蛋白互作网络
蛋白互作网络分析显示猪AURKC 与10 个蛋白可能存在相互作用,包括AURKB、BIRC5、INCENP、CDCA8、NDC80、BUB3、CENPA、TACC2、KIF11 和HIST2H3PS2,其 中 与AURKB蛋白相互作用最为密切,与BIRC5、INCENP、CDCA8、NDC80、BUB3 相互作用较为密切(见图4)。
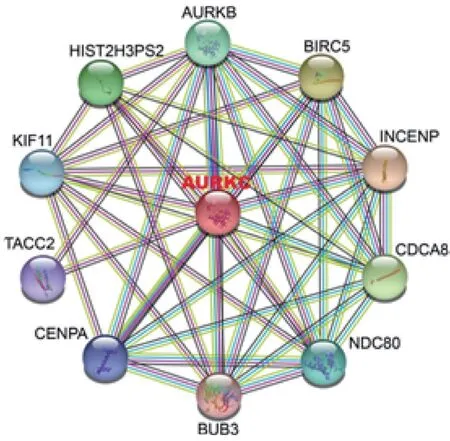
图4 AURKC 蛋白互作网络Fig.4 Interacting network of AURKC protein
2.5 猪AURKC 功能注释及ceRNA 调控网络
分析AURKC的注释结果共发现26 个GO:在生物学过程(Biological process),主要涉及调节胞质分裂、有丝分裂、精子生成、蛋白质磷酸化、减数分裂、纺锤体中央组装、卵母细胞发育、磷酸化、细胞分裂、细胞周期、组蛋白-丝氨酸磷酸化等11个GO;在细胞组分(Cellular component),主要涉及浓缩染色体、纺锤体中央、纺锤体微管、中心体、染色体过客复合体、中间体等6 个GO;在分子功能(Molecular function)方面,主要涉及一系列蛋白激酶活性以及ATP 结合和核苷酸结合等9 个GO(见图5)。
miRNAs 调控网络分析发现,BMIAURKC受4个miRNAs(ssc-miR-28-3p、ssc-miR-202-3p、ssc-miR-1296-5p 和ssc-miR-361-5p)靶向调控;有1 个lncRNA 与AURKC竞争性结合ssc-miR-28-3p,有2 个lncRNAs 与AURKC竞 争 性 结 合sscmiR-202-3p,有6 个lncRNAs 与AURKC竞 争 性结合ssc-miR-1296-5p,有1 个lncRNA 与AURKC竞争性结合ssc-miR-361-5p(见图5)。
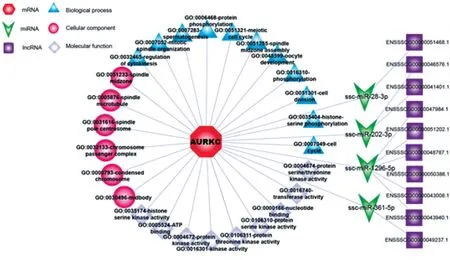
图5 猪AURKC 的功能注释及ceRNA 调控网络Fig.5 Functional annotation of porcine AURKC and ceRNA regulatory network
3 结论与讨论
3.1 BMI AURKC 序列特征及与其他物种的进化关系
本研究通过睾丸转录组测序获得了BMI 睾丸中AURKC基因的表达量,获悉了其对应Ensembl 数据库的转录本为ENSSSCT00000051895.2;利用BMI睾丸cDNA 扩增获得了AURKC基因序列1 063 bp,其中CDS 序列894 bp,编码297 个氨基酸,序列提交GenBank 获得基因登录号为OK042305。蛋白功能域预测发现,AURKC 蛋白包含S_TKc 结构域,该结构域是丝氨酸/苏氨酸蛋白激酶催化结构域,从原核生物的大肠杆菌到真核生物的人类都较为保守,特别其催化亚基更高度保守[12],表明AURKC蛋白功能在物种间较为保守。氨基酸序列比对发现BMI 与马、双峰骆驼、羊驼、北海狮、北海狗的相似度均在90%以上,与除绵羊、大鼠、小鼠外的其它哺乳动物的相似度均大于80%。在进化上BMI与马、双峰骆驼、羊驼的亲缘关系较近,说明BMI AURKC 与其他哺乳动物在进化上具有高度的保守性和序列同源性。
3.2 AURKC 蛋白互作网络
蛋白互作网络分析发现AURKC 与AURKB、BIRC5、INCENP、CDCA8、NDC80、BUB3、CENPA、TACC2、KIF11、HIST2H3PS2 等10 个蛋白存在相互作用,其中与AURKB 的相互作用最密切,AURKB 和AURKC 在功能上能相互补偿,从而确保哺乳动物正常的精子发生,在雄性小鼠中,双敲AURKB和AURKC后,会导致精母细胞无法协调联会复合体侧向元件的分解与染色体的浓缩和分离,进而影响了减数分裂的进程[2]。AURKC 和AURKB 通过与IN box 结构域的相互作用和磷酸化稳定内着丝粒蛋白(INCENP),确保染色体的正常分离[13],此外,AURKB 可通过负调控AURKC 来防止非整倍体的产生,而AURKC 可挽救AURKB沉默的多核表型[14]。BIRC5 是凋亡抑制蛋白IAP家族成员,可影响细胞分裂、增殖并抑制细胞凋亡,其作为染色体乘客复合物(CPC)的组成成分,通过与AURKC 相互作用在G2/M 期调节微管的动力并促进细胞的有丝分裂[13]。INCENP 与BIRC5 一样可作为CPC 的一个组成部分,不仅可靶向着丝粒,而且可与AURKB 形成复合物,在有丝分裂期间协调染色体分离、纺锤体出现和胞质分裂[15]。CDCA8 也是脊椎动物CPC 的重要组成部分,与减数分裂期间纺锤体的形成和染色体的分离密切相关,在肿瘤发生中起调节作用,其高表达与多种癌症的发生、侵袭相关,是多种癌症潜在预后的生物标志物[16]。NDC80 是位于动粒外层的异源四聚体蛋白复合物,在有丝分裂中起关键作用,NDC80 磷酸化影响其独立于微管的降解,进而影响动粒蛋白的降解[17]。BUB3 是有丝分裂纺锤体组装复合体的保守组分,还是早期胚胎发生过程中纺锤体检验点激活通路的重要部分,在维持哺乳动物卵母细胞中染色体的正确分离和染色体的稳定性方面起重要作用[18]。CENPA 位于活性着丝粒上,能影响动粒形成和染色体分离,CENPA 核小体是着丝粒活跃的表观遗传标记[19]。TACC2 在促进细胞分裂和维持有丝分裂中染色质的稳定性方面发挥作用,TACC2 还是雄激素调节基因,通过调节有丝分裂的细胞周期来促进前列腺癌的细胞增殖,是前列腺癌和乳腺癌中的促癌因子[20]。KIF11 不仅是一种有丝分裂调节器,还是维持两极纺锤体有丝分裂的驱动蛋白,能在有丝分裂后介导中心体迁移并移动高尔基体[21]。
3.3 AURKC 基因突变与精子畸形症
AURKC的突变会引起人类罕见的大头精子症,Dieterich 等[9]研究发现,AURKC外显子3 中的C.144delc 纯合突变产生了移码突变,致使AURKC翻译提前终止,最终生成了具有不完全催化结构域的无功能截短蛋白质,从而导致了大头精子症。Ben Khelifa 等[6]发现AURKC外显子6 中的无义突变c.744C>G(p.Y248*)与C.144delc 具有相似的效果,会导致AURKC蛋白质功能缺失。Dieterich等[22]发现AURKC 外显子6 的错义突变c.686G>A(p.C229Y)虽然没有前面2 个突变严重,可以保留AURKC的部分功能,但不足以支持减数分裂的进程。Ben Khelifa 等[23]发现位于AURKC外显子5 的受体共剪接位点的杂合突变c.436-2A>G,会导致第5 外显子跳跃,进而产生缺乏部分S_TKc 结构域的截短蛋白。Hua 等[24]发现位于AURKC外显子3 的纯合错义突变c.269G>A 与大头精子症相关。BMIAURKC基因编码区与野猪、大白猪、长白猪、巴克夏猪、汉普夏猪、皮特兰猪、八眉猪、金华猪、梅山猪、荣昌猪、藏猪等11 个猪品种进行比对未发现变异位点,表明BMI 雄性不育及大头精子不是由AURKC基因编码区序列突变引起,可能由于表达差异、转录及翻译后修饰或者其他基因异常所引起,有待进一步研究。
3.4 AURKC 相关ceRNA 调控网络
对BMIAURKC基因功能注释及构建ceRNA 调控网络图发现,有4 个miRNAs 靶向调控AURKC,分别是ssc-miR-28-3p、ssc-miR-202-3p、ssc-miR-1296-5p 和ssc-miR-361-5p。miRNA 是高度保守的内源性小非编码RNA,可以在转录后或翻译水平调节基因表达,它没有开放阅读框(ORF),也不编码蛋白质,主要通过与其靶标mRNA 的3'UTR 结合来干扰基因表达,且miRNA 在物种间高度保守。sscmiR-28-3p 属于mir-28 家族,参与磷脂酰肌醇信号通路,与许多肿瘤的发生、发展相关[25]。ssc-miR-202-3p 能通过靶向Wnt/β-catenin 信号通路的LRP6和Cyclin D1调控细胞的增殖和凋亡,从而决定细胞的命运[26];miR-202 在雄性小鼠精原干细胞中是细胞周期的负调控因子,敲除miR-202 能够加速细胞周期进程,引起分化后的精原细胞大幅度凋亡[27]。ssc-miR-1296-5p 位于猪miR-1296 的5’臂,可通过靶向DDIT4基因作用于mTOR 和PI3K-Akt 信号通路[28]。ssc-miR-361-5p 位于ssc-miR-361 的5’臂,可通过靶向信号传感器和转录激活因子6(STAT6)在前列腺癌中发挥抑癌作用[29]。这4 个miRNAs 的发现为BMIAURKC基因进一步的功能研究指明了方向。

