云南沟谷雨林建群种绒毛番龙眼居群的遗传多样性
陈少瑜 李江 陈伟 冯弦




摘要:【目的】了解云南特有的溝谷雨林主要建群种绒毛番龙眼居群的遗传多样性及遗传结构特征,为雨林植被恢复的采种规划和恢复效果评估提供重要参考。【方法】采用SSR分子标记技术,检测分析云南省西双版纳州和普洱市思茅区的绒毛番龙眼3个天然居群和1个人工居群共82个样本的SSR多态性,采用POPGENE v.1.32、GenAlEx 6.501和STRUCTURE 2.3.3等进行遗传多样性及遗传结构分析。【结果】19对SSR引物共检测到88个等位基因,每对引物平均4.632个,19个位点的Shannon’s信息指数(I)变化范围为0.264~1.687,平均为0.879;多态信息指数(PIC)变化范围为0.128~0.729,平均为0.422,期望杂合度(He)的变化范围为0.138~0.765,平均为0.473;4个居群I和He的均值分别为0.785,0.437。19个位点的遗传分化系数(F)均值为0.069,基因流(Nm)的均值为3.363。STRUCTURE分析将绒毛番龙眼4个居群82个样本分为4个类群,基于遗传距离的UPGMA聚类显示4个居群聚为2个分支,菜阳河天然林居群、普文人工林居群和普文天然林居群聚为一支,勐养镇天然林居群单独为另一支。【结论】绒毛番龙眼的遗传多样性处于中等水平;居群间的遗传分化较低,遗传变异主要来源于居群内;建议特别关注居群内个体的选择和保护,在沟谷雨林植被恢复中设定天然居群多样性水平的恢复目标,并通过科学的采种规划实现这一目标。
关键词: 绒毛番龙眼;SSR分子标记;珍稀濒危树种;沟谷雨林建群种;遗传多样性
中图分类号: S718 文献标志码: A 文章编号:2095-1191(2022)03-0850-09
Genetic diversity of Pometia tomentosa, a major constructive species of valley rainforest in Yunnan
CHEN Shao-yu LI Jiang CHEN Wei FENG Xian
(1Yunnan Academy of Forestry and Grassland/Laboratory for Conservation and Breeding for Rare, Endangered and Endemic Forest Plants of State Forestry Administration/Yunnan Provincial Key Laboratory for Cultivation and Utilization of Forest Plants, Kunming, Yunnan 650201, China; 2Institute of Forest Research,Yunnan Academy of Forestry and Grassland, Kunming, Yunnan 650201, China; 3Institute of Forest industry, Yunnan Academy of Forestry and
Grassland, Kunming, Yunnan 650201, China)
Abstract:【Objective】Pometia tomentosa is a rare and precious local tree species in Yunnan Province of China and it is also one of the major constructive species for valley rainforests. Understanding the genetic diversity and genetic structure of Pometia tomentosa could provide a guide for a seed collecting strategy to effect vegetation restoration in valley rainforests. 【Method】Four populations with a total of 82 samples of Pometia tomentosa from the Xishuangbanna prefecture were studied. Simple sequence repeat(SSR) molecular technique was used to detect polymorphism of allelic fragments. POPGENE, version, 1.32 GenAIEx 6.501 and STRUCTURE 2.3.3 were used to analyze the genetic diversity and genetic structure of populations. 【Result】Eighty-eight alleles were detected by 19 pairs of SSR primers with an average of 4.632 per pair. The range of Shannon’s information index(I) was 0.264-1.687 with a mean of 0.879. Polymorphism information content (PIC) varied from 0.128 to 0.729 with a mean of 0.422, while expected heterozygosity (He) varied from 0.138 to 0.765 with a mean of 0.473. The average values of I and He for the 4 populations were 0.785 and 0.437 respectively. The average of genetic differentiation (F) was 0.069 and gene flow (Nm) was 3.363,which means a relatively low genetic differentiation among the populations. The 4 populations were divided into 4 groups based on STRUCTURE analysis. UPGMA clustering indicated that the 4 populations were clustered into 2 branches, one of which consisted of a natural population in Caiyanghe, an artificial population in Puwen and a natural population in Puwen. The other branch contained only a natural population in Mengyang. 【Conclusion】 The genetic diversity of Pometia tomentosa is only medium at both the species and the population levels. Genetic variation comes mainly from within populations, so individuals within populations should be targeted for selection and conservation. In the recovery of valley rainforests, the genetic diversity of natural populations could be set as a target, which can be achieved by scientific planning of the seed collection.
Key words: Pometia tomentosa; SSR molecular markers; rare and precious species; constructive species for valley rainforest; genetic diversity
Foundation items: Central Financial Forestry Science and Technology Promotion Demonstration Project(Yun 2021-TG02);Team Building Project of Yunnan Academy of Forestry and Grassland Sciences(LKYTD-2020-003)
0 引言
【研究意义】绒毛番龙眼[Pometia tomentosa(Bl.) Teysm. et Binn]为无患子科(Sapindaceae)番龙眼属(Pometia)常绿大乔木,濒危种,国家三级保护植物,在我国主要分布于云南西南部的永德和沧源、南部的景洪、思茅、勐腊和金平以及东南部的麻栗坡等地区海拔475~1500 m的沟谷季雨林中,为云南南部季节性雨林的主要建群树种。绒毛番龙眼树体高大、生长速度快,材质优良,抗腐抗虫能力强,成为云南特有的优质珍贵乡土用材树种,但由于生境的破坏、人为的滥砍乱伐已导致该物种的濒危(文彬等,2002)。群体遗传多样性和遗传结构的研究对物种科学保护及合理利用具有重要的理论和实践意义(Wuyun et al.,2015),对制定诸如优先保护种群的确定、有效的取样策略等物种保护策略及近交衰退种群的恢复策略起重要作用(Millar and Westfall,1992),也可为揭示物种演化历史和进化潜力,探讨物种濒危原因提供科学参考(Hedrick,2004)。由此,开展绒毛番龙眼居群遗传多样性研究将为其合理保护及植被恢复中采种策略的制定、效果评价提供科学依据。【前人研究进展】对绒毛番龙眼的研究仅见种子萌发(文彬等,2002;闫兴富和曹敏,2008a)、育苗技术(邱琼等,2014,2015)、生理生态(闫兴富和曹敏,2007,2008b;于洋等,2007)及植被恢复(段宗亮等,2012)等方面的报道,遗传多样性研究尚为空白。尽管基于分子标记的绒毛番龙眼遗传多样性研究尚未见报道,但对于无患子科其他属植物的相关研究有较多报道。刁松锋等(2016)利用12条ISSR引物分析了无患子18个天然居群共265单株的遗传多样性,表明无患子以自交为主,其天然居群遗传多样性丰富,居群内的遗传多样性高于居群间;姜翠翠等(2016)应用ISSR分析了来自5个不同种源地的73份无患子种质的遗传多样性,结果显示其遗传多样性较为丰富,各种源地种质间的遗传差异较大;李冬波等(2020)利用40对SSR引物对广西原产和引种的88份荔枝种质资源进行遗传多样性和聚类分析,结果表明SSR分子标记是一种适用于荔枝资源遗传多样性分析的理想分子标记,广西荔枝种质资源遗传背景相对较窄,遗传多样性较低;胡福初等(2021)利用SRAP分子标记对30份早熟荔枝种质资源进行多样性分析及聚类,结果表明30份资源具有较为丰富的遗传多样性,大部分特早熟资源聚在一起。关于龙眼属种质的遗传多样性已有较多报道(陈虎等,2012;郑姗等,2016)。胡文舜等(2019)基于龙眼品种香脆果实的转录组测序数据库,开发出50对龙眼EST-SSR多态性引物,在无患子属、韶子属、荔枝属和龙荔属等8份近缘属材料上进行通用性鉴定,并用之进行了无患子科5个属55份种质的遗传多样性分析,结果表明LYP1-3、LYP2-8和LYP2-11等13对引物在全部5个属上均有扩增条带,具有共同通用性,表明龙眼EST-SSR引物在无患子科植物中具有良好的属间通用性;同时,UPGMA聚类分析将55份材料在遗传相似系数0.561处明显地划分为龙眼、龙荔、荔枝、无患子和红毛丹5个大类。然而,本课题组将上述龙眼开发出的50对引物用于绒毛番龙眼的SSR扩增,却未得到理想的扩增效果。【本研究切入点】鉴于绒毛番龙眼所处的濒危状态、特殊的生态地位及重要的经济价值,尽管已有育苗和生理生态方面的研究报道,但至今却没有对之进行基于分子标记的遗传多样性分析,不了解其遗传多样性状况,更是缺乏在此基础上的合理保护和科学利用。【拟解决的关键问题】在绒毛番龙眼转录组测序及引物开发的基础上,利用SSR标记技术对绒毛番龙眼4个居群82份资源进行遗传多样性及聚类分析,揭示绒毛番龙眼居群的遗传多样性状况及遗传结构特征,为其保护及植被恢复中采种策略的制定、效果评价提供参考。
1 材料与方法
1. 1 试验材料
于2020年5月从西双版纳傣族自治州普洱市思茅区采集绒毛番龙眼4个居群共82单株的叶样。各居群的名称、样本数量及地理信息见表1。将野外分单株采集的叶片置样品袋中,放入装有硅胶的密封盒中干燥,备用。
1. 2 试验方法
1. 2. 1 基因组DNA提取 用TSINGKE植物DNA提取试剂盒(擎科生物技术有限公司)提取绒毛番龙眼叶片的基因组DNA,具体步骤参照试剂盒说明书。
1. 2. 2 SSR引物的筛选 对绒毛番龙眼的转录组进行测序,根据转录组测序的生物信息学分析得到的SSR位点引物信息,按照SSR引物筛选原则初步选择并合成200对引物,分别以4个居群随机抽取的3个DNA样本为模板进行PCR扩增。用最后扩增良好的荧光PCR产物进行3730xl测序仪检测,采用GeneMapper V4.1对检测到的数据进行分析,筛选出的具有多态性的引物用以所有样本的扩增。
1. 2. 3 扩增体系及扩增程序 按照表2的反应体系进行SSR-PCR扩增,扩增程序如下:94 ℃预变性5 min;94 ℃ 5 min,60 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃延伸10 min,4 ℃保存。扩增产物(2 µL)与6 µL溴酚蓝混匀进行琼脂糖电泳(300 V,12 min),得到電泳胶图,由胶图判断模板浓度,基于模板浓度将扩增产物用水稀释至毛细管电泳所需浓度。
1. 2. 4 毛细管电泳检测 按130∶1的比例将HiDi与GS500的内标混合成mix,将mix以每孔10.0 µL分装于国产96孔反应板,之后按编号在96孔板中依次加入0.5 µL检测样品,离心至4000 r/min即停。将孔板在金属浴加热器上95 ℃预变性5 min,取出立即放入-20 ℃冷却,取出后4000 r/min离心,混匀,于3730测序仪上进行毛细管电泳,获得电泳图谱。
1. 3 遗传分析方法
1. 3. 1 数据矫正及整理 以条带大小(bp)的方式统计每个个体的扩增位点,根据GeneMapper V4.1分析得到的峰图,以信号值大于400,无杂峰干扰,且同一位点的峰形相似进行位点统计,建立原始数据矩阵。
1. 3. 2 遗传多样性分析 用POPGENE v.1.32和GenAlEx 6.501(Peakall and Smouse,2012),分别计算SSR位点和居群的遗传多样性指标,包括观测等位基因(Na)、有效等位基因(Ne)、Shannon’s信息指数(I)、多态信息指数(PIC)、观测杂合度(Ho)、期望杂合度(He)。
1. 3. 3 遗传分化分析 用GenAlEx 6.501计算各位点的近交系数(FIS)、基因流(Nm)及居群间的遗传分化系数(FST)。
1. 3. 4 遗传结构分析 用STRUCTURE 2.3.3(Pritc-hard et al.,2000)的贝叶斯聚类法分析群体的遗传结果。用马尔科夫链(Markov chain monte carlo,MCMC)方法预设群体分组(K),K值设置为2~10,每个K值重复运行20次,每个循环的重复抽样次数设置为100000次,采用Evanno等(2005)的方法计算最适K值,结果通过STRUCTURE HARVESTER方法(Earl and Vonholdt,2012)在线计算(http://taylor0.biology.ucla. Edu/STRucture Harvester/),用Distruct(Rosenberg et al.,2002)绘制群体遗传结构图。
1. 3. 5 聚类分析 基于Nei’s遗传距离,采用NTSYS 2.10对4个居群进行非加权成组配对算数平均法(Unweighted pair-group method with arithmetic means,UPGMA)的聚类分析。
2 结果与分析
2. 1 EST-SSR引物及SSR位点的多态性分析结果
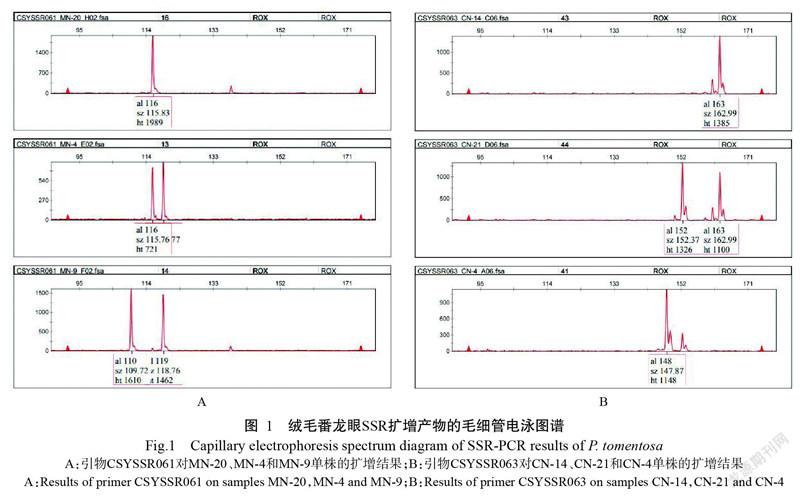
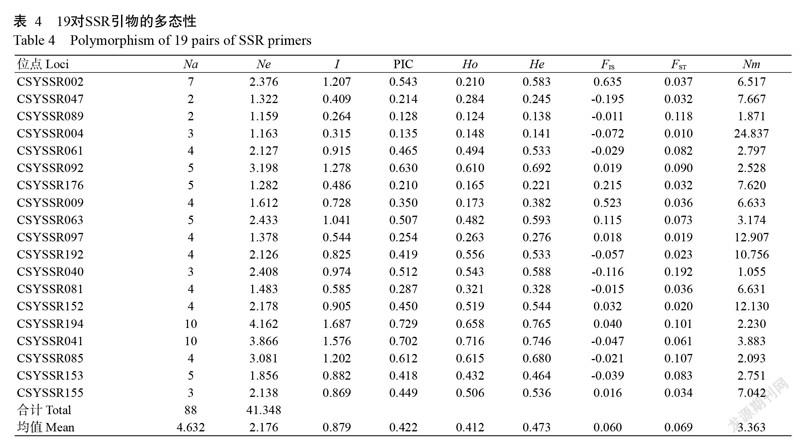
基于绒毛番龙眼转录组测序及序列信息分析合成200对引物,经初选和复选确定稳定性高及多态性强的19对引物(表3)对所有样本进行SSR扩增。图1为引物CSYSSR061和CSYSSR063分别对6个单株样品扩增结果的毛细管电泳图谱,图谱显示了扩增出的位点及片段大小。19对引物对4个居群82份样本进行SSR扩增,共检测到88个等位基因,Na为4.632个。每对引物检测到的等位基因数2~10个,其中,引物CSYSSR194和CSYSSR041检测到的等位基因数最多,各10个,而引物CSYSSR047和CSYSSR089则均只检测到2个。Ne总数为41.348,数值范围为1.159(CSYSSR089)~4.162(CSYSSR194),Ne的均值为2.176个(表4)。
由表4还可看出,19个位点PIC变化范围为0.128~0.729,均值为0.422,其中位点CSYSSR194和CSYSSR041的PIC最高,分别达0.729和0.702,位点CSYSSR089显示的多态信息最低,其PIC为0.128。19个位点中有15个位点的PIC>0.250,具有较高的多态信息,占总数的78.9%。19个位点的I变化范围为0.264~1.687,均值为0.879;Ho和He的变化范围分别为0.124~0.716和0.138~0.765,均值分别为0.412和0.473。位点的杂合度可反映群体的遗传多样性,当观测杂合度小于期望杂合度时(Ho<He),说明分析群体内存在一定的近亲交配,物种缺乏足够的杂合子,本研究中,Ho<He表明绒毛番龙眼群体内存在近亲交配,导致杂合子不足。
2. 2 绒毛番龙眼居群的遗传多样性及遗传分化分析结果
绒毛番龙眼4个居群(PN,CN,MN和PA)的遗传多样性分析结果见表5。I和He可很好地反映居群的遗传多样性水平。由表5可看出,4个居群I和He的变化范围分别为0.752~0.823及0.421~0.455,均值分别为0.785和0.437;各居群间的I和He,即遗传多样性水平存在差异,其中CN居群的遗传多样性相对较高(I=0.823,He=0.455),而PN居群的遗传多样性水平则相对较低(I=0.752,He=0.0.421)。4个居群的Ho均值为0.410,小于He的均值0.437,总体上看,群体缺乏杂合子,而分别比较各居群的Ho和He可见,4个居群中只有PA居群的Ho(0.455)大于其He(0.444),说明PA居群内杂合子占有较高的比例。
绒毛番龙眼4个居群的群体内FIS的变化范围为-0.055~0.109,均值为0.049,其中只有PA居群的FIS(-0.055)小于0,其他3个居群的F均大于0,19个位点的F均值为0.060,F均值为0.069(表4)。F统计量是反映居群间遗传分化和居群遗传结构的重要指标,其中FIS常用以衡量居群内偏离哈迪—温伯格平衡的程度,当F>0时,居群内发生近亲交配,杂合子不足,反之,则说明居群内为异型交配,杂合子过量。本研究19个位点F的均值大于0,在种的水平上,绒毛番龙眼群体内存在近亲交配,杂合子不足,但其中有10个位点小于0,说明这10个位点上杂合子过剩;在居群水平上,4个居群中只有PA居群的F小于0,PA居群内行异型交配,杂合子过剩,其他3个居群内存在近亲交配。F可很好地反映居群间遗传分化状况,当F≤0.15时,居群间的遗传分化较低,绒毛番龙眼4个居群F均值为0.069,說明绒毛番龙眼居群间的遗传分化较低,遗传变异主要来源于群体内的个体,只有6.9%的遗传变异来源于居群间。
2. 3 绒毛番龙眼居群的遗传结构特征
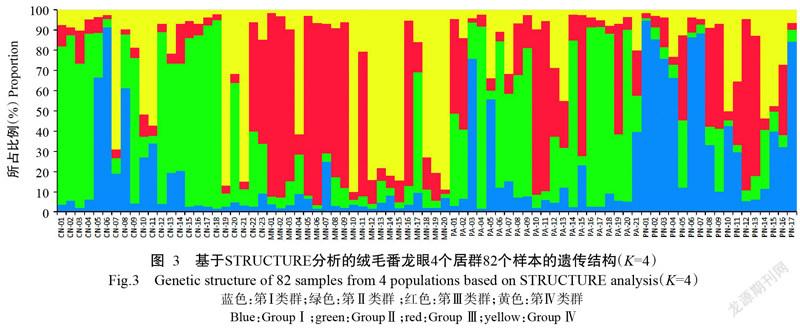
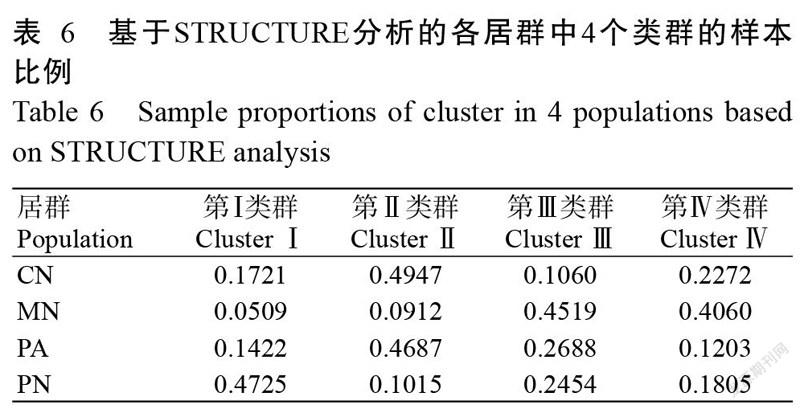
通过STRUCTURE 2.3.3的贝叶斯聚类法对绒毛番龙眼进行居群的遗传结构分析,得到当K=4时ΔK值最大(图2),表明4个居群82个样本中存在4个不同类群(图3),其中,第Ⅰ类群主要包含PN和CN居群的个体,第Ⅱ类群主要包含CN和PA居群的个体,第Ⅲ类群主要包含MN和PA居群的个体,第Ⅳ类群则主要包含MN和CN居群的个体。表6给出了各居群中4个类群的样本比例,当K=4时,CN居群样本的基因组成主要来源于第Ⅱ类群(49.47%)和第Ⅳ类群(22.72%),MN基因组成主要来源于第Ⅲ类群(45.19%)和第Ⅳ类群(40.60%),PA基因组成主要来源于第Ⅱ类群(46.87%)和第Ⅲ类群(26.88%),PN的基因组成主要来源于第Ⅰ类群(47.25%)和第Ⅲ类群(24.54%),可见,4个居群是非均质的,存在一定的基因交流现象,但仍具有明显的遗传结构。从各居群包含的基因池组分来看,CN和PA居群具有更为相似的遗传组分,二者的基因组成近50.00%来源于第Ⅱ类群。
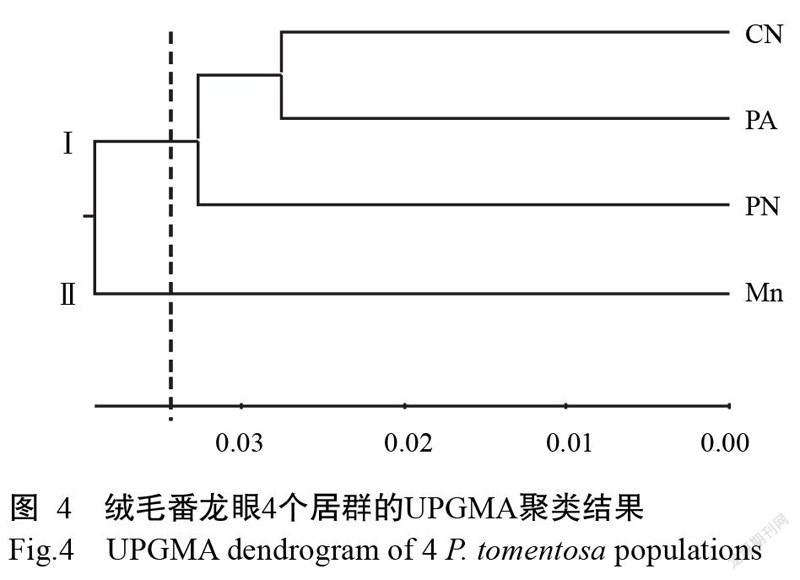
2. 4 绒毛番龙眼4个居群的遗传关系及UPGMA聚类
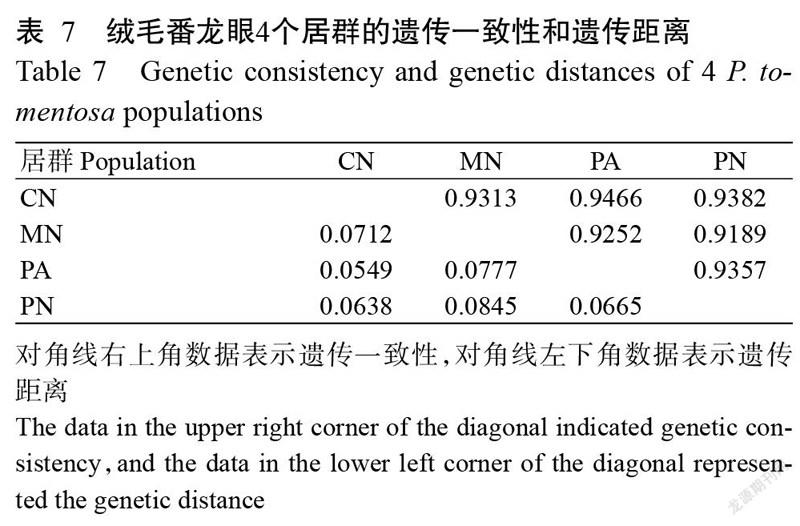
为进一步了解居群间的遗传关系,利用POPGENE v.1.32对居群间的遗传距离进行计算,结果显示,4个居群中CN与PA间遗传距离最小,为0.0549,MN与PN间遗传距离最大,为0.0845(表7)。基于Nei’s遗传距离进行4个居群的UPGMA聚类,聚类结果显示在遗传距离为0.035处4个居群聚成2组,组Ⅰ中,CN与PA聚为一个亚组,PN单独为一个亚组,组Ⅱ只包含MN一个居群(图4)。可见,CN与PA居群的遗传相似性较高,而MN与其他3个居群的遗传相似性均较低。
3 讨论
PIC是遗传多样性研究中常用的一个指标,用以评估引物的鉴别能力,反映引物提供信息的可靠性(Barkley et al.,2006;Uzun et al.,2011)。通常,当PIC >0.50时,引物的多态性高,提供的信息量丰富,可很好地反映遗传多样性;当0.25<PIC<0.50时,引物的多態性及信息量较高,可提供较合理的信息;当PIC<0.25时,引物的多态性低,提供的信息量较少(Wang et al.,2014;李静等,2020)。本研究中使用的19对引物PIC均值为0.422,其中15对引物的PIC >0.25,占总数的78.9%,因此,研究采用的引物多态性较高,可为揭示绒毛番龙眼的遗传多样性提供丰富而有效的信息。
I 和He是衡量种质资源遗传多样性的重要参考指标(Wang et al.,2015)。无患子科其他树种已有一些遗传多样性相关报道,无患子18个天然居群265个样本的I值分别为0.2569(物种水平)和0.1998(居群水平),He分别为0.3909(物种水平)和0.2980(居群水平)(刁松锋等,2016),46份荔枝种质资源的He为0.355(曾淇等,2010),我国96份荔枝种质资源的I和He分别为0.425和0.270(向旭等,2010);Nybom(2004)总结了国内外利用SSR分子标记研究长寿命、多年生、远交、风力传播种子的林木群体的He均值,为0.647;王滑(2010)对具有相同生活史的核桃(Juglans regia)、深纹核桃(J. sigillata)研究结果显示,二者He分别为0.605和0.678。本研究中4个绒毛番龙眼居群I=0.785,He=0.437,比较采用相同分子标记的上述相关研究结果,绒毛番龙眼的遗传多样性高于无患子科其他树种,却低于林木群体的均值及具有相同生活史的核桃,可见绒毛番龙眼具有中等水平的遗传多样性。遗传多样性是物种生存和进化的基础,其水平的高低是物种的进化史、分布区、生物学特性、生境及人为活动等因素综合作用的结果(Souza et al.,2013)。通常,特有种、濒危种、分布范围狭窄、自交为主的物种其遗传多样性水平较低(Hamrick and Godt,1996),绒毛番龙眼属于特有种、濒危种,分布也相对狭窄,其遗传多样性水平却处于中等水平,究竟其原因应与其多年生、长寿命、虫媒传粉及异交为主的繁殖方式有关,长期的演化及异交积累了丰富的遗传变异。自然保护区的建立对于防止生境破坏和分布片段化导致的遗传多样性降低也起到了积极作用。本研究中CN居群位于菜阳河国家级自然保护区内,其遗传多样性在4个居群中最高便是一个很好的例证。另外,研究结果显示4个居群的遗传多样性由高至低依次为CN、PA、MN、PN,PA是人工居群,其遗传多样性并不低于天然居群(MN和PN)的遗传多样性,说明通过科学合理的构建绒毛番龙眼人工居群可维持其物种的遗传多样性。
绒毛番龙眼居群间的遗传分化较低,遗传变异主要来源于居群内的个体。植物居群的遗传结构受多个内外因素影响,其中繁育系统、基因流和演替阶段等显著影响居群的遗传分化,是影响遗传结构的最重要因素(葛颂,1994)。当基因流Nm>1时,说明居群间的基因流较大,能一定程度上降低遗传漂变产生的居群间遗传分化;当Nm<1时则说明居群间的基因流较小,居群间因遗传漂变及选择作用而产生分化(Hamrick et al.,1992)。本研究居群间的Nm= 3.363(Nm>1),较大的基因流有效地阻止了遗传漂变,从而降低了绒毛番龙眼居群间的遗传分化。绒毛番龙眼为异花传粉,昆虫是主要的传粉者,这样的传粉特性导致了其较高的遗传变异,且变异主要存在于居群内。另外,绒毛番龙眼的分布区域较小,昆虫利于长距离传粉及种子较易于散布等因素为居群间的基因交流提供了可能性。
基于遗传距离的UPGMA分析结果CN与PA居群间的遗传距离最小,二者首先聚为一个亚组,之后与PN居群聚为组Ⅰ,MN单独为组Ⅱ,说明CN与PA居群在遗传组成上更为相似,即遗传相似性更高,STRUCTURE分析也表明CN和PA居群样本的基因组成主要来源于第Ⅱ类群(分别为49.47%和46.87%),可推测PA居群的种质主要来源于CN居群。从地理距离上看,PN与CN居群的地理距离较近,二者与MN居群的地理距离较远,可见3个天然居群的遗传距离关系和地理距离关系相吻合,结果为资源保护策略及育种策略的制定提供有益参考。另外,尽管MN居群与其他3个居群的遗传距离较大,但4个居群的遗传组成为非均质,居群间存在基因交流。
4 结论
绒毛番龙眼的遗传多样性处于中等水平,居群间分化较小,其遗传变异主要来源于居群内。在迁地保护时应特别关注居群内个体的选择和保护;此外在沟谷雨林植被恢复中可设定近似天然林居群多样性水平的恢复目标,并通过科学的采种规划实现这一目标。
参考文献:
陈虎,何新华,黄桂香,李峰,姜建初,朱建华. 2012. 不同龙眼资源遗传多样性的SCoT和ISSR比较分析[J]. 广西植物,32(4):536-541. [Chen H,He X H,Huang G X,Li F,Jiang J C,Zhu J H. 2012. Comparison and analysis of SCoT and ISSR markers for genetic diversity of Longan[J]. Guihaia,32(4):536-541.] doi:10.3969/j.issn.1000-314.01.04.01.
刁松锋,邵文豪,陈涛,姜景民,段文彬. 2016. 无患子天然居群遗传多样性研究[J]. 林业科学研究,29(2):176-182. [Diao S F,Shao W H,Chen T,Jiang J M,Duan W B. 2016. Genetic diversity of Sapindus mukorossi natural populations in China based on ISSR[J]. Forest Research,29(2):176-182.] doi:10.13275/j.cnki.lykxyj.2016.02.004.
段宗亮,杨德军,邱琼,陈显兵,陈勇. 2012. 景洪市废弃石矿区人工植被恢复技术研究[J]. 水土保持应用技术,(5):23-24. [Duan Z L,Yang D J,Qiu Q,Chen X B,Chen Y. 2012. Study on artificial vegetation restoration technology in wasted stone mining area of Jinghong[J]. Technology of Soil and Water Conservation,(5):23-24.] doi:10.3969/ j.issn.1673-5366.2012.05.08.
葛頌. 1994. 酶电泳资料和系统与进化植物学研究综述[J]. 武汉植物学研究,12(1):71-84. [Ge S. 1994. Electrophoresis data and studies of plant systematics and evolution[J]. Journal of Wuhan Botanical Research,12(1):71-84.]
胡福初,吴小波,陈哲,吴凤芝,周文静,冯学杰,范鸿雁,周瑞云,王祥和. 2021. 基于SRAP分子标记的特早熟荔枝种质资源遗传多样性分析[J]. 热带作物学报,42(4):920-926. [Hu F C,Wu X B,Chen Z,Wu F Z,Zhou W J,Feng X J,Fan H Y,Zhou R Y,Wang X H. 2021. Genetic diversity analysis of very early maturing litchi germplasm resources based on SRAP molecular markers[J]. Chinese Journal of Tropical Crops,42(4):920-926.] doi:10.3969/j.issn.1000-2561.2021.04.002.
胡文舜,陈秀萍,郑少泉. 2019. 龙眼EST-SSR标记开发及无患子科5个属种质遗传多样性分析[J]. 园艺学报,46(7):1359-1372. [Hu W S,Chen X P,Zheng S Q. 2019. EST-SSR markers developed from Dimocarpus longan and their application in genetic diversity analysis of five genera of Sapindaceae[J]. Acta Horticulturae Sinica,46(7):1359-1372.] doi:10.16420/j.issn.0513-353x.2018-0753.
姜翠翠,卢新坤,叶新福. 2016. 能源植物无患子种质的分子遗传多样性分析[J]. 分子植物育种,14(10):2888-2895. [Jiang C C,Lu X K,Ye X F. 2016. Molecular genetic diversity analysis of energy plant Sapindus mukorossi germplasm resources[J]. Molecular Plant Breeding,14(10):2888-2895.] doi:10.13271/j.mpb.014.002888.
李冬波,徐宁,秦献泉,李鸿莉,侯延杰,邱宏业,张树伟,朱建华,彭宏祥. 2020. 广西原产和引种荔枝种质资源的遗传多样性分析及核心种质构建[J]. 南方农业学报,51(7):1537-1544. [Li D B,Xu N,Qin X Q,Li H L,Hou Y J,Qiu H Y,Zhang S W,Zhu J H,Peng H X. 2020. Genetic diversity and construction of core collections of litchi(Litchi chinensis Sonn.)germplasm originated and introduced in Guangxi[J]. Journal of Southern Agriculture,51(7):1537-1544.] doi:10.3969/j.issn.2095-1191.2020.07. 004.
李静,田思雨,毛威涛,姚国新. 2020. 基于SSR分子标记的红安县省沽油遗传多样性分析[J]. 河南农业科学,49(10):42-47. [Li J,Tian S Y,Mao W T,Yao G X. 2020. Genetic diversity of Staphylea bumalda DC in Hong’an County based on SSR markers[J]. Journal of Henan Agricultural Sciences,49(10):42-47.] doi:10.15933/j.cnki. 1004-3268.2020.10.006.
邱琼,姜远标,杨斌,杨德军,徐玉梅,刘庆云. 2014. 不同育苗基质对绒毛番龙眼幼苗生长的影响[J]. 种子,33(2):67-69. [Qiu Q,Jiang Y B,Yang B,Yang D J,Xu Y M,Liu Q Y. 2014. Effect of substrates on seedling growth of Pometia tomentosa[J]. Seed,33(2):67-69.] doi:10.3969/j.issn.1001-4705.2014.02.018.
邱瓊,裴艳辉,杨德军. 2015. 乡土珍贵用材树种绒毛番龙眼的育苗造林技术[J]. 四川林业科技,36(6):139-141. [Qiu Q,Pei Y H,Yang D J. 2015. Seedling raising and afforestation techniques of precious timber tree species Pometia tomentosa[J]. Journal of Sichuan Forestry Scien-ce and Technology,36(6):139-141.] doi:10.16779/j.cnki. 1003-5508.2015.06.030.
王滑. 2010. 西藏核桃种质资源遗传多样性研究[D]. 北京:中国林业科学研究院. [Wang H. 2010. Genetic diversity of germplasm resources on walnut in Tibet region[D]. Beijing:Chinese Academy of Forestry.]
文彬,殷寿华,兰芹英,杨湘云. 2002. 绒毛番龙眼种子萌发生态特性的研究[J]. 广西植物,22 (5):408-412. [Wen B,Yin S H,Lan Q Y,Yang X Y. 2002. Ecological characteri-stics of seed germination of Pometia tomentosa[J]. Guihaia,22(5):408-412.] doi:10.3969/j.issn.1000-3142.2002. 05.006.
向旭,欧良喜,陈厚彬,孙清明,陈洁珍,蔡长河,白丽军,赵俊生. 2010. 中国96个荔枝种质资源EST-SSR遗传多样性分析[J]. 基因组学与应用生物学,29(6):1082-1092. [Xiang X,Ou L X,Chen H B,Sun Q M,Chen J Z,Cai C H, Bai L J, Zhao J S. 2010. EST-SSR analysis of gene-tic diversity in 96 Litch(Litch chinensis Sonn.) germplasm resources in China[J]. Genomics and Applied Bio-logy,29(6):1082-1092.] doi:10.3969/gab.029.001082.
闫兴富,曹敏. 2007. 不同光照梯度的遮荫处理对绒毛番龙眼幼苗生长的影响[J]. 热带亚热带植物学报,15(6):465-472. [Yan X F,Cao M. 2007. Effects of shading treatment on the growth of Pometia tomentosa seedlings[J]. Journal of Tropical and Subtropical Botany,15(6):465-472.] doi:10.3969/j.issn.1005-3395.2007.06.001.
闫兴富,曹敏. 2008a. 光照对绒毛番龙眼种子萌发的影响[J]. 云南植物研究,30(2):183-189. [Yan X F,Cao M. 2008a. Effects of light treatment on the germination of Pometia tomentosa (Sapindaceae) seeds[J]. Acta Botanica Yunnanica,30(2):183-189.] doi:10.3969/j.issn.2095-0845.2008.02.009.
闫兴富,曹敏. 2008b. 林窗对热带雨林冠层树种绒毛番龙眼幼苗生长的影响[J]. 应用生态学报,19(2):238-244. [Yan X F,Cao M. 2008b. Effects of forest gap on the seedling growth of a canopy trees pecies Pometia tomentosa in tropical rainforest of Xishuangbanna[J]. Chinese Journal of Applied Ecology,19(2):238-244.]
于洋,曹敏,郑丽,盛才余. 2007. 光对热带雨林冠层树种绒毛番龙眼种子萌发及其幼苗早期建立的影响[J]. 植物生态学报,31(6):1028-1036. [Yu Y,Cao M,Zheng L,Sheng C Y. 2007. Effects of light on seed germination and seedling establishment of a tropical rainforest canopy tree,Pometia tomentosa[J]. Chinese Journal of Plant Eco-logy,31(6):1028-1036.] doi:10.17521/cjpe.2007.0130.
曾淇,李明芳,鄭学勤. 2010. 基于SSR标记的荔枝种质遗传多样性分析[J]. 植物遗传资源学报,11(3):298-304. [Zeng Q,Li M F,Zheng X Q. 2010. Analysis of genetic diversity within litchi varieties based on SSR markers[J]. Journal of Plant Genetic Resources,11(3):298-304.] doi:10.13430/j.cnki.jpgr.2010.03.023.
郑姗,张小艳,张立杰,谢丽雪,李韬. 2016. 40个龙眼品种(品系)DNA指纹图谱构建及遗传关系分析[J]. 中国农学通报,32(1):107-112. [Zheng S,Zhang X Y,Zhang L J,Xie L X,Li T. 2016. DNA fingerprinting of 40 Dimocarpus longan germplasms by ISSR markers and analysis of genetic relationship[J]. Chinese Agricultural Science Bulletin,32(1):107-112.] doi:10.19303/j.issn.1008-0384. 2012.12.008.
Barkley N A,Roose M L,Krueger R R,Federici C T. 2006. Assessing genetic diversity and population structure in a citrus germplasm collection utilizing simple sequence repeat markers(SSRs)[J]. Theoretical and Applied Gene-tics,112(8):1519-1531. doi:10.1007/s00122-006-0255-9.
Earl D A,Vonholdt B M. 2012. STRUCTURE HARVESTER:A website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conservation Genetics Resources,4(2):359-361. doi:10.1007/s12686-011-9548-7.
Evanno G,Regnaut S,Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:A simulation study[J]. Molecular Ecology,14(8):2611-2620. doi:10.1111/j.1365-294X.2005.02553.x.
Hamrick J L,Godt M J W,Sherman-Broyles S L. 1992. Gene flow among plant populations:Evidence from genetic markers[C]//Missouri Botanical Garden. 5th International Symposium of Experimental and Molecular Approaches to Plant Biosystematics:215-232.
Hamrick J L,Godt M J W. 1996. Effects of life history traits on genetic diversity in plant species[J]. Philosophical Transaction of the Royal Society,351(1345):1291-1298. doi:10.1098/rstb.1996.0112.
Hedrick P W. 2004. Recent developments in conservation genetics[J]. Forest Ecology and Management,197(1-3):3-19. doi:10.1016/j.foreco.2004.05.002.
Millar C I,Westfall R D. 1992. Allozyme markers in forest genetic conservation[J]. New Forests,6:347-371. doi:10.1023/A:1018330906758.
Nybom H. 2004. Comparison of different nuclear DNA mar-kers for estimating intraspecific genetic diversity in plants[J]. Molecular Ecology,13(5):1143-1155. doi:10.1111/j.1365-294X.2004.02141.x.
Peakall R,Smouse P E. 2012. GenALEx 6.5:Genetic analysis in excel. Population genetic software for teaching and research—An update[J]. Bioinformatics,28(19):2537-2539. doi:10.1093/bioinformatics/bts460.
Pritchard J K,Stephens M,Rosenberg N A,Donelly P. 2000. Association mapping in structured populations[J]. The American Journal of Human Genetics,37:170-181. doi:10.1086/302959.
Rosenberg N A,Pritchard J K,Weber J L,Cann H M,Kidd K K,Zhivotovsky L A,Feldman M W. 2002. Genetic structure of human populations[J]. Science,298(5602):2381-2385. doi:10.1126/science.1078311.
Souza I G B,Souza V A B,Lima P S C. 2013. Molecular characterization of Platonia insignis Mart.(“Bacurizeiro”) using inter simple sequence repeat(ISSR) markers[J]. Molecular Biology Reports,40(5):3835-3845. doi:10. 1007/s11033-012-2462-6.
Uzun A,Yesilogu T,Polat I,Aka-Kacar Y,Gulsen O,Yildirim B,Tuzcu O,Tepe S,Canan I,Anil S. 2011. Evaluation of genetic diversity in lemons and some of their relatives based on SRAP and SSR markers[J]. Plant Molecular Biology Reporter,29(3):693-701. doi:10.1007/s11105-010-0277-y.
Wang H,Pan G,Ma Q G,Zhang J P,Pei D. 2015. The genetic diversity and introgression of Juglans regia and Juglans sigillata in Tibet as revealed by SSR markers[J]. Tree Genetics & Genomes,11:804. doi:10.1007/s11295-014-0804-3.
Wang S Q,Liu Y,Ma L Y,Liu H B,Tang Y,Wu L P,Wang Z,Li Y Y,Wu R L,Pang X M. 2014. Isolation and characterization of microsatellite markers and analysis of genetic diversity in Chinese Jujube(Ziziphus jujube Mill.)[J]. PLoS One,9(6):e99842. doi:10.1371/journal.pone. 0099842.t002.
Wuyun T,Amo H,Xu J S,Ma T,Uematsu C,Katayama H. 2015. Population structure of and conservation strategies for wild Pyrus ussuriensis Maxim. in China[J]. PLos One,10(8):1-20. doi:10.1371/journal.pone.0133686.
(責任编辑 邓慧灵)

