基于cox1序列的中国6个花鲈野生群体遗传多样性
范嗣刚,黄 皓,2,王鹏飞,闫路路,赵 超,张 博,邱丽华,2
基于序列的中国6个花鲈野生群体遗传多样性
范嗣刚1,黄 皓1,2,王鹏飞1,闫路路1,赵 超1,张 博1,邱丽华1,2
(1. 中国水产科学研究院南海水产研究所 / 广东省渔业生态环境重点实验室 / 农业农村部水产品加工重点实验室,广东 广州 510300;2. 上海海洋大学水产与生命学院,上海 201306)
【】利用细胞色素C氧化酶1基因()序列片段研究中国6个花鲈()野生群体的遗传多样性和遗传结构。用PCR方法获得花鲈个体的序列,测序后,用MEGA X、DnaSP 6.12和Arlequin 3.01等软件进行数据分析。【】在6个花鲈群体229个样品共检测到48个多态性位点和50个单倍型。单倍型多样性为0.799 ~ 0.888,核苷酸多样性为0.001 6 ~ 0.002 7。6个花鲈群体遗传分化指数(st)为- 0.008 1 ~ 0.176 7。分子方差分析显示,遗传分化90.98%来自群体内,9.02%来自群体间。邻接系统进化树显示6个花鲈群体分为两大支。单倍型邻接树和单倍型网络关系图结果表明,未检测到有地理谱系结构的单倍型。中性检验显示,花鲈群体可能经历过种群扩张。
花鲈;序列;遗传多样性;遗传结构
花鲈()隶属于鲈形目(Perciformes)鮨科(Serranidae)花鲈属,俗称鲈鱼、七星鲈、花寨等[1],属广温、广盐性鱼类,广泛分布于中国、朝鲜半岛和日本沿海[2]。花鲈生长速度快,肉质鲜美,无肌间刺,营养价值和经济价值较高,是我国重要海水养殖鱼种。目前,我国花鲈产业繁育和养殖模式主要为北方培育和提供亲鱼,福建和浙江育苗,广东和福建养成,导致花鲈种质混乱[2-3],加上多年的人工繁殖和高强度捕捞,中国花鲈近亲繁殖加剧,种质退化严重,良种缺乏,急需开展遗传选育[3]。遗传多样性评估是遗传选育的重要环节,分析花鲈遗传多样性对花鲈遗传选育研究极为重要。
线粒体DNA(mtDNA)是一种核外遗传物质,有分子质量小、结构简单、进化速度快等特点,是评估群体遗传和生物进化的有效标记[4-7],已广泛应用于水生动物的群体遗传学研究[7-10]。已有少量基于线粒体序列的花鲈群体遗传学研究。Liu等[11]用细胞色素b(Cytochrome b,Cytb)基因序列分析了9个地域的花鲈遗传结构。刘明月等[12]用Cytb分析了我国6个沿海地区花鲈野生群体;Gong等[13]用线粒体基因组序列研究了中国沿海12个花鲈群体的遗传结构,认为存在两个地理群体;Wang等[14]用细胞色素c氧化酶亚基Ⅰ(Cytochrome c oxidase subunit I,cox1)基因部分序列研究了中国5个花鲈地理群体遗传多样性。除线粒体DNA方法外,还有基于SSR和基因组重测序等方法的研究[15-19]。但地理群体、采集时间、研究手段不同,花鲈遗传多样性会有差异。本研究利用线粒体基因序列,分析中国沿海6个花鲈群体遗传多样性,探讨其群体关系,为花鲈遗传选育研究提供参考依据。
1 材料和方法
1.1 样本采集与DNA提取
2020年在我国沿海6个地区海域捕获野生花鲈229尾,其中在天津(TJ)(118°0′E,38°50′N)、上海(SH)(122°29′E,31°17′N)、长岛(CD)(120°3′E,38°7′N)各采集39尾,青岛(QD)(120°30′E,35°58′N)38尾,厦门(XM)(118°14′E,24°21′N)40尾,北海(BH)(108°56′E,21°24′N)34尾。分别取每个个体的鳍条,用无水乙醇固定,运回实验室后于-20℃下保存。用海洋动物组织基因组DNA提取试剂盒 [天根生化科技(北京)有限公司,中国] 提取各样品DNA。用琼脂糖凝胶电泳和NanoDrop 2000(Thermo Scientific,美国)检测DNA的完整性和浓度。
1.2 PCR扩增与测序
根据花鲈线粒体基因组序列(KP408212.1),用Primer premier 5软件设计基因片段扩增引物:-F,ATATAGCGTTCCCTCGAATG;-R,AAAAGGATTACCGCTACCAG。PCR体系为20 μL,其中ExTaq酶(Takara,日本)0.2 μL,10×ExTaq Buffer(含Mg2+)2 μL,dNTP Mix 1.6 μL,正、反向引物(10 μmol/L)各0.8 μL,DNA(50 ng/μL)0.6 μL,去离子水14 μL。反应程序:95℃ 5 min;95℃30 s,52℃30 s,72℃ 60 s,30个循环;72℃7 min。用琼脂糖凝胶检测反应产物。用胶回收试剂盒(广州美基生物科技有限公司)回收目的片段,送至擎科生物技术有限公司进行双向测序。
1.3 数据处理分析
用MEGA X[20]比对cox1序列,用DnaSP 6.12[21]软件计算多态位点数、单倍型数量及单倍型多态性(Haplotype diversity,d)、核苷酸多态性(Nucleotide diversity,)和Tajima’s检测。用MEGA X[20]的Maximum Composite Likelihood模型计算单倍型间的遗传距离,用邻接(Neighbor-Joining,NJ)法构建单倍型系统发育树。
用软件Arlequin 3.01中的分子方差分析(Analysis of molecular variance,AMOVA)计算群体间和群体内的差异和遗传分化指数(st)。根据公式m= (0.5/st)‒0.5计算基因流[22]。用PopArt 1.7[23]构建单倍型进化网络图。
2 结果与分析
2.1 序列特征与遗传多样性
花鲈229个个体基因部分序列长984 bp,含GC 49.1%。在这984个核苷酸位点中,有48个多态性位点(占4.88%),其中单突变点23个,简约信息位点25个。
共检测到50个单倍型(表1),其中Hap2、9、12为6个群体共有单倍型,Hap1为5个群体共有,Hap3、16为4个群体共有,Hap10、14、24为3个群体共有,Hap11、18、21、22、31为2个群体共有。其余28个单倍型为各群体独有,上海群体独有的单倍型最多(9个),其次为青岛群体(7个),厦门群体独有的单倍型最少(1个)。单倍型Hap1被51个个体共享,包含个体数最多;其次为Hap12,被45个个体共享;Hap9被25个个体共享。所有的单倍型序列已递交到GenBank数据库,登记号为OK274345-OK274394。

表1 花鲈6个野生群体cox1序列的单倍型的分布情况
6个花鲈群体序列d为0.799 ~ 0.888,其中厦门群体最高,长岛群体最低(表2)。6个花鲈群体的为0.001 6 ~ 0.002 7,其中北海群体最高,青岛群体最低。整体上,6个花鲈群体的d为0.892,为0.002 3。各群体的Tajima’s均为负值(表2)。

表2 6个花鲈群体的遗传多样性参数
说明:“*”表示有显著性差异(0.05)
Note: “*”means significant difference(0.05)
2.2 种群遗传结果
花鲈6群体间遗传分化系数st为-0.008 1 ~ 0.176 7(表3),仅天津群体与长岛群体间st值为负,其余群体间st均为正值。天津群体与上海群体的st值最大(0.176 7)。除天津群体与长岛群体,上海群体与青岛群体的st无显著差异外,其余群体间st均有显著差异(0.05)。花鲈6群体间基因流m为-62.228 4 ~ 999.500 0(表3)。

表3 6个花鲈群体间遗传分化系数Fst(对角线下)和基因流Nm(对角线上)
说明:“*” 表示有显著差异(0.05)
Note: “*”means significant difference(0.05)
AMOVA结果(表4)显示,6群体的总st为0.090 2,差异不显著。其中90.98%的遗传变异来自群体内,仅有9.02%的遗传变异来自群体间。

表4 6个花鲈群体遗传变异的分子方差分析
2.3 单倍型系统进化树与网络图
50个单倍型的NJ系统进化树(图1)表明,单倍型无明显的谱系分化,6个群体所有单倍型无规律地均匀分布,未发现与地理分布相对应的分支。
部分序列的单倍型网络关系(图2)显示,Hap12处于网络图的中心位置,可能是最原始的单倍型,其次为Hap1和Hap9。各群体的单倍型在网络中散乱混杂分布,单倍型的进化关系与地理分布无明显联系,与单倍型系统进化树结果相符。

只显示Bootstraps > 50%的点
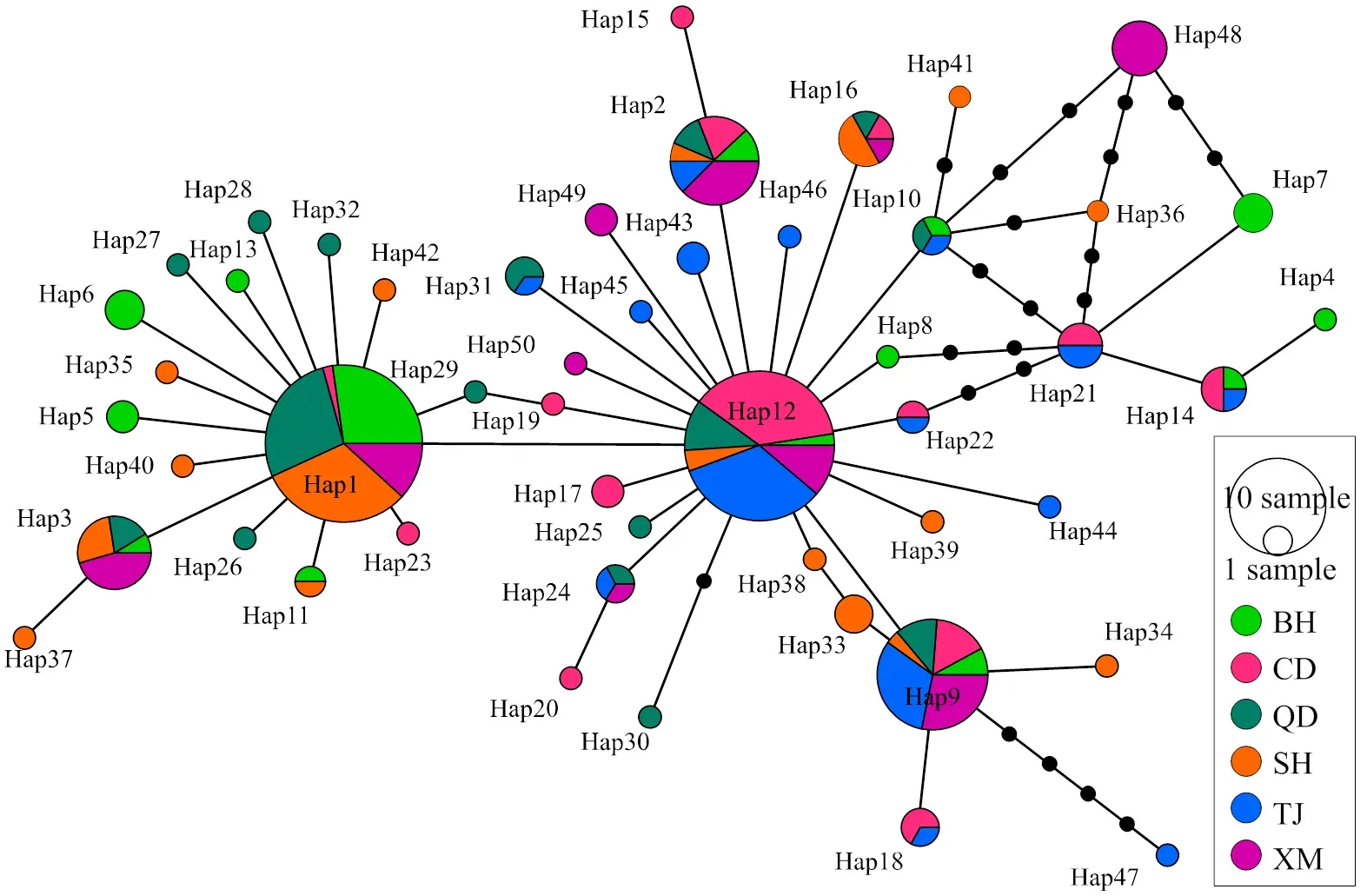
图2花鲈cox1基因序列的单倍型网络
3 讨论
遗传多样性是物种生存、进化和适应环境的基础,物种遗传多样性高则适应环境能力强,进化潜力大。序列遗传多态性较高,已用于各种鱼类遗传多样性研究[9,12-13]。本研究获得花鲈基因的部分序列,长984 bp,其中多态性位点有48个(占4.88%)。Wang等[14]研究的花鲈群体基因部分序列长586 bp,多态性位点21个(占3.58%),单倍型20个,与之比对,本研究序列仅有339 bp为同一段序列。因此本研究有645 bp的cox1序列是首次用于花鲈的群体遗传多样性分析。另外,本研究的多态性位点数量和比例均高于Wang等[13],说明多态位点在整个序列中均有分布。本研究序列的单倍型是Wang等[13]的2倍,与本研究选择的序列更长有关。
单倍型多样性和核苷酸多样性是反应遗传多样性高低的重要指标。本研究中,花鲈各群体d总体为0.892,为0.002 3(表3),与Wang等[14]的结果相似,均为高单倍型多样性(d> 0.5),低核苷酸多样性(< 0.005)。这种结果也在其他鱼类、甲壳类和海参中检测到[18,24-27],暗示花鲈种群近期可能经历了快速扩张事件,随着种群数量的增加,单倍型多样性显著提高,但无时间使核苷酸发生变异[28],因此核苷酸多样度较低。这在中性测试结果中也得到证实:6个中国花鲈群体的Tajima’s均为负值,花鲈群体可能经历过种群扩张。
Liu等[11]的基于线粒体Cytb基因部分序列的中国花鲈6野生群体遗传多样性结果表明,d为0.96 ~ 1.00,为0.008 ~ 0.015,均高于本研究和Wang等[14]的结果,说明20多年来,花鲈野生群体遗传多样性是下降的,原因可能是野生个体被大量捕捞。在韩国也发现,近20 a花鲈野生群体的遗传多样性有所下降[25]。另外,本研究的6个花鲈野生群体与Wang等[14]的5个花鲈野生群体有相同或相近的采样地点:均采集了青岛群体和上海群体,另外采集点北海和防城港相邻,厦门和列屿相邻。经比较,本研究的群体遗传多样性均高于Wang等[14]的结果,如本研究青岛群体d为0.848,上海群体d为0.825;在Wang等[14]的青岛群体d为0.572,上海崇明群体d为0.656。这可能是由所采集样品不同所致。另外,随着花鲈的人工繁育、养殖技术和养殖模式日趋成熟,已能满足人们的消费需求,缓解了花鲈野生群体的捕捞。
本研究的3个北方群体(天津、青岛和长岛)的值均较低(其中青岛群体的最低),低于其他3个南方群体(上海、厦门和北海),Wang等[14]也发现青岛群体的值最低。这可能说明花鲈在北方被大量捕捞。花鲈在中国北方海域比在南方海域生长性和环境适应性更佳[29]。因此大量的野生花鲈个体被捕捞并运送到中国南方地区甚至到日本、韩国等国家[29],从而导致北方的花鲈群体种质资源受到严重破坏。因此长期的过度捕捞使北方的花鲈群体经历了瓶颈事件,使值降低。
st是衡量群体间遗传分化程度的重要指标。根据Wright[30]研究,st为0~0.05,群体间遗传分化很小,可忽略;st为0.05~0.15,群体间存在中等程度的遗传分化;st为0.15~0.25,群体间遗传分化较大;st>0.25时,群体间存在明显的遗传分化,st为负值,说明群体间无遗传分化,基因交流非常频繁。本研究中,中国沿海花鲈群体总体遗传分化指数为0.090 2,表明中国花鲈群体遗传分化水平总体处于中等程度。在6个花鲈群体间,天津群体与上海群体、青岛群体、北海群体的遗传分化程度较大(0.15 本研究发现,中国沿海6花鲈群体的遗传变异主要来自群体内(90.98%)。Liu等[11]的基于线粒体Cytb和控制区序列的花鲈群体遗传变异有93.72%来自群体内。王桂兴等[17]用花鲈微卫星标记研究中国6个花鲈群体的遗传多样性,也证实中国花鲈群体有91.96%遗传变异来自群体内。这些结果均表明中国沿海花鲈的遗传变异主要来自群体内。线粒体DNA单倍型是研究物种群体遗传变化程度的一个重要参数。本研究共检测到50个单倍型,所有单倍型无规则地分布在整个系统进化树和网络图中,未形成与地理分布相符合的谱系结构,说明目前中国花鲈群体的遗传分化程度不高。Hap12和Hap1为主要单倍型,被多个群体共享且分布频率高,在单倍型网络图中处于中心位置,可能是花鲈群体祖先单倍型,是对外界环境适应能力强且能在花鲈群体中稳定存在的优势单倍型。Hap12、Hap1通过数个碱基的替换演变成其他单倍型。Wang等[14]也发现可能有两种祖先单倍型。同时,花鲈各群体均有特有单倍型,表明花鲈群体的遗传多样性丰富。在花鲈种质资源管理中,需重视特有单倍型的保护,使其保持多样化。 [1] 苏跃鹏. 海鲈养殖新技术[M]. 北京: 中国农业出版社, 2014: 1-6. [2] 温海深, 李昀, 张美昭, 等. 花鲈: 家喻户晓的中国海鲈[J]. 中国水产, 2020(8): 110-111. [3] 温海深, 于朋, 李昀, 等. 花鲈南北方养殖群体的形态学比较研究[J]. 中国水产, 2020(4): 81-84. [4] 张顺, 廖健, 郭昱嵩, 等. 基于线粒体cox1基因序列的弹涂鱼类系统进化关系[J]. 广东海洋大学学报, 2017, 37(1): 21-27. [5] 廖健, 张顺, 龙水生, 等. 5种虾虎鱼类线粒体COI基因序列变异及系统进化[J]. 广东海洋大学学报, 2016, 36(1): 7-12. [6] 刘楚吾, 黎锦明, 彭银辉, 等. 星洲银罗非鱼线粒体DNA的RFLP研究[J]. 广东海洋大学学报, 2008, 28(3): 16-20. [7] 彭敏, 韩耀全, 王大鹏, 等. 基于线粒体Cytb基因序列的西江流域广西境内卷口鱼遗传多样性分析[J]. 南方水产科学, 2020, 16(5): 10-18. [8] 李敏, 黄梓荣, 许友伟, 等. 基于线粒体cytb序列的花斑蛇鲻种群遗传结构研究[J]. 南方水产科学, 2019, 15(6): 41-48. [9] 匡天旭, 帅方敏, 陈蔚涛, 等. 西江鲫的遗传多样性与群体结构[J]. 南方水产科学, 2018, 14(5): 29-35. [10] 杨喜书, 章群, 余帆洋, 等. 华南6水系与澜沧江-湄公河攀鲈线粒体ND2基因的遗传多样性分析[J]. 南方水产科学, 2017, 13(3): 43-50. [11] LIU J X, GAO T X, YOKOGAWA K, et al. Differential population structuring and demographic history of two closely related fish species, Japanese sea bass () and spotted sea bass () in Northwestern Pacific[J]. Molecular Phylogenetics and Evolution, 2006, 39(3): 799-811. [12] 刘明月, 蒋琦辰, 杨家新. 不同海域中国花鲈的细胞色素b序列的遗传分析[J]. 南京师大学报(自然科学版), 2010, 33(1): 102-106. [13] GONG J, CHEN B H, LI B J, et al. Genetic analysis of whole mitochondrial genome of(Perciformes: Moronidae) indicates the presence of two populations along the Chinese coast[J]. Zoologia, 2020, 37: 1-12. [14] WANG W, MA C Y, CHEN W, et al. Population genetic diversity of Chinese sea bass () from southeast coastal regions of China based on mitochondrialgene sequences[J]. Biochemical Systematics and Ecology, 2017, 71: 114-120. [15] AN H S, LEE J W, KIM H Y, et al. Genetic differences between wild and hatchery populations of Korean spotted sea bass () inferred from microsatellite markers[J]. Genes & Genomics, 2013, 35(5): 671-680. [16] 江鑫. 花鲈与日本鲈群体遗传结构与多样性研究[D]. 青岛: 中国海洋大学, 2009: 75-76. [17] 王桂兴, 侯吉伦, 任建功, 等. 中国沿海6个花鲈群体的遗传多样性分析[J]. 中国水产科学, 2017, 24(2): 395-402. [18] WANG J, XUE D X, ZHANG B D, et al. Genome-wide SNP discovery, genotyping and their preliminary applications for population genetic inference in spotted sea bass ()[J]. PLoS One, 2016, 11(6): e0157809. [19] ZHAO Y F, PENG W Z, GUO H Y, et al. Population genomics reveals genetic divergence and adaptive differentiation of Chinese Sea bass ()[J]. Marine Biotechnology, 2018, 20(1): 45-59. [20] KUMAR S, STECHER G, LI M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. [21] ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution, 2017, 34(12): 3299-3302. [22] ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992, 9(3): 552-569. [23] LEIGH J W, BRYANT D. Popart: full-feature software for haplotype network construction[J]. Methods in Ecology and Evolution, 2015, 6(9): 1110-1116. [24] 冯梦霞, 汤永涛, 刘如垚, 等. 基于线粒体COI基因的河南境内四水系日本沼虾种群遗传多样性[J]. 中国水产科学, 2021, 28(12): 1548-1558. [25] 郜星晨, 章群, 薛丹, 等. 基于线粒体控制区部分序列的南海大斑石鲈遗传多样性分析[J]. 海洋科学, 2016, 40(7): 41-45. [26] ZHANG Y R, YANG F, WANG Z L, et al. Mitochondrial DNA variation and population genetic structure in the small yellow croaker at the coast of Yellow Sea and East China Sea[J]. Biochemical Systematics and Ecology, 2017, 71: 236-243. [27] 杨明柳, 徐岩, 高霆炜, 等. 基于COⅠ基因片段的中国南部沿海近亲拟相手蟹的群体遗传多样性研究[J]. 海洋学报, 2021, 43(2): 105-115. [28] BOWEN B W, BASS A L, ROCHA L A, et al. Phylogeography of the trumpetfishes (): ring species complex on a global scale[J]. Evolution, 2001, 55(5): 1029-1039. [29] 江鑫, 杨官品, 危起伟, 等. 中国沿海花鲈群体遗传结构分析[J]. 中国海洋大学学报(自然科学版), 2009, 39(2): 271-274. [30] WRIGHT S. Evolution in Mendelian populations[J]. Genetics, 1931, 16(2): 97-159. Population Genetic Diversity of Spotted Sea Bass () from China Based on MitochondrialGene Sequence FAN Si-gang1, HUANG Hao1,2, WANG Peng-fei1, YAN Lu-lu1, ZHAO Chao1, ZHANG Bo1, QIU Li-hua1,2 (1.,,,510300,; 2.,201306,) 【】To study the genetic diversity and genetic structure of six wildpopulations from China by the cytochrome c oxidase subunit 1 () genes of mitochondrial DNA.【】cox1 sequence was amplified with PCR technology and analyzed by MEGA X, DnaSP 6.12 and Arlequin 3.01 softwares.【】More than 48 polymorphic sites and 50 haplotypes were detected from 229 individuals from six populations. The haplotype diversity (d) was 0.799 - 0.888, and the nucleotide diversity () was 0.001 6 - 0.002 7, the genetic diversity index (st) was - 0.008 1-0.176 7. The analysis of molecular variance (AMOVA) showed that 90.98% genetic variation occurred within populations and 9.02% occurred among populations. The analysis of N-J tree and network of haplotypes indicated that there were no geographic haplotype. Neutrality test indicated that these populations might have undergone a population expansion. ;gene sequence; genetic diversity; genetic structure 范嗣刚,黄皓,王鹏飞,等. 基于序列的中国6个花鲈野生群体遗传多样性[J]. 广东海洋大学学报,2022,42(3):11-17. Q75;S917.4 A 1673-9159(2022)03-0011-07 10.3969/j.issn.1673-9159.2022.03.002 2021-10-09 国家自然科学基金(31802281);广东省重点领域研发计划项目(2021B0202020002);中国水产科学研究院基本科研业务费专项 (2020TD21) ;广州市科技计划项目(202102020487);广州市农村科技特派员项目(20212100051);中央级公益性科研院所基本科研业务费专项(2021TS03;2021SD11) 范嗣刚(1982―),男,博士,副研究员,从事海洋生物功能基因研究。E-mail: fansigang@scsfri.ac.cn 邱丽华(1971―),女,博士,研究员,从事海洋生物功能基因研究。E-mail: qiugroup_bio@outlook.com (责任编辑:刘庆颖)

