基于人工神经网络的凡纳滨对虾分子标记育种值预测
杨 琼,刘青云,李强勇,彭 敏,杨春玲,童艳梅,曾地刚,陈秀荔,陈晓汉,赵永贞
基于人工神经网络的凡纳滨对虾分子标记育种值预测
杨 琼,刘青云,李强勇,彭 敏,杨春玲,童艳梅,曾地刚,陈秀荔,陈晓汉,赵永贞
(广西壮族自治区水产科学研究院/广西水产遗传育种与健康养殖重点实验室,广西 南宁 530021)
【】探讨逆传播人工神经网络(BPANN)算法用于预测分子标记育种值的可行性。采用高通量测序技术对284尾F1代凡纳滨对虾及其父母本进行特定长度扩增片段测序(SLAF-seq),随机取200尾对虾样品的数量性状基因座(QTL)基因型和体质量数据,构建BPANN预测模型,利用该模型分别对其余84尾凡纳滨对虾进行体质量性状预测。构建了1个高密度的单核苷酸多态性(SNP)遗传连锁图谱,鉴定出6个与体质量相关的QTL,对此QTL的BPANN育种值预测结果显示,育种值的平均误差为0.032 0±0.006 4,低于贝叶斯线性回归模型预测的平均误差值(0.046 2±0.005 6)。BPANN用于预测凡纳滨对虾分子标记育种值效果良好。
人工神经网络; 凡纳滨对虾; 分子标记; 育种值
新品种选育是动物养殖业最重要的工作之一。分子标记辅助选择(Marker-assisted selection,MAS)技术可直接选择基因型进行育种,显著提高选育效率[1]。MAS主要用分子标记构建高密度遗传连锁图谱,再鉴定与目标性状关联的数量性状基因座(QTL)。用QTL选择育种时需准确预测分子标记基因型组合的育种值。传统的分子标记育种值的预测方法主要是线性回归分析,如岭回归分析(Ridge regression,RR)、贝叶斯估计(Bayesian estimation,BE)、最佳无偏预测(Best linear unbiased prediction,BLUP)等[2]。岭回归线性预测忽略了分子标记与目标性状的交互作用和非线性[3]。贝叶斯预测、BLUP预测等模型允许通过差异收缩估计分子标记效果,可更灵活描述复杂分子标记与目标性状的关系[4]。人工神经网络(Artificial neural network,ANN)是一种由大量处理单元连接组成的非线性、自适应数据处理算法,可模仿人类神经单元网络进行分布式并行信息处理[5]。逆传播人工神经网络(Back propagation artificial neural network,BPANN)是目前应用最广泛的神经网络[6]。近年来ANN已广泛用于构建非线性复杂问题的优化解计算模型[7-9],在小鼠[10]、奶牛[11]和小麦[12]等物种分子标记育种值预测方面已取得一定进展,但在水产动物分子标记育种值预测方面未见报道。
凡纳滨对虾()又称南美白对虾,是世界上养殖产量最大的虾种。生长性状是对虾最重要经济性状之一,构建凡纳滨对虾遗传连锁图谱并鉴定生长性状的QTL,开发用于育种的分子标记,建立预测分子标记育种值方法,对凡纳滨对虾新品种选育较为重要。过去遗传图谱构建主要基于随机扩增多态性DNA(RAPD)、限制性片段长度多态性(RFLP)、扩增片段长度多态性(AFLP)、简单序列重复(SSR)和简单序列重复区间(ISSR)等[13-14]传统分子标记,这些标记生成的遗传图谱密度相对较低。随着高通量测序技术的发展,基因分型测序(GBS)、限制性位点相关DNA测序(RAD-seq)、特定长度扩增片段测序(Specific length amplified fragment sequencing,SLAF-seq)等第2代测序技术开始用于构建单核苷酸多态性(Single nucleotide polymorphism,SNP)遗传连锁图谱,这些标记比传统标记更密集,更一致,更有效,成本更低[15]。
本研究采用第2代高通量测序技术对凡纳滨对虾进行SLAF-seq,构建SNP遗传连锁图谱,鉴定体质量相关QTL,构建BPANN预测模型并进行分子标记育种值预测,探讨BPANN用于预测分子标记育种值的可行性,为凡纳滨对虾MAS育种提供基础。
1 材料与方法
1.1 动物材料
凡纳滨对虾由广西水产科学研究院凡纳滨对虾遗传育种中心提供。用人工授精方法,使1尾雄性亲虾和1尾雌性亲虾交配,孵化的F1代群体用作遗传连锁图谱的作图群体。
1.2 样品采集
随机采集作图群体的284尾凡纳滨对虾及其父母本,用电子称测量体质量,用剪刀剪下背部的肌肉,放入液氮中保存。
1.3 DNA提取
用海洋动物基因组DNA提取试剂盒(天根生物,中国)提取肌肉DNA。用NanoDrop分光光度计和琼脂糖凝胶电泳对DNA进行定量。
1.4 遗传图谱构建和QTL分析
用HaeⅢ和Hpy166Ⅱ消化对虾的基因组DNA,将测序接头通过T4连接酶连接到消化获得的DNA片段,PCR扩增这些片段,纯化扩增产物。在Illumina HiSeq系统上进行SLAF测序。对原始测序读数进行质量控制,以获得高质量序列。将高质量序列与凡纳滨对虾基因组(https://www.ncbi.nlm.nih. gov/genome/?term=Vannamei)比对,鉴定基于SNP的多态性SLAF标记。将多态性的SLAF标记用HighMap软件构建遗传连锁图谱[12]。使用R/qtl软件包进行QTL分析,每个数据集的检测限(limit of detection,LOD)阈值基于排列组合测试(1 000个排列组合,< 0.05)建立。LOD值高于此阈值的QTL是显著的QTL。估计QTL解释的表型变异:1–10–2LOD/n(为样本数)[16]。遗传图谱构建和QTL分析由百迈客生物技术公司(北京)进行。
1.5 分子标记育种值的预测模型
每个QTL LOD值选择最大SNP作为分子标记,并随机选择作图群体的200个凡纳滨对虾样品,SNP基因型及体质量数据分别用于构建BPANN预测和贝叶斯线性回归(Bayesian linear regression)模型[3]。
BPANN预测模型:用MATLAB7.0的人工神经网络程序包构建BPANN模型,该模型包括1个输入层、2个隐含层和1个输出层(图1)。用凡纳滨对虾样品SNP基因型及体质量数据训练神经网络1 000次。
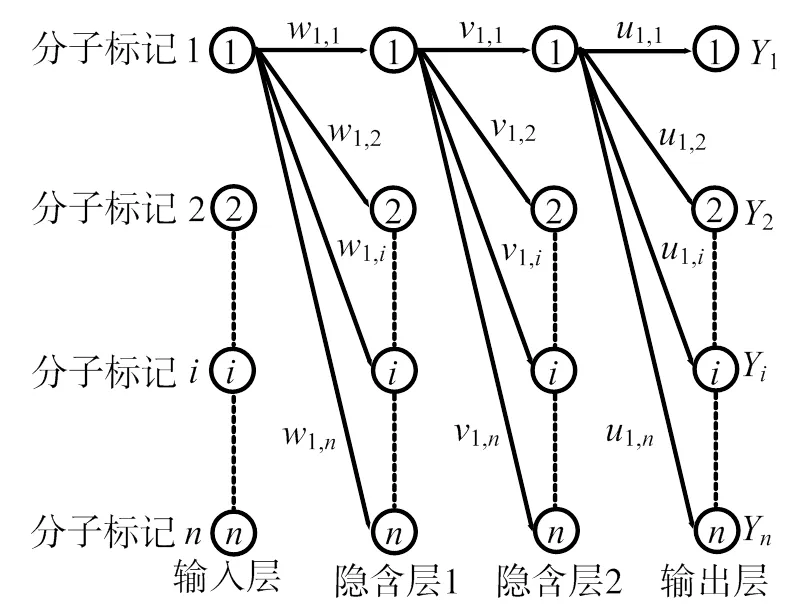
w、v、u分别为各层的各个单元的连接权;Y为输出的值
贝叶斯线性回归预测模型:用R/BLR程序包(http://cran.r-project.org/web/packages/BLR/index.html)建立贝叶斯线性回归预测模型(Bayes A):Ŷ=+1i1+2i2+e,其中是总体平均值,Y为群体中第个体的表型值(= 1, 2, 3, ...,),1和2为分子标记基因型,对于QQ基因型,1= 1,2= 0;对于Qq基因型,1= 0,2= 1;对于qq基因型,1= -1,2= 0;为分子标记的遗传效应;为残差。分别用建立的BPANN预测模型和贝叶斯线性回归预测模型对剩余的84尾凡纳滨对虾进行体质量预测,比较两者的预测效果。
2 结果与分析
2.1 遗传图谱构建及QTL分析
SLAF测序产生439.77 Gb数据,平均30为95.81%,有57.83%的序列被成功匹配到凡纳滨对虾基因组(数据已上传NCBI数据库,登录号:PRJNA545592)。结果表明,SLAF文库的构建和测序正常。过滤并聚类所有序列,鉴定出807 505个SLAF标记,用SLAF的多态性标记构建遗传连锁图谱。结果共有17 338个SLAF标记定位在遗传连锁图谱上。总图距为6 360.12 cM,标记间平均图距为0.37 cM,包含44个连锁群。
利用遗传图谱,对凡纳滨对虾体质量性状进行QTL分析。LOD阈值确定为5.2,在连锁群7和16鉴定了2个与体质量相关的QTL(图2)。
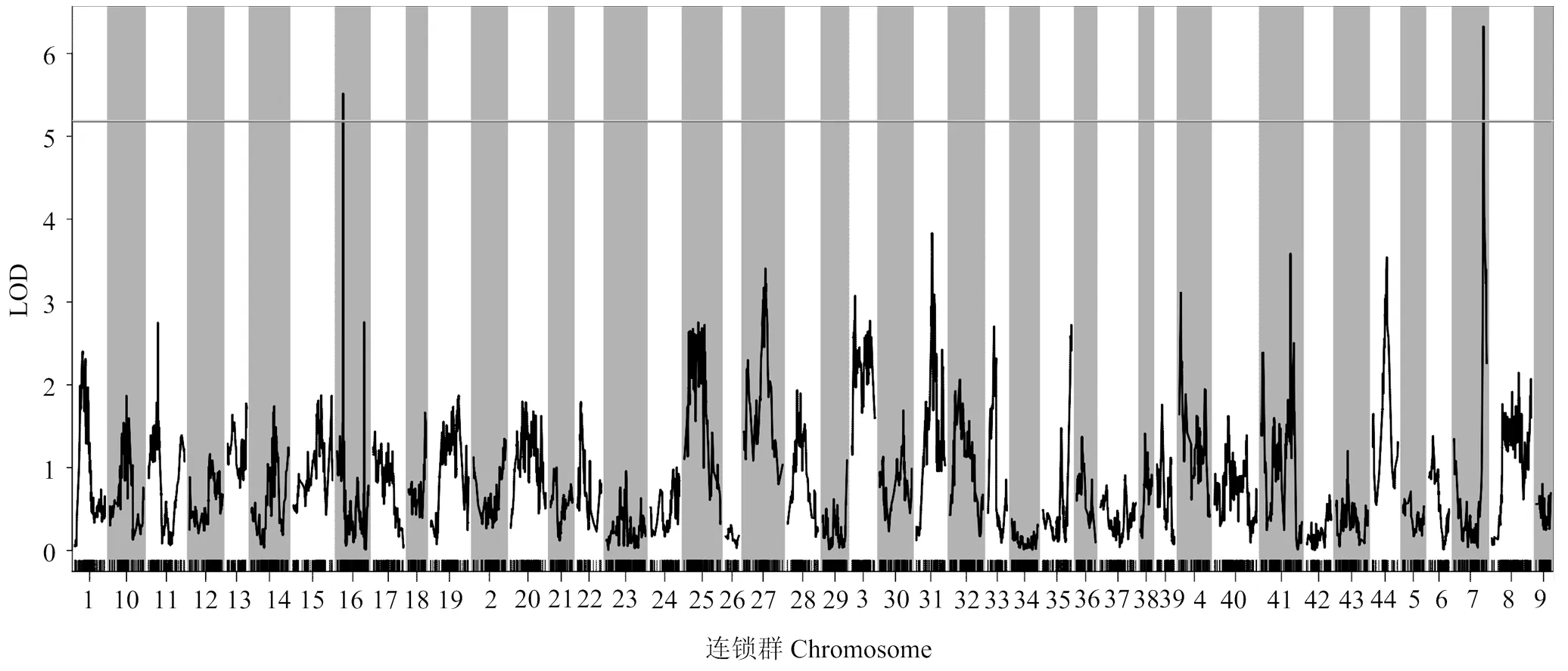
灰色横线显示LOD阈值The grey horizontal line shows the LOD threshold
Fig .2 Quantitative trait loci for weight in
2.2 分子标记育种值的贝叶斯线性回归预测和BP神经网络预测
为用更多分子标记进行预测分析,将LOD降至3.5,鉴定得6个QTL。在6个鉴定的体质量相关的QTL区间中,分别取LOD最大的6个SLAF标记(Marker10241515、Marker4729146、Marker2125004、Marker3571091、Marker1700932、Marker4067002)。剩余84尾对虾的标记基因型数据育种值(体质量)预测结果见表1。表1可见,贝叶斯线性回归预测的平均误差为0.046 2 ± 0.005 6,BP神经网络预测的平均误差为0.032 0 ± 0.006 4。

表1 用贝叶斯线性回归和BP神经网络预测的凡纳滨对虾育种值
3 讨论
3.1 遗传图谱构建和QTL分析
与传统的育种方法相比,分子标记辅助育种可提高育种效果,加快育种进程。本研究用SLAF-seq构建凡纳滨对虾的高密度遗传图谱,相邻标记间平均距离为0.37 cM,而用RAPD、FLP和SSR开发的凡纳滨对虾遗传图谱的相邻标记间平均距离为1 ~ 5 cM[17-20],本研究构建的凡纳滨对虾遗传连锁图谱密度更高。本研究的遗传连锁图谱包括44个连锁群,与之前报道的凡纳滨对虾遗传连锁图谱的连锁群数量一致[21],表明凡纳滨对虾有44对染色体。本研究鉴定了2个与生长相关的QTL,而之前报道的凡纳滨对虾生长相关QTL数量不同[21],可能由所用凡纳滨对虾群体不同,QTL阈值不同所致。
3.2 分子标记育种值的BP神经网络预测
准确预测分子标记育种值对于分子标记辅助选育较为重要。González-Recio等[22]用人工神经网络预测基于全基因组数据的荷斯坦种公牛寿命,发现比用贝叶斯算法更准确。Okut等[10]用人工神经网络预测使用密集分子标记的小鼠体质量指数,认为人工神经网络至少与其他预测方法效果相当,其捕获非线性关系的潜在能力对研究复杂基因控制的数量性状较为有用。Yao等[23]用人工神经网络算法识别影响奶牛采食量的QTL,显示了机器学习方法的巨大灵活性。Ehret等[24]使用人工神经网络预测德国奶牛的产奶育种值,预测效果与GBLUP(基因组最佳线性无偏预测)相当。本研究进行了284尾凡纳滨对虾及其父母本的SLAF测序,并利用其中200个样品的分子标记基因型数据和体质量数据建立了贝叶斯线性回归预测和BP神经网络预测模型。利用模型对剩余的84个样品进行6个分子标记育种值的预测,结果表明BP神经网络预测的误差小于贝叶斯预测,这可能是因为本研究采用的6个分子标记间存在非线性叠加关系,而BP人工神经网络算法有很强的预测复杂非线性关系的能力[6]。本研究结果显示了人工神经网络算法在分子标记育种值预测的潜力。不过,本研究神经网络预测误差仍较大,这可能与分子标记数量较少有关;同时,本研究用于建模的样本和用于预测的样本来源于同一群体,因此可能会存在预测准确性偏高的问题。此外,神经网络算法类型、隐含层数量、神经单元数量、用于机器训练的样本选择等均对预测的效果有一定的影响,还需进一步研究优化。
4 结论
本研究应用高通量测序技术构建了高密度的凡纳滨对虾遗传连锁图谱,鉴定了生长相关的QTL,并探索应用神经网络预测分子标记育种值的可行性,结果表明,BPANN用于预测凡纳滨对虾分子标记育种值效果良好。本研究结果可为凡纳滨对虾分子标记辅助育种研究提供基础数据。
[1] 孙效文, 鲁翠云, 贾智英, 等. 水产动物分子育种研究进展[J]. 中国水产科学, 2009, 16(6): 981-990.
[2] MEUWISSEN T H E, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819-1829.
[3] OKUT H, WU X L, ROSA G J M, et al. Predicting expected progeny difference for marbling score in Angus cattle using artificial neural networks and Bayesian regression models[J]. Genetics, Selection, Evolution: GSE, 2013, 45(1): 34.
[4] GIANOLA D, DE LOS CAMPOS G, HILL W G, et al. Additive genetic variability and the Bayesian alphabet[J]. Genetics, 2009, 183(1): 347-363.
[5] ARYA S, HO CHUNG Y. Artificial neural network estimation of data and channel characteristics in free-space ultraviolet communications[J]. Applied Optics, 2020, 59(13): 3806-3818.
[6] YAO L T, ZHONG Y F, WU J Y, et al. Multivariable logistic regression and back propagation artificial neural network to predict diabetic retinopathy[J]. Diabetes, Metabolic Syndrome and Obesity: Targets and Therapy, 2019, 12: 1943-1951.
[7] CHON K H, COHEN R J. Linear and nonlinearmodel parameter estimation using an artificial neural network[J]. IEEE Transactions on Bio-Medical Engineering, 1997, 44(3): 168-174.
[8] ZHANG W, BAO Z M, JIANG S, et al. An artificial neural network-based algorithm for evaluation of fatigue crack propagation considering nonlinear damage accumulation[J]. Materials (Basel, Switzerland), 2016, 9(6): 483.
[9] TALEBI N, NASRABADI A M, MOHAMMAD- REZAZADEH I, et al. nCREANN: nonlinear causal relationship estimation by artificial neural network; applied for autism connectivity study[J]. IEEE Transactions on Medical Imaging, 2019, 38(12): 2883-2890.
[10] OKUT H, GIANOLA D, ROSA G J M, et al. Prediction of body mass index in mice using dense molecular markers and a regularized neural network[J]. Genetics Research, 2011, 93(3): 189-201.
[11] GIANOLA D, OKUT H, WEIGEL K A, et al. Predicting complex quantitative traits with Bayesian neural networks: a case study with Jersey cows and wheat[J]. BMC Genetics, 2011, 12: 87.
[12] LIU D Y, MA C X, HONG W G, et al. Construction and analysis of high-density linkage map using high-throughput sequencing data[J]. PLoS One, 2014, 9(6): e98855.
[13] MAUGHAN P J, BONIFACIO A, JELLEN E N, et al. A genetic linkage map of quinoa () based on AFLP, RAPD, and SSR markers[J]. TAG Theoretical and Applied Genetics Theoretische and Angewandte Genetik, 2004, 109(6): 1188-1195.
[14] KUIPER M T. Building a high-density genetic map using the AFLP technology[J]. Methods in Molecular Biology (Clifton, N J), 1998, 82: 157-171.
[15] SUN X W, LIU D Y, ZHANG X F, et al. SLAF-seq: an efficient method of large-scale de novo SNP discovery and genotyping using high-throughput sequencing[J]. PLoS One, 2013, 8(3): e58700.
[16] BROMAN K W, GATTI D M, SIMECEK P, et al. R/qtl2: software for mapping quantitative trait loci with high-dimensional data and multiparent populations[J]. Genetics, 2019, 211(2): 495-502.
[17] GARCIA D K, DHAR A K, ALCIVAR-WARREN A. Molecular analysis of a RAPD marker (B20) reveals two microsatellites and differential mRNA expression in[J]. Molecular Marine Biology and Biotechnology, 1996, 5(1): 71-83.
[18] PÉREZ F, ORTIZ J, ZHINAULA M, et al. Development of EST-SSR markers by data mining in three species of shrimp:,, andbirdy[J]. Marine Biotechnology (New York, N Y), 2005, 7(5): 554-569.
[19] ANDRIANTAHINA F, LIU X L, HUANG H. Genetic map construction and quantitative trait locus (QTL) detection of growth-related traits infor selective breeding applications[J]. PLoS One, 2013, 8(9): e75206.
[20] ZHANG L S, YANG C J, ZHANG Y, et al. A genetic linkage map of Pacific white shrimp (): sex-linked microsatellite markers and high recombination rates[J]. Genetica, 2007, 131(1): 37-49.
[21] YU Y, ZHANG X, YUAN J, et al. Genome survey and high-density genetic map construction provide genomic and genetic resources for the Pacific White Shrimp[J]. Scientific Reports, 2015, 5: 15612.
[22] GONZÁLEZ-RECIO O, WEIGEL K A, GIANOLA D, et al. L2-Boosting algorithm applied to high-dimensional problems in genomic selection[J]. Genetics Research, 2010, 92(3): 227-237.
[23] YAO C, SPURLOCK D M, ARMENTANO L E, et al. Random Forests approach for identifying additive and epistatic single nucleotide polymorphisms associated with residual feed intake in dairy cattle[J]. Journal of Dairy Science, 2013, 96(10): 6716-6729.
[24] EHRET A, HOCHSTUHL D, GIANOLA D, et al. Application of neural networks with back-propagation to genome-enabled prediction of complex traits in Holstein-Friesian and German Fleckvieh cattle[J]. Genetics, Selection, Evolution: GSE, 2015, 47(1): 22.
Prediction of Breeding Value of Molecular Markers inUsing Artificial Neural Network
YANG Qiong, LIU Qing-yun, LI Qiang-yong, PENG Min, YANG Chun-ling, TONG Yan-mei, ZENG Di-gang,CHEN Xiu-li,CHEN Xiao-han, ZHAO Yong-zhen
(/,530021,)
【】To explore the feasibility of the back propagation artificial neural network (BPANN) algorithm for predicting the breeding value of molecular markers,【】High-throughput sequencing technology was used to perform specific length amplified fragment sequencing (SLAF-seq) on 284 F1 generation ofand their parents, and the QTL genotype and weight data of 200 shrimp samples were randomly selected to construct a BPANN prediction model. The model was used to respectively predict the weight traits of the remaining 84 shrimps.【】A high-density single nucleotide polymorphism (SNP) genetic linkage map was constructed, and 6 weight-related QTLs were identified, and used to predict breeding values by the BPANN. The average error of the breeding value predicted by the BPANN prediction model was 0.032 0 ± 0.006 4, which was lower than the average error value of the Bayesian linear regression model (0.046 2 ± 0.005 6).【】The BPANN algorithm has a good effect on predicting the breeding value of molecular markers in.
artificial neural network;; molecular marker; breeding value
杨琼,刘青云,李强勇,等. 基于人工神经网络的凡纳滨对虾分子标记育种值预测[J]. 广东海洋大学学报,2022,42(3):122-126.
Q959.223+.633
A
1673-9159(2022)03-0122-05
10.3969/j.issn.1673-9159.2022.03.016
2021-11-09
广西创新驱动发展专项资金项目(桂科AA17204080);国家现代农业产业技术体系广西创新团队建设任务书(nycytxgxcxtd-14-01);国家虾产业技术体系建设任务书(CARS-48)
杨琼(1968―),女,学士,高级工程师,主要研究方向为科技管理。E-mail: 421059417 @qq.com
赵永贞(1978―),男,博士,研究员,研究方向为水产遗传育种。E-mail:fisher1152002@126.com。
(责任编辑:刘庆颖)

