印度洋深海沉积物样品纯培养细菌的分离鉴定
姜钊 ,张卫花 *
( 1. 西藏民族大学 藏药检测技术教育部工程研究中心,陕西 咸阳 712082;2. 西藏民族大学 西藏高原相关疾病分子遗传机制与干预研究省级重点实验室,陕西 咸阳 712082)
1 引言
海洋环境复杂多样,理化性质独特,孕育了丰富的海洋微生物,从海洋表层到海底几千米的地方都有其生命活动。海洋微生物不仅能进行光合作用,也能通过同化海洋环境中的各种营养物质为大型生物提供营养;同时,也能以分解者的角色为海洋植物提供无机营养,维持整个海洋生态系统的良性可持续的循环[1]。然而,随着海洋资源的过渡开采,人类正面临着诸如资源短缺、环境污染和人类健康危机等日益严重的生存与发展危机。在这种大背景下,海洋微生物的重要性不仅仅体现在元素循环、物质转化和能量流动方面,也在生物的演化研究、CO2的减排、节能以及资源的可持续发展与利用等方面发挥积极作用[2]。
2010年由麦切·索基恩负责的全球海洋生物普查计划顺利完成,该计划分析了超过1 200个区域的海洋样本,最终估计海洋微生物种类达到10亿种左右,微生物的总重量相当于2 400亿头非洲象,这些微生物无论是从多样性还是丰富度方面都达到了令人吃惊的程度[3]。但是,海洋微生物的纯培养数量依然较少,科学家们开展了包括太平洋、印度洋、南海、Woodlark海盆、日本海、秘鲁边缘、卡斯卡底古陆边缘海域、鄂霍次克海、地中海等一系列海洋环境微生物的纯培养及多样性分析工作[4-11],统计发现,被描述的海洋来源微生物物种只占全球全部已描述微生物的9.7%,每年增加速率仅为0.93%,大多数海洋微生物还没有实现试验室的纯培养[12]。环境样品中可培养微生物的多样性及出菌率与菌种分离时所用的样品预处理方法、选择培养基的种类以及培养条件等息息相关。比如,样品稀释法可减少优势微生物的竞争,利于寡营养微生物的分离[13];稀有碳氮源的添加,可获得更多稀有放线菌和细菌类群[14];新鲜湿样经过自然风干稀释后,进行瞬时湿热处理,适合放线菌类群的分离培养[15-16]。因此,不同分离方法的探索、培养基的设计,在海洋微生物纯培养分离中显得格外重要。
为调查印度洋深海沉积物放线菌资源的丰富性,同时探索放线菌分离条件,本研究以采自印度洋深海沉积物样品为研究对象,利用多种分离培养基,采用湿热处理法,在前期响应面法优化的样品处理条件基础上,对采自印度洋深海沉积物样品中放线菌进行纯培养分离,争取最大程度分离得到纯培养放线菌及其他类群,以期为后期放线菌代谢产物分离及相关研究提供材料。
2 材料与方法
2.1 材料
2.1.1 样品来源
本实验样品于2013年采自印度洋,为深海沉积物样品,采用抓斗、箱式取样方法获得,沉积表层约5 cm深度作为取样区域,共取样12份,-20℃保存,取样点的经纬度及深度信息见表1,图1。
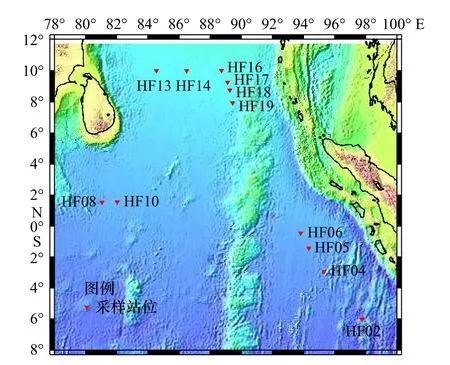
图1 取样点地理分布Fig. 1 The geographical distribution of sampling sites
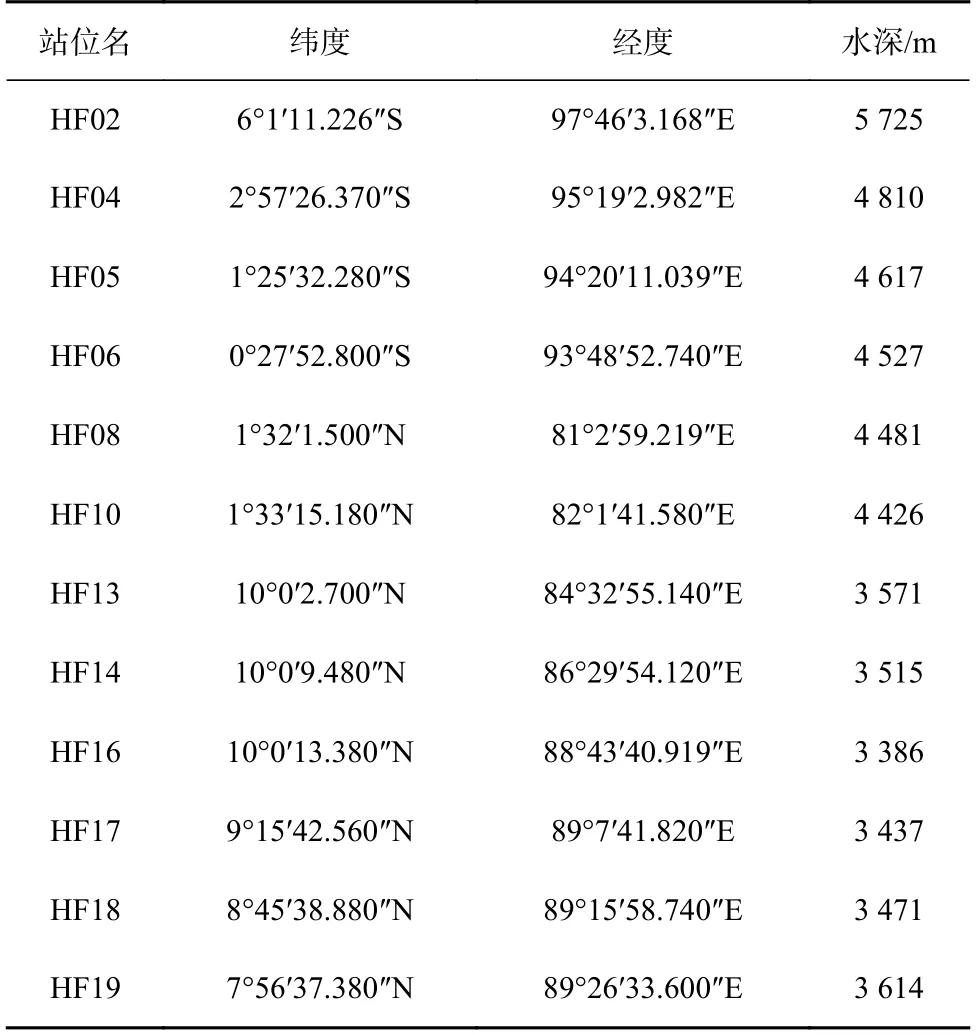
表1 印度洋沉积物采样点信息Table 1 Information of sampling sites from Indian Ocean sediment
2.1.2 分离培养基
依照预实验结果,选取14种分离培养基(表2)进行培养分离,每种培养基添加2.12%海盐(海彩海水晶,上海海彩生物科技有限公司)以模拟海水环境,同时添加50 mg/L制霉菌素和25 mg/L萘啶酮酸,以抑制真菌和革兰氏阴性菌的生长,从而获得更多放线菌类群。
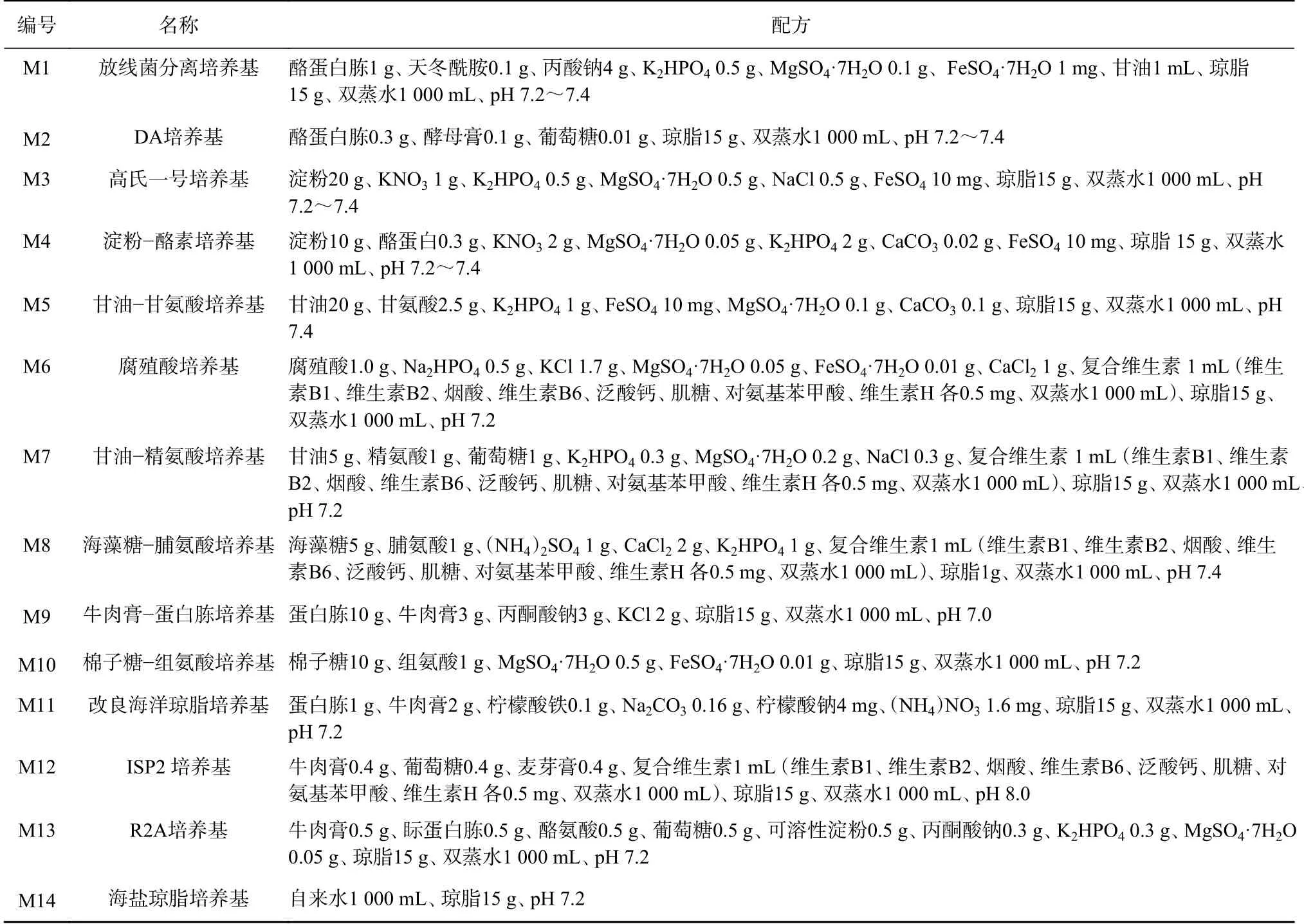
表2 14种分离培养基Table 2 14 kinds of culture media
2.2 菌株分离、纯化及保藏
为获得更多海洋放线菌类群,根据前期优化获得样品的最优处理条件,采用湿热处理方法进行分离:(1)沉积物样品于无菌培养皿风干1.7 d;(2)称取2 g样品至18 mL无菌水中混匀,水浴锅湿热处理(湿热处理温度为55℃,湿热处理时间为1.3 min);(3)置于28℃摇床摇匀20 min,分别取100 μL样品稀释液涂布于14种分离培养基(均加2.12%海盐,同时添加50 mg/L制霉菌素和25 mg/L萘啶酮酸),每组3个重复,并置于28℃恒温培养箱培养30 d;(4)挑取每个平板不同形态菌落于TSA及IPS2培养基中划线、纯化、编号,记录分离来源及分离培养基信息;(5)纯化培养后收集每株菌菌体并利用牛奶管保藏。
2.3 16S rRNA基因扩增及测序
利用酶解法提取收集的各菌株DNA[17],采用细菌16S rRNA基因的通用引物[18]进行PCR扩增(PA:5′-CAGAGTTTGATCCTGGCT-3′; PB:5′-AGGAGGTGATCCAGCCGCA-3′),引物由生工生物工程(上海)股份有限公司合成。PCR 扩增体系:10×Buffer(Mg2+2.5 mmol)5 μL、正向引物 PA(0.25 μmol)1 μL、反向引物 PB(0.25 μmol)1 μL、dNTP 1 μL、DNA 1 μL、Taq DNA聚合酶0.3 U,用ddH2O定容至50 μL。PCR扩增反应条件:95℃预变性4 min;95℃变性1 min,55℃退火1 min,72℃延伸1 min(循环30次);72℃延伸10 min。PCR产物用0.8 %琼脂糖凝胶进行电泳检测,用λ-EcoT14 Idigest DNA Marker为对照,产物检测回收后送昆明泊尚生物有限公司进行测序,先测一个反应1~800 bp(V1-V4)判断种属相似性,若低于98.5 %再进行反向测序。
2.4 基于16S rRNA基因的系统发育分析
通过观察返回的16S rRNA基因序列峰图,选取测序结果好的序列,双向测序则利用Seqman进行拼接,得到1 500 bp左右全长序列。利用EzBioCloud BLAST(http://eztaxon-e.ezbiocloud.net/)[19]进行序列相似性分析;选取同源性较高的模式菌株的16S rRNA基因序列作为参比对象,采用Clustal X软件[20]进行多序列比对,比对结果利用MEGA 7.0[21]软件分析,基于邻近法(NJ)[22]进行系统发育树的构建。
3 结果与讨论
3.1 菌株的分离结果
分离平板培养30 d后,根据菌落大小、形态、颜色等特征,挑取分离平板上的单菌落进行纯化、去重,总共从12个分离样品中纯化得到343株放线菌和细菌纯培养物,并对其16S rRNA基因进行了测序。测序结果经EzBioCloud数据库比对分析,鉴定为4个门:放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria);分属5个纲:放线菌纲(Actinobacteria)、噬纤 维 菌 纲(Cytophagia)、芽 孢 杆 菌 纲(Bacilli)、α-变形菌纲(Alphaproteobacteria)和γ-变形菌纲(Gammaproteobacteria);13个目:棒杆菌目(Corynebacteriales)、微球菌目(Micrococcales)、丙酸杆菌亚目(Propionibacteriales)、弗兰克氏菌目(Frankiales)、链霉菌目(Streptomycetales)、链孢囊菌目(Streptosporangiales)、嗜纤维菌目(Cytophagales)、芽孢杆菌目(Bacillales)、鞘脂单胞菌目(Sphingomonadales)、根瘤菌目(Rhizobiales)、红 细 菌 目(Rhodobacterales)、假 单 胞 菌 目(Pseudomonadales)和海洋螺菌目(Oceanospirillales)下的26个科39个属121个种,如表3所示。可以看出:分离菌株中放线菌门占比最高,占13个科,20个属,71个种(占比58.7%),这可能是因为样品处理和培养条件更利于放线菌的选择性培养;其次为变形菌门和厚壁菌门(分离种数分别占21.5%和17.4%);拟杆菌门最少。各属中分布的菌种数见表4,其中链霉菌属(Streptomyces)、芽孢杆菌属(Bacillus)、考克氏菌属(Kocuria)、葡萄球菌属(Staphylococcus)、迪茨氏菌属(Dietzia)、红球菌属(Rhodococcus)、微杆菌属(Microbacterium)、节细菌属(Arthrobacter)、类诺卡氏菌属(Nocardioides)、拟诺卡氏菌属(Nocardiopsis)为所采印度洋深海沉积物样品中的优势微生物类群,其中链霉菌属和芽孢杆菌属分布最多。同时分离得到了部分数量较少类群,如:食烷菌属(Alcanivorax)、嗜冷杆菌属(Psychrobacter)、叶杆菌属(Phyllobacterium)、鞘脂单胞菌属(Sphingopyxis)、紫杆菌属(Porphyrobacter)、芽球菌属(Blastococcus)、刘志恒菌属(Zhihengliuella)、柠檬球菌属(Citricoccus)、拉氏杆菌属(Rathayibacter)、威廉姆斯菌属(Williamsia)等。将分离到的放线菌、细菌及近缘物种建立系统发育树,见图2和图3。
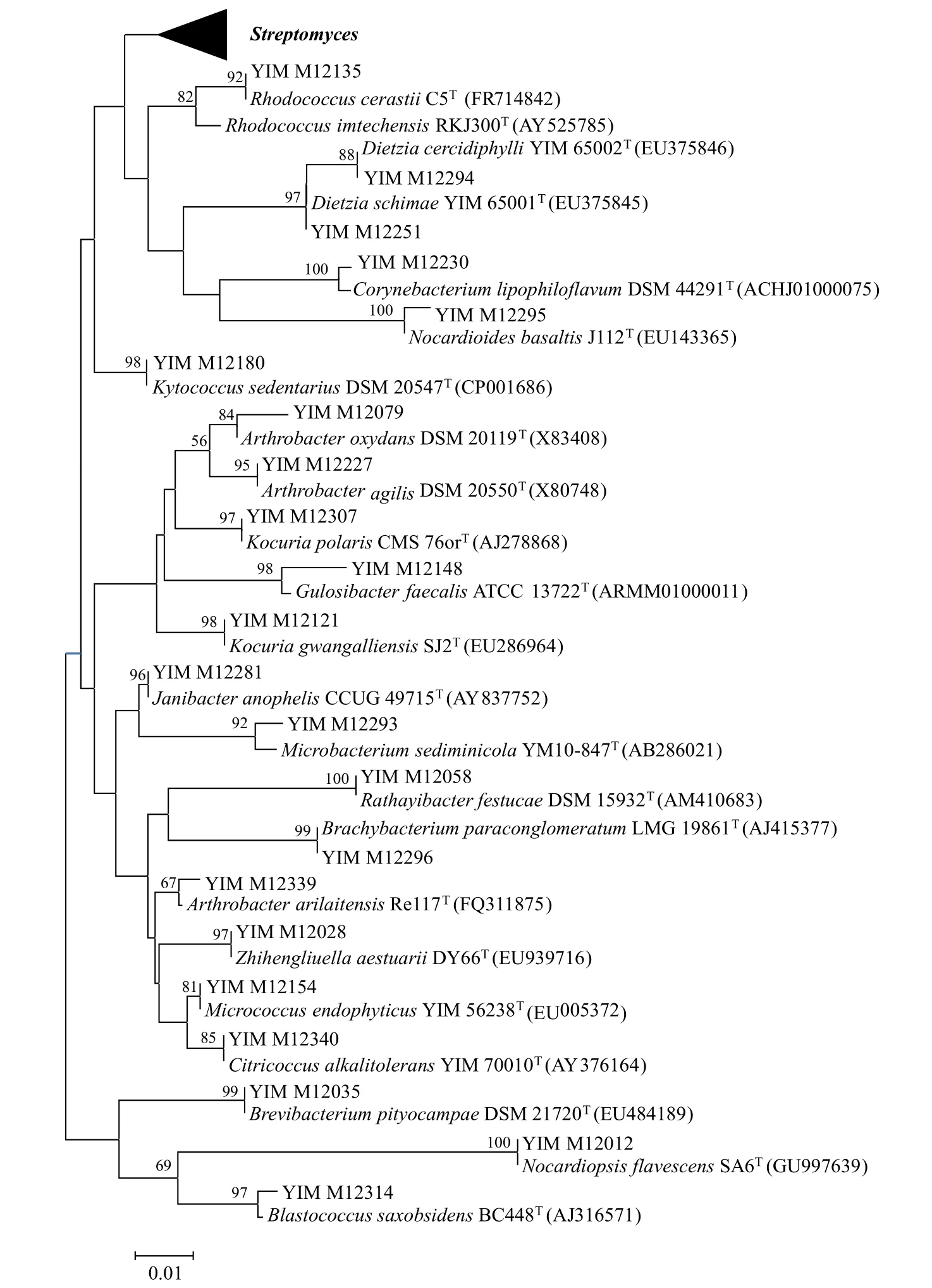
图2 印度洋沉积物中放线菌16S rRNA基因序列邻近系统进化树Fig. 2 Neighbour-joining phylogenetic tree based on 16S rRNA gene sequences of actinobacterial isolates collected from the Indian Ocean sediment
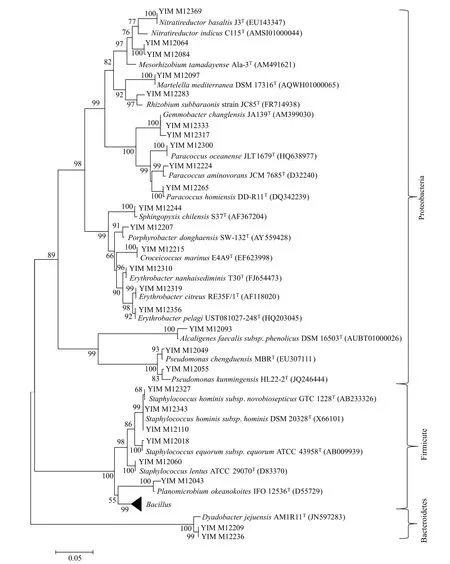
图3 印度洋沉积物中细菌16S rRNA基因序列邻近系统进化树Fig. 3 Neighbour-joining phylogenetic tree based on 16S rRNA gene sequences of bacterial isolates collected from the Indian Ocean sediment
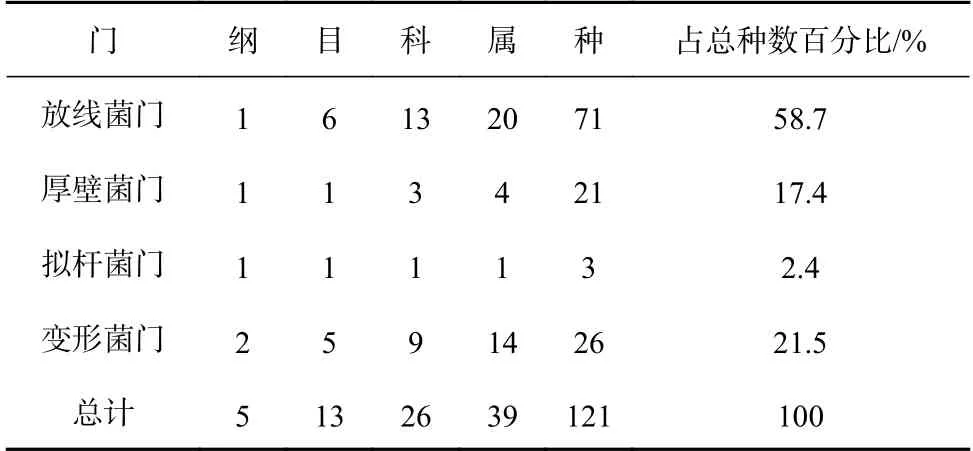
表3 分离菌株整体分布表Table 3 Overall distribution of isolated strains
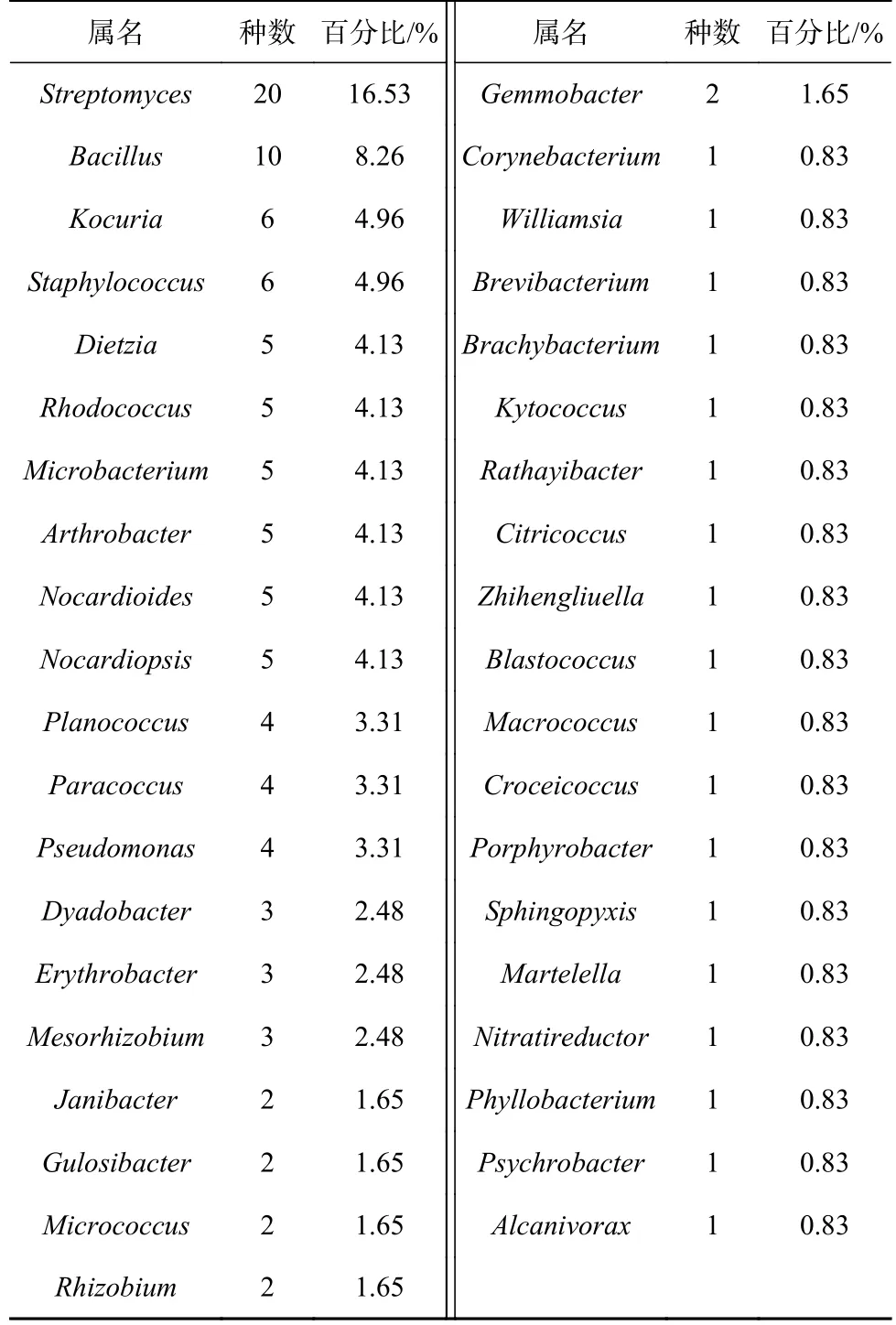
表4 种分布百分比Table 4 Percentages of species distribution
3.2 不同样点的菌株分离比较
统计各样点菌株分离结果见图4,由图可知HF06、HF13、HF14、HF16、HF17、HF18、HF19采样点样品的多样性较丰富,这7个采样点都分离出了大于11个属,18个种的菌株。其中HF17采样点样品的多样性最丰富,可能因为样品的分离及处理条件更利于其样品纯培养菌株的分离。链霉菌属、微杆菌属在这12个样品中都分离到,为海洋沉积物微生物优势类群。部分菌株只在极少数样品中分离得到,例如:拉氏杆菌属只在HF02采样点样品中分离得到;威廉姆斯菌属只在HF06采样点样品中分离得到;地中海菌属(Martelella)、叶杆菌属只在HF10采样点样品中分离到;短杆菌属(Brevibacterium)只在HF13采样点样品中分离到;嗜冷杆菌属只在HF14采样点样品中分离得到;刘志恒菌属只在HF17采样点样品中分离得到;芽球菌属、类诺卡氏菌属只在HF19采样点样品中分离得到;食烷菌属只在HF10采样点和HF19采样点样品中分离得到;两面神菌属(Janibac-ter)、鞘脂单胞菌属只在HF02采样点和HF17采样点样品中分离到;中慢生根瘤菌属(Mesorhizobium)只在HF10采样点和HF17采样点样品中分离得到,不同采样点中细菌分离多样性差异明显。
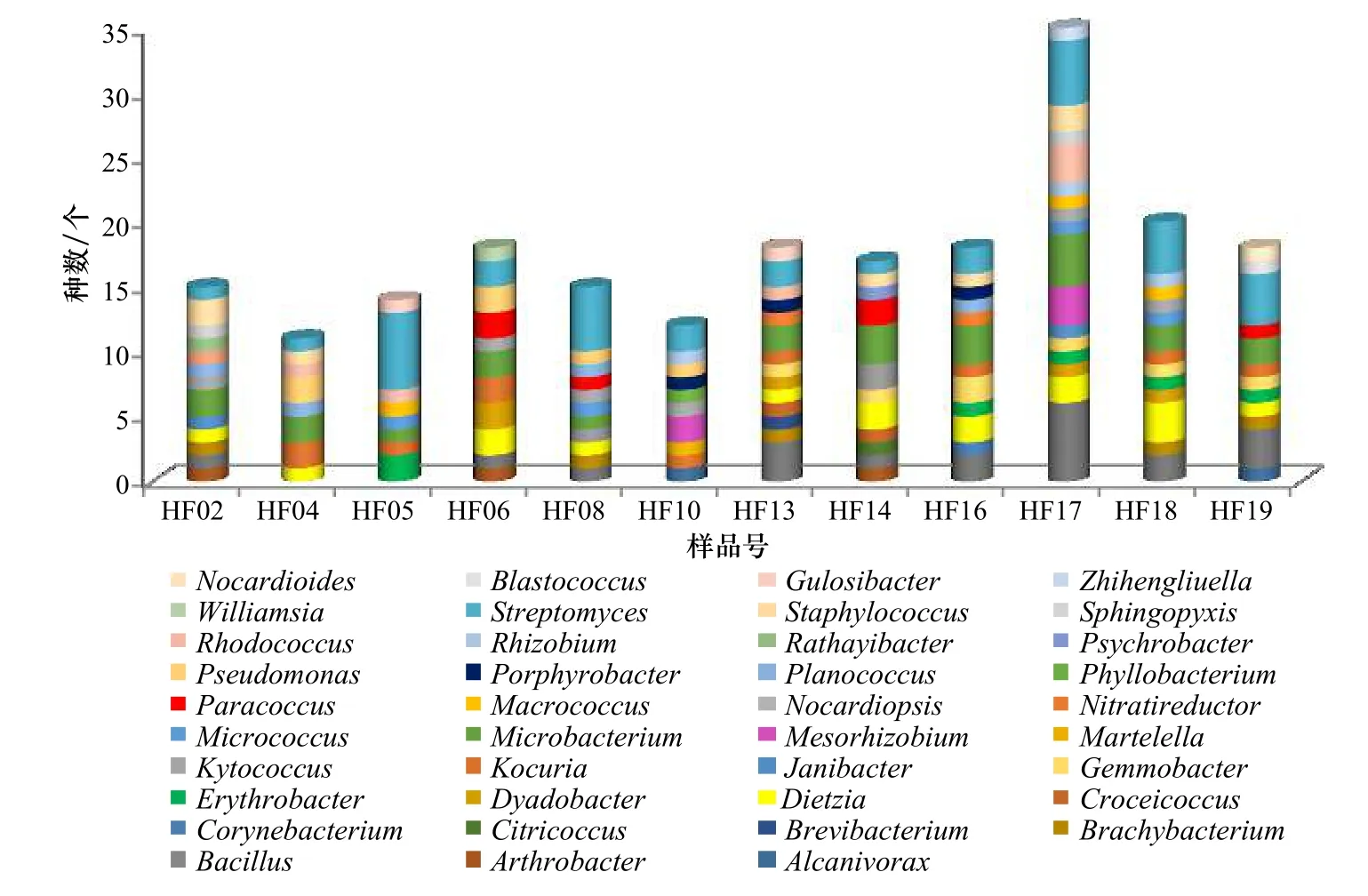
图4 所有沉积物样品中不同类群细菌菌株数目Fig. 4 Strain numbers of different bacteria groups isolated from all sediment samples
3.3 菌株的多样性与分离培养基的关系
在分离的所有菌株中,由于对不同培养基的营养需求不同,每种培养基分离出的菌株数量不同,统计各培养基分离菌株数目见图5。由图可知,在14种分离培养基中,M2、M3、M7、M9、M10、M12、M13这7种培养基分离效果较好,分离菌株数都在15个种以上,M7培养基分离的菌株数超过20个种,M12超过30个种。显而易见,培养基M7和M12分离的菌株多样性最丰富,因此,包含不同营养物的培养基设计对菌株的分离极为重要。
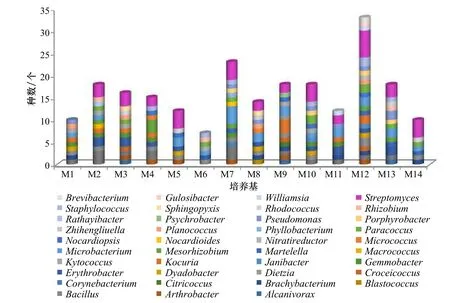
图5 不同培养基中分离的细菌菌株类群及数目Fig. 5 Strain numbers of different bacteria groups isolated from different culture media
3.4 潜在的新分类单元
目前普遍认为16S rRNA 基因序列相似性小于95%,且在系统进化树上形成的稳定独立分支又具有特征性碱基,可以大体作为属的界限;16S rRNA基因序列相似性小于98%且DNA杂交同源性小于70%为“种”的界限[23-24]。因此,将16S rRNA序列相似性低于98%的菌株确定为潜在新分类单元,本研究共发现12个潜在分类单元(表5)。主要分布在食烷菌属、节细菌属、成对杆菌属(Dyadobacter)、芽殖杆菌属(Gemmobacter)、球菌属(Macrococcus)、中慢生根瘤菌属、叶杆菌属、动性球菌属(Planococcus)、假单胞菌属(Pseudomonas)9个属。其中,菌株YIM M12122、YIM M12139和YIM M12140与最相近的菌株16S rRNA基因相似性均低于95%,是潜在的新属。由此可见,印度洋深海沉积物中存在着大量的未被发现的微生物类群,是巨大的资源宝库。
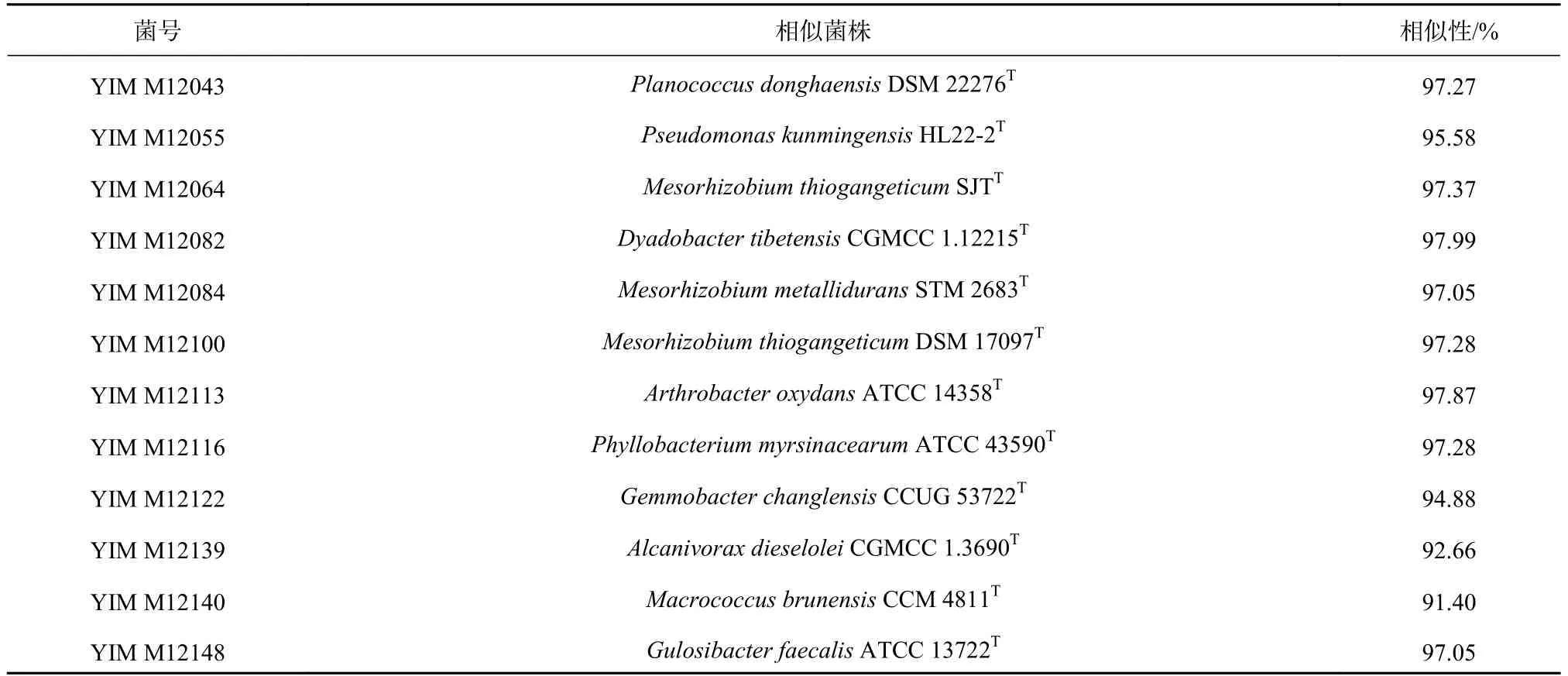
表5 潜在新物种的16S rRNA基因序列比对结果Table 5 Comparison of 16S rRNA gene sequences of potential new species
4 结论
采自印度洋的12个样点中共分离得到343株放线菌和细菌纯培养物,共分布在39个属121个种。其中,放线菌门占58.7%,变形菌门占21.5%,厚壁菌门占17.4%,拟杆菌门占2.4%。对比以往海洋微生物分离结果(图6):Goodfellow和Fiedler[25]的研究统计发现,在海洋环境中发现的放线菌共有49个属,包括12个新属,这12个新的分类单元全部是2000年后才从海洋环境中首次发现和描述的;徐盈[26]从南海沉积物环境中分离得到40个科64个属,其中18个属与Goodfellow和Fiedler分离相同。在本研究中分离得到的39个属中分别有12个属和19个属与文献[25-26]分离的细菌属相同,共有8个属在3次分离中均得到,而本研究中柠檬球菌属等16个属在以上研究中并未分离得到。其中,人们很容易从海洋分离得到的优势类群如小单胞菌属、红球菌属、链霉菌属及迪茨氏菌属[27]的后3个在本研究中大量分离得到,为本次分离得到的优势类群。而专属海洋菌盐细菌属(Salinibacterium)和盐水孢菌属(Salinispora)[14]未被分离到,说明了在纯培养条件的局限下得到的微生物种类仍然有限。在本次分离中,得到部分数量较少的类群,如食烷菌属、嗜冷杆菌属、叶杆菌属、鞘脂单胞菌属、紫杆菌属、芽球菌属、刘志恒菌属、柠檬球菌属、拉氏杆菌属、威廉姆斯菌属等,可能设计的培养基和样品处理条件不利于上述类群的生长。不同的培养基分离差异比较中发现,不同培养基分离得到的物种数差异较大,也证明了不同的针对性培养基的设计及样品处理方法的探索是得到更多可培养细菌的前提和保障。本研究共发现12个潜在分类单元,其中菌株YIM M12122、YIM M12139和YIM M12140与最相近的菌株16S rRNA基因相似性均低于95%,是潜在的新属。研究表明,不同的培养方法和培养基的选择直接影响到样品中纯培养微生物获得的几率和数量,后期可利用免培养的测序数据讨论样品所含微生物的多样性,并结合其他分子生物学手段更好地设计和改造纯培养方法及培养基,以加大纯培养微生物的获得率。

