功能性菌群构建的研究进展
黄佳城,张瑷珲,付友思,方柏山
(1 厦门大学化学化工学院,福建 厦门 361005;2 厦门市合成生物技术重点实验室,福建 厦门 361005)

1 功能性菌群
经历了20 年的不断发展,合成生物学逐渐成为了一个综合学科融合之所,一个交叉学科碰撞之处。研究人员创建了形形色色的“微生物细胞工厂”,不仅实现了许许多多化学品的绿色制造,而且开发了生物修复、生物材料合成等多种功能[1]。其中,美国加州大学伯克利分校(UCB)化学工程系Keasling 课题组[2]改造大肠杆菌和酿酒酵母合成青蒿素前体是合成生物学最经典的里程碑案例之一。他们历经20 年,研究过程涉及载体构建、酶异源表达、底盘细胞开发、启动子与核糖体结合位点高通量筛选、通量平衡分析和转录后调控等,最终实现了青蒿素的半合成。该项研究成果在国际上产生重大影响,被Discovery杂志评为当年的十大科学进展之一。
然而,单一生物体工程在复杂化合物合成上面临诸多矛盾:第一,复杂目标产物合成所需的大容量外源基因与底盘细胞接收能力的矛盾[3];第二,复杂目标产物合成途径对细胞中资源需求与细胞本体生长代谢的矛盾[4-5];第三,单一生物体目标产物合成过程所需的稳定微环境与环境扰动的矛盾(例如环境变化或产物/副产物胁迫)[6-7]。为了解决上述产物合成和生产中单一生物体工程面临的矛盾,研究人员正在开发动态微生物群落中多物种工程改造平台替代单一生物体工程:从最初的菌株代谢通路的模块化构建的“单兵作战”快速发展为合成微生物群落共同协作的“联动共赢”来面对复杂的产物合成难题[8-10]。
微生物群落一般由多种微生物组成,相比于单一菌株,这些微生物组成的群体具备多种优势[8]:第一,群体中成员的分工协作减轻了外源载体的容量;第二,分工和资源交换减轻群落中个体的代谢负担;第三,较高的鲁棒性减轻了环境变化的胁迫压力。因此,相对于单一菌株,整个群落成员的协作使其具有更强的代谢能力,同时群落中物种之间的信息交流使其具有更强的稳定性[9-11]。正是由于这些特性,微生物群落在复杂基质和污染物的降解[12-18],药物、生物燃料和蛋白质复合物的生物法生产[13,19-24],功能化生物材料的制备[18,25-27],分布式逻辑计算/存储器[28-33]、生物传感器的构建[34-35]等方面具有巨大的潜力。
但是微生物群落平台的发展带来诸多优势的同时,也提升了工作量和操作难度[8]。幸运的是,宏组学(meta-omics)的发展为我们带来了微生物群落的大数据[36],日益成熟的人工智能理论和技术显著提高了其处理和分析大数据的能力[37]。通过引入人工智能技术有望从大数据中探究客观规律,并利用这些规律快速寻找特定问题的最佳解决方案。
本文详细介绍了功能性菌群“自下而上(bottom-up)”和“自上而下(top-down)”的两种设计策略,以及功能性菌群的分析工具--“宏组学”的发展现状,总结了功能性菌群构建过程中面临的挑战,并展望了以“智能设计”为核心的发展方向。
2 功能性菌群的设计策略
在构建功能性菌群的探索中,研究人员提出两种不同的研究策略:分别是“自下而上”和“自上而下”。这两种研究策略对建立功能性菌群的研究提出不同的见解[38]。
2.1 “自下而上”的设计策略
“自下而上”的研究策略一般是通过获取微生物组中单个成员的基因组来重建它们的代谢网络,并使用建模或网络分析工具来指导设计微生物群落,使群落具有一定的功能(图1)。这是合成生物学家所关注和努力的方向。该研究策略可以分为以下两个部分:合成微生物群落工具的开发;利用工具促使群落目标功能的实现。

图1 使用“自下而上”策略构建功能菌群Fig.1 “Bottom-up”strategy for constructing functional microbial communities
2.1.1 构建合成微生物群落工具的开发
微生物群落具有“群落内部通信”、“感受外界环境变化”以及“交换群落内资源”的功能。为了模拟以上三方面的功能,研究人员建立了“群体感应(quorum sensing,QS)”“诱导性元件”“共营养化”3 种方法来开发适用于合成微生物群落的工具。
群体感应[39]是一种生物通信系统:在原核物种中,细胞产生的自体诱导物分子能够作为种群感知群落密度的一个信号,随着细胞群的增长,细胞会根据自诱导剂浓度来调节基因表达以控制群体水平的行为。然而直接利用天然的群体感应系统作为合成微生物群落的工具会存在信号串扰的问题,因此通过对现有QS 系统的改造,研究人员建立了一系列具有正交性的QS 系统。加州大学旧金山分校(UCSF)的Hasty 课题组通过启动子和蛋白质修饰,将Rpa 和Tra 群体感应系统和Lux和Las 系统进行整合,构建具有完全正交性的系统[40],并提出可诱导群体感应(inducible quorum sensing system,iQS)的概念[41]。中科院深圳先进技术研究院娄春波课题组[42]通过对元件挖掘、理性设计以及定向进化等手段,开发了10 套全新的群体感应系统,形成了一整套具有通用性高、正交性强、可以跨生物界通信的合成生物学工具箱。
诱导性元件使得群落中的成员能够感应环境条件并做出一系列的反应。有的研究通过外源添加分子来改变环境条件进而控制群落中成员的基因表达。麻省理工学院(MIT)的Collins 课题组[43]在Lactococcus lactisNZ9000 中构建不同诱导物诱导的回路,通过控制诱导物的添加模拟群落的社交关系;同时环境条件的改变也包括了调整营养浓度和培养条件来改变种群特异性基因表达[44]。
群落成员间相互依赖的功能性菌群可以通过共养相互作用的原理进行设计,使得群落中的某一生物体依赖其他群落成员所产生的代谢物,进而构建成员更多、鲁棒性更高的功能性菌群。加州大学圣地亚哥分校(UCSD)的Zengler 课题组[45]系统性地描述如何通过营养缺陷型策略构建复杂群落的原理。根据这一原理,哥伦比亚大学的Wang 课题组[46]构建了14 种不同缺陷型大肠杆菌群落,它们之间相互依赖、共同生存。
上述这三方面的先驱性的工作可以帮助我们构建正交性更好的“群体感应”系统、调控更为精准的“诱导性元件”和更为复杂的“共营养化”策略,有利于我们建立具有高信噪比的菌种通信,完成高精准的群落内基因表达和构建高鲁棒性的功能性菌群,也为群落行为的实现提供了可靠的工具。
2.1.2 利用工具使得群落表现出目标功能
在群落行为的设计中,合成生物学家们利用2.1.1所述的工具驱动微生物群落表现的3种最具代表的行为“菌群的数量控制”、“分布式代谢”和“菌群的空间编程”。
在群落的代谢过程中,某些特定代谢物的相对产量可能会在代谢流层面上影响最终产物的产出或者是对底盘生物造成毒性[47],而通过“控制微生物群体大小水平”则可以改善该问题。Hasty课题组[48]将两套正交性的群体感应系统和裂解毒蛋白结合,构建了两个能够维持竞争关系的沙门菌群。
“分布式代谢”则是解决产物合成路线过长的终极利器。它通过将冗长的代谢回路合理地分割成不同的部分,并导入最佳宿主中,可以越过本来在单一生物工程中由于代谢负担过大而不能生产的难题来合成复杂的天然产物。MIT 的Stephanopoulos课题组利用分布式代谢的策略,将生产紫杉醇前体--氧化紫杉烷(oxygenated taxanes)的回路分段导入大肠杆菌和酿酒酵母中,利用共培养技术将氧化紫杉烷的产量提高到33 mg/L,同时证明了该体系也适用于其他氧化类异戊二烯的生产[19]。
“菌群的空间编程”则通过在空间上形成有序的组织结构来帮助整个群落应对环境的扰动[49]。斯坦福大学的Riedel-Kruse 课题组[50]通过建立正交、可组合的黏附素库,结合不同的诱导性元件在E.coliMG1655 和E.coliS1 间建立了晶格状、相分离和不同的黏附模式,为多细胞的空间编程提供了一个工具箱,研究人员可以利用该工具箱构建各种不同的结构来提升群落的稳定性。
虽然已经能够利用“自下而上”策略建立发挥特定功能的中小型功能性菌群[16,46,48-50],同时适用于合成微生物群落的工具的研发在早期合成生物学发展中已经初具雏形,元件的正交性和通用性也已经获得保障[41-42,50],并且已经完成对群落个体的精准控制目标[16,43,48-50]。但是由于群落设计原理、模型和计算工具的缺失,导致了群落行为设计的难度大、设计完成的菌群稳定性差等问题,并且合成群落中菌种数量的递增只能通过更复杂的营养化策略,随之带来的是工程菌株中构建的回路大多数是为了维持群落稳定而非产品的工业生产。以上这一系列问题给利用该策略建立大型功能性菌群带来了诸多麻烦。
2.2 “自上而下”的设计策略
“自上而下”的研究策略则是微生物学家和组学家的主战场。“自下而上”的研究策略擅长构建中小型功能性菌群,相比之下“自上而下”的设计策略在分析和建立大型、复杂的功能性菌群方面更具优势。这一策略的研究有明显的前期后期分界线。
2.2.1 通过驯化获得功能性菌群
该策略前期的研究工作集中在菌群驯化(图2),通过物理化学环境的设计引导现有的微生物组(自然发生或接种)发生生态选择,以执行期望的生物过程。成功的例子包括:来自瓦格宁根大学的Lettinga 课题组[51]通过改变反应器内的流体动力特征来调整硫酸盐废水中硫酸盐还原菌和甲烷产生菌之间的竞争关系;南洋理工大学的Liu 课题组[52]系统性地阐述了在生物膜和活性污泥形成过程中,生物反应器内水的剪应力对于微生物群落的形成、结构和代谢过程的重要作用等。同时在这一时期发展了一系列相关的数学模型,用来量化解析系统中化学物质和相关微生物的质量平衡关系,并模拟了化学和生物化学转化率,探究了系统内的微生物群落的空间关系:昆士兰大学的Batstone 课题组建立了厌氧消化过程的数学模型,推动了传统废水循环中反应器的设计与新兴厌氧过程的发现[53];通过结合物理输运过程模型,来自代尔夫特理工大学的van Loosdrecht课题组[54]建立了多物种参与的生物膜模型来分析不同环境条件下产生的生物膜的性质。这一系列与时间、空间相关的模型推动了“自上而下”功能性菌群设计策略的发展,加深了研究者们对于自然环境下微生物群落的形成条件和培养要求的理解,反过来指导了一系列菌群驯化相关的反应器的设计以及培养条件的设置。
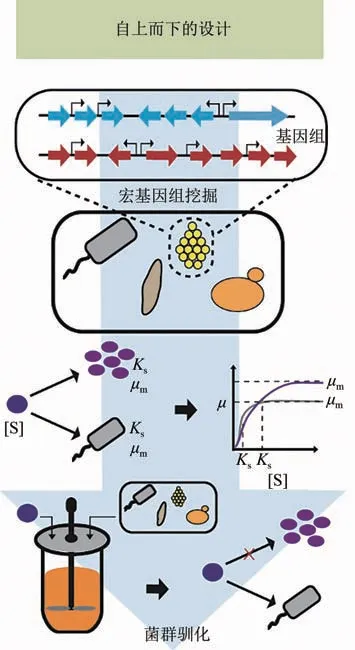
图2 使用“自上而下”策略构建功能菌群构建Fig.2 “Top-Down”strategy for construct functional microbial communities
2.2.2 通过挖掘与原位改造技术打开“环境菌群黑箱”
该策略后期研究目标慢慢转向如何打开环境菌群的“黑箱”,向大自然寻求智慧。这个时期的主要工作集中于两部分:挖掘和原位改造由大自然环境长期进化产生的生态系统(图2)。
利用宏基因组技术进行信息的挖掘。研究者们通过环境微生物的采样,不仅在大自然中发现了新的微生物物种[如加州大学伯克利分校(UC Berkeley)的Banfield课题组[55]利用宏基因组技术从地下水中发现了一类由于16S rRNA 的特殊性无法被传统方法检测出的原核生物,并命名其为CPR],而且发现了一系列的新生物砖,以及新的化学品合成回路(美国洛克菲勒大学Brady团队[56]利用NRPS 基因宏基因组测序和BAC 文库筛选的方法从2000 多个土壤样本中发现了一种未知抗生素合成基因簇)。他们还成功发现了新的蛋白质(来自曼彻斯特大学的Turner课题组[57]在英国海岸的环境样品中发现了300 多种新的亚胺还原酶)、新的CRISPR 系统[诺贝尔奖获得者,加州大学伯克利分校(UC Berkeley)的Doudna 课题组[58]从地下水、底泥、酸性尾矿废水生物膜和土壤、婴儿肠道等微生物样品中挖掘出第1 个来自古菌的Cas9 蛋白和2 个新的CRISPR-Cas 系统]以及一系列的特殊调控元件(哥伦比亚大学的Wang团队[59]通过宏基因组挖掘的方式从数据库中发现大量5'端调控元件,经高通量实验验证后发现一系列能够实现物种特异性表达的元件)。随着第三代测序(例如Nanopore和PacBio等)的兴起,测序的读长的增加使得我们能够挖掘到更为完整的群落信息,获得更多大自然进化中的瑰宝。
摒弃在实验室中进行实验的方式,发展原位工程的改造策略。原位工程上的发展给予了研究者们更自由的操作空间和更有希望的应用前景,但相比实验室的非原位工程而言也具有更高的难度。首先,要保证有方法可以递送质粒进入环境:Wang 课 题 组[60]利 用 接 合 型 质 粒(conjugative plasmids),成功将荧光蛋白基因导入小鼠的肠道菌群中,建立了在哺乳动物肠道微生物中进行原位工程的方式方法;其次,要确保具有群落内精准基因编辑的工具:UCSF 的Turnbaugh 课题组[61]利用噬菌体M13 完成了群落中大肠杆菌的精准基因编辑。上述两项工作,分别对标代谢工程中的基因工程改造和基因编辑方法,让我们在原位水平上修改群落属性成为可能。当然也有作为早期工作的延伸,通过改变环境条件进行原位菌落驯化的研究:上海交通大学的张晨虹课题组[62]通过高纤维饮食引起整个肠道微生物群落的变化,使得群落发酵产生短链脂肪酸。类似的工作还有最近吸引众多科研者目光的粪便微生物移植技术(FMT)[63]。
利用自上而下的策略设计功能性菌群的研究中,早期菌群的驯化已经能够解决很大一部分的生态问题(其中代表性的生态污泥的研究[51-52]也已趋近完善,并具有应用价值),后期在打开菌群这个“黑箱”的过程中,元件的挖掘与原位技术也取得了一定的进展[56-59]:可以看见自上而下的研究方法为自下而上的元件设计研究提供了不少新思路,两者的界限正在慢慢地被打破,一些新的研究范式正在被催生[38]。但是,在后期的研究中,环境样本的收集和元件的验证需要高通量的实验技术,巨大的实验工作量对有限的人力物力造成了极大挑战。除此之外,原位工程技术的发展不仅受到接合型质粒转入范围较小且不稳定的影响,也受限于噬菌体工程[64]:是否存在广谱噬菌体或是通过噬菌体改造来改变噬菌体的专一性,进而加强原位工程的精准性。同时该策略虽然在生态治理中具有广泛的应用前景,但是对于工业生产来说可操作性差,难以保障生物安全[38]。
3 功能菌群的分析手段
“自下而上”和“自上而下”两种微生物组研究策略之所以能够获得迅猛的进步,除了合成生物技术的发展,还要归功于它们的另一个共性之处--广泛使用组学技术作为群落的分析手段:“自下而上”的研究方法利用了培养组学技术结合16S 扩增子测序技术,解析合成群落的结构以及研究群落动态变化[46],而“自上而下”的研究策略利用宏基因组技术从环境中获得基因组信息[55-61]、研究群落的动态变化过程及原因[62]。而在此之前,研究人员已经开发了一系列的方法、流程和软件来完成复杂的组学分析问题。
3.1 利用宏组学分析功能性菌群
在群落研究中广泛使用的组学技术包括宏基因组(metagenomics)、宏转录组(metatranscriptomics)、宏蛋白组(metaproteomics)、宏代谢组(metametabolomics)等。其中,扩增子测序[65]生成菌群结构谱,用于研究菌群中微生物种类、丰度和比例;宏基因组[66]解析了菌群的功能性问题;宏转录组[65]、宏蛋白质组[67]、宏代谢组[68]则揭示了在采集样本时的理化环境条件下,菌群准备发挥什么功能、菌群正在发挥什么功能、菌群已经发挥了什么功能。通过对宏组学数据的处理,我们可以获得整个微生物群落组成、功能、相互作用关系的图谱(图3)。
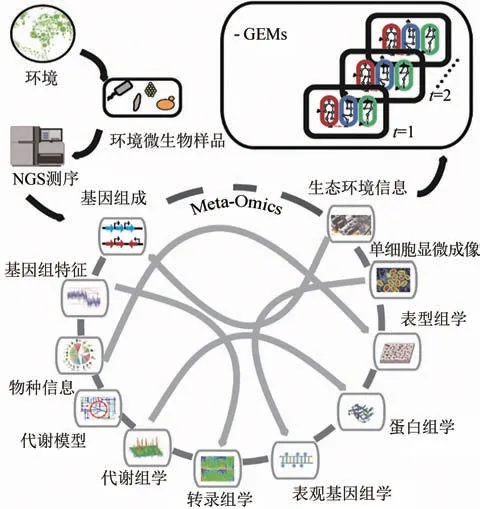
图3 使用“宏组学”分析功能性菌群Fig.3 Analyzing functional microbial communities using meta-omics methodology
早期的宏组学作为功能性菌群的分析工具,主要集中于利用单一组学对群落进行分析。科罗拉多大学Pace[69]在1985年最早提出了从环境样品中直接克隆DNA 的想法,而后扩增子测序和鸟枪测序法的出现推动了这一学科的发展,一系列大型的测序计划接踵而至:由美国国立卫生研究院(NIH)发起的人类微生物组计划(iHMP)通过扩增子测序(阶段一)和宏基因组测序(阶段二)的方法加深了人们对于微生物菌群相关疾病的理解[70]。同时,研究人员也将目光从地球环境转向生物反应器,如,来自康奈尔大学的Angenent 课题组[71]通过对9 个生物反应器长达1 年的采样并对样品进行扩增子测序,绘制了每个生物反应器中的群落图谱,分析结果表明了每个生物反应器中都具有独特的高稳定性群落结构。近两年来,渐渐有课题组开始利用多组学联用技术对功能菌群的进行分析:卢森堡大学的Willems 课题组[72]通过宏基因组、宏转录组、宏蛋白质组和宏代谢组学联用的方法,对来自生物废水处理厂的含油微生物种群进行了分析,发现了群落中存在着广泛的表型可塑性和生态位互补性,并利用培养实验揭示了微生物生态系统是如何应对干扰的;香港大学张彤课题组[73]建立了宏基因组、宏转录组、靶向代谢组学联用的分析流程,从具有双酚A(BPA)生物降解功能的功能性菌群中识别出了参与种间相互作用的底物,并通过分离培养和共培养的方式证明了基于多组学整合分析方法构建出的复杂降解途径。相较于单一组学,尤其是在扩增子测序和宏基因组测序方面,多组学联用能够更为精准地刻画功能性菌群的时空景观,但是同时产生的海量数据也为分析带来极高的难度,大幅增加的数据量带来了算力需求提升以及如何进行多组学之间的融合分析[74]等诸多问题。
3.2 宏组学数据的主要处理流程与分析软件
宏组学数据的主要处理软件见表1。

表1 宏组学数据的主要处理软件Tab.1 Major software for processing meta-omics data
宏组学数据的处理难点主要产生于宏基因组和宏转录组的处理过程中如何从扩增子和鸟枪测序法的数据中解析出整个生物群落的整体概况--主要是针对基因序列的处理。宏蛋白组、宏代谢组两种组学的处理方式和一般蛋白组学和代谢组学方法较为一致,本文此处不再赘述。
目前,处理“宏基因组”数据的主要流程软件 包 括QIIME[75]、QIIME2[76]、bioBakery[77]和metaWRAP[78]等。其中,由UCSD 的Knight 课题组开发的QIIME2,是QIIME 的升级版本,由开源软件Python 和R 编写,具有便利的cli、Artifact API、Studio三种操作模式。其主要对扩增子数据进行分析,功能包括:下机数据的预处理、质量控制、聚类获得操作分类单元(operating taxonomic units,OTUs)、利用DADA2[89]降噪获得扩增子序列变体(amplicon sequence variant,ASV)、特征表的生成、群落多样性指标的计算、群落发育树的建立等,涵盖了扩增子测序数据处理和分析的方方面面。bioBakery 和metaWRAP 主要针对宏基因组数据,分别采用基于比对和基于组装两种不同的分析方式。由哈佛大学Huttenhower 课题组研开发的bioBakery 支持多组学的数据整合,同时支持扩增子数据分析。整体流程整合了Kneaddata[90]用于对宏基因组测序数据进行质量控制;MetaPhlAn[91]用于从宏基因组鸟枪测序数据中分析微生物群落的组成;HUMAnN[92]用于从宏基因组或宏转录组测序数据中准确地分析大量微生物代谢途径和其他分子功能;StrainPhlAn[93]用于群落内菌株级别解析度的分析和群落内系统发育树的建立与分析。metaWRAP通过组装的方式对宏基因组测序数据进行分析:metaWRAP-Read_qc模块对原始数据进预处理和质量控制,MegaHit[94]、metaSPAdes[95]或metaWRAP-Assembly 用于序列组装,Kraken[96]用来对组装的序列进行快速物种分类和丰度分析、MaxBin2[97]、metaBAT2[98]、CONCOCT[99]3 个软件用于对组装的序列进行进一步分箱。在宏基因组学数据处理的基础上,基因组尺度代谢模型(genome-scale metabolic model,GEM)重建可以由CarveMe 完成[100]。在基因组尺度代谢模型重建的基础上,目前能够实现直接静态模拟功能(例如,通量平衡分析FBA、动态通量平衡分析dFBA等)的工具只有metaGEM 这一种[101]。群落动态模型可以由广义Lotka-Volterra 模型实现,但是尚未有人提出从分析时序性的多组学数据到生成群落含时的动态模型与模拟的标准处理流程,也尚未有人提出与从分析多组学的空间面板数据到生成群落区域关系的标准处理流程。
在组学算法不完善和样本质量不够稳定的影响下,利用宏组学研究群落动态存在如下缺点[65];代谢网络重建不准确和不完整;许多基因、蛋白质和代谢物的功能未知;对驱动个体和群落水平表型的进化的原因缺乏理解;对基因、代谢和生态系统调节体系(例如,群体感应信号-响应系统)的理解有限。而且越来越大的数据量亟需一个高效的信息处理流程。
4 挑战与展望
尽管利用上述两种策略(bottom-up&top-down)设计功能菌群的研究已经取得较大的成功,但是由于技术所限,更多、更广泛的功能菌群的设计与合成存在很大的挑战:诸如计算能力、数学模型的缺失以及巨大的实验工作量等。而功能性菌群作为一个复杂群体,单独的“自下而上”和“自上而下”已经不足以用来设计这样复杂的系统。威斯康星大学麦迪逊分校的McMahon 教授[38]通过总结两种策略设计功能性菌群的现有研究,提出“整合设计(integrated design)”的概念来解决两种策略的固有缺点:将基于过程的模型(topdown 策略)与利用代谢组学重构的代谢模型(bottom-up策略)合并,以模拟生态系统过程、质量平衡关系和代谢通量,并使用基因组及其衍生信息以制定菌群的设计策略;但是McMahon 教授并没提出可行的流程。
近些年来人工智能技术[37,102]的兴起,为解决“宏组学”产生的菌群大数据带来了良兵利器[103]。结合了人工智能和整合设计的方针为功能性菌群的建立引入了“智能设计”策略。以下3点问题有望在短时间内获得较好的解决方案:
(1)区域范围内功能性菌群的时空变化关系
区域是多尺度的,从反应器级别到市、省、国家、地球区域,对于各尺度区域中菌群的时空变化的理解,有利于研究人员开展微尺度区域下功能性菌群的时空控制与培养和进行大尺度区域下功能性菌群的采集。然而,对于此方向的研究,研究人员一般采用解释性强但泛化性差的线性模型[104]。近期,诸如一系列基于深度学习的时空数据模型[105]以及神经网络解释性的研究[106]的发展,有助于提高现有模型的泛化性并保持一定的解释性。
(2)结合“宏组学”与深度学习的多组学群落分析流程
随着测序价格下降带来的海量组学数据,需要一个可行、高效的信息处理流程。近年来,深度学习在解决超高维问题中的优越性使其在生物学问题中具有广泛的应用:研究人员使用机器学习设计蛋白质序列[107]、处理组学数据[108]等。机器学习在“宏组学”数据的处理上,包括OUT 聚类、分箱等已经有了许多的应用[103]。可以预见的是,接下来的重心应该向“如何对特征表或是其他传统算法输出结果的进行整合”这一方向发展。宏组学数据分析产生了一系列的图数据[109],包括代谢网络、蛋白互作网络等,随着近两年来图神经网络的发展[110],正在逐渐地转化为一个可操作的数据源;多模态学习[111]的发展则给予了多组学数据融合问题一个可以接入的框架。
(3)功能性菌群内分布式代谢回路的计算设计
传统合成生物学通过基于模型化的思想,构建了一系列通量平衡分析模型来对底盘生物的代谢回路进行建模、模拟、预测与改造,发展了基于约束的代谢回路设计的计算方法[112]。原位工程的发展也已经给予研究人员工程化复杂群落的能力[60-64],但反观在复杂群落中设计代谢回路,由于群落中的微生物种类和数量、关系复杂性的上升,基于约束的代谢回路设计方式已经不再适用[38]。但随着近年来强化学习的发展[113],当把已经构建好的群落动态模型作为一个交互环境,我们仍然有希望在群落级别下的高维优化问题中获得令人满意的结果。
功能性菌群的构建作为一个热门的话题,该方面的研究现如今已经有了令人可喜的成果,但也存在一些问题和挑战。随着组学技术的不断发展和“智能设计”的引入,相信不久的将来这个领域将获得更大的突破。

