基于CRISPR 序列的肠道沙门氏菌分离株CSST 分型
徐 越,张丽霞,王茜月,管飘萍,刘文博
(1.扬州大学兽医学院,江苏扬州 225009;2.江苏省动物重要疫病与人兽共患病防控协同创新中心,江苏扬州 225009)
沙门氏菌是一种常见的食源性致病菌[1]。污染的生鸡蛋、蛋制品和未充分加热的肉品是人类感染沙门氏菌的主要途径,在我国70%~80%的食源性细菌病是由沙门氏菌引起的[2]。沙门氏菌属是肠杆菌科的重要成员,有2 个种、42 个血清群、2 500 多个血清型,我国已报道的血清型达240 种以上[3]。不同血清型沙门氏菌的侵袭力与致病力显著不同,所以探寻沙门氏菌的分型方法对鉴别控制由其引起的疾病具有重要意义。
成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)是细菌防御外源遗传物质入侵的“免疫机制”[4]。CRISPR 系统的结构可以简单描述为由前导序列(leader)、重复序列(direct repeats)、间隔序列(spacers)以及CRISPR相关蛋白基因(cas)组成[5],其中重复序列和间隔序列相互交错组成CRISPR 位点。而本试验的分类依据——间隔序列,来源于外源核苷酸,且可以用来抵御同源的外源核苷酸入侵,所以间隔序列中包含着细菌遗传进化信息[6]及其特有的生态学、地理学信息。CRISPR 序列的长度取决于间隔序列的数量,其在不同有机体之间以及不同细菌血清型或菌株之间差异很大,而这些信息均可以用来对含有此结构的细菌进行分型[7]。
沙门氏菌基因组中也存在这种“免疫机制”,其包含着沙门氏菌的遗传进化信息,因而能够用于沙门氏菌分型[8]。沙门氏菌存在2 个CRISPR 位点——CRISPR1 和CRISPR2。根据这两个位点的各种信息可以对沙门氏菌进行分型,尤其是因为间隔序列的插入或丢失,形成了间隔区特有的多态性,间接造成了细菌CRISPR 位点的多态性。所以利用CRISPR 序列中间隔序列的顺序作为细菌分型依据的方法逐渐得到应用。这种方法分辨力高,在亚型的分型中也有较好的前景。传统CRISPR 分型方法需要测出沙门氏菌2 个CRISPR 位点的全部间隔序列,并且需将全部间隔序列绘制成间隔序列图谱,再根据图谱进行细菌分型,虽然分辨率较高,但耗时耗力。目前已出现了许多新型的CRISPR 分型方法。例如:CRISPR 基因可以与毒力基因结合形成CRISPR-MVLST,从而对细菌进行分型,其在对肠道沙门氏菌分型中优于PFGE 分型方法;还有CLSPT(CRISPR loci spacer-pair typing)分型方法,各取CRISPR1 和CRISPR2 中靠近leader 的1 个间隔序列绘制图谱进行分型[9],但分型效果不佳;经改良还出现了各取3 个距离leader 最远或者是菌株进化过程中最先插入3 个间隔序列的CSST(CRISPR initial three spacers sequence typing) 分型方法[8]。这种方法既保留了CLSPT 分型方法快速、高效、简单的优点,又补足了CLSPT 分型方法的不足,而且还延续了传统CRISPR 分型方法的准确性,同时又不需要耗时耗力地测出并绘制全部间隔序列。为验证CSST 分型方法的可行性,本试验先采用传统CRISPR 分型方法进行测序,然后在数据分析阶段分别用传统CRISPR 分型方法和CSST 分型方法对20 株肠道沙门氏菌进行分型。
1 材料
1.1 菌种
24 株鸡源肠道沙门氏菌,2013—2017 年从江苏省分离(由于试验操作等原因,有4 株被剔除,只有20 株参与分型分析),由扬州大学兽医学院传染病实验室分离鉴定保存,信息见表1。
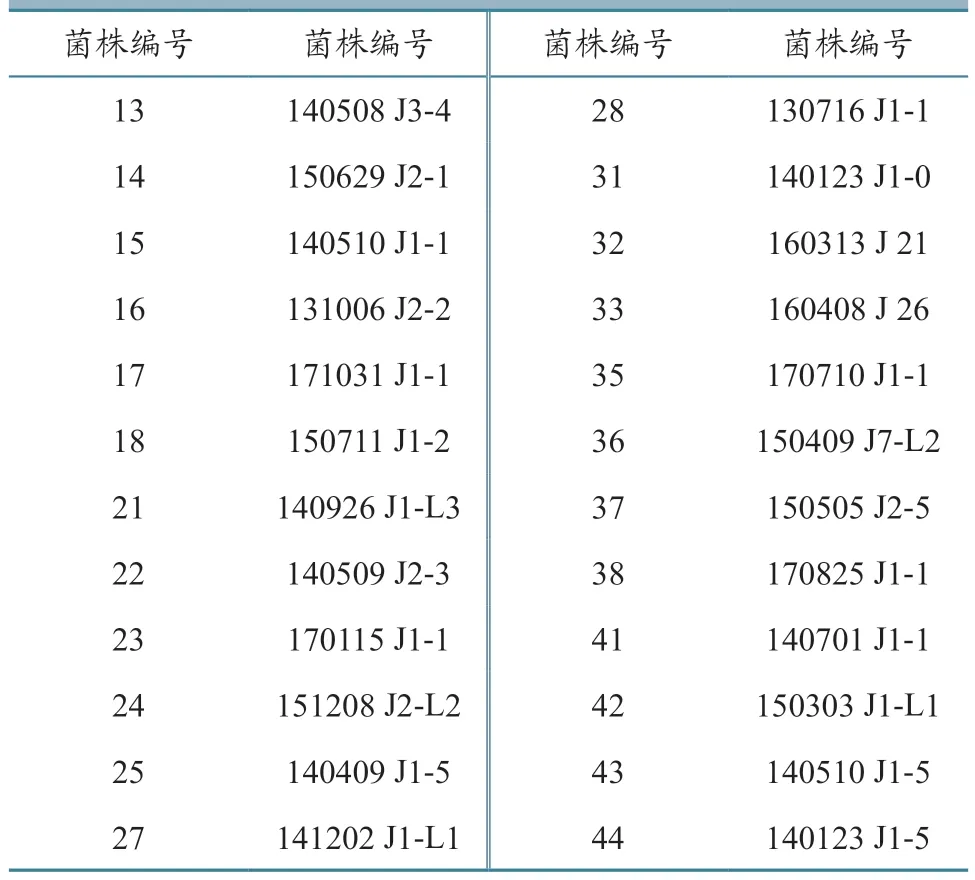
表1 菌株信息
1.2 主要仪器
生物安全柜,型号CLASS2 YPEA2,购自Thermo 公司;电子天平,型号FA2004,购自上海精密仪器仪表有限公司;奥林巴斯显微镜,型号BX53,购自日本奥林巴斯公司;高压灭菌锅,型号SX500,购自Tomy 公司;PCR 仪,型号9902,购自美国ABI 公司;电泳仪电泳凝胶成像系统,型号PowerPacTMBasicUniversal Hood II,购自美国Bio-Rad 公司。
1.3 主要试剂
SS 琼脂,购自青岛海博生物技术有限公司;16S rDNA Bacterial Identification PCR Kit,购自宝生物工程(大连)有限公司。
1.4 CRISPR 引物
参照庄孝飞[7]设计的引物(表2),送生工生物工程(上海)股份有限公司合成。

表2 CRISPR 引物序列信息
2 方法
2.1 菌株复苏
挑取解冻菌液划线接种于无菌SS 琼脂培养基上,37 ℃培养24 h,然后选取单个菌落继续纯化培养。
2.2 菌株鉴定
采用16S rRNA 基因扩增和BLAST 序列分析方法进行菌株鉴定[10]。PCR 扩增体系(总体积为25.00 μL):Mix 12.50 μL,引物各0.25 μL,模板DNA 2.00 μL,ddH2O 10.00 μL。PCR 扩增程序:95 ℃预变性10.0 min;94 ℃变性1 min,60.5 ℃/52.0 ℃(CRISPR1、2 位点引物的退火温度见表2)退火1.0 min,72 ℃延伸1.5 min,共50 个循环;72 ℃延伸5.0 min,4 ℃保存。
采用煮沸裂解法制备细菌基因组模板:挑取纯培养后的单个菌落放于装有200 μL 灭菌双蒸水的灭菌管中,100 ℃处理10 min,5 000 r/min 离心5 min,取上清作为PCR 模板,根据TaKaRa 16S rDNA Bacterial Identification PCR Kit 说明进行PCR扩增;取5 μL PCR 扩增产物,经1%琼脂糖凝胶电泳检测,将大小正确的扩增产物,送南京擎科生物工程有限公司测序。
2.3 CRISPR 分型
参照2.2 的PCR 方法,以及CRISPR1、CRISPR2 位点引物的退火温度(表2)进行PCR扩增;配制1%琼脂糖凝胶,取5 μL 扩增产物电泳;电泳结束后,EB 染色,凝胶成像系统中观察记录结果;将纯化的DNA,经1%琼脂糖凝胶电泳验证后,送南京擎科生物工程有限公司测序。
2.4 数据分析
将20 株不同血清型沙门氏菌菌株CRISPR1和CRISPR2 位点序列拼接得到相关序列。
借助CRISPR 数据库中的在线工具CRISPR finder(http://crispr.u-psud.fr/Server/)找出序列中的间隔序列及重复序列;将其中的重复序列去掉,保留间隔序列且赋予不同的背景颜色,再将其按顺序组合在一起,绘制成CRISPR1 和CRISPR2 间隔序列图谱。对于相同血清型来说,一种颜色表示一种间隔序列,不同颜色表示不同的间隔序列,重复的间隔序列则用相同的颜色表示;对于不同血清型而言,不同间隔序列用不同形状和颜色表示,与其他血清型序列相同的间隔序列用已有颜色表示,但不同血清型之间,表示间隔序列的形状不同。
本试验所用菌种皆为肠道沙门氏菌,所以间隔序列均选用矩形表示。传统CRISPR 分型,取CRISPR1 和CRISPR2 的全部间隔序列绘成图谱;而CSST 分型,取CRISPR1 和CRISPR2 序列中前3 个间隔序列绘成图谱。
3 结果与分析
3.1 菌株复苏
SS 琼脂培养基上出现中间黑色、边缘灰白色的透明小菌落,且没有杂菌生长。
3.2 菌株鉴定
测序序列经过BLAST 序列分析,确定20 株细菌均为肠道沙门氏菌,分为4个血清变异亚型(表3)。
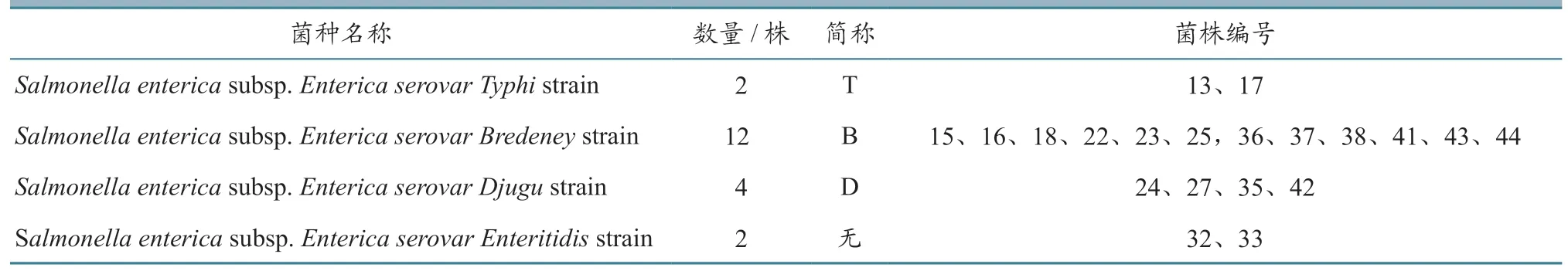
表3 20 株沙门氏菌分离株的血清亚型
3.3 CRISPR 分型
3.3.1 琼脂糖凝胶电泳 结果(图1~2)显示:20 株肠道沙门氏菌的CRISPR1、CRISPR2 条带大小差异较小,其中CRISPR1 均为650 bp 左右,CRISPR2 均为450 bp 左右(10 号孔D24 菌株条带略小)。
3.3.2 重复序列 20 株沙门氏菌的CRISPR1 位点处第一个重复序列均为5'-CGTGTTTATCCCCGCTGACGCGGGGAAACT-3',固定重复序列(repeat sequence)均为5'-CCGGTTTATCCCCGCTGGCGCGGGGAACAC-3';CRISPR2 位点处的第一个重复序列均为5'-ACGGCTATCCTTGTTGGCGCGGGGAACAC-3',固定重复序列(repeat sequence)均 为5'-CGGTTTATCCCCGCTGACGCGGGGAACAC-3'。本试验的20 株菌株,根据16S rRNA 基因分型方法[10]分为4 种亚型。各菌株CRISPR1、CRISPR2 位点处的第一个重复序列和固定重复序列(repeat sequence)均相同,说明这两个重复序列在沙门氏菌种内相对保守[11]。
3.3.3 间隔序列 本试验共测出4 种CRISPR1 间隔序列,41 种CRISPR2 间隔序列(图3)。在同种肠道沙门氏菌中,CRISPR2 间隔序列的种类比CRISPR1 丰富的多(图4),说明CRISPR2 是同种亚型沙门氏菌进化较快的序列。此外还发现,许多间隔序列同源性很高,例如2.8、2.12、2.33、2.40间隔序列,还有2.9、2.13、2.18、2.34、2.41 间隔序列(表4),与Fei 等[12]试验得出的一些间隔序列有较高同源性的结果相一致。

表4 部分肠道沙门氏菌菌株测出的CRISPR 基因中相似间隔序列
3.3.4 传统CRISPR 分型 20 株临床肠道沙门氏菌被分成了15 种亚型,其中CR4 和CR9 为最优势CR 型,分别占总数的20%和15%,其他CR型呈零星分布(表5、图5)。
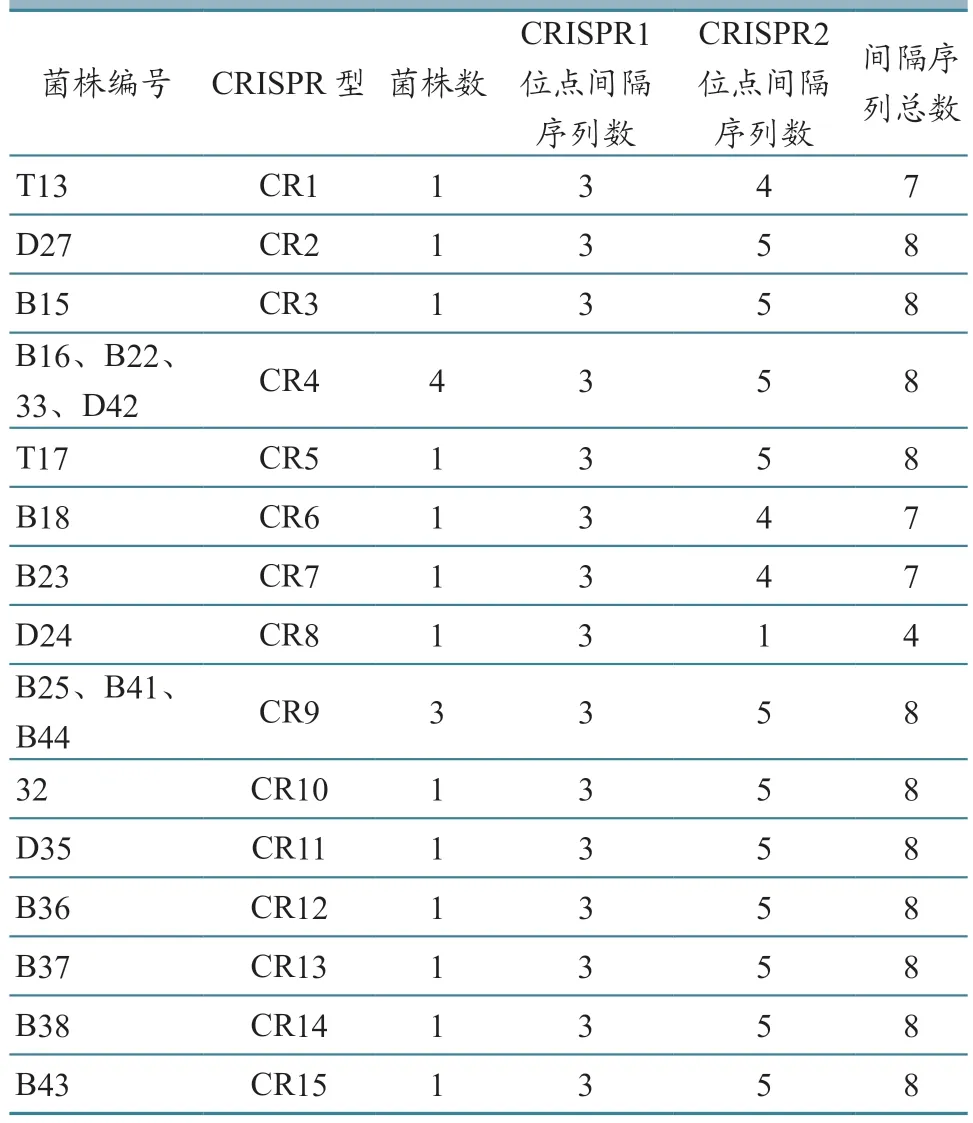
表5 20 株沙门氏菌15 种CRISPR 分型结果
3.3.5 CSST 分型 20 株临床肠道沙门氏菌被分成了15 种亚型,其中CT4 和CT9 为最优势CT 型,其他CT 型均呈零星分布(表6、图6),与传统CRISPR 分型完全一致。
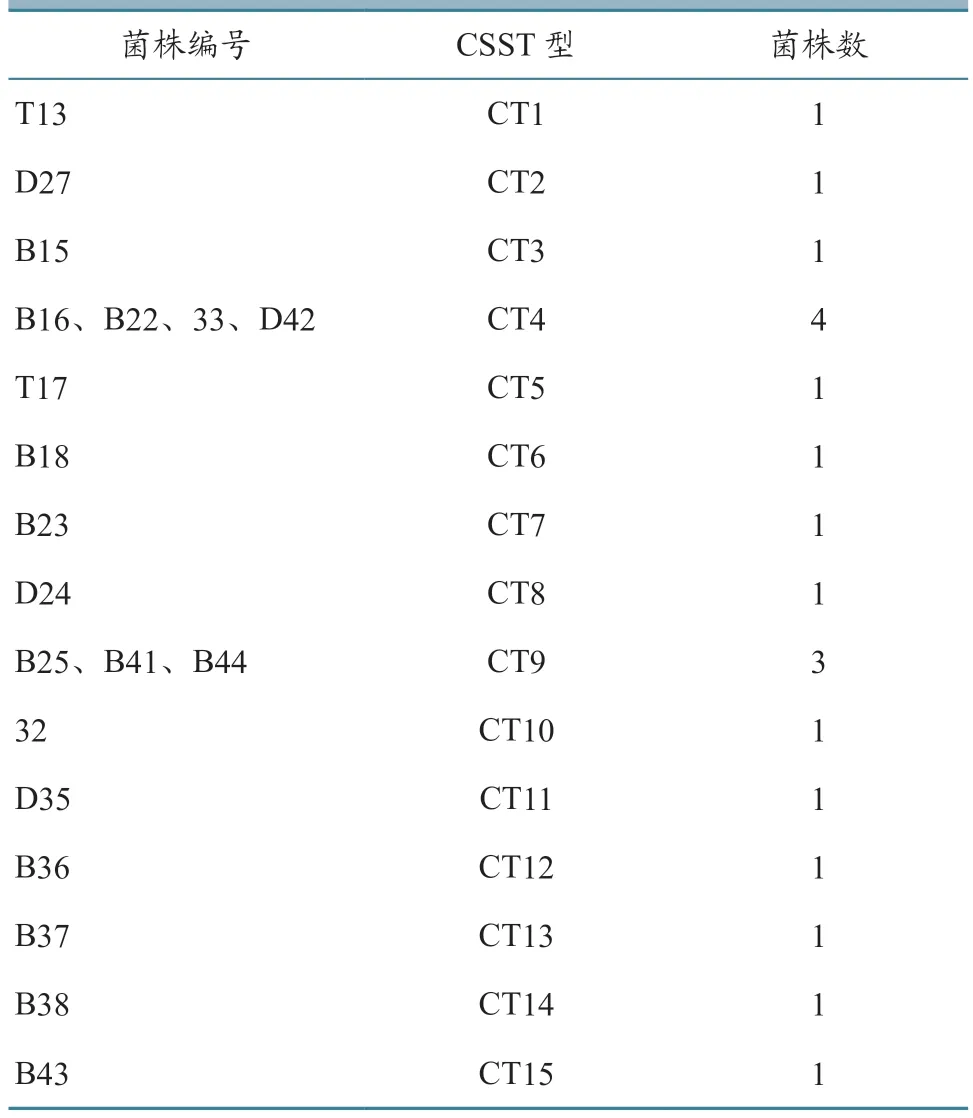
表6 20 株沙门氏菌CSST 分型结果
4 讨论
CRISPR 序列不仅可以作为沙门氏菌的“免疫机制”,而且其中的重要结构——间隔序列也在沙门氏菌分型中有着重要作用。间隔序列在进化中具有良好的“时钟”性质,所以其包含着沙门氏菌的独特分子“指纹”遗传进化等信息,使其除了能够用于沙门氏菌与其他表型类似菌种的区分,还能用于沙门氏菌种间的分型。本试验是根据沙门氏菌存在2 个CRISPR 位点(CRISPR1 和CRISPR2)的间隔序列信息进行20 株沙门氏菌的种间分型:先采用传统CRISPR 分型方法,然后在数据分析阶段分别用传统CRISPR 分型方法和CSST 分型方法,对20 株肠道沙门氏菌进行分型,均成功将20 株临床肠道沙门氏菌分成了15 种亚型,验证了CSST分型方法取代传统CRISPR 分型方法的可行性。
由本试验结果得知,肠道沙门氏菌CRISPR1的保守性较CRISPR2 高,推测CRISPR2 进化的发生较CRISPR1 晚。根据CRISPR2 间隔序列具有一定同源性,猜测在沙门氏菌获得外源性DNA 且用其来防御同源序列的入侵者过程中。每个细菌最初获得的外源DNA 并不完全一样,随后根据环境以及细菌生存情况留下最适宜的外源核苷酸片段,所以会留下同源性较高的CRISPR2 间隔序列,但这还需进一步证明。从图3 可以看出,CRISPR 间隔序列的大小差异不大,具有一定保守性,最大的为38 bp(编号2.34 的间隔序列),最小的为32 bp。
通过图5~6 可以发现:20 株肠道沙门氏菌CRISPR1 的3 个间隔序列相似性很高,可以用其作为肠道沙门氏菌与其他细菌的分型依据;而CRISPR2 的间隔序列种类很多,同源性较CRISPR1 略低,但其依旧具有一定的保守性。因此,CRISPR1 适合用于细菌种的分型,而CRISPR2 可以用于更细致的分型,例如亚型的分型,但这需更进一步研究。
由试验可知,使用传统CRISPR 分型方法,20 株肠道沙门氏菌被分成了15 种CRISPR 型(简称CR 型)。由于试验菌株较少,无法得出16S rRNA 分型方法与CRISPR 分型方法的联系与不同,但CSST 分型方法的分型结果与传统CRISPR 分型方法完全一致。
本试验所用的20 株菌株为本实验室在不同时间从江苏省不同养殖场分离得到的,其CRISPR 分型和CSST 分型结果与时间、地点等因素没有明显关联。但不同时间或地点的菌株属于同一CR/CT型(CR4/CT4 型、CR9/CT9 型),说明同种CR/CT 型的肠道沙门氏菌在江苏省持续存在,且沿着某种传播途径传播,这为控制肠道沙门氏菌进一步传播提供了指导。菌株的CR/CT 型相同,即菌株CRISPR 的间隔序列相同,其中包含的遗传进化信息、生态学和地理学信息也相同,这为研究菌株的起源和进化提供了重要信息。
本试验证明,采用CSST 分型方法替代传统CRISPR 分型方法是可行的,在专属于CSST 分型方法的引物出现之前,二者的试验方法是一致的,在试验阶段并无优劣之分。但在数据分析阶段,传统CRISPR 分型需要取CRISPR1 和CRISPR2 的全部间隔序列绘成图谱,而绘制图谱则需要去掉重复序列并将每一种间隔序列赋予不同的背景颜色,再将其按顺序组合在一起;而对于不同血清型而言,不同间隔序列还要用不同的形状来表示。因此,在用CRISPR 间隔序列对沙门氏菌进行分型时,所选取的间隔序列越少越好,但间隔序列太少又不能保证分型方法的准确性和精确度,所以需要取CRISPR1 和CRISPR2 序列中距离leader 最远或者是菌株进化过程中最先插入的3 个间隔序列绘成图谱的CSST 分型方法成为最优的尝试。本试验结果显示,CSST 分型方法与传统CRISPR 分型方法的分型结果完全一致,在试验方法相同的前提下,CSST 分型方法数据分析更加省时省力,有着快速、高效、简单的优点,且还延续了传统CRISPR分型方法的准确性,可替代数据分析繁琐的传统CRISPR 分型方法,成为流行病学溯源的重要技术方法。

