氧化应激状态下心肌细胞中长链非编码RNA的差异表达谱分析
李哲 刘子倩 王胤 曾焕玉 李培峰

摘要:
为了寻找氧化应激状态下与心脏疾病有关的lncRNA,采用基因芯片法对正常心肌细胞和H2O2诱导的心肌细胞进行lncRNA和mRNA表达图谱分析,通过基因本体论和通路分析探索差异表达基因的功能,构建lncRNA和mRNA共表达网络,从差异表达的lncRNA中选取10组上调和10组下调差异显著的lncRNA进行RT-qPCR验证。研究结果表明,在正常心肌细胞和氧化应激下的心肌细胞中,lncRNA和mRNA的表达均有差异,在差异上调显著的10组lncRNA中,RT-qPCR检测结果与芯片结果一致,这表示它们可能是心脏疾病的潜在治疗靶点。
关键词:
芯片分析;lncRNA;基因本体论分析;通路分析;共表达网络;RT-qPCR
中图分类号: R735.7
文献标志码:A
收稿日期:2021-04-25
基金项目:
国家自然科学基金海外青年基金(批准号:82050410450、81850410551)资助;山东省自然科学基金(批准号:ZR2019BH014)资助。
通信作者:
曾焕玉(AUNG Lynn Htet Htet),女,博士,主要研究方向为利用高通量测序,生物信息学,细胞生物学方法研究衰老及心脑血管疾病的机理机制;李培峰,男,博士,教授,主要研究方向为神经元衰老,自由基生物医学研究,细胞凋亡与心血管疾病的研究,心血管疾病的分子机制研究等。E-mail:peifli@qdu.edu.cn
氧化应激诱导的心肌细胞凋亡在冠状动脉疾病[1]、心肌病[2]、心肌炎[3]、心力衰竭[4]和高血压[5]等多种类型心脏病的病理过程中起着重要作用[6],心肌细胞的凋亡通常始于心肌中活性氧的产生[7],而活性氧的产生会造成细胞积累大量的自由基[8],自由基不仅损伤组织和细胞[9],还能诱发心脏病[10]。目前,保护H9c2等心肌细胞免受氧化应激诱导凋亡的分子机制尚不清楚[11-12],因此寻找与氧化应激有关的潜在的治疗靶点迫在眉睫。长链非编码RNA(Long noncoding RNA,lncRNA)是指长度大于200 nt[13],序列保守且表达水平低的RNA[14],lncRNA在表观遗传[15]、转录[16]和转录后[17]水平上调节多种生物学功能,或直接调节蛋白活性[18],近年来,许多研究发现lncRNA是心脏类疾病的治疗靶点[19],在心脏再生[20]和修复[21]中起着至关重要的作用。在H2O2介导的氧化应激反应中,02 mmol H2O2仅诱导心肌细胞凋亡[22-23]。本文采用生物信息学方法筛选出差异表达的lncRNA和mRNA,对差异表达的基因进行富集分析,通过RT-qPCR实验对预测结果进行验证,为寻找心脏疾病的潜在治疗靶点、探究心脏疾病的发生发展提供理论基础。
1 材料与方法
1.1 样本
实验所用H9c2细胞来源于大鼠心肌细胞系,由中国科学院(上海)细胞库提供。从每组样本中提取总的RNA,通过琼脂糖凝胶电泳评估RNA完整性,采用Nanodrop ND-1000检测RNA的质量和数量。
1.2 微阵列分析
设计大鼠lncRNA阵列来识别lncRNA和蛋白编码基因。从Rat lncRNA Orthologo数据库、NCBI Refseq数据库和UCSC ALL-mRNA记录数据库中筛选出约9 000个lncRNA。
1.3 RNA標记和阵列杂交
按照安捷伦单色微阵列基因表达方法进行样品标记和阵列杂交。mRNA从总RNA中纯化,去除rRNA。对每个样本进行扩增,采用随机引物法转录成荧光cRNA。标记的cRNA用RNeasy Mini Kit纯化,用NanoDrop ND-1000测定标记cRNA的浓度和比活性。加入阻断剂和破碎缓冲液,将标记的cRNA片段化,60 ℃加热30 min,最后加入杂交缓冲液稀释标记cRNA。将杂交溶液组装到lncRNA芯片载玻片上,载玻片在杂交仪中65°C孵育17 h后用DNA微阵列扫描仪对杂交阵列进行清洗、固定和扫描。
1.4 数据分析
应用Agilent Feature Extraction软件(版本11011)分析采集的微阵列图像。使用genspring GX v1151软件包分析lncRNA和mRNA。筛选标准:P<005,logFC>2。
1.5 LncRNA的筛选和验证
从下调和上调差异表达的lncRNA中分别选出差异显著的10组lncRNA,进行RT-qPCR验证,分析其表达变化。
2 结果与讨论
2.1 两组样本中lncRNA和mRNA表达的变化
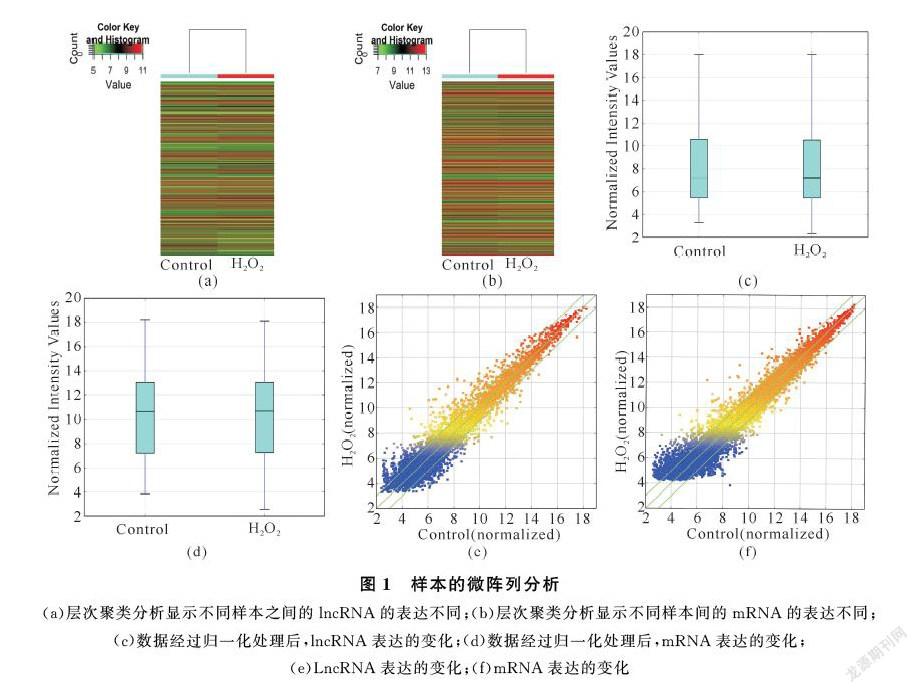
用基因芯片法对正常H9c2和H2O2诱导的H9c2两组样本中lncRNA和mRNA的表达图谱进行分析,如图1所示。图中“绿色”表示低相对表达,“红色”表示高相对表达。箱式图表示数据经归一化处理后,所有lncRNA和mRNA的数据分布基本相同。散点图中X轴和Y轴的值是两组样本数据归一化后的平均值,绿色的线代表变化倍数(默认的变化倍数值是20)。结果显示,正常H9c2细胞和H2O2处理的H9c2细胞中lncRNA的表达存在显著差异,共有1 097个显著失调的lncRNA,包括662个上调和435个下调的lncRNA。
2.2 差异表达基因的GO分析
GO分析包括生物过程分析、细胞成分分析和分子功能分析三个领域,如图2所示。在生物过程分析中,单生物过程、单生物细胞过程以及生物调控是与下调mRNA相关的三个最重要的过程;而单个生物体的过程、自然调节和对刺激的反应是与上调mRNA相关的三个最重要的过程。在细胞成分分析中,细胞外周、质膜以及细胞外区是与下调mRNA相关的三个最重要的过程;而膜部分、膜和细胞外周的内在组成部分与上调mRNA相关的三个最重要的过程。在分子功能分析中,结合,蛋白质结合以及离子结合是与下调mRNA相关的三个最重要的过程;而结合、蛋白质结合和转运蛋白活性是与上调mRNA相关的三个最重要的过程。
2.3 差异表达基因的通路分析
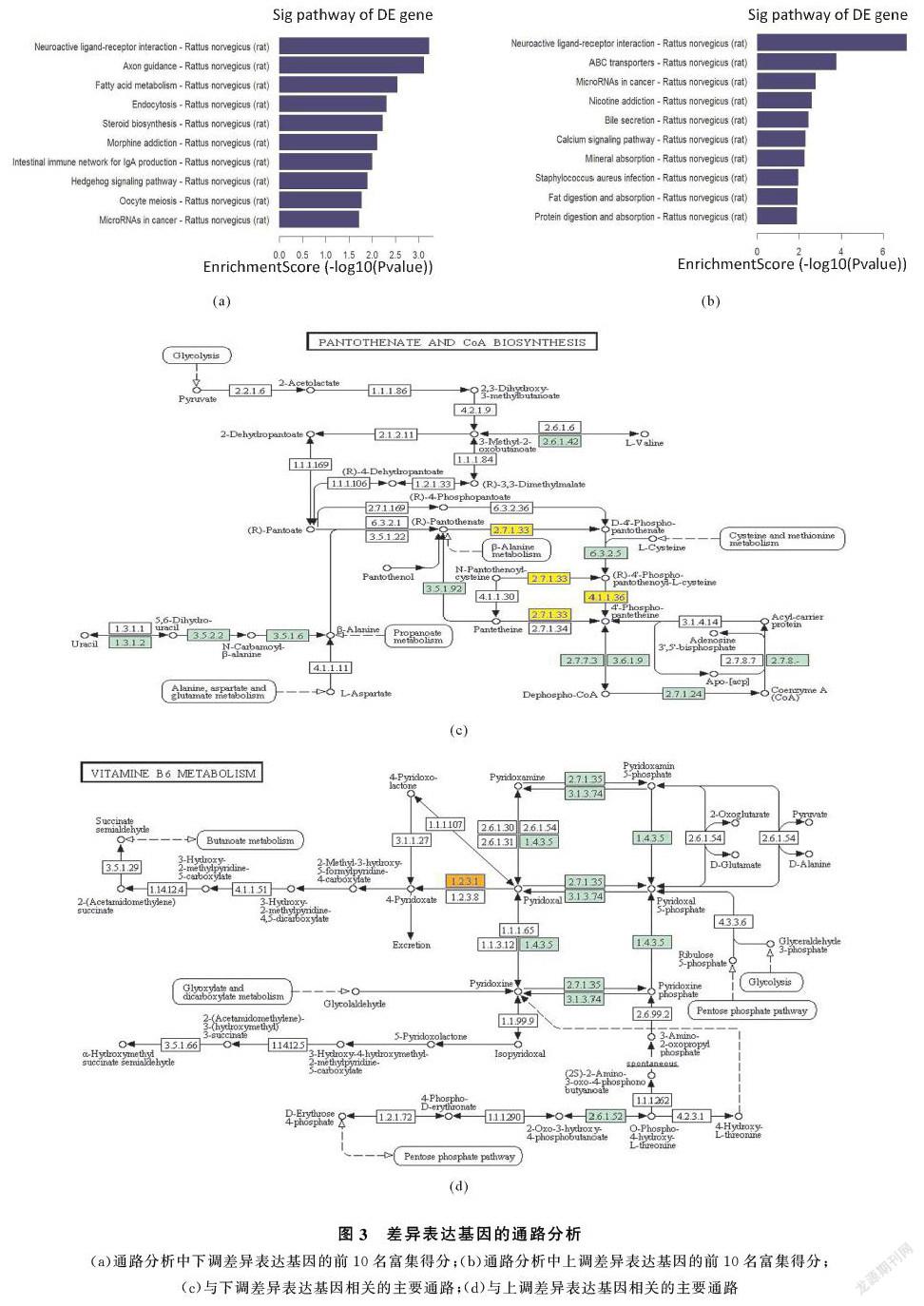
差异表达上调和下调的lncRNA的富集分析和通路分析如图3。富集分析显示,差异表达的lncRNA主要与细胞受体有关。通路分析鉴定了27条和51条由上调和下调的差异表达基因靶向的基因通路。
2.4 共表达网络的构建
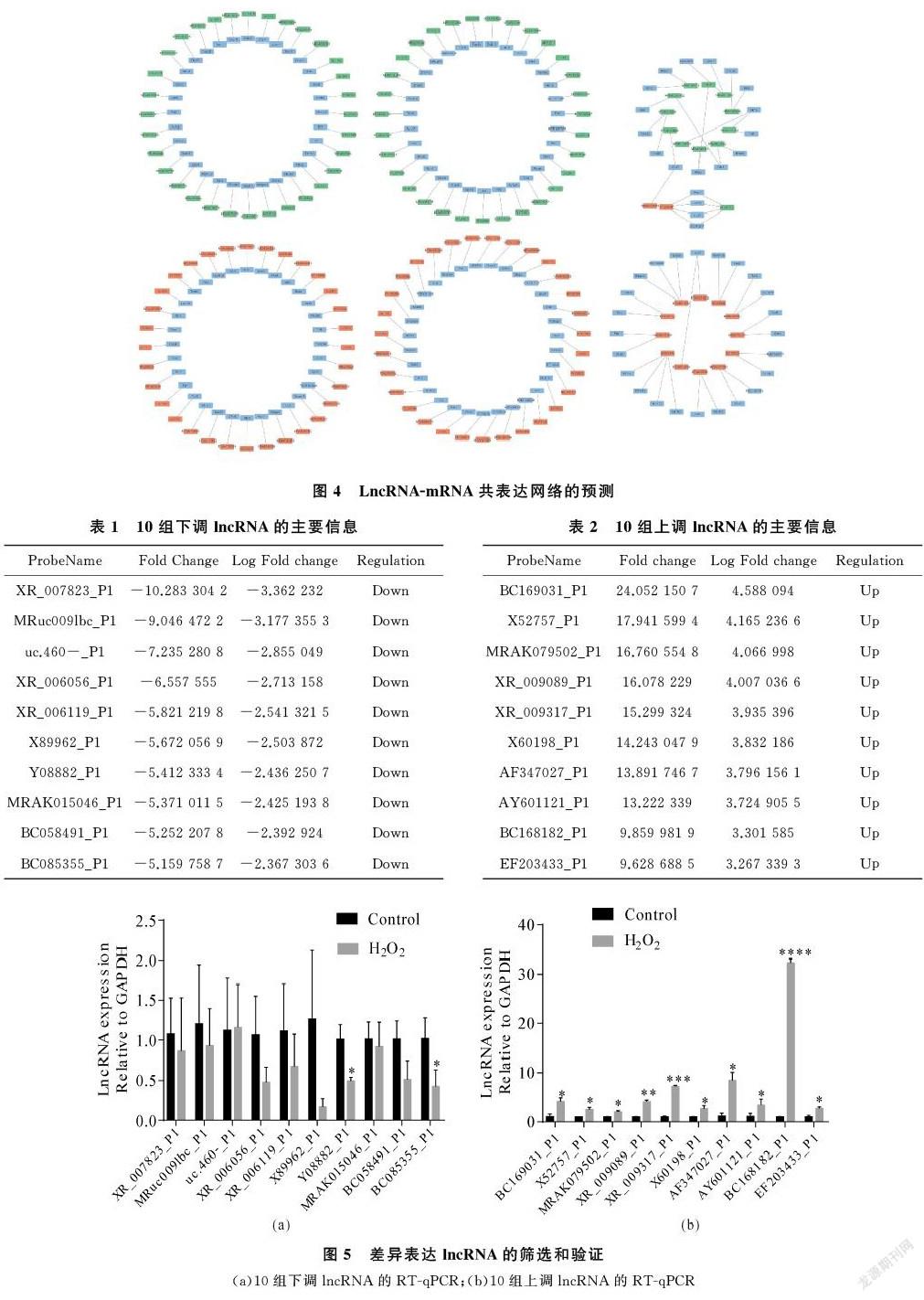
选取数个归一化高表达、高倍数变化的mRNA,将其叠加在lncRNA-mRNA相关网络上,以确定它们与lncRNA的关联。差异显著的基因被用于构建lncRNA-mRNA调控网络,如图4所示。在网络中,红色节点代表显著上调的基因,绿色节点代表显著下调的基因,蓝色节点表示lncRNA的编码基因。
2.5 差异表达lncRNA的筛选和验证
从下调和上调的差异lncRNA中分别选出10组差异显著的lncRNA,如表1、表2所示。用RT-qPCR对lncRNA的表达进行验证,如图5所示。RT-qPCR结果显示,在10组下调显著的lncRNA中有2组与芯片结果一致,其余的无意义。在上调显著的10组lncRNA中,RT-qPCR结果与芯片结果基本一致,因此可能是氧化应激的潜在治疗靶点。*P<005,** P <001,*** P <0001。
2.6 讨论
心脏疾病发病过程漫长,很难治愈,对人类健康造成了严重的威胁。随着生活水平的提高,心脏疾病的患病率和死亡率逐年增加,心脏疾病会造成心肌细胞的凋亡[24-25],而氧化应激在心肌细胞凋亡过程中扮演着重要的角色[26-27],因此研究氧化应激状态下心肌细胞凋亡的靶点尤为重要。
本研究使用基因芯片法,对两组样本的lncRNA和mRNA进行分析,筛选出1 097个差异表达的lncRNA,包括662个差异上调的lncRNA和435个差异下调的lncRNA。基因本体论分析显示,这些差异表达的lncRNA可能与蛋白质结合,从而参与细胞的代谢;RT-qPCR结果显示,在下调显著的10个lncRNA中,只有Y08882_P1和BC085355_P1在两组样本中的表达与芯片结果一致,而上调显著的10个lncRNA在两组样本中的表达均与芯片结果一致。
在动脉粥样硬化中lncRNA有表达调控的作用[28-29],通过与下游靶点的结合,从而调控与动脉粥样硬化有关的基因的表达[30]。在分子功能分析中,结合、蛋白质结合以及转运蛋白活性是与上调基因相关的三个最重要的过程,这提示10组差异上调的lncRNA可能通过与下游蛋白结合继而参与调控与心脏疾病有关基因的表达。
3 结论
本研究通过生物信息学和RT-qPCR等方法对样本进行分析,最终筛选出10个可能与心脏疾病有关的lncRNA,可能是心脏疾病潜在的治疗靶点,这为研究心脏疾病的机制提供了可靠的理论依据。本文还未详细研究与10组上调lncRNA有关的下游靶基因,今后将重点研究其下游通路。
参考文献
[1]MALAKAR A K, CHOUDHURY D, HALDER B, et al. A review on coronary artery disease, its risk factors, and therapeutics[J]. Journal of Cellular Physiology, 2019, 234(10): 16812-16823.
[2]SHI X X, LI W J, LIU H H, et al. The ROS/NF-kappa B/NR4A2 pathway is involved in H2O2 induced apoptosis of resident cardiac stem cells via autophagy[J]. Oncotarget, 2017, 8(44):77634-77648.
[3]HKIMIAN G, COMBES A. Myocarditis[J]. Revue Medecine Interne, 2017, 38(8): 531-538.
[4]MISHRA P, SAMANTA L. Oxidative stress and heart failure in altered thyroid states[J]. Scientific World Journal, 2012, 2012: 741861.
[5]OLIVERAS A, DE LA SIERRA A. Resistant hypertension: Patient characteristics, risk factors, co-morbidities and outcomes[J]. Journal of Human Hypertension, 2014, 28(4): 213-217.
[6]TSUTSU H, KINUGAWA S, MATSUSHIMA S. Oxidative stress and heart failure[J]. American Journal of Physiology Heart and Circulatory Physiology, 2011, 301(6):H2181-H2190.
[7]YU B W, LI J L, GUO B B, et al. Chlorogenic acid analogues from gynura nepalensis protect H9c2 cardiomyoblasts against H2O2-induced apoptosis[J]. Acta Pharmacologica Sinica, 2016, 37(11): 1413-1422.
[8]黃海兰, 徐波. 米口袋清除自由基活性及其成分研究[J]. 青岛大学学报(自然科学版), 2006, 19(4): 30-35.
[9]劉召, 刘玉琳. 中医药清除自由基的研究现状[J]. 西部中医药, 2007, 20(10): 55-57.
[10] 胡博路, 孟洁. 30种中草药清除自由基的研究[J]. 青岛大学学报(自然科学版), 2000, 13(2): 38-40.
[11] REN G, QIAO H X, YANG J, et al. Protective effects of steroids from allium chinense against H2O2-induced oxidative stress in rat cardiac H9c2 cells[J]. Phytotherapy Research, 2010, 24(3): 404-409.
[12] LIU C H, LU D Y, YOU J R, et al. Efficacy of water fraction from dioscorea cirrhosa on oxidative stress and apoptosis in H9c2 cardiomyocytes induced by H2O2[J]. Journal of Traditional Chinese Medicine, 2021, 41(1): 51-58.
[13] KOPP F, MENDELL J T. Functional classification and experimental dissection of long noncoding RNAs[J]. Cell, 2018, 172(3): 393-407.
[14] ST LAURENT G, WAHLESTEDT C, KAPRANOV P. The landscape of long noncoding RNA classification[J]. Trends in Genetics, 2015, 31(5): 239-251.
[15] MERCER T R, MATTICK J S. Structure and function of long noncoding RNAs in epigenetic regulation[J]. Nature Structural & Molecular Biology, 2013, 20(3): 300-307.
[16] CHOI S W, KIM H W, NAM J W. The small peptide world in long noncoding RNAs[J]. Briefings in Bioinformatics, 2019, 20(5): 1853-1864.
[17] SUN X, ZHENG H X, SUI N. Regulation mechanism of long non-coding RNA in plant response to stress[J]. Biochemical and Biophysical Research Communications, 2018, 503(2): 402-407.
[18] HUANG Y. The novel regulatory role of lncRNA-miRNA-mRNA axis in cardiovascular diseases[J]. Journal of Cellular and Molecular Medicine, 2018, 22(12): 5768-5775.
[19] HE L, CHEN Y, HAO S Q, et al. Uncovering novel landscape of cardiovascular diseases and therapeutic targets for cardioprotection via long noncoding RNA-miRNA-mRNA axes[J]. Epigenomics, 2018, 10(5): 661-671.
[20] CAI B Z, MA W Y, DING F Z, et al. The long noncoding RNA CAREL controls cardiac regeneration[J]. Journal of the American College of Cardiology, 2018, 72(5): 534-550.
[21] PONNUSAMY M, LIU F, ZHANG Y H, et al. Long noncoding RNA CPR (cardiomyocyte proliferation regulator) regulates cardiomyocyte proliferation and cardiac repair[J]. Circulation, 2019, 139(23): 2668-2684.
[22] AUNG L H H, LI Y Z, YU H, et al. Mitochondrial protein 18 is a positive apoptotic regulator in cardiomyocytes under oxidative stress[J]. Clinical Science, 2019, 133(9): 1067-1084.
[23] KEMP T J, CAUSTON H C, CLERK A. Changes in gene expression induced by H2O2 in cardiac myocytes[J]. Biochemical and Biophysical Research Communications, 2003, 307(2): 416-421.
[24] SU Q, LV X. Revealing new landscape of cardiovascular disease through circular RNA-miRNA-mRNA axis[J]. Genomics, 2020, 112(2): 1680-1685.
[25] QI W C, LI Z H, XIA L J, et al. LncRNA GABPB1-AS1 and GABPB1 regulate oxidative stress during erastin-induced ferroptosis in HepG2 hepatocellular carcinoma cells[J]. Scientific Reports, 2019, 9(1): 16185.
[26] LI M, DUAN L W, LI Y X, et al. Long noncoding RNA/circular noncoding RNA-miRNA-mRNA axes in cardiovascular diseases[J]. Life Sciences, 2019, 233: 116440.
[27] ZENG J, ZHU L, LIU J, et al. Metformin protects against oxidative stress injury induced by ischemia/reperfusion via regulation of the lncRNA-H19/miR-148a-3p/Rock2 axis[J]. Oxidative Medicine and Cellular Longevity, 2019, 2019: 8768327.
[28] KUMAR S, WILLIAMS D, SUR S, et al. Role of flow-sensitive microRNAs and long noncoding RNAs in vascular dysfunction and atherosclerosis[J]. Vascular Pharmacology, 2019, 114: 76-92.
[29] REN K, XU X D, YU X H, et al. LncRNA-modulated autophagy in plaque cells: A new paradigm of gene regulation in atherosclerosis?[J]. Aging (Albany NY), 2020, 12(21): 22335-22349.
[30] PIERCE J B, FEINBERG M W. Long noncoding RNAs in atherosclerosis and vascular injury: Pathobiology, biomarkers, and targets for therapy[J]. Arteriosclerosis Thrombosis and Vascular Biology, 2020, 40(9): 2002-2017.
Abstract:
In order to search for lncRNA related to heart disease under oxidative stress, the expression profiles of lncRNA and mRNA in normal cardiomyocytes and H2O2-induced cardiomyocytes were analyzed by microarray analysis. Then, the function of differentially expressed gene was explored through Gene Ontology (GO) and pathway analysis, and construct a lncRNA-mRNA co-expression network. Among the differentially expressed lncRNA, 10 lncRNA significantly differentially up-regulated and 10 down-regulated groups were selected for RT-qPCR verification. The results show that the expressions of lncRNA and mRNA were different in normal cardiomyocytes and in cardiomyocytes under oxidative stress. In the ten groups of significantly upregulated lncRNA, RT-qPCR detection results were consistent with the microarray results, indicating that they may be potential therapeutic targets for heart disease.
Keywords:
microarray; lncRNA; Go analysis; pathaway analysis; co-expression network; RT-qPCR

