基于GEO数据库分析影响口腔鳞状细胞癌的关键基因
赵军 刘丽娜
中图分类号:R739.8 文献标志码:A doi:10.3969/j.issn.1001-3733.2021.06.018
口腔癌是世界上第六大常见恶性肿瘤,死亡率高,预后差。口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是口腔癌的主要病理类型,约占口腔癌的90%,可发生于口腔的不同部位,如舌、牙龈、口腔黏膜、硬腭、嘴唇和口腔底[1]。尽管外科手术、放化疗和免疫治疗取得了一定的进展,但由于其侵袭、转移和复发,OSCC的5年生存率只有大约50%。OSCC可以早期预防和治愈,但大多数OSCC患者直到晚期才被确诊[2-3]。因此,寻找潜在的生物标志物对口腔鳞状细胞癌的早期诊断和治疗迫在眉睫。
目前,在高通量实验方法中,如微阵列分析已广泛应用于差异表达基因分析[4-5]。大量的微阵列数据被存储在公共数据库中。这些储存库便于研究人员通过整合多个微阵列数据集来识别疾病相关的生物标志物,有助于理解OSCC形成的分子机制。有学者利用SEER数据库分析影响腺样囊性癌患者预后的临床病例因素[6]。本研究中从GEO数据库下载了3 个微阵列数据集来识别OSCC和正常对照组的差异表达基因(differentially expressed genes,DEGs), 用于寻找与OSCC发生、发展有关的关键基因,这些关键基因可能作为诊断和治疗OSCC的潜在生物标志物。
1 资料与方法
1.1 微阵列数据信息
从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载基因表达数据集GSE74530、GSE78060和GSE138206。这3 个数据集的平台都是GPL570(Affymetrix Human Genome U133 Plus 2.0 Array),GSE74530和GSE138206分别含有6 个OSCC样本和6 个正常样本,GSE78060包含26 个OSCC样本和4 个正常样本。微阵列数据集汇总如表1。

表1 3 个微阵列数据集的汇总信息Tab 1 Summary of the 3 microarray datasets from the GEO database
1.2 DEGs数据处理与识别
GEO2R(www.ncbi.nlm.nih.gov/geo/geo2r/)在线分析工具可用于通过比较两组或多组样本来识别DEGs。使用GEO2R对OSCC样本和正常样本间的DEGs进行分析,筛选标准是P<0.01和|logFC|>1,并对DEGs用火山图可视化。随后用维恩图在线工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)确定3 个数据集中共同上调和下调的DEGs。
1.3 DEGs的GO和KEGG富集分析
DAVID(https://david.ncifcrf.gov/)在线分析工具可以为大规模基因或蛋白质提供全面的生物功能注释信息。基因本体论(gene ontology,GO)包括生物过程(gene process,BP)、分子功能(molecular function,MF)和细胞成分(cellular component,CC)。京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)数据库从基因组测序和其他高通量实验技术生成的大规模分子数据集中了解生物系统的高级功能和效用。利用DAVID对DEGs进行GO功能和KEGG通路富集分析,P<0.05认为具有统计学意义。
1.4 PPI网络构建与hub基因鉴定
STRING(https://string-db.org)在线数据库用于搜索已知蛋白质和预测蛋白质-蛋白质相互作用(protein-protein interaction,PPI), 包括蛋白质之间的直接物理相互作用和间接功能相关性。DEGs的PPI分析采用STRING数据库,置信度≥0.4,结果用Cytoscape软件进行分析和可视化。此外,利用Cytoscape的cytoHubba插件计算每个蛋白节点的连接度,排名前10的基因被鉴定为hub基因。
1.5 hub基因的表达
UCLCAN(https://ualcan.path.uab.edu/index.html)是一个基于TCGA数据库的分析和挖掘癌症数据的网站工具。通过UCLCAN验证hub基因的表达,P<0.05被认为具有统计学意义。
1.6 生存分析
Kaplan-Meier plotter(http://kmplot.com/analysis/)在线的肿瘤分析工具中可以获得基因表达与多种癌症患者的生存率相关的信息。采用Kaplan-Meier对OSCC中hub基因表达的生存状态进行分析验证,P<0.05认为具有统计学意义。
2 结 果
2.1 DEGs的识别
从GSE74530中获得2 783 个DEGs,包括2 009 个上调基因和774 个下调基因。在GSE78060中,鉴定出2 774 个DEGs,包括1 115 个上调基因和1 659 个下调基因。从GSE 138206中共鉴定出1 265 个DEGs,包括569 个上调基因和696 个下调基因(图1)。用维恩图求出3 组间的重叠部分,最终得到182 个上调DEGs和75 个下调DEGs(图2)。
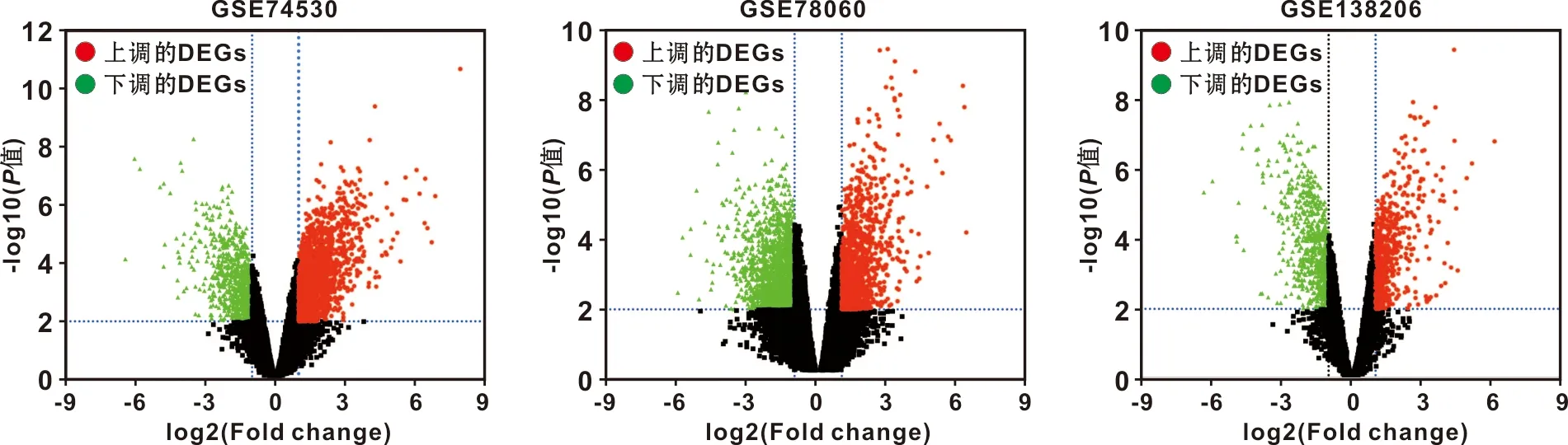
图1 3 个数据集中OSCC组织与正常组织之间DEGs的火山图

图2 3 个数据集中共有的DEGs的韦恩图
2.2 DEGs的GO和KEGG富集分析
2.2.1 上调DEGs的GO和KEGG富集分析 BP分析表明,DEGs主要富集细胞外基质组织、细胞黏附、胶原分解代谢、血管生成和细胞外基质分解。在CC中,DEGs主要分布在胞外区、胞外体、胞外间隙、细胞外基质和蛋白质类细胞外基质。MF分析显示,DEGs在钙离子结合、细胞外基质结构成分、胶原结合、金属内肽酶活性和血小板源性生长因子结合等方面均显著富集。另外,KEGG途径分析表明,DEGs主要集中在细胞外基质受体相互作用、PI3K-Akt信号通路、局灶性粘附、阿米巴病、蛋白质消化吸收等方面。
2.2.2 下调DEGs的GO和KEGG富集分析 BP分析表明,DEGs主要富集凋亡过程的阳性调节、细胞-底物粘附的阳性调节、突触组装的阳性调节、跨膜受体蛋白酪氨酸激酶信号通路、CD4阳性、α-βT细胞分化。在CC中,DEGs主要集中在细胞外体、蛋白质类细胞外基质、细胞外基质中。MF分析显示,DEGs在肝素结合中明显富集。此外,KEGG途径分析表明,DEGs主要富集于脂肪酸降解、缬氨酸、亮氨酸和异亮氨酸降解。
2.3 PPI网络构建与hub基因鉴定
用STRING数据库构建了DEGs的PPI网络,包括247 个节点和1 132 个边,有10 个基因没有被发现。Cytoscape显示的结果如图3,根据蛋白节点的连接度得分,前10 位的hub基因分别为FN1、COL1A1、COL1A2、COL3A1、CD44、COL4A1、COL5A1、CXCL8、POSTN和CXCL10(表2)。所有这些hub基因在OSCC样本中均上调。

表2 连接度排名前10的基因Tab 2 Top 10 genes ranked by degree

图3 DEGs的PPI网络
2.4 hub基因的表达验证
除了在UALCAN数据库中未发现CXCL8外,其他hub基因在OSCC样本中的表达均显著高于正常组织(P<0.001)(图4)。
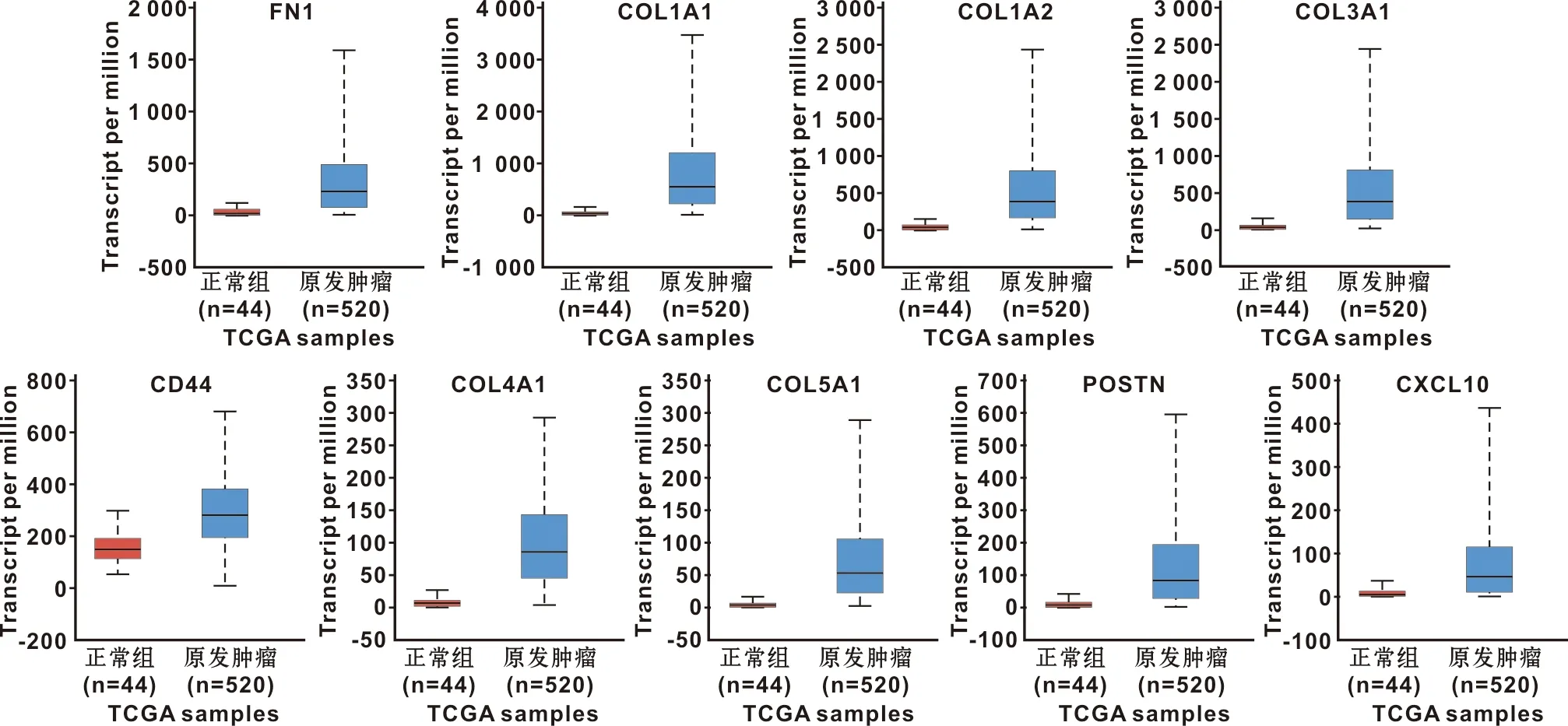
图4 hub基因在口腔鳞癌组织和正常组织中的表达
2.5 hub基因的生存分析验证
FN1、CD44、COL5A1、CXCL8的高表达与OSCC患者较差的总体生存率(overall survival,OS)显著相关,而COL3A1的高表达与OSCC患者的良好OS显著相关。然而,COL1A1、COL1A2、COL4A1、POSTN和CXCL10的表达与OSCC患者的预后无关(图5)。

图5 OSCC患者hub基因表达的生存分析
3 讨 论
在全球范围内,OSCC是一个重要的公共卫生问题,每年约有30万新病例被诊断[7]。尽管有研究发现NEK2、CCNB1等基因在OSCC的发生过程中起到一定作用[8-9],但OSCC的病理机制尚不完全清楚。为了进一步探索OSCC发病机制,本文从GEO数据库中下载OSCC样本和正常组织样本的基因表达数据,通过GEO2R在线分析,成功挖掘到差异表达的基因,用STRING数据库构建了差异表达基因的PPI网络,同时,用Cytoscape软件寻找hub基因,最后通过UALCAN基因表达验证以及通过Kaplan-Meier plotter生存分析验证,最终获得了FN1、COL3A1、COL5A1、CD44和CXCL8等5 个hub基因。
FN1属于细胞外基质的糖蛋白家族,已被证明参与细胞粘附和迁移过程[10]。据报道, FN1的过度表达与OSCC侵袭有关,并且与较差的预后有关[11-12]。这与此文研究结果一致。COL3A1编码III型胶原的pro-alpha1链。有研究表明COL3A1与肿瘤有关,高表达的COL3A1意味着较差的预后[13]。然而,在本研究中,COL3A1的高表达是OSCC的一个有利的预后因素。目前,在OSCC中对COL3A1的研究较少,因此,对COL3A1的分子机理还有待进一步研究。COL5A1可以编码V型胶原的α链。已有研究表明COL5A1与胃癌、乳腺癌有关,是肿瘤发生、发展和预后的不利因素[14-15]。然而,COL5A1在OSCC中的研究鲜有报道。在本研究中,COL5A1在OSCC中高表达,且高表达是OSCC患者的负面预后因素。CD44属于细胞表面糖蛋白家族,参与细胞与细胞的相互作用、细胞粘附和迁移。研究表明高表达的CD44参与了对化疗诱导的细胞凋亡的抵抗,这可能导致OSCC复发[16-17]。本研究发现CD44在OSCC中的表达明显高于正常组织,并且CD44高表达患者的生存率较差。CXCL8编码的蛋白质属于参与炎症反应的CXC趋化因子家族。CXCL8/CXCL8R轴在人类癌症中具有负作用,并可通过各种方式导致肿瘤的进展[18-19]。有研究证明,CXCL8与其受体CXCR2结合与OSCC的进展有关[20]。本研究发现高水平的CXCL8与OSCC患者的阴性预后有关。
综上所述,本研究从GEO 公共数据库下载OSCC表达谱芯片数据,在样本量相对充足的情况下,筛选OSCC组织和正常组织之间DEGs,并最终确定了与OSCC预后密切相关的hub基因。根据以上内容,通过生物信息学的方法获得的结果,具有一定的可靠性,如FN1、CD44和CXCL8在OSCC的研究中已有一些报道,而且本研究的结果和之前的研究结果是一致的。然而COL3A1和COL5A1在OSCC 研究中,鲜有报道,后续需要进一步通过相关实验对其进行验证。

