利用SNP标记分析淳安花猪群体遗传多样性和群体结构
戴丽荷,褚晓红,陈晓宇,杨娜娜,黄孙平,徐如海
(浙江省农业科学院畜牧兽医研究所/浙江省畜禽遗传育种工程技术研究中心,浙江 杭州 310021)
淳安花猪是浙江省地方猪品种之一,原产于淳安县境内,距今有500多年的饲养历史。1980年,淳安花猪与安徽的皖南花猪归并,称为皖浙花猪[1]。淳安花猪具有大多数中国地方猪种的特点,如繁殖力强、肉质优良、耐粗饲、生长速度慢、瘦肉率低等,曾是当地农户饲养的当家猪种,1983年全县淳安花猪能繁母猪存栏量达到8 319头。但由于地方猪普遍存在生长速度慢、瘦肉率低等特点,加上近30多年来瘦肉型猪种的冲击,淳安花猪饲养量急剧减少,2005年存栏仅248头,已濒临灭绝[2]。
要评估猪保种群体的现状,最重要的是分析当前群体的遗传多样性。目前猪群体遗传多样性的评估最常使用的是SNP(单核苷酸多态性)分子标记。SNP标记具有密度高、遗传稳定、可自动化分析等特点,在猪、鸡等畜禽上都有相应的SNP芯片。随着SNP芯片技术的成熟和价格的降低,SNP芯片愈来愈广泛地应用于猪的遗传多样性分析以及其他遗传学研究中[3-7]。
近年以来,淳安花猪的生存形势愈加严峻,现有保种群体几经变动,系谱记录缺失,养殖数量继续下降。目前淳安花猪保种群体的遗传多样性如何?群体近交情况怎么样?家系数量到底有几个?能否重构群体系谱?只有解决这些问题,才能更科学有效地保护淳安花猪这一宝贵地方猪种。本研究就是利用SNP芯片技术鉴定淳安花猪保种群体中现有的全部能繁公母猪的个体SNP位点,在此基础上分析淳安花猪群体的遗传多样性和亲缘关系,并重新划分家系,以指导淳安花猪种群后续的恢复扩群以及种质资源保护工作。
1 材料与方法
1.1 试验动物
2021年4月13日至6月30日,在浙江省杭州市淳安县采集54头浙江省杭州市淳安县淳安花猪现有保种群全部能繁公母猪的前腔静脉血样,其中母猪47头,公猪7头,置于-80 ℃冰箱保存。
1.2 试验方法
1.2.1 淳安花猪群体基因组DNA的提取 采用磁珠法血液DNA提取试剂盒(康为世纪,CW2531S)提取淳安花猪的DNA。所有样品的DNA利用0.8% 琼脂糖凝胶电泳和Nanodrop ND-2000进行定量分析,DNA的浓度调整至50 ng/μL。
1.2.2 SNP芯片分型 所有合格的DNA样品利用Illumina CAUPorcine 50K SNP芯片进行基因型分型,然后对获得的芯片扫描数据按以下标准进行质控:先除去性染色体上的标记,只使用常染色体上的位点,再选择SNP检出率大于0.9、个体检出率大于0.9的位点,进行后续分析。
1.2.3 遗传多样性分析 遗传多样性分析包括有效群体含量(Effective population content, Ne)分析[8]、群体的多态性标记比例(Proportion of polymorphic marker, PN)分析[9]、群体的期望杂合度(Expected heterozygosity, He)和观察杂合度(Observed heterozygosity, Ho)分析[10]。利用Plink(V1.90)软件分析多态性标记比例、期望杂合度和观察杂合度,利用SNeP(V1.1)软件分析群体有效含量。
1.2.4 种群基因组亲缘关系分析 利用Plink(V1.90)软件计算个体间的遗传距离,构建状态同源(identity by state, IBS)矩阵,进行IBS遗传距离分析。通过全基因组标记信息构建基因组关系G矩阵[11],利用Gmatrix(V2)软件进行G矩阵分子亲缘关系分析,绘制热图展现个体之间的基因组亲缘关系。
1.2.5 基于长纯合片段的近交系数分析 使用Plink1.9b软件进行基因组长纯合片段(Runs of homozygosity, ROH)分析。首先使用Plink(V1.90)计算得到每个样本的ROH长度[9],然后通过计算个体中ROH片段的总长度占常染色体基因组总长的比例得到基于ROH的近交系数[12]。
1.2.6 聚类分析构建淳安花猪群体家系 基于IBS遗传距离分析结果和G矩阵分析结果,利用Mega X(V10.0)软件构建邻接法进化发育树(Neighbor-Joining tree, NJ tree),进行聚类分析,构建淳安花猪保种群体家系。
2 结果
2.1 基因组DNA检测
54份淳安花猪DNA样品的浓度均大于100 ng/μL,OD260nm/OD280nm为1.7~2.0,基因组DNA样品质量较好,无蛋白质污染,也无降解,达到基因芯片的分析要求,可以用于后续研究。
2.2 SNP分型及质控
利用Illumina CAUPorcine 50K SNP芯片分析54头淳安花猪的基因组DNA,共检测到44 739个SNPS位点,个体的基因型检出率为98.25% ~99.14%,平均值为98.49%。通过质量控制,总共得到32 713个高质量的SNP位点,在染色体上的分布情况如表1所示。这些位点的分布密度在1号染色体最多,为3 835个,说明1号染色体的遗传变异相对较丰富;18号染色体的SNP数目最少,为742个。

表1 质控前后的SNP在染色体上的分布
2.3 遗传多样性分析
54头淳安花猪共检测到了73.187个有效等位基因,平均有效等位基因数为1.355,最小等位基因频率平均为0.143,分布比较均匀,其中0.1~0.2之间的相对较多,占25.05%,分布情况如图1A。SNP位点的多态性标记比例(PN)为0.543,表明54.3% 的SNP位点具有多态性,SNP位点的多态信息含量为0.018~0.375,平均多态信息含量为0.193,其中,14 582个SNP位点的多态信息含量大于0.25(用于计算多态信息含量的SNP位点共35 679个),为中度多态性SNP位点,如图1B所示。淳安花猪有效群体含量(Ne)为5.0,平均观察杂合度(Ho)为0.254,平均期望杂合度(He)为0.257,平均观察杂合度略小于平均期望杂合度,但两者十分接近。
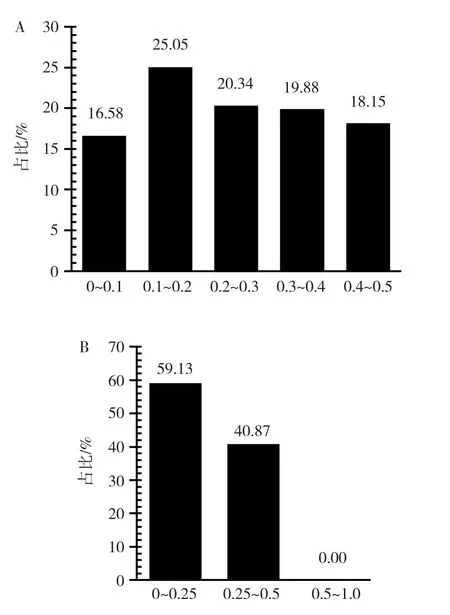
图1 淳安花猪遗传多样性分析
2.4 IBS距离矩阵分析
54头保种淳安花猪之间的IBS遗传距离在0.083 3~0.388 7之间,平均为0.212 9;7头种公猪的IBS遗传距离在0.104 2~0.286 2之间,平均为0.233 9。淳安花猪保种群体IBS距离矩阵的结果如图2所示,大部分方格颜色较浅(呈中等程度的亲缘关系),小部分个体间的IBS遗传距离较近,存在较高的亲缘关系。
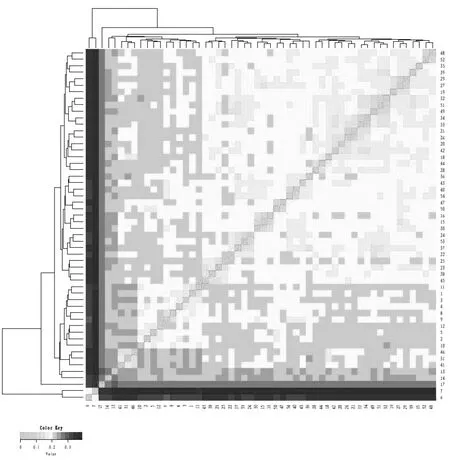
图2 淳安花猪种群的状态同源(IBS)矩阵
2.5 G矩阵分析
利用SNP位点构建的基因组关系G矩阵,进一步分析淳安花猪保种群体的亲缘关系,结果如图3所示。大部分淳安花猪个体间呈中等程度的亲缘关系(图3中颜色较浅的方格),而部分个体之间存在较近的亲缘关系(图3中颜色较深的方格),与IBS距离结果一致。

图3 淳安花猪种群的G矩阵
2.6 ROH分析
在54头淳安花猪保种群体中共检测到2 646个ROH片段,长度在1~10 Mb之间的ROH数量最多,占72.54% (图4A),其中,最短的ROH的长度为1.12 Mb,位于15号染色体上,最长的ROH的长度为135.85 Mb,位于13号染色体上。ROH的分布比较均匀,1号染色体上的ROH数量最多,为313个;12号染色体上的ROH数量最少,为65个(图4B)。每头淳安花猪的ROH总长度在2.93~1 050.05 Mb之间,平均ROH总长度为488.91 Mb,ROH总长度在400~700 Mb之间的个体数最多,共有35头(图4C),占65%。
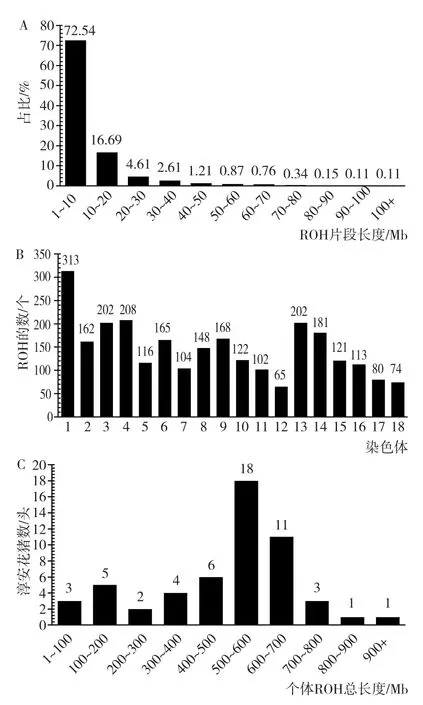
图4 淳安花猪ROH分析
淳安花猪保种群体基于ROH计算的平均近交系数为0.199,大部分集中在0.1~0.3,最低0.001 2,最高0.428 5,表明淳安花猪保种群体出现了近交现象。
2.7 淳安花猪保种群体家系结构分析
一般来讲,猪保种群体的家系划分是基于公猪。同时结合G矩阵和遗传距离分析结果,将这7头种公猪划分为5个家系,分别命名为家系A、家系B、家系C、家系D、家系E,分别含有2、2、1、1、1头种公猪。
根据47头淳安花猪母猪与不同家系公猪亲缘关系的远近程度,将母猪划分进上面已构建的5个公猪家系中。另外,在这5个家系分类以外,有25头母猪与所检测的公猪血缘关系都比较远,将其合并构建为家系F,最终整个淳安花猪保种群体家系分析结果见表2,共分为6个家系。
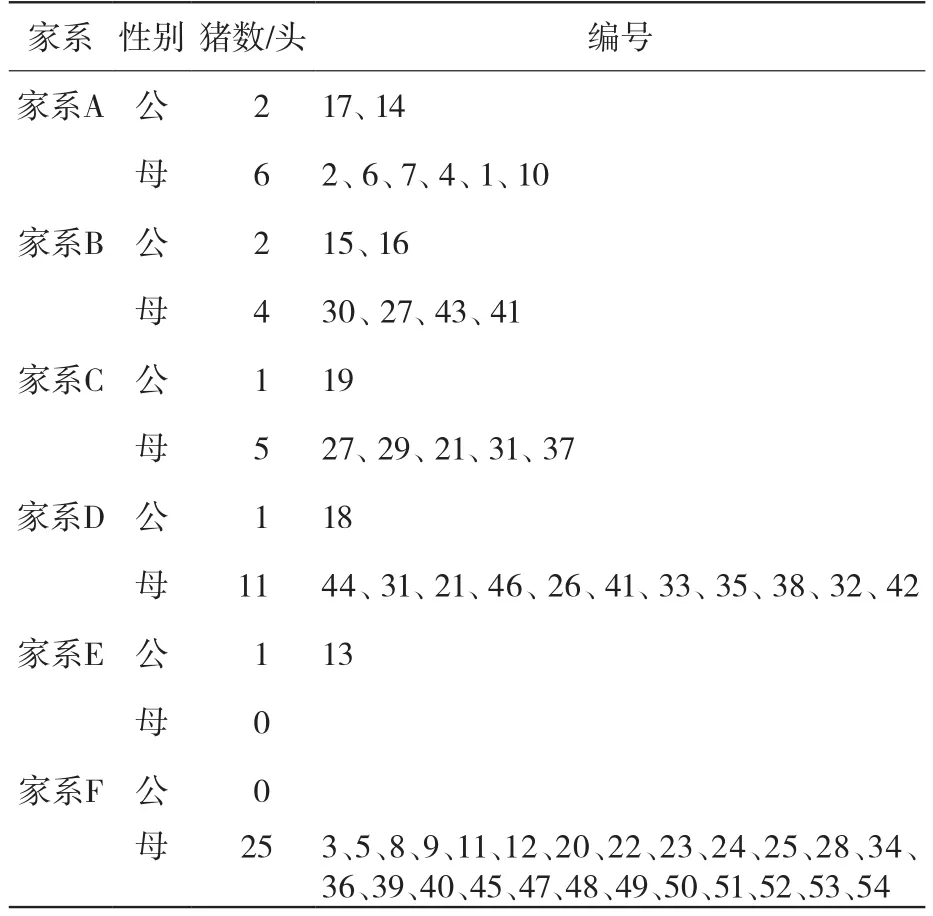
表2 群体家系构建结果
3 讨论
猪群体遗传多样性研究一般使用微卫星标记或SNP标记。章学东等[13]利用9对微卫星标记检测淳安花猪群体遗传多样性,认为淳安花猪群体遗传多样性丰富。但随着技术的发展,SNP芯片突显出了更大的优势,与微卫星标记相比,SNP标记覆盖密度大,分布广泛,数量丰富,突变率低,遗传稳定性高。有研究表明,采用SNP标记比采用微卫星标记估计亲缘关系的准确性更高[14]。本研究主要利用中国地方猪种的Illumina CAUPorcine 50K SNP芯片,发现个体的基因型检出率为98.25% ~99.14%,平均值为98.49%,且这些位点几乎覆盖全部基因组,分布相对均匀,具有很好的代表性。利用同样的芯片,蓝塘猪个体基因型检出率94.88% ~98.43%,平均为98.24%[7]。马身猪个体基因型检出率为97.75% ~98.16%,平均为98.01%[4]。这些研究表明,该款SNP芯片适用于我国地方猪种的SNP检测。
黄树文[5]利用SNP芯片对中国华南地区(云南、广东、广西、福建、海南)21个地方猪种和6个西方猪种的分析表明,27个猪种的多态性标记比例为0.795~0.995。蔡春波等[4]发现马身猪的多态性标记比例为0.724。本研究中淳安花猪SNP位点的多态性标记比例为0.543,与第三世代青峪猪群体的0.536 7[3]基本相当,说明淳安花猪的遗传多样性较少。淳安花猪并没有像西方商业猪种那样经受过高强度的选育,但却没有表现出更高的遗传多样性,这反映了大多数中国地方猪种保种群规模较小、遗传多样性丢失严重的现象[15-16]。在杂合度方面,27个猪种的观测杂合度为0.25~0.43,中国地方猪种中,以官庄花猪的观测杂合度最低,为0.298;期望杂合度为0.295~0.421,中国地方猪种中,闽北花猪最低,为0.295[5]。本研究中淳安花猪的平均观察杂合度(Ho)为0.254,平均期望杂合度(He)为0.257,均低于华南地区的地方猪种,原因可能是由于一直以来淳安花猪的保种群体小,近亲繁殖普遍存在而使群体发生纯合反应,导致杂合度降低。此外,淳安花猪的平均观察杂合度略小于平均期望杂合度,但两者十分接近,表明现有的淳安花猪群体未经受过严格的选育,猪群几乎处于自由交配状态[17],这与近几年淳安花猪保种群变动剧烈有关。因此,在淳安花猪保种工作中,应尽量引入新的血统,避免近亲繁殖,以提高遗传多样性。
衡量群体遗传关系的指标主要看遗传距离和根据基因组G矩阵分析的亲缘关系。一般来说群体遗传距离越近,遗传相似系数越大,则亲缘关系越近[18]。陈究成[19]利用简化基因组测序方法发现浙江省地方猪群体平均遗传距离为0.151,其中淳安花猪遗传距离为0.159。本研究发现淳安花猪遗传距离为0.212 9,这与SNP标记检测方法、样本分析数量、保种群体变动等有关。G矩阵分析结果也同样表明,大部分淳安花猪个体间呈中等程度的亲缘关系,部分个体之间存在较近的亲缘关系,与IBS距离结果一致,表明淳安花猪保种群体存在近交风险。
ROH是个体基因组内纯合基因型的连续片段,蕴含着群体近交的信息,其长度和频率可以反映群体历史。基于ROH计算的基因组近交系数是个体真实的近交系数,不依赖于系谱记录的准确性和完整性,能准确地反映两个配子间的关系。本研究基于ROH值计算的群体平均近交系数为0.199,比马身猪(0.237)[4]低,但比青峪猪(0.075)[3]、恩施黑猪(0.069)[6]和深县猪(0.009 3)[20]都要高,表明淳安花猪群体出现了近交现象,这与IBS矩阵以及G矩阵结果相一致。后续工作中应引入新的血统,扩大群体含量,进行科学选配,避免近亲繁殖。
实际育种工作中,在系谱的准确性和完整性上出现误差或缺失很难避免,尤其是在大部分的地方猪保种场,由于规模和技术力量受限,系谱的完整性、连续性及准确性等问题尤为突出。淳安花猪保种群体由于这几年变动较大,存在系谱缺失的情况,本研究将54头淳安花猪分为6个家系,其中5个家系均含有种公猪,但不同家系中个体数目差异很大,家系E没有母猪,家系F没有公猪,在后续的保种工作中,需要关注各家系种猪的数量,尽量维持一个平衡的家系结构。
总之,淳安花猪保种群体的杂合度低,近交程度高,家系少,各家系的个体数量差异大,容易引起遗传多样性的丢失。结合研究结果,笔者认为切实可行的淳安花猪保种策略,一是根据家系划分结果,在实际选配中可采取不同家系间的轮回配种,实现避开全同胞、半同胞的不完全随机交配;二是可利用回交法造就新的公猪血统,增加群体有效含量;三是延长世代间隔,控制近交增量;四是加强档案管理,及时建立电子档案及规范耳号编号、系谱记录流程,对遗传数据做到可及时跟踪和监测,定期分析,及时调整保种方案。本研究对淳安花猪的保种工作具有实际指导意义。