FFAR4基因多态性与藏族先天性心脏病关系的研究*
李爽林, 张 辉, 王 洋, 高 慧, 杨应忠, 刘永年
(青海大学医学院,青海西宁 810001)
先天性心脏病(congenital heart disease,CHD)的发病机制中遗传因素占重要作用[1],主要包括染色体异常、单基因病和多基因病等。流行病学数据显示,高原地区CHD 发病率明显高于平原地区且具有民族差异[2]。本课题组前期利用Genome-Wide Human SNP Array 6.0芯片对部分高原藏族CHD患者单核苷酸多态性(single nucleotide polymorphism,SNP)位点进行全基因组关联分析(genome-wide association study,GWAS),发现易感位点rs11187529 存在于游离脂肪酸受体4(free fatty acid receptor 4,FFAR4)基因上。FFAR4 是一种G 蛋白偶联受体,在找不到配体以前被称为G 蛋白偶联受体120(G-protein-coupled receptor 120,GPR120)[3],作用于长链脂肪酸包括以二十碳五烯酸(eicosapentaenoic acid,EPA)和二十二碳六烯酸(docosexaenoic acid,DHA)为代表的ω3 多不饱和脂肪酸(polyunsaturated fatty acids,PUFAs)家族[4]。研究显示,FFAR4 在大鼠心脏组织中存在表达,高脂饮食可明显上调心脏组织中的FFAR4基因转录物[5]。后期研究进一步表明,FFAR4 在小鼠的心肌细胞和成纤维细胞中表达,且可通过信号传导介导EPA 而防止心脏成纤维细胞的纤维化[6]。本研究选择了前期研究新发现的易感位点所在基因FFAR4,进一步验证FFAR4基因多态性与藏族CHD的关系。
材 料 和 方 法
1 研究对象
选取2018 年9 月~2019 年9 月在青海大学附属医院、青海省人民医院等就诊的藏族CHD 患者103例,同期参加体检的藏族健康对照者267 例。病例组纳入标准均经病史资料采集,临床体格检查诊断,心电图、心脏彩超检查或经心导管及心血管造影检查后临床确诊为CHD,所有病例组CHD 患者排除无其他先天性遗传疾病。同期参加体检的年龄、性别相匹配的健康藏族对照者,经病史资料采集,临床体格检查诊断,心电图和心脏彩超检查证实为健康者,所有健康对照者均排除CHD 和其他先天性遗传疾病。本研究通过青海大学医学院伦理委员会的审批,经研究对象同意并签署知情同意书。
2 候选tag SNP位点的筛选
研究表明,部分SNP 位点可提供遗传单位单体型(haplotype)内大部分信息,这些位点称为标签SNP(tag SNP)。通过千人数据库中基因数据库查找FFAR4基因的常见SNP 位点,使用HaploView 4.2 软件通过连锁不平衡(linkage disequilibrium)分析挑选tag SNP 位点,设置参数最小等位基因频率(minor allele frequency,MAF)>0.05,重新筛选SNP 位点后,根据SNP 之间连锁不平衡相关程度R2>0.08 选择tag SNP,单体型块的划分采用表示位点间连锁不平衡程度的D值置信区间(confidence interval,CI)法,D值的95%CI在0.70~0.98 的相邻SNP 归于同一个单体型块,根据筛选结果选择了5 个tag SNP 位点,即rs12219199、rs12220062、rs77999136、rs12243124 和rs10882282,见图1及表1。

表1 候选tag SNP位点信息表Table 1. Information about candidate tag SNP loci

Figure 1. Selection of candidate tag SNP loci. The black triangle box in the figure represents the enclosed monomer block,the red panel shows that there is a strong linkage balance between adjacent SNPs,and the white panel indicates that the linkage disequilibrium is weak or non-existent.图1 候选tag SNP位点的筛选
3 实验方法
3.1 基因提取 使用用乙二胺四乙酸二钾抗凝管采集CHD 患者和健康对照者外周静脉血5 mL,1 788.8×g离心10 min 后吸取血浆层至冻存管,置冰箱保存(-80 ℃);提取白细胞层至15 mL 的离心管中,加入红细胞裂解液至12 mL,混匀后1 788.8×g离心10 min 倒掉红细胞层,重复上述步骤充分去除红细胞,最后加入1.5 mL白细胞裂解液室温保存。
使用Gentra Puregene Blood Kit(Qiagen)按照标准步骤提取基因组DNA,提取后的DNA 经琼脂糖凝胶(1.5%~2.0%)电泳40 min 后,在凝胶成像曝光仪中观察提取的DNA 结果。从全部研究对象的血液标本中提取了基因组DNA 后,使用核酸定量分析仪测定其浓度和纯度,DNA纯度结果均在1.8<A260/A280<2.0,代表纯度高符合使用要求,浓度稀释至50 μg/L后冻存于冰箱(-20 ℃)备用,见图2。

Figure 2. Image of agarose gel electrophoresis of some DNA.The PCR products were exposed by gel imaging system,and the results showed that the bands were clear and bright,without heterobands and tails,indicating that the extracted DNA was in ideal condition.图2 部分DNA样本琼脂糖凝胶电泳图
3.2 引物设计 采用Genotyping Tools 及MassARRAY Assay Design 软件(Agena)为每个候选tag SNP位点自动设计PCR 扩增引物及单碱基延伸引物,由北京博奥晶典生物技术有限公司合成,序列见表2。

表2 候选tag SNP位点的引物序列Table 2. The sequences of the primers for candidate tag SNP loci
3.3 PCR 扩增 将反应体系[DNA 1 μL 和反应混合物4 μL(HotStarTaq DNA 聚合酶0.1 μL,引物1 μL,dNTPs 0.1 μL,缓冲液0.5 μL,MgCl20.4 μL,灭菌双蒸水1.9 μL 补齐)]共5 μL 加至384 孔PCR 反应板中。在PCR 扩增仪上将设置反应条件为:94 ℃预变性4 min,循环1 次;94 ℃变性20 s,56 ℃退火30 s,72 ℃延伸1 min,循环45次;72 ℃复延伸。
3.4 PCR 产物的虾碱性磷酸酶(shrimp alkaline phosphatase,SAP)处理 在PCR 产物中加入SAP 处理以去除扩增产物中剩余的、非结合的dNTPs,SAP通过切断5'端的磷酸基,使未结合的DNA 磷酸化。将反应体系[PCR 产物5 μL 和SAP 混合液2 μL(包括SAP 0.5 U 和缓冲液0.17 μL)]共7 μL分配到384孔PCR 反应板的每个孔中。将处理过的培养板放置在37 ℃的培养箱中40 min,然后再放置在85 ℃的培养箱中5 min(反应条件为:37 ℃预变性40 min;85 ℃变性5 min;4 ℃复延伸),以使SAP失活。
3.5 单碱基延伸反应 反应体系(9 μL)包含SAP处理后PCR 产物7 μL 和延伸反应混合物2 μL(引物0.94 μL,iPLEX 酶0.041 μL,iPLEX 缓冲液0.2 μL,iPLEX 终止液0.2 μL,灭菌双蒸水0.619 μL 补齐),加入到PCR 反应孔上。将反应程序设置为:94 ℃预变性30 s,循环1 次;52 ℃变性5 s,80 ℃退火5 s,循环4 次;94 ℃预变性5 s,循环1 次;52 ℃变性5 s,80 ℃退火5 s,循环39次;延伸3 min,4 ℃维持。
3.6 树脂纯化 这一清理步骤对于优化延长反应产物的质谱分析非常重要。将6 mg Clean Resin树脂平铺到树脂板中,在单碱基延伸反应的产物对应孔内加入16 μL的水,将干燥后的树脂添加到引物扩增反应产物中,使树脂与反应物充分接触,离心使树脂沉入孔底部。经过酸性试剂预处理的阳离子树脂,去除Na+、K+和Mg2+等盐类,如果不去除,这些离子会在质谱中产生高背景噪声。
3.7 芯片点样 通过基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-TOF MS)技术,检测延伸产物分子量的大小,将SNP 引起的碱基差异通过分子量的差异而体现。将树脂纯化后的延伸产物移至芯片上,将芯片放入质谱计中,然后用真空下的激光通过基质辅助激光解吸电离飞行时间方法对每个点进行拍摄,在MALDI-TOF 质谱中,激光束作为解吸和电离源,基质吸收了激光的光能,并导致部分被照亮的基质汽化,迅速膨胀的基质羽状物将一些分析物带入真空中,基质分子吸收大部分入射激光能量,使样品分子被汽化和电离,它们就被静电转移到飞行时间质谱仪(time-of-flight mass spectrometer,TOF-MS)中,在那里它们从基质离子中分离出来,根据它们的质量-电荷比(m/z)单独检测,检测结果使用TYPER 4.0软件(Agena)进行基因分型。
4 统计学处理
数据分析采用SPSS 19.0 软件。计量资料采用均数±标准差(mean±SD)表示,两组间比较采用独立样本t检验;计数资料两组间比较采用χ2检验。使用Hardy-Weinberg 平衡检验样本的群体代表性,以P>0.05 代表样本基因型符合遗传平衡,具有群体代表性。两组间基因型和等位基因频率的比较使用χ2检验或Fisher 精确概率检验,以P<0.05 为差异有统计学意义。连锁不平衡和单体型通过SHEsis(http://analysis.bio-x.cn)在线网站分析,分析结果使用P值、优势比(odds ratio,OR)及其95%CI 表示,以P<0.05为差异有统计学意义,OR=1表示该因素对疾病的发生不起作用,OR>1表示该因素可能为危险因素,OR<1表示该因素可能为保护因素。
结果
1 病例组和对照组一般资料比较
样本资料包括CHD 患者103 例和健康对照者267例,病例组平均年龄为(22.0±17.0)岁,对照组为(22.2±12.3)岁,组间年龄差异无统计学意义(P>0.05)。病例组男性42例,女性61例,对照组男性99例,女性168 例,χ2检验分析结果显示组间性别构成差异无统计学意义(P>0.05)。
2 Hardy-Weinberg平衡检验结果
使用Hardy-Weinberg 平衡定律检验Excel 小软件在线检验样本的基因多态性是否符合遗传平衡。结果显示,FFAR4基因筛选的5 个候选tag SNP 位点rs12219199、rs12220062、rs77999136、rs12243124 和rs10882282 在病例组和对照组均符合Hardy-Weinberg 平衡定律(P>0.05),所选位点具有样本代表性,可进行下一步分析,见表3。

表3 候选tag SNP位点的Hardy-Weinberg平衡检验Table 3. Hardy-Weinberg equilibrium test for candidate tag SNP loci
3 候选tag SNP位点基因型和等位基因型频率分析
FFAR4基因SNP分型结果如表4所示,rs1221919的3 种基因型CC、CT 和TT 在病例组分别是51.5%、36.9%和11.7%,在对照组分别是50.2%、42.7%和7.1%,CT 和TT 基因型频率分布在两组间无显著差异(P>0.05);等位基因C 和T 在病例组分别是66.9%和30.1%,在对照组分别是71.5%和28.5%,两组间T 等位基因频率分布无显著差异(P>0.05)。rs1220062的3种基因型GG、GA 和AA 在病例组分别是46.6%、38.8% 和14.6%,在对照组分别是51.3%、38.6%和10.1%,GA 和AA 基因型频率分布在两组间无显著差异(P>0.05);等位基因G 和A 在病例组分别是66.0%和34.0%,在对照组分别是70.6%和29.4%,两组间A 等位基因频率分布无显著差异(P>0.05)。rs77999136 的3 种基因型CC、CT和TT在病例组分别是89.3%、9.7%和1.0%,在对照组分别是90.6%、9.4%和0.0%,CT 和TT 基因型频率分布在两组间无显著差异(P>0.05);等位基因C和T 在病例组分别是94.2%和5.8%,在对照组分别是95.3%和4.7%,两组间T 等位基因频率分布无显著差异(P>0.05)。rs12243124 的3 种基因型TT、TC和CC 在病例组分别是33.0%、7.6%和19.4%,在对照组分别是33.7%、51.3%和15.0%,TC 和CC 基因型频率分布在两组间无显著差异(P>0.05);等位基因T和C在病例组分别是56.8%和43.2%,在对照组分别是59.4%和40.6%,两组间C 等位基因频率分布无显著差异(P>0.05)。rs10882282 的3 种基因型GG、GC 和CC 在病例组分别是26.2%、46.6% 和27.2%,在对照组分别是30.7%、51.3%和18.0%,GC 和CC 基因型频率分布在两组间无显著差异(P>0.05);等位基因T 和C 在病例组分别是68.0%和32.0%,在对照组分别是68.7%和31.3%,两组间C等位基因频率分布有显著差异(P<0.05)。
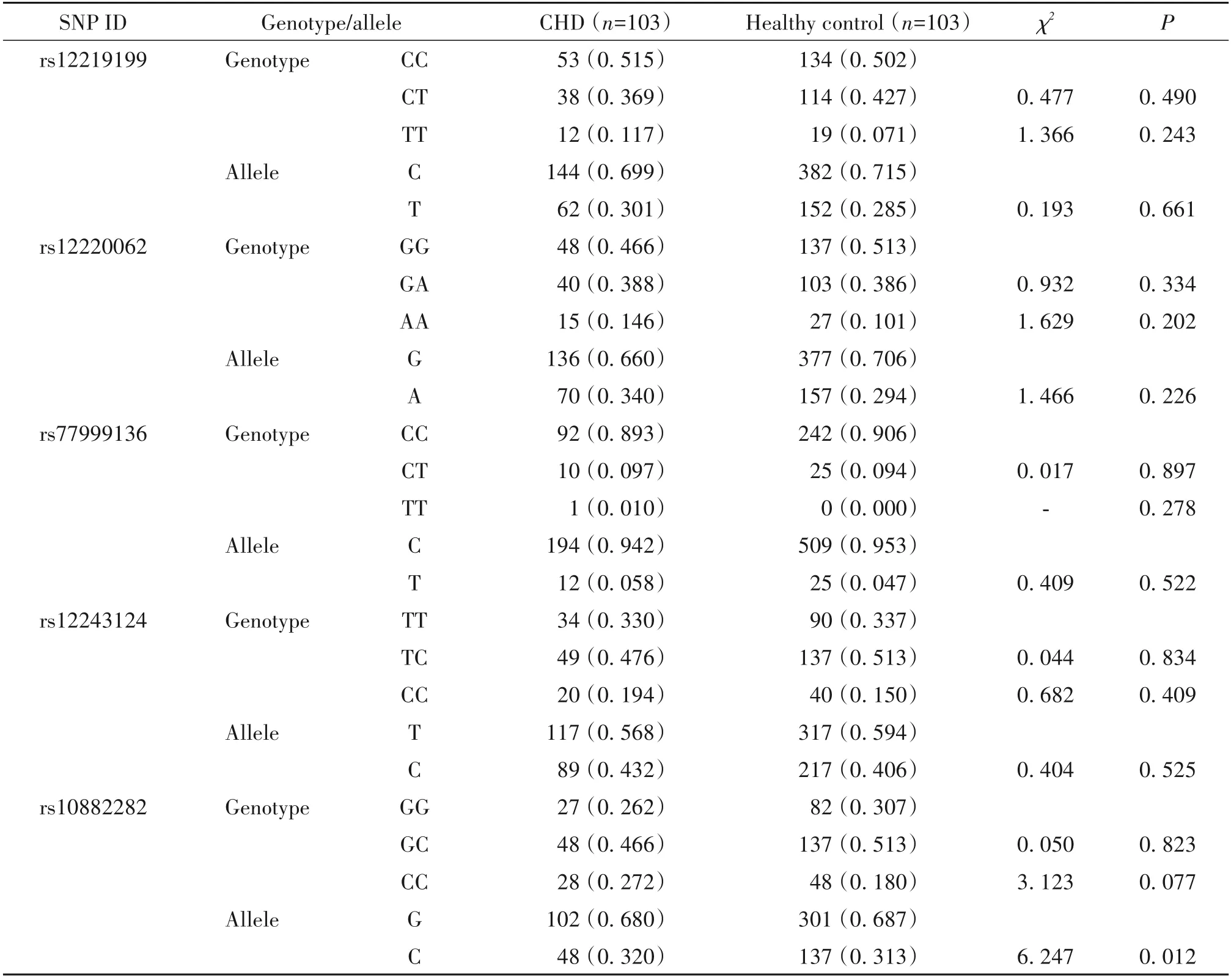
表4 候选tag SNP位点在CHD组和健康对照组中基因型及等位基因频率的分布Table 4. Distribution of the genotypic and allelic frequencies of the candidate tag SNP loci between congenital heart disease(CHD)group and healthy control group
4FFAR4 基因候选tag SNP 位点的连锁不平衡和单体型分析结果
候选tag SNP 位点按rs12219199-rs12220062-rs-77999136-rs12243124-rs10882282在染色体上的先后顺序进行排序,这些SNP位点间的遗传连锁程度以D值表示,FFAR4基因非编码区候选tag SNP 位点连不平衡图显示,这些SNP位点之间存在较强的连锁不平衡,见图3。
单体型分析结果显示有7 类常见单体型,其中单体型CACCC 在两组间无显著差异(P>0.05),OR=1.463(95% CI:0.915~2.340);单体型CGCCC 在两组间无显著差异(P>0.05),OR=0.918(95% CI:0.445~1.896);单体型CGCTC 在两组间有显著差异(P<0.05),OR=2.849(95%CI:1.400~5.798),可能为CHD 的危险因素;单体型CGCTG 在两组间有显著差异(P<0.05),OR=0.702(95%CI:0.506~0.974),可能为CHD 的保护因素;单体型TACCC 在两组间无显著差异(P>0.05),OR=1.054(95% CI:0.695~1.598);单体型TGCCC 在两组间无显著差异(P>0.05),OR=0.640(95% CI:0.281~1.457);单体型TGTTG 在两组间无显著差异(P>0.05),OR=1.223(95%CI:0.586~2.549),见表5。
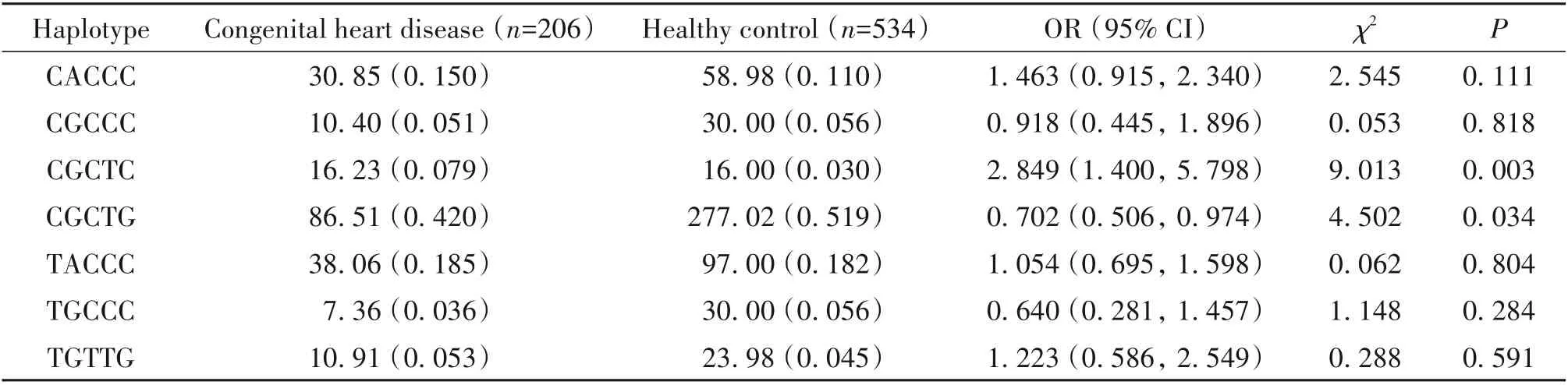
表5FFAR4基因非编码区单体型分析Table 5. Haplotype analysis of noncoding area of theFFAR4 gene
讨论
CHD 是国内外新生儿发病率最高的先天性出生缺陷,占所有先天性畸形的25%[8],也是围产期和婴儿死亡的主要原因[9],在活产新生儿中CHD 的发病率约为0.8%~0.9%,死产或流产胎儿中的发病率达1%[10]。CHD 的确切发病机制目前仍没有完全阐明,研究表明遗传因素和环境因素的相互作用是其主要的致病危险因素[11]。流行病学统计调查显示高原地区的CHD 发病率较平原地区升高,且发病率与海拔高度成正相关[12],海拔4 000 米以上的藏族儿童患病率0.874%[13],明显高于国内低海拔地区[14-15],且患病率在不同民族存在差异性,青海省黄南洲儿童CHD在藏族患病率高于汉族[16],可能与长期生活环境引起的遗传差异有关。
FFAR4基因位于人类第10 号染色体上,最初是从人类基因数据库的DNA 片段中克隆所认识,被命名为GPR120;随着研究发展揭示了其可以作为长链脂肪酸的受体,重新命名为FFAR4基因[17]。研究发现,FFAR4基因有两个受体序列,既可以形成对应BC-101175 受体序列,编码361 个氨基酸蛋白质的包含1 086 个核苷酸的短转录物,称为FFAR4短序列(FFAR4-S)[18],也可以交替剪接形成对应NM-181745受体序列,包含361 个氨基酸的蛋白质FFAR4-S 和含有一个额外外显子3 的377 个氨基酸(包含1 134个核苷酸)的独特较长同工型,称为FFAR4长序列(FFAR4-L)[19]。FFAR4-L 中编码的额外16 个氨基酸间隙,位于FFAR4-L 异构体的第3 个细胞内环,这是一个对G 蛋白偶联以及拮抗剂诱导的受体磷酸化和随后的脱敏都至关重要的受体结构域[20]。这个额外的间隙含有4 个磷酸不稳定的丝氨酸/苏氨酸残基,在没有激动剂的情况下,较短的变异体表现出更明显的基础磷酸化,表明FFAR4-S 和FFAR4-L 可能具有不同的激动剂刺激的磷酸化谱,FFAR4-L 中的额外间隙可能有助于掩盖结构性磷酸化位点[21]。至今的研究表明,小鼠和大鼠中只存在FFAR4-S 序列[22],且非人灵长类动物恒河猴和食蟹猴研究模型显示,同样只存在较短的361 个氨基酸蛋白质的受体序列,与人类FFAR4-S 的同源性为97.5%[23],FFAR4-L序列只存在于人类。因此,人类FFAR4基因的分子研究具有特异性。

Figure 3. Linkage disequilibrium among the 5 candidate SNP loci ofFFAR4 gene. The sequencing of SNP loci is in the order of chromosome position from small to large,and the degree of genetic linkage between these SNPs is estimated asD values. The red panel indicates that there is a strong linkage balance between adjacent SNPs,while the white panel indicates that the connection imbalance is weak.图3FFAR4基因5个候选tag SNP位点的连锁不平衡图
研究表明,FFAR4 通过介导受体EPA,抑制转化生长因子β1 诱导的小鼠心脏成纤维细胞纤维化,从而预防心力衰竭[24],且不会在心肌细胞或成纤维细胞中积累[25]。FFAR4 的小分子激动剂GW9508 以浓度依赖的方式在心脏成纤维细胞中阻止了转化生长因子β1 促纤维化信号传导,敲除FFAR4后GW9508的抗纤维化作用被阻断,间接证明了FFAR4 在心脏保护中的介导作用[26]。FFAR4 与ω3 PUFAs 的多种作用相一致。ω3 PUFAs 的有益作用首次被认识到是当时流行病学显示的富含ω3 PUFAs 饮食人群的心肌梗死发生率较低,随后几项大规模随机临床试验证明了ω3 PUFAs 对心脏的保护作用。许多动物研究也表明,ω3 PUFAs 对心血管系统具有多效有益作用,包括抗心律失常、降低血浆甘油三酯、抗血栓形成、抗动脉粥样硬化、抗炎和抗纤维化作用[27]。多个研究结果表明,ω3 PUFAs 在包括缺血再灌注[28]、心力衰竭[29]和心律失常[30]在内的多种病理生理情况下具有心脏保护作用。其中EPA 的心脏保护作用是最明显的[31]。EPA 的增加可减少线粒体基质的氧化应激,通过保护线粒体极大地保护了心脏[32]。内源性生成的ω3 脂肪酸脂可通过激活FFAR4 形成ω3 PUFAs-FFAR4通路,减轻血管炎症[33]、动脉血栓形成和血管内膜增生[34],从而发挥有益的心脏保护作用。FFAR4与心脏的直接作用机制目前研究较少。
本研究提取了无血缘关系的100 例藏族CHD 患者和100 例藏族健康对照者的基因组DNA 信息,筛选了FFAR4基因非编码区的5 个tag SNP 位点,通过分析基因型和等位基因型频率在CHD 病例组和健康对照组的分布差异发现,rs10882282的等位基因C在两组间的差异有统计学意义,提示rs10882282 位点的等位基因C与藏族CHD 相关。在多因素复杂疾病CHD 中,多个变异位点组合构成的单体型分析同样具有重要的意义。连锁不平衡分析显示,筛选的5个候选tag SNP 位点之间存在连锁不平衡。单体型分析结果显示,常见单体型CGCTC 在两组间的频率有显著差异(P<0.05),OR=2.849(95% CI:1.400~5.798)表明CGCTC 可能为CHD 的危险因素;单体型CGCTG 也与CHD 显著相关(P<0.05),OR=0.702(95% CI:0.506~0.974)表明CGCTG 可能为CHD 的保护因素。单体型CGCTG 这几个基因型均为FFAR4基因的野生基因型,提示FFAR4基因突变后可能导致CHD 的发病,且单体型CGCTG 中易感等位基因G 突变为等位基因C 后,单体型CGCTC 即变成了CHD 的危险因素,再次表明FFAR4基因中携带等位基因C 可能是藏族CHD 的致病因素,FFAR4可能为藏族CHD的易感基因。
明确CHD 的遗传病因对疾病的预防和预后非常重要,早发现、早诊断是治疗CHD的关键。FFAR4基因多态性与CHD 的研究之前并未开展,本课题首次选择人类FFAR4基因多态性与高原藏族CHD 的关系研究进行验证,结果提示FFAR4有可能是CHD的易感基因。不过本研究样本量有限,存在一定局限性,有待进一步进行深入研究。

