玉米SNAC转录因子的基因结构特征与逆境调控预测
罗平,庞博,崔进鑫,于爽,王晓楠,程名,陈勇,高文伟*,郝转芳*
(1.新疆农业大学农学院,乌鲁木齐 830000;2.中国农业科学院作物科学研究所,北京 100081)
随着全球气候变化,降雨量不均衡导致干旱等一系列逆境胁迫的发生。在非生物逆境胁迫中,干旱胁迫是制约农作物生长发育和产量的重要因素[1]。逆境会导植物一系列形态、生理、生化和分子组成的改变[2]。在细胞和分子水平,植物通过ABA途径和非ABA途径调控一系列耐旱基因表达来响应干旱胁迫。这些基因不仅通过产生重要的功能蛋白保护细胞避免受到伤害,还通过特定基因调控信号转导来响应逆境胁迫,比如转录因子[3]。转录调控是基因表达的重要机制[4],转录因子等调控蛋白通过与基因启动子中的顺式元件相互作用,激活或抑制应激相关基因,在应激反应中发挥重要作用[5]。NAC转录因子家族是一类植物特有的参与耐逆响应的转录因子。NAC家族的命名最早源于牵牛花(Pharbitis)NAM、拟南芥(Arabidopsisthaliana)ATAF1/2和CUC2(cup-shaped cotyledon)基因[6],取三个基因的首字母命名为NAC基因[7]。NAC转录因子N端具有由150个氨基酸组成的保守NAC结构域,高度保守,C端的转录激活区则高度可变。NAC结构域主要包含5个亚域[8],其中,A、C、D亚域非常保守,而B、E在不同基因中有所差异。NAC转录因子C端通常作为转录激活物或阻遏物发挥作用,这些蛋白质通过与顺式作用的启动子元件结合而起作用,从而为启动靶基因的转录提供了触发器[9]。
植物生长和农业生产力不断受到干旱、高盐和极端温度等各种环境因素的威胁。植物已经在分子和生理水平上进化出适应胁迫的有效机制,从而使它们能够在胁迫条件下生存。近年来,已发现多个NAC转录因子直接参与或通过调控参与干旱、高盐、高温、冷害等应答基因的表达,在植物非生物逆境胁迫响应中发挥着重要作用[10]。NAC转录因子的过表达株系多表现为较强的非生物逆境耐受性,并且可以提高两种或以上的耐逆境能力。例如,过表达OSNAC10提高了水稻耐旱性[11];过表达SNAC1和ONAC045提高了水稻耐旱和耐盐性[12-13];过表达SNAC2、OsNAC5、OsNAC6提高了水稻抵御冷、旱和盐胁迫的能力[14-16]。小麦中过表达TaNAC2L可以提高转基因拟南芥的耐高温能力[17];过表达TaNAC2和TaNAC8可以提高转基因拟南芥抵御非生物的能力[18-19]。有研究在ZmNAC111基因启动子中插入微型反转重复转座因子(MITE),结果显示,MITE与玉米抗旱性的自然变异显著相关,通过在拟南芥中异源表达,82 bp 的MITE通过RNA定向DNA甲基化和H3K9二甲基化抑制ZmNAC111的表达,增强了ZmNAC111在转基因玉米中的表达,提高苗期的耐旱性,提高水分利用效率,诱导干旱响应基因在水分胁迫下的上调[20]。拟南芥中过表达ZmSNAC1(ZmNAC010312)、ZmNAC33(ZmNAC030295)和ZmNAC55(ZmNAC050436)导致种子萌发期对ABA和渗透胁迫的敏感性,提高了拟南芥的耐旱性[21-23]。同时,ZmNAC84在烟草中过表达可以提高转基因植株耐旱性,减轻干旱诱导的氧化损伤,在此过程中,ABA产生的H2O2首先诱导ZmCCaMK和ZmNAC84表达并激活ZmCCaMK,随后激活的ZmCCaMK在S113位点磷酸化ZmNAC84,从而通过激活下游基因来诱导抗氧化防御[24]。大豆GmSNAC49在拟南芥中过表达导致干旱和ABA信号通路相关基因上调,提高了干旱胁迫的耐受性[25]。葡萄VvNAC17的过表达增强了拟南芥的抗旱性,同时上调了与ABA和胁迫相关基因的表达[26]。芒属植物MLNAC10通过调节ABA信号通路发挥抗氧化酶的作用,减轻ROS的损伤,是干旱和耐盐胁迫的重要调节因子[27]。在耐干旱作物高粱中也有研究表明,13个SbNAC在开花后的干旱胁迫反应中起着积极或消极的转录调控作用[28]。
上述研究都表明,NAC基因是参与植物非生物胁迫反应的关键调控基因家族之一。玉米是主要的粮、经、饲兼用的作物,对我国农牧业有重要的支撑作用。因此,本研究利用生物信息学手段分析和鉴定玉米SNAC家族基因,系统地对成员的理化性质、基因结构、系统进化、磷酸化位点、启动子及二级结构等进行预测分析。这些信息将为进一步研究SNAC基因家族的耐逆境功能提供理论参考。
1 材料与方法
1.1 供试材料
16个玉米SNAC转录因子蛋白序列从PlantTFDB(http://PlantTFDB.cbi)获取,命名及分类参照Li等[29]方法。水稻、拟南芥、大豆等已知的与非生物胁迫相关的NAC基因组序列从NCBI数据库(https://www.ncbi.nlm.nih.gov/genome)下载。
1.2 玉米SNAC的系统发育分析
将SNAC序列通过ClustalX 1.83软件进行比对。然后使用PhyML软件(版本2.4.3)[30]中最大似然法对所有对齐的NAC序列进行系统发育分析。通过FigTree1.4.0对进化树进行可视化[31]。
1.3 玉米SANC蛋白结构分析
通过GSDS工具(http://GSDS.cbi.pku.edu.cn/)对SNAC基因的内含子和外显子结构图谱进行可视化。利用ExPASy(https://web.expasy.org/cgi-bin/protparam/protparam)数据库统计SNAS蛋白的氨基酸数目、理论等电点、分子量、不稳定参数、脂肪族氨基酸指数等理化性质信息,SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsaautomat.pl?page=npsa_sopma.html)软件对SNAC家族成员的氨基酸序列进行二级结构预测。DNAMAN 软件对SNAC家族的16个成员进行氨基酸多序列比对分析。利用MEME(http://MEME.nbcr.net/meme3/MEME.html)鉴定SNAC蛋白中存在的保守序列模体。
1.4 玉米SANC基因家族生物信息学预测分析
通过TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)对SNAC家族成员的氨基酸序列进行分析,预测其有无跨膜结构;利用 NetPhos 3.1 Server (http://www.cbs.dtu.dk/services/NetPhos/)软件对SNAC家族成员的氨基酸序列进行潜在磷酸化位点分析;利用植物顺式元件数据库PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析SNAC基因启动子区的顺式作用元件。
2 结果与分析
2.1 玉米SNAC基因家族的系统发育分析
利用PhyML软件对玉米SNAC以及其他物种中典型的耐胁迫NAC基因进行系统分析,以探讨SNAC基因之间的进化关系,结果(图1)表明,ZmNAC050302与SNAC1、ZmSNAC1(ZmNAC010312)和ZmNAC55(ZmNAC050436)同属一个亚群,SNAC1、ZmSNAC1和ZmNAC55三个基因已有被证实具有耐胁迫功能。ZmNAC030295与TaNAC6关系较近,属同一亚群。CarNAC3亚群中ZmNAC100475和ZmNAC080398关系较近,有研究发现,ZmNAC111(ZmNAC100475)基因在玉米中与耐旱相关,与其同一类群的ZmNAC080398可能具有相似的耐逆境机制。

图1 玉米、拟南芥、水稻、大豆等SNAC基因聚类分析Fig.1 Cluster analysis of SNAC genes in maize,Arabidopsis,rice and soybean
2.2 玉米SNAC基因家族氨基酸序列对比分析
SNAC转录因子在逆境胁迫中能提高植物抵抗非生物胁迫的能力,从而增加产量。利用DNAMAN软件对16 个玉米SNAC 蛋白序列进行多重比对发现,这些 SNAC 蛋白序列的N端均含有高度保守的NAC域,长度约为 150~163 个氨基酸,可以分为5个子域(A~E),其中ZmNAC070395不含有子域A。
2.3 玉米SNAC家族成员理化性质分析
SNAC家族成员的理化性质如表1所示。可以看出,理论等电点最高的是ZmNAC060339,为9.35,而ZmNAC060339的酸性氨基酸的总数比碱性氨基酸的总数少,为碱性蛋白。ZmNAC010373、ZmNAC030295、ZmNAC050436、ZmNAC060339、ZmNAC070359、ZmNAC080308、ZmNAC090241都为碱性蛋白。脂肪族氨基酸指数反映了蛋白质的热稳定性,分布在57.51(ZmNAC010373)~88.37(ZmNAC031467)之间,总体表明蛋白质间热稳定性差异较小。其中ZmNAC030295和ZmNAC080398的不稳定参数较高,分别为55.48和53.50。

表1 SNAC家族成员的理化性质Table 1 Physico-chemical properties of SNAC family members
2.4 SNAC氨基酸序列跨膜结构预测
TMHMM 综合了跨膜区疏水性、螺旋长度和膜蛋白拓扑学限制等性质[32]。用 TMHMM-2.0对16个玉米SNAC基因家族成员的氨基酸序列进行跨膜结构域分析,结果表明,这些蛋白不存在跨膜结构域,位于膜外区的可能性较大,为非跨膜蛋白类,推测均在细胞内表达和调控。
2.5 SANC基因家族成员的潜在磷酸化修饰位点预测
磷酸化和去磷酸化在多种真核细胞中的新陈代谢、细胞分裂以及信号转导等方面都发挥着至关重要的作用[33]。通过NetPhos2.0 Server 软件对玉米SNAC家族16个成员的氨基酸序列进行潜在磷酸化位点的分析,结果如表2所示。可以看出,玉米SNAC家族16个成员的氨基酸序列均有潜在的磷酸化位点,这些潜在的磷酸化位点分别位于丝氨酸Ser、苏氨酸Thr和酪氨酸Tyr上,该结果表明,SNAC家族的成员能够被丝氨酸激酶、苏氨酸激酶和酪氨酸激酶所磷酸化,从而实现其功能的调控。玉米SNAC家族存在着大量的磷酸化位点,推测SNAC基因家族可能参与着大量的翻译后修饰。由表中可以看出,SNAC家族成员中ZmNAC031467潜在的磷酸化位点数最多,其次是ZmNAC100475和ZmNAC080398,最少的是ZmNAC020303,只有13个潜在的磷酸化位点。
2.6 SNAC家族成员蛋白质二级结构
采用SOPMA 对SNAC家族成员的氨基酸序列进行蛋白质二级结构的预测,结果如表2和图2所示。可以看出,组成SNAC家族成员的多肽链的二级结构主要有4种类型,分别为α-螺旋(alpha-helix)、β-转角(beta-turn)、延伸链结构(etended strand)以及无规则卷曲(rndom coil),以α-螺旋和无规卷曲为主,并且无规则卷曲占的比例最高,而β-转角和延伸链结构所占的比例则相对较少。对SNAC家族成员二级结构的预测和分析有助于认识蛋白的空间结构。

表2 SNAC家族成员的二级结构 4 种类型所占的比例及磷酸化位点预测Table 2 Proportion of 4 types of secondary structure of SNAC family members and prediction of phosphorylation sites

图2 SNAC家族成员的二级结构Fig.2 Secondary structure of SNAC family members
2.7 玉米SNAC蛋白的保守结构域分析
利用MEME工具来识别玉米SNAC蛋白中10个不同的保守模体(图3)。根据10个预测基序的分布,16个SNAC成员与系统发育分析的分类一致。NAC转录因子家族成员N端都具有高度保守的结构域,由大约150个高度保守的氨基酸残基组成,可以形成螺旋-转角-螺旋的三维结构,并能特异结合目标DNA和蛋白[34]。图3中16个SNAC蛋白都具有基序2和基序3,而ZmNAC070359不具有基序1,在氨基酸序列分析中ZmNAC070359不具有子域A,但还是与剩余15个SNAC蛋白共享子域B和C。此外,尽管转录激活区相对不同,但在一些SNAC亚群中,转录激活区还是存在一些保守的子域。基序6在ZmNAC010312、ZmNAC050436和ZmNAC050302中都存在。同时,ZmNAC010312存在的基序5也与ZmNAC020303、ZmNAC050302、ZmNAC050436、ZmNAC070300共享。基序8也存在于多个基因的转录激活区域,如ZmNAC010373、ZmNAC031467、ZmNAC070395、ZmNAC080308、ZmNAC080398和ZmNAC100475。ZmNAC020303、ZmNAC031467、ZmNAC070300和ZmNAC080308都具有基序9。耐旱因子ZmNAC100475上的基序9也存在于ZmNAC031467、ZmNAC040359和ZmNAC080308中。

图3 SNAC家族成员的基因结构分布及蛋白基序分布Fig.3 Distribution of gene structure and protein sequence of SNAC family members
2.8 玉米SNAC基因结构分析
对玉米SNAC基因的核苷酸结构分析可知,ZmNAC050436不存在上游调控区域,其余基因均存在上下游调控区域。在内含子方面,6个基因(ZmNAC010312、ZmNAC020303、ZmNAC050436、ZmNAC050302、ZmNAC070300、ZmNAC070359)由一个内含子构成,6个基因(ZmNAC010373、ZmNAC030295、ZmNAC060339、ZmNAC080308、ZmNAC080398、ZmNAC100475)由两个内含子构成,3个基因(ZmNAC040359、ZmNAC070395、ZmNAC090241)由三个内含子组成,而ZmNAC031467由8个内含子构成,这可能与基因序列较长有关。玉米SNAC基因内含子较少,这可能与启动下游基因表达有关,越少的内含子可以更快的启动转录激活,从而加快相关基因的表达或抑制。ZmNAC050302、ZmNAC050436和ZmNAC070300三个基因结构相似度较高,分别与ZmNAC070300基因的相似度为50.88%和53.76%,同时ZmNAC050302和ZmNAC050436两个基因相似度最高达93.58%。结合系统发育分析,很明显同一亚群中最密切相关的成员具有相似的基因结构。
2.9 启动子分析
SNAC基因的转录调控与启动子区应激反应调控元件的存在有关,如DRE(dehydration-responsive elements)、ABRE(ABA-responsive elements)、茉莉酸反应元件、水杨酸反应元件、LTRE(low-temperature responsive elements)、MYB(myeloblastosis)和MYC(myelocytomatosis)结合位点[35]。通过生物信息学分析和预测,玉米SNAC基因家族启动子区域均含有大量的逆境响应相关元件(表3),这些元件参与逆境胁迫应答(干旱)和脱落酸(ABA)诱导的表达调控,其中所有成员都含有DRE、MYB两种调控元件;15个SNAC启动子含有ABRE调控元件。ZmNAC030295、ZmNAC031467、ZmNAC090241的启动子含有较多ABRE元件,分别含有10、14、12个,说明对脱落酸诱导的表达调控较为敏感。这三种转录因子与抗非生物逆境调控有关,另外还发现有MYC调控元件。此外,某些基因还具有独特的顺式作用元件,如ZmNAC030295含有多个参与光响应的顺式作用元件(G-box、TCT-motif、TCCC-motif);ZmNAC050302含有赤霉素反应元件(GARE-motif);ZmNAC080398含有参LTRE顺式作用元件 。以上结果表明,玉米SNAC家族基因通过这些元件的调控可能在玉米逆境响应中发挥着重要的作用。
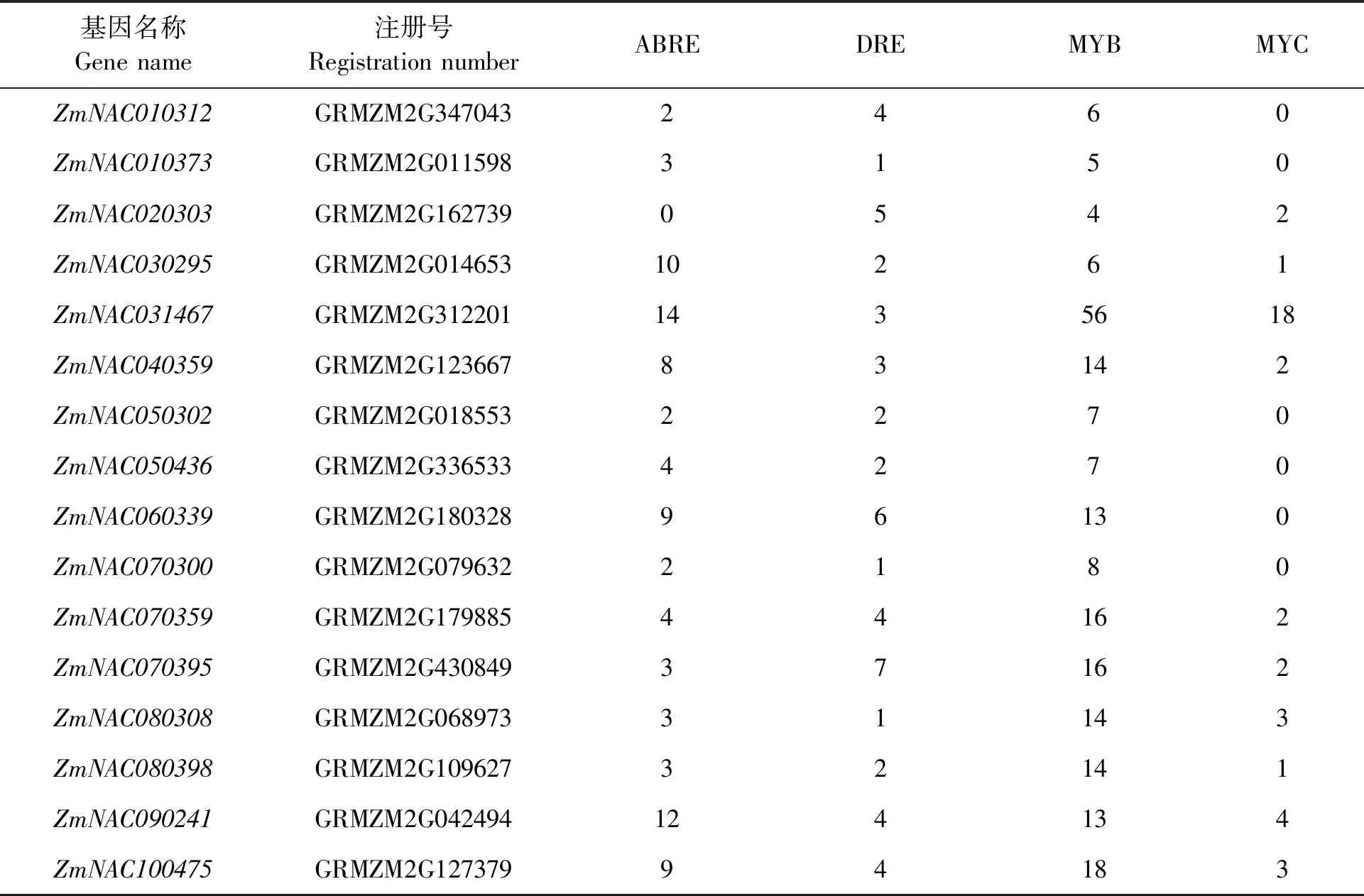
表3 玉米SNAC基因家族启动子ABRE、DRE、MYB、MYC元件的数量分布Table 3 Distribution of ABRE,DRE,MYB,MYC components in promoters of SNAC gene family in maize
3 讨论
玉米是我国三大粮食作物之一,是高效的经济作物,对我国经济发展有着十分重要的影响,也是调整我国农业生产结构的核心。非生物胁迫引发一系列植物反应,包括基因表达模式和细胞代谢的改变。转录因子调节介导多种非生物胁迫反应和生物过程的基因表达[36]。本研究对玉米SNAC家族进行了生物信息学分析,结果表明,玉米SNAC家族存在着大量的磷酸化位点,同时因不具有跨膜结构,推测均在细胞内表达和调控。此外,脂肪族氨基酸指数分布区域范围较小,总体表明蛋白质间热稳定性差异较小,而ZmNAC030295和ZmNAC080398的不稳定参数较高,因此推测该基因可能还具备其他功能,有助于提高玉米SNAC基因家族功能的多样化。启动子分析表明,玉米SNAC家族启动子区域均含有大量的逆境响应相关元件,这些元件通过与相应的转录因子结合来调控下游基因的表达从而参与到植物抗逆响应。二级结构分析表明,玉米SNAC的转录调控区具有高度的内在混乱性,使得SNAC蛋白不能有一个单一稳定的结构,而这种灵活性使SNAC蛋白能与不同的靶蛋白相互作用,同时也使SNAC蛋白成为转录因子功能和结构系统分析的模式蛋白,造就了SNAC转录因子不同的功能作用。
根据在拟南芥、水稻、大豆中报道的几个典型耐胁迫NAC基因,16个玉米SNAC家族蛋白共有4个保守基序,构成一个NAC结构域,在识别和结合特定DNA序列中起作用,而位于C末端的转录区域被认为是反式激活结构域[37]。据报道,SNAC1、GmSNAC49和CarNAC3所在非生物和生物胁迫耐受性中起着至关重要的作用。例如,水稻SNAC1基因导入小麦中的过表达株系与野生型相比,过表达的植株表现出对干旱和高盐的耐受性显著增强,正调控与非生物胁迫ABA信号转导途径关联基因的表达[38]。大豆GmSNAC49的过表达导致干旱和ABA信号通路相关基因的上调,在耐旱性中发挥积极作用;鹰嘴豆CarNAC4在拟南芥中的过量表达提高了拟南芥对干旱和盐胁迫的耐受性[39]。这些结果表明,进化关系密切的NAC基因在植物中可能具有相似的生物学功能。这些信息可用于探索植物,特别是主要作物玉米中潜在的胁迫相关SNAC基因。
系统进化分析表明,ZmNAC030295与TaNAC6基于同一亚群,同时有研究揭示,TaNAC6在提高小麦耐逆性和调控小麦生长发育方面的潜在作用[40]。同时ZmNAC050302与三个耐胁迫因子ZmNAC050436、SNAC1和ZmNAC010312在同一亚群,ZmNAC050302、ZmNAC050436和ZmNAC010312都仅有1个内含子。结构域分析表明,ZmNAC050302、ZmNAC050436、ZmNAC010312三个基因具有相同的基序,ZmNAC050302和ZmNAC010312共享基序6。这些结果预测ZmNAC050302在抗逆境方面有一定的正向调控作用。同时,ZmNAC080398与ZmNAC111(ZmNAC100475)的进化关系较近,具有相似的基因结构,在具有A~E亚域的基础上还共享基序8,说明ZmNAC080398也可能在玉米胁迫环境中起着一定作用。同时,过量表达SNAC2和OsNAC6能提高转基因水稻的抗旱和抗盐能力[15-16],本研究中16个SNAC基因家族通过玉米抗旱自交系Tie 7922经过水分胁迫之后,各个基因在不同时间不同部位都发生显著变化,比如,GRMZM2G430849_P01(ZmNAC050302)基因在胁迫后24 h表达量最高,而GRMZM2G162739_P01(ZmNAC080398)基因在1 h这个时间段表达最为明显[43]。所以预测它们在非生物胁迫中参与了响应。
总之,本研究系统分析了玉米SNAC蛋白的系统发育关系和保守基序。在胁迫相关亚群中,一些预测的应激反应SNAC基因被筛选分离出来。生物信息学的数据加深了对玉米SNAC基因参与植物对非生物胁迫反应的理解,为进一步揭示玉米SNAC基因家族的耐逆境功能,抗逆分子育种和挖掘SNAC基因应用潜力提供参考。

