食品中沙门氏菌恒温隔绝式PCR检测方法的建立
杨若璇,佟尧,赵燕英,汤承,刘骥,朱成林,曾英杰,于基成,唐俊妮*
(1.西南民族大学食品科学与技术学院,四川成都 610041)(2.西南民族大学青藏高原动物遗传资源保护与利用教育部重点实验室,四川成都 610041)(3.西南民族大学畜牧兽医学院,四川成都 610041)(4.大连民族大学生物技术与资源利用教育部重点实验室,沈阳大连 116600)
沙门氏菌(Salmonella)是一种革兰氏阴性的杆状细菌,属肠杆菌科,是常见的食源性病原菌。自1885年首次被分离出来,沙门氏菌血清型已发展到2637种。沙门氏菌是一种严重危害动物和人类健康的病原菌,在人类及畜禽中均曾有过沙门氏菌病爆发的记录[1]。由沙门氏菌感染而引发的病症有败血症、伤寒及肠胃炎等,婴儿、老年人及免疫力低下者还可能出现菌血症,少数还会伴随脑膜炎或骨髓炎。全球每年有1600万沙门氏菌感染病例,其中约60万死亡,据相关报道,国内外沙门氏菌污染引起食物中毒的人数频占首位[2]。近年来,许多分离菌株出现了抗生素高度耐药,给公众健康带来了巨大健康威胁[3]。因此,对沙门氏菌的检测非常重要。
对食品中沙门氏菌进行检测是预防沙门氏菌感染的有效手段。目前,沙门氏菌的检测方法主要有传统检测方法、免疫学方法、分子生物学方法以及生物传感器检测的方法等。传统检测方法依据GB 4789.4-2016《食品安全国家标准食品微生物学检验沙门氏菌检验》[4]的标准方法,检测步骤为预增菌、增菌、分离培养、生化实验以及血清学鉴定分型等,存在耗时长,操作繁琐等缺点[5]。近年来,学者们一直在寻找更快速的检测方法,如李倩影等[6]利用pH响应比色酶联免疫吸附法检测牛奶中猪霍乱沙门氏菌,所构建的比色ELISA方法适用于牛奶中不同浓度的猪霍乱沙门氏菌快速定量检测。Li等[7]用荧光DNAzyme和通用封堵连接剂、超聚合酶链反应形成视觉生物传感器,实现信号放大来检测沙门氏菌等。虽然这些方法快速,但也存在一定缺陷,如免疫学方法易出现假阳性或者假阴性,生物传感器在稳定性和精确度上还有待改善,并且对设备的要求非常高[8,9]。乐振窍等[10]用等温多自配引发扩增技术(Isothermal Multiple Self-matching-initiated Amplification,IMSA),建立了检测沙门氏菌的IMSA法,适合用于食品中沙门氏菌快速检测。相比较而言,分子生物学具有特异性好、灵敏度高、快速准确等优点,已经广泛应用于微生物检测中[11,12]。但是目前存在最大问题是适用于现场快速检测方法还比较匮乏。因此,针对沙门氏菌探索适用于现场快速检测的方法非常有必要。
恒温隔绝式PCR技术(Insulated isothermal PCR,iiPCR)是一种新型的分子生物学快速检测技术,基于Rayleigh-Benard的对流原理,通过反应管底部液体持续加热产生上部液体与下部液体的温度差,控制散热的速率与对流的时间,在反应管内部形成稳定温度梯度,从而提供PCR扩增所需的反应条件[13]。该技术最早在2002年,由Krishnan[14]首次报道,结合POCKITTM系列手持式核酸检测仪,体积小、自带电源,加样后一键操作,反应时间短,灵敏度高,方便快捷,可达到现场快速检测的目的。
当前国内外报道iiPCR技术的文献主要应用于虾白斑病病毒[15]、尖孢镰刀菌[16]、滑膜支原体[17]、无乳链球菌[18]、猪流行性腹泻病毒[19]、牛冠状病毒[20]等。利用该技术对食源性病原菌的研究还较少。本实验的目的是基于恒温隔绝式PCR技术建立一种快速检测沙门氏菌的方法,并应用于实际食品样品检测,为沙门氏菌现场快速检测平台建设提供参考。
1 材料与方法
1.1 菌株
本研究所用到的沙门氏菌H9812、单增李斯特菌ATCC19115、金黄色葡萄球菌ATCC6538、大肠杆菌ATCC25922、蜡样芽孢杆菌ATCC11778 均由本实验室保存。
1.2 主要试剂与仪器
亚硒酸盐胱氨酸增菌液(SC)、亚硫酸铋琼脂(BS)、缓冲蛋白胨水(BPW)、胰蛋白胨大豆琼脂(TSA)、1%亚碲酸钾卵黄增菌液,青岛海博生物技术有限公司;Premix Ex Taq(Probe qPCR)、TB Green Premix Ex TaqⅡ、核酸染料,GELVIEW 宝生物工程(大连)有限公司;50 bp DNA Ladder、Taq PCR Master Mix (2x,blue dye),北京擎科新业生物技术有限公司;氯化钠,天津市致远化学试剂有限公司;G-10 电泳琼脂糖凝胶,Biowest 公司;Tris-HCl、EDTA,北京博奥拓科技有限责任公司。
POCKITTM小型智能型核酸分析仪、R-tube(48),厦门金瑞鸿捷生物科技有限公司;CFX96 荧光定量PCR 仪、VerSaDoc2000 凝胶成像系统,美国Bio-Rad有限公司;PCR 仪,TSNENEN031445 基因(美国)有限公司;Galanz WD800B 型微波炉,顺德市格兰仕微波炉电器有限责任公司;SC-15 数控超级恒温槽,宁波新芝生物科技股份有限公司;Eppendorf 5804R 型冷冻离心机,Eppendorf 公司;DYY-6C 电泳仪,北京六一仪器厂。
1.3 引物及探针的设计与合成
选取沙门氏菌的invA基因作为目标基因,利用Primer Premier 5 软件设计一对引物及探针,并对引物与探针进行BLAST 比对。另一对常规PCR 引物参考Chen 等[21],详细引物信息见表1。引物和探针均由生工生物工程(上海)股份有限公司合成。
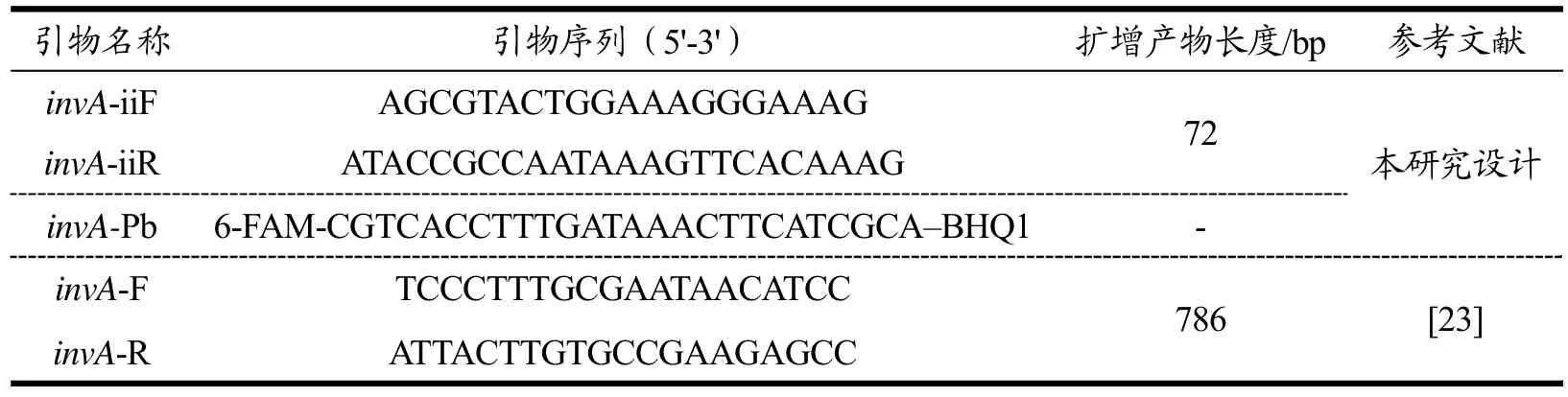
表1 invA 特异性引物和探针基本信息 Table 1 invA specific primers and probes
1.4 模板DNA 的提取
本实验参考陈光丽等[22]水浴提取法提取细菌DNA。
1.5 引物及探针的验证
采用实时荧光定量PCR 仪(CFX96)对沙门氏菌、单增李斯特氏菌、金黄色葡萄球菌、蜡样芽孢杆菌及大肠杆菌及阴性对照进行扩增检测,以验证引物和探针的可行性。反应体系及条件:总反应体积为20 μL:10 μL Premix Ex Taq,1.2 μL 探针(10 μmol/L),上、下游引物各0.4 μL(10 μmol/L),1.0 μL DNA 模板,其余用无菌水补齐。反应条件:95℃变性1 min 30 s,以95℃ 5 s,55 30℃ s ,72 30℃ s 扩增40 个循环,在55℃结束开始收集荧光信号。
1.6 iiPCR 反应体系的摸索
以李凡等[20]建立的牛冠状病毒iiPCR 方法进行参考,采25 μL 体系分别对已知浓度的Taq 酶(预混酶)5 U/μL(11~13.5 μL)上下游引物10 μmol/μL(0.5~2 μL)、探针10 μmol/μL(0.25~1 μL)、模板DNA(1~4 μL)、无菌水的用量进行条件探索,确定各自用量的最佳值。
1.7 特异性试验
采用上述扩增体系,结合POCKITTM手持式核酸分析仪对上述提取的沙门氏菌和其他细菌的DNA 作为模板进行检测,判定方法的特异性。
1.8 灵敏性试验
将纯培养的沙门氏菌菌液,通过十倍梯度稀释,稀释成不同的浓度梯度(100~10-9),并同时进行平板计数,实验设置三个平行。采用POCKITTM手持式核酸分析仪进行样品的检测,确定最低检出限。
1.9 稳定性试验
参考刘健新等[23]荧光定量RT-PCR 检测方法,分别对过夜培养液的3 个稀释度(10-6、10-7、10-8)提取的模板DNA 采用POCKITTM手持式核酸分析仪进行三次重复性试验,以评价该方法的稳定性。
1.10 实际食物样品的采集
市场上采集14份食品样品,样品采集信息见表2。

表2 样品采集信息 Table 2 The sample collection information
1.11 食物样品前增菌培养时长探索
将食物样品按照每份10 g直接分装至缓冲蛋白胨水中富集培养,于培养第2 h 开始,平均每隔1 h 采集一次培养液,直至第8 h,采集前需静置5 min,利用iiPCR 和传统PCR 对上述采集点的培养液进行检测,探索实际样品最小培养时长。
传统PCR 反应体系及反应条件:Taq PCR Master Mix(2×,blue dye)10 μL、上游引物0.4 μL、下游引物0.4 μL、模板DNA1 μL、无菌超纯水补足至20 μL体系;反应扩增条件:95℃预变性5 min、95℃变性40 s,54℃退火50 s,72℃延伸40 s,35 个循环;72℃延伸10 min。产物于4℃保存或进行凝胶电泳检测。
产物的琼脂糖电泳检测:配置的琼脂糖凝胶的浓度为3%,取4.5 μL 扩增产物进行点样,90 V、72 A条件下电泳40 min,在凝胶成像系统下观察结果。
1.12 实际采集食物样品检测
针对市场上采集的14 份食品样品,采用传统PCR方法、iiPCR 方法对样品进行检测,比较其检出率,同时采用传统培养法对结果进行验证。并将建立的iiPCR 方法,与国标法、传统PCR 方法和荧光定量PCR从样品采集到结果检出整个流程制作流程对比图。
1.13 数据处理
数据采用Microsoft Excel、SPSS 25 软件进行分析处理。
2 结果与分析
2.1 引物及探针验证结果
成功建立iiPCR 检测方法的前提是选择合适的靶基因和探针设计[24],因此,本实验的引物和探针设计至关重要。肠侵袭蛋白与沙门氏菌的侵袭能力紧密相关[25],其中invA是主要的毒力因子,可以作为沙门菌属的特异性基因[26]。因此,本实验选择invA基因作为研究的靶基因。通过本实验设计引物和探针,采用传统PCR 和实时荧光定量PCR,分别对沙门氏菌、单增李斯特氏菌、金黄色葡萄球菌、蜡样芽孢杆菌及大肠杆菌进行扩增,结果见图1,从图中可以看出只有沙门氏菌能够出现特异性扩增,其他4 株菌未见任何扩增,证明设计的沙门氏菌引物与探针具有良好的特异性。
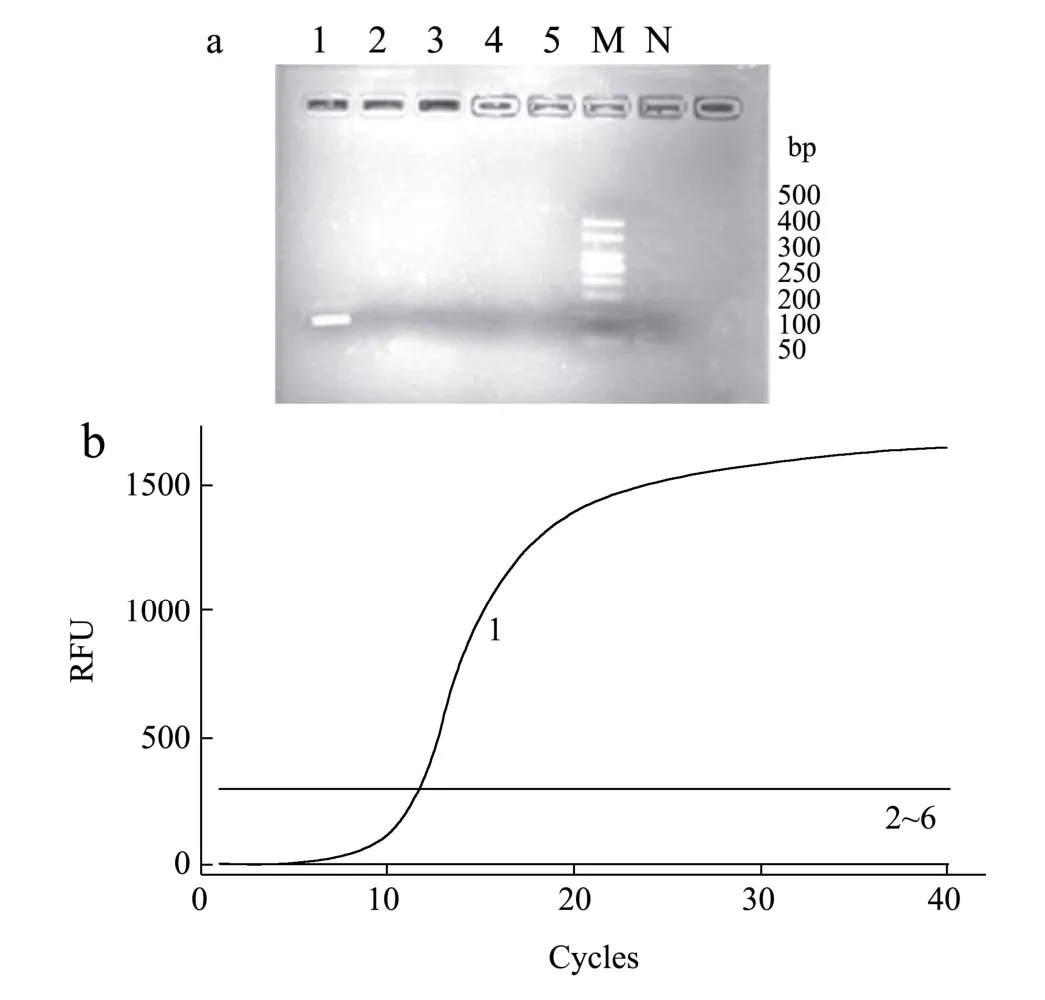
图1 沙门氏菌的引物、探针验证结果 Fig.1 The primer and probe verification for Salmonella
2.2 反应体系优化探索结果
通过对上、下游引物区间范围用量、探针区间范围用量、模板区间范围用量进行优化筛选,摸索出沙门氏菌iiPCR 最终反应体系为:5 U/μL Taq 酶(预混酶)12.5 μL,10 μmol/μL 上、下游引物各1 μL,10 μmol/μL 探针0.5 μL,模板2 μL,补足无菌水至25 μL。
2.3 特异性试验结果
进一步将沙门氏菌、单增李斯特氏菌、金黄色葡萄球菌、大肠杆菌、蜡样芽孢杆菌的DNA 按上述程序进行iiPCR 扩增,结果见图2,图中POCKITTM手持式核酸分析仪界面上显示只有沙门氏菌检出为阳性(+),单增李斯特氏菌、金黄色葡萄球菌、大肠杆菌、蜡样芽孢杆菌和阴性对照均检出为阴性(-),初步表明针对沙门氏菌建立的iiPCR 检测方法特异性良好。

图2 沙门氏菌特异性试验 Fig.2 Specific test for Salmonella
2.4 灵敏度和稳定性试验结果
将纯培养浓度为7.5×108CFU/mL 沙门氏菌菌液,通过梯度稀释至不同的浓度,然后采用上述扩增体系对不同稀释浓度的培养液(7.5×102CFU/mL、7.5×101CFU/mL、7.5 CFU/mL)进行检测,从图3 中可以看出,POCKITTM手持式核酸分析仪界面上3 号孔显示为阴性(-),1 号孔和2 号孔显示为阳性(+),三次重复实验的结果一致,表明所建立的iiPCR 方法稳定性良好,对沙门氏菌纯培养液的最低检出限为75 CFU/mL。
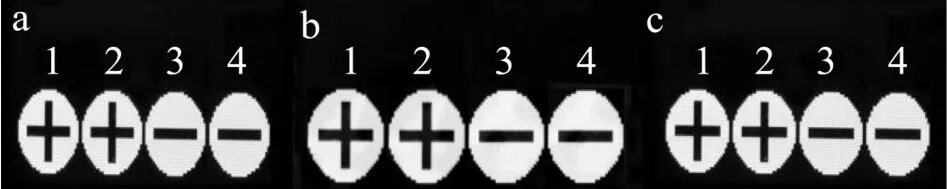
图3 沙门氏菌稳定性和灵敏度试验 Fig.3 The stability and sensitivity test evaluation for Salmonella
进一步将纯培养浓度为7.5×108CFU/mL 的沙门氏菌菌液通过十倍梯度稀释培养液,分别采用传统PCR 方法及实时荧光定量PCR 方法进行灵敏度检测对比,结果见图4a 和4b。由图4a 可得,传统PCR方法对沙门氏菌的最低检出限为泳道6 中的7.5×103CFU/mL;由图4b 可得,实时荧光定量PCR 方法对沙门氏菌的最低检出限为7.5×102CFU/mL;而本研究建立的iiPCR 方法对沙门氏菌的最低检出限为75 CFU/mL,灵敏度比传统PCR 灵敏100 倍,比实时荧光定量PCR 灵敏10 倍。
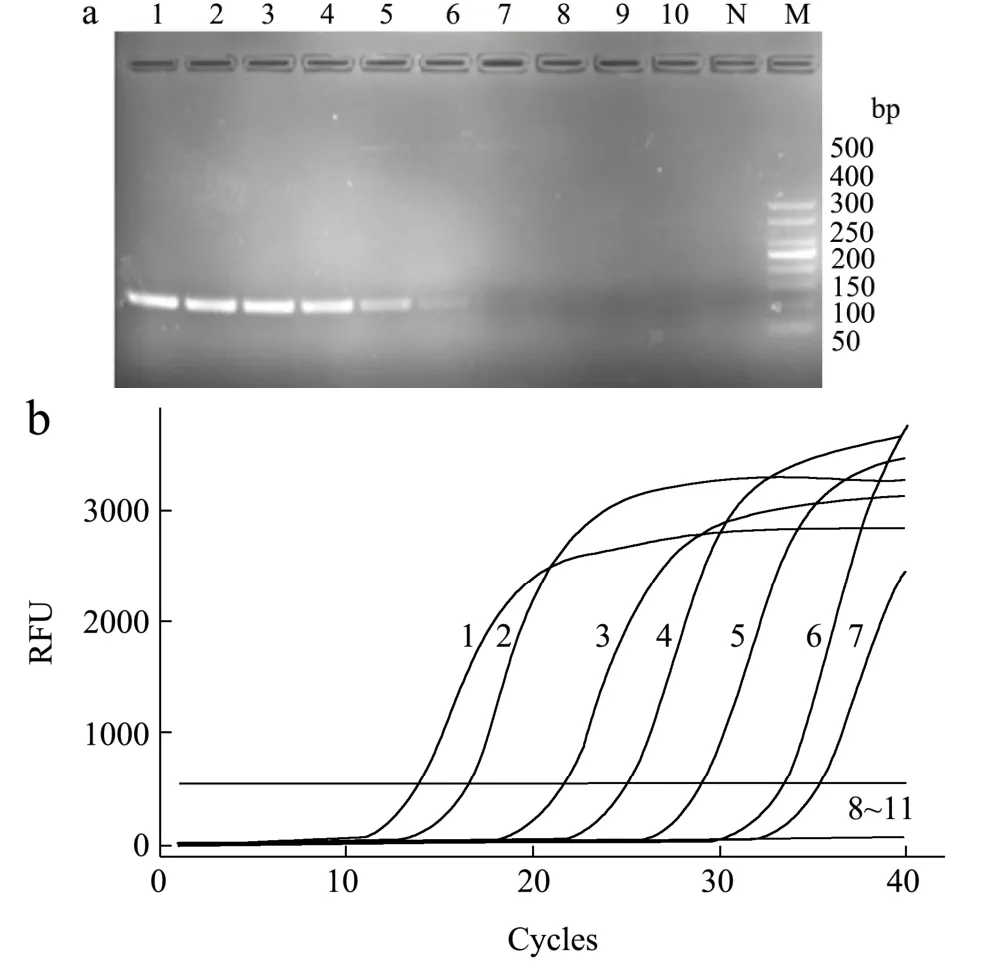
图4 沙门氏菌的传统PCR 和实时荧光定量PCR 敏感性试验 Fig.4 Sensitivity test by traditional PCR and real-time fluorescence quantitative PCR for Salmonella
2.4 食物样品前增菌培养时长的探索结果
对实际采集的阳性样品在缓冲蛋白胨水(BPW)中进行预增菌,通过对2、3、4、5、6、7 和8 h 时的培养液进行检测,结果见图5。由图5a 可以看出,采用传统PCR 检测,在8 h 预增菌的培养点上可以检测出阳性条带。由图5b 可以看出,iiPCR 在5 h、6 h、7 h 和8 h 的预增菌培养点上都可以检测出阳性结果,说明建立的iiPCR 方法检测的灵敏度更高。
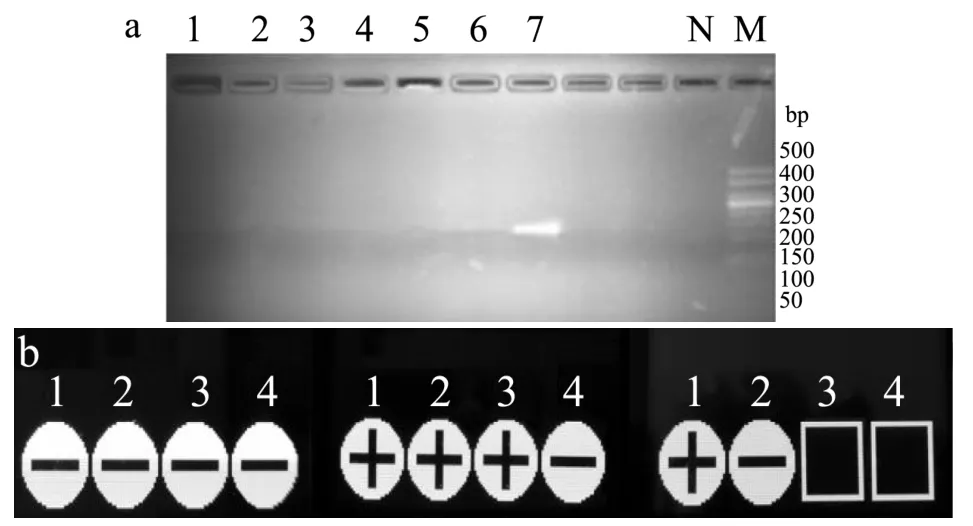
图5 实际样品前增菌培养时间探索 Fig.5 Exploring the pre-enrichment and cultivation time for actual samples detection
2.5 方法的初步应用
采用建立的iiPCR方法对采集的14份实际样品进行检测,在5 h 增菌培养点,iiPCR 方法扩增结果见图6,iiPCR 方法对2 份样品检出沙门氏菌呈阳性结果,检出率为14%(2/14)。

图6 食品样品iiPCR 在5 h 培养点的检出结果 Fig.6 The iiPCR detection results for food samples at 5 h
2.6 三种检出方法的比较及检测流程对比
采用建立的iiPCR方法与传统PCR方法以及传统培养分离法对采集样品进行检出结果比较,结果统计见表3。在8 h 培养点时传统PCR 方法扩增结果见图7,传统PCR 方法也可对2 份样品(11 号和12 号)检出沙门氏菌阳性结果,检出率为14%(2/14)。根据GB 4789.4-2016 对18 h 培养点的样品进行分离鉴定,结果见图8a,发现所检出的2 份阳性样品分离的菌落,可在BS 培养基上生长为圆形、呈黑色并带有金属光泽的菌落,在传统培养法分离的菌落进行常规PCR 鉴定,检测结果见图8b,图中也显示只有11 号和12 号检测结果呈阳性,其余均为阴性,与5 h 培养点时的iiPCR 方法检测出阳性结果一致。
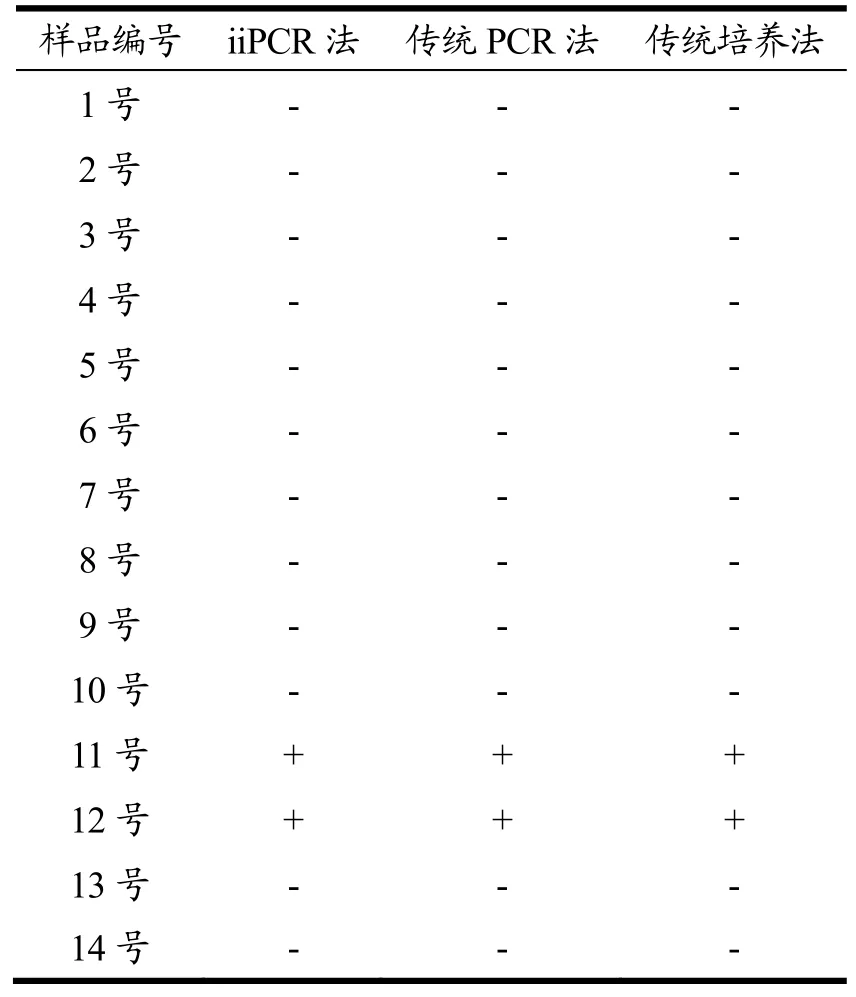
表3 三种检测方法的比较 Table 3 The comparison of three detection methods
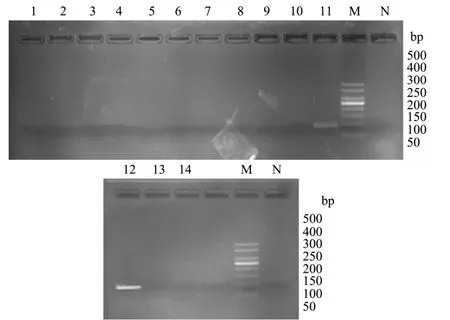
图7 食品样品经过8 h 预增菌的PCR 检出结果(沙门氏菌)Fig.7 Food sample detection results of traditional PCR by 8 h pre-enrichment culture (Salmonella)

图8 传统培养法分离的菌落(a)和分离细菌的PCR 鉴定结果(b)Fig.8 The traditional culture method validation (a) and PCR identification results (b)
对建立的iiPCR 方法和传统培养法、传统PCR 方法、荧光定量PCR 进行对比,从样品的采集到结果的检出,传统培养法整个鉴定检测流程至少需要4~6 d[27],传统PCR 方法检测整个流程需12~16 h 甚至更久,荧光定量PCR 方法检测整个流程需10~12 h 左右,而iiPCR 方法检测整个流程可在6~8 h 完成,比其他检测方法节省很多时间,并且可直接得出结果,操作更便捷,如图9。
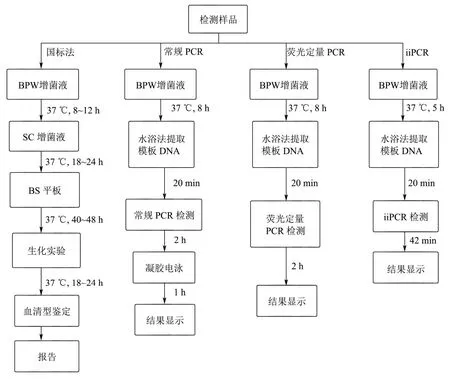
图9 iiPCR 方法和其他检测方法对比流程图 Fig.9 The comparison flow chart of iiPCR and other detection methods
细菌在接种培养后,前1~4 h 生长一般迟缓,繁殖数量较少,迟缓期后,进入对数期,这个时期细菌开始大量繁殖。本研究通过探索对于食品样品中沙门氏菌前增菌培养时间,只需在缓冲蛋白胨水中培养5 h后配合水浴法提取模板DNA,利用建立的iiPCR 方法即可检测出样品中是否含有沙门氏菌。研究结果和Tsen 等[28]及Du 等[29]探索结果相似,Tsen 等[28]针对鸡肉中沙门氏菌检测,验证鸡肉样品经过富集以及DNA提取步骤,可利用iiPCR 在8 h 内完成检测结果。Du等[29]针对餐饮中的沙门氏菌和志贺氏菌进行iiPCR 方法的开发和评估,说明该技术对食品污染致病菌的检测应用是可行的。本实验建立的iiPCR 方法灵敏度比Du 等建立的iiPCR(最低检出限103CFU/mL)灵敏度高,本实验检出限为75 CFU/mL。结合POCKITTM系列手持式核酸检测仪42 min 的反应和结果显示,大大节省时间,从目标细菌富集,细菌模板DNA 提取,靶特异性基因扩增,到检测结果显示,整个检测流程可在6~8 h 左右完成。
3 结论
3.1 本研究建立了检测沙门氏菌的iiPCR 方法,克服了仪器昂贵、不方便携带、操作人员要求高等缺点。选择沙门氏菌invA 基因作为本方法的靶基因,设计了引物和探针,对iiPCR 反应体系进行优化,获得最佳反应体系。经实验得出建立的iiPCR 方法灵敏度高,沙门氏菌的最低检出限可达75 CFU/mL,采用iiPCR检测其他细菌(单增李斯特氏菌、金黄色葡萄球菌、大肠杆菌、蜡样芽孢杆菌等)结果无交叉反应,说明特异性良好。并分别对沙门氏菌培养液的三个稀释度进行了三次重复实验,结果完全一致,说明建立的iiPCR 方法稳定性良好。
3.2 利用上述建立的方法对实际采集食品样品进行应用检测,将采集的14 份食品样品直接在缓冲蛋白胨水中富集培养,采集2、3、4、5、6、7、8 h 的培养液,同时采用建立的iiPCR 方法和传统PCR 方法进行样品检测和比对,结果表明在5 h 的培养点,建立的iiPCR 方法能对2 份样品可检测出沙门氏菌阳性结果,检测率为14%(2/14);而采用传统PCR 方法在8 h培养点时才能对这2 份样品检测出阳性结果,检出率为14%(2/14)。虽然实时荧光定量PCR 灵敏度要高于传统PCR 2~3 个数量级,但本研究建立的iiPCR 方法比实时荧光定量PCR 的灵敏度高10 倍,且基于4通道POCKITTM 手持核酸分析仪,自带电源,体型小,方便携带并且一键操作,加样后直接反应,检测过程仅42 min,反应后结果直接显示于液晶屏,省去分析及凝胶电泳等繁琐过程,整体耗时要远比传统PCR 和荧光定量PCR 检测方法少。可见,针对实际样品的检测,建立的iiPCR 更加快捷、灵敏和实用。但本研究也存在不足,特别是方法中涉及的阳性菌株和阴性菌株数量不够多,为更好地评价建立的iiPCR方法的特异性和灵敏度,我们会在后续的研究中进一步增加食品样品采集数量,并与传统法和荧光实时定量PCR 方法进行比较,以达到方法的推广使用。
3.3 综上,本研究建立的一种基于恒温隔绝式PCR技术检测沙门氏菌的方法,对方法进行部分应用,初步证明该方法灵敏度高,特异性好,稳定性好。将该方法结合水浴法快速提取模板DNA 以及POCKIT™手持式核酸分析仪,通过设计特异性引物和探针,可实现对沙门氏菌现场快速检测。从目标细菌富集,到模板DNA 提取,特异性基因扩增到出现检测结果,整个过程可在6~8 h 完成。相比其它检测方法,至少节省6 h 检测时间,省去凝胶电泳过程,操作更便捷。

