测序仪性能评价用脱氧核糖核酸国家参考品的建立
曲守方,黄传峰,孙楠,于婷,黄杰
·论著·
测序仪性能评价用脱氧核糖核酸国家参考品的建立
曲守方*,黄传峰*,孙楠,于婷,黄杰
100050 北京,中国食品药品检定研究院非传染病诊断试剂室(曲守方、孙楠、于婷、黄杰),理化检测室(黄传峰)
建立测序仪性能评价用脱氧核糖核酸国家参考品。分别将扩增培养后的永生化的人细胞系、培养达到 108数量级的转染 HPV11 质粒的细菌、大肠杆菌和欧陆森氏菌高 GC 菌株收集后提取基因组 DNA,然后进行 DNA 浓度测定。接着将人基因组 DNA、HPV 病毒基因组 DNA、大肠杆菌基因组 DNA 和高 GC 菌株基因组 DNA 分别按照 20、5、50、50 ng/μl 终浓度要求,进行分装,制备国家参考品。在不同的基因测序仪进行上机测序,包括 BGISEQ-500、NextSeq CN500、NovaSeq 6000 和 BioelectronSeq 4000等。构建人全基因组变异标准集和一致性序列。采用不同测序平台进行协作标定,评价变异标准集和一致性序列的适用性。制备的国家参考品经不同平台测序后构建了人全基因组变异标准集和一致性序列。经过不同测序平台标定,结果符合人全基因组变异标准集和一致性序列的要求。测序仪性能评价用脱氧核糖核酸国家参考品可以用于高通量基因测序仪的性能评价,为产品注册检定及上市后监督管理提供依据。
高通量基因测序仪; 测序通量; 标准集; 一致性序列; 单核苷酸多态性; 插入缺失
高通量测序技术在临床上已经被广泛应用于遗传病、肿瘤和传染病的诊断、预后和治疗选择等[1-3]。随着测序成本降低,越来越多患者受益于这些基于高通量测序的检测技术。
目前国内市场上存在着多种二代测序仪。Illumina 公司的测序平台采用边合成边测序(sequencing by synthesis,SBS)技术原理,是基于 DNA 簇(DNA cluster)、桥式 PCR 和可逆阻断等核心技术[4]。Thermo Fisher 公司拥有 Solid 测序平台、Ion PGM 和 Ion Proton 平台,采用的是半导体测序原理,在半导体芯片孔中的微球上固定 DNA 链,随后依次掺入碱基(A 腺嘌呤、C 胞嘧啶、G 鸟嘌呤、T 胸腺嘧啶),每加入一个碱基就释放出氢离子,反应池中的酸碱度(pH)发生改变,离子感受器就会感受到化学信号,从而读出 DNA 序列[5]。华大基因公司收购了美国 Complete Genomics 公司并改进其技术,推出了 BGISEQ-500、BGISEQ-50、MGISEQ-200、MGISEQ-2000 和 MGISEQ-T7 测序仪,测序原理是以改进的 DNA 纳米球技术来扩增 DNA 序列,并使用联合探针锚定聚合技术(combinatorial probe-anchor synthesis,cPAS)进行测序[6-7]。
不同测序平台之间存在系统性差异,Illumina 测序平台在读取高 AT 或高 GC 富集片段的时候错误率差强人意;Ion Torrent 测序平台对于检测同一碱基连续出现时的数量可能会有所误差;华大测序平台在敏感性与特异性之间做到平衡优化方面存在不足[8-10]。随着精准临床诊断的兴起,基因测序准确性也面临着巨大的挑战。因此对测序平台的性能进行综合评估,能够有效规范临床上平台使用和其应用开发。中国食品药品检定研究院研制测序仪性能评价用脱氧核糖核酸国家参考品,包括4 种基因组 DNA 样本,分别是人基因组 DNA 样本、大肠杆菌基因组 DNA 样本、高 GC 含量细菌基因组 DNA 样本、人乳头瘤病毒(human papillomavirus,HPV)11 型基因组DNA 样本,用于一代、二代测序仪的测序准确率、重复性等性能评价。
1 材料与方法
1.1 材料与仪器
正常男性外周血白细胞构建永生化细胞系样本、感受态大肠杆菌(ATCC 8739)、高 GC 含量的欧陆森氏菌(Olsenella)、HPV11重组质粒均由中国食品药品检定研究院(简称中检院)提供。
QIAsymphony DNA Midi Kit 购自美国 Qiagen公司;Axygen®AxyPrep Easy-96 Plasmid Kit 购自美国Corning 公司;MGI Easy 微生物 DNA 提取试剂盒(磁珠法)购自深圳华大智造科技股份有限公司;Qubit®dsDNA HS Assay Kit 和Qubit®ssDNA Assay Kit 均购自美国 Invitrogen 公司;KAPA Library Quantification Kits 购自美国 Roche 公司;文库构建试剂盒分别购自深圳华大智造科技股份有限公司、杭州贝瑞和康基因诊断技术有限公司、美国Illumina 公司和博奥生物集团有限公司;BGISEQ-500RS 高通量测序试剂套装均购自深圳华大智造科技股份有限公司;NextSeq CN500 测序仪反应通用试剂盒购自杭州贝瑞和康基因诊断技术有限公司;NovaSeq 6000 S4 Reagent Kit(300 cycles)高通量测序试剂购自美国Illumina 公司;测序反应通用试剂盒(半导体法)购自博奥生物集团有限公司。
Qubit®3.0 荧光定量仪和 Applied Biosystems StepOnePlus 均购自美国Thermo Fisher Scientific公司;超声波破碎仪购自比利时Diagenode 公司;Covaris E210 超声波破碎仪购自美国 Covaris 公司;BGISEQ-500 基因测序仪购自深圳华大智造科技股份有限公司;NextSeq CN500 基因测序仪购自杭州贝瑞和康基因诊断技术有限公司;NovaSeq 6000 基因测序仪购自美国Illumina 公司;BioelectronSeq 4000 基因测序仪购自博奥生物集团有限公司。
1.2 方法
1.2.1 制备 按照试剂盒说明书,采用 QIAsymphony DNA Midi Kit 提取正常男性外周血白细胞构建的永生化细胞系样本,获得人基因组 DNA;转染 HPV11 质粒的细菌培养达到 108的数量级,采用 Axygen®AxyPrep Easy-96 Plasmid Kit获得 HPV 病毒基因组 DNA;大肠杆菌细菌数培养达到 108的数量级,采用十六烷基三甲基溴化铵法获得大肠杆菌基因组 DNA;Olsenella 高 GC 菌株,经厌氧培养后,细菌数量级达到 108,采用 MGI Easy 微生物 DNA 提取试剂盒(磁珠法),获得高 GC 菌株基因组 DNA。用 Qubit®3.0 荧光定量仪通过 Qubit®dsDNA HS Assay Kit 进行 DNA 浓度测定,每个样本进行 5 次浓度检测取平均值。将人基因组 DNA、HPV11 病毒基因组 DNA、大肠杆菌基因组 DNA 和 Olsenella 高 GC 菌基因组 DNA 分别按照 20、5、50、50 ng/μl终浓度要求,进行样本稀释和分装,制备国家参考品。
1.2.2 测序 采用文库构建试剂盒对基因组样本进行文库制备。先使用酶切打断或者超声打断,将国家参考品 DNA 片段化处理。经过接头连接和文库扩增等步骤,获得待测序分析的文库。使用Qubit®3.0 荧光定量仪通过 Qubit®dsDNA HS Assay Kit 或者 Applied Biosystems StepOnePlus 通过 KAPA Library Quantification Kit测定各个文库的浓度,按照测定的浓度混合文库。采用上述的测序反应通用试剂盒并按照试剂盒说明书在不同基因测序仪进行上机测序,包括 BGISEQ-500、NextSeq CN500、NovaSeq 6000 和BioelectronSeq 4000 基因测序仪等。
1.2.3 人全基因组变异标准集和一致性序列构建 在参考序列为 hs37d5 的人基因组区域,使用不同的全外显子芯片选择 27.1 Mb 的外显子区域,包括安捷伦公司的 Agilent V6 捕获芯片、NimbleGen 公司的Nimblegen Human Exome v3 芯片,IDT 公司芯片以及 BGI 公司 V4 芯片。使用软件 SOAP nuke 对所有原始数据进行低质量过滤得到有效数据,使用软件 BWA 和 GATK Haplotype Caller 对各数据集分别进行变异检测,获得两类文件:文件 gVCF,用于生成候选变异区间;文件 VCF,用软件 GATK VQSR 处理初步过滤得到候选变异集。然后合并各平台的候选变异集,使用软件 vcf annotate 将各候选变异区间注释到合并的变异集上。对于每一个候选变异集,随机取 40000 个在候选变异区间内的变异位点作为训练集,使用 R 语言中“e1071”软件包的“one-classSVM”进行分类,得到各候选变异集的极端变异阈值,用于变异筛选。对于未通过筛选的变异,在其两侧加减 50 碱基对,生成低置信变异区间。最后合并候选变异区间,与低置信变异区间取补集,再与参考基因组非 N 区间取交集,建立高置信单核苷酸多态性和插入缺失(single nucleotide polymorphism/insertion-deletion,SNP/Indel)位点标准集。
1.2.4 一致性序列构建 大肠杆菌基因组数据来源于美国国家生物技术信息中心。Olsenella 高 GC 菌的原始数据使用 BGISEQ-500 测序后进行 de novo 组装获得。HPV11 基因组数据使用 Sanger测序进行拼接组装。使用软件 BWA + SamTools +GATK Haplotype Caller 对各数据集分别进行比对、排序和变异检测得到 gVCF 文件,将所有样本的 gVCF 合并;进行 vcf 文件过滤,最后产生一致性序列。
1.2.5 协作标定 分别使用 BGISEQ-500、NovaSeq 6000、NextSeq CN500 和 BioelectronSeq 4000 基因测序仪,对这 4 个基因组 DNA 进行协作标定。与人基因组样本的全基因组变异标准集进行比对,并与大肠杆菌、Olsenella 高 GC 菌和 HPV 11 基因组样本的一致性序列进行比对。
2 结果
2.1 变异标准集和一致性序列
将各个平台的人全基因组变异集参考集进行汇总,得到不同区域的人类全基因组变异标准集,包括全基因组 2.7 Gb 区域和 1 Gb 区域,外显子27 Mb 区域以及乳腺癌易感基因1_2(breast cancer susceptibility gene1_2,BRCA1_2)17 Kb 区域。每个区域的大小以及包含 SNP 和 Indel 的数量(表 1),并产生一致性序列(Human.consensus.fa,HighGC.consensus.fa,HPV11.consensus.fa,. consensus.fa)。
2.2 协作标定
采用 BGISEQ-500、NovaSeq 6000、NextSeq CN500 和 BioelectronSeq 4000 基因测序仪等不同测序平台对国家参考品进行检测。检测人基因组 DNA 参考品,对人全基因组(2.7 Gb 区域)变异标准集的比对结果表明,不同测序仪对SNP、Indel检测的准确率和灵敏度存在着差别,尤其是 BioelectronSeq 4000 测序仪对 Indel 检测的准确率和灵敏度仅为 33.57% 和47.85%,但是对 SNP 检测的准确率和灵敏度均超过 86%;而其他 3 个测序仪对 SNP、Indel 检测的准确率和灵敏度均超过 89%(表 2)。检测人基因组 DNA 参考品,将结果与指定的全基因组 1 Gb 参考序列、指定的全外显子27 Mb 区域和指定的 BRCA1_2 基因 17 Kb 区域序列进行比对;检测细菌和病毒 DNA参考品,将结果与对应的参考序列进行比对,4 个测序仪的测序一致序列准确率均不低于 99.0%(表 3)。

表1 人全基因组变异标准集
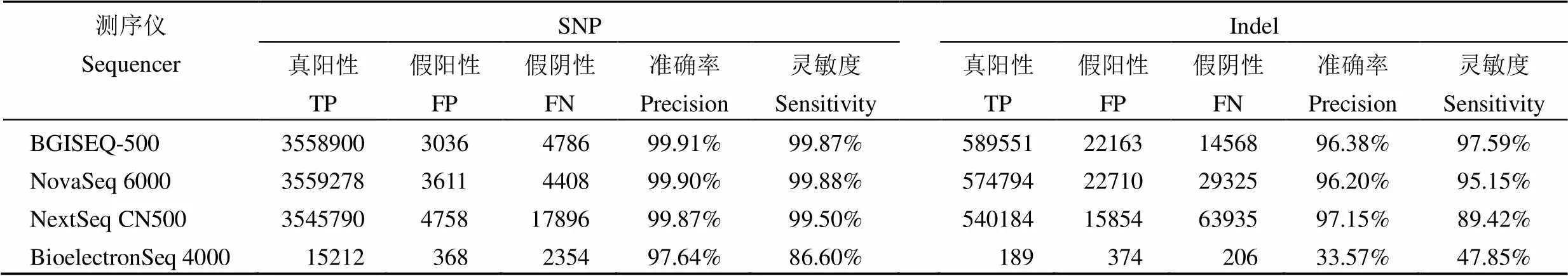
表2 人全基因组(2.7 Gb 区域)变异标准集的比对结果

表3 一致性序列比对结果
3 讨论
二代测序仪主要的质控参数包括比对到基因组上唯一位置的 base 比率(unique map rate)、重复的 reads 所占比例(duplication)、测序深度(average sequencing depth)和覆盖率(coverage)等。不同平台的测序仪有各自特异性的参数,对其性能评价应关注于测序通量、测序读长、碱基识别质量百分比、测序覆盖率、测序平均深度、测序准确率和重复性等指标。高通量测序仪的测序通量不同,临床的用途也有区别。体外诊断试剂标准物质对于保证检测的标准化具有重要的意义[11-12]。为了对高通量测序仪进行有效的性能评估,中检院建立测序仪性能评价用脱氧核糖核酸国家参考品。根据测序仪检测不同类型样本的临床用途,在设计国家参考品时,要求参考品的组成包括人基因组、细菌基因组以及病毒基因组样本,同时也增加干扰性的高 GC 细菌样本。
单核苷酸变异(SNV)和插入/缺失突变(Indel)是生物 DNA 常见的突变类型。小长度范围内的变异以及较长的缺失突变,目前都能够较好地检测出来。但对于大多数较长的插入突变和更复杂的结构性变异(structural variation,SV),由于二代测序的 reads 很短,难以定位在基因组上,所以难以检测出。在测序仪性能评价时,其测序准确率(包括对 SNP/Indel 和一致性序列)是非常重要的指标。在检测人基因组 DNA 国家参考品时,与 SNP、Indel 的参考数据集比较后,对测序仪的 SNP、Indel 检测的准确率和灵敏度有相应的要求。从国家参考品协作标定数据可以看出,对 SNP、Indel 检测的准确率和灵敏度在不同测序平台上有不同的表现,反映了不同平台性能上的差异,这个差异主要由平台本身的技术原理和技术性能决定[13]。因此对测序平台的 SNP、Indel 检测的准确率和灵敏度应有要求,但并未进行统一要求,只是要求制造商应给出各自平台的具体要求。这种评价方式和国际上评价测序仪的方式是一致的。在检测人基因组 DNA 国家参考品,结果与指定的全基因组 1 Gb 参考序列、指定的全外显子27 Mb 区域和指定的 BRCA1_2 基因 17 Kb 区域序列进行比对;在检测细菌和病毒 DNA 国家参考品,结果与对应参考序列进行比对,要求测序仪的测序一致序列准确率应均不低于 99.0%。结果表明各个测序仪的测序一致序列准确率均≥ 99.0%,能够满足国家参考品的要求。建立的国家参考品可以用于二代测序仪的测序准确率等性能评价,它采用实物检测和与参考序列比对的两种方式进行评价,具有重要的应用价值。
[1] Lalonde E, Wertheim G, Li MM. Clinical impact of genomic information in pediatric leukemia. Front Pediatr, 2017, 5:263.
[2] Gutowska-Ding MW, Deans ZC, Roos C, et al. One byte at a time: evidencing the quality of clinical service next-generation sequencing for germline and somatic variants. Eur J Hum Genet, 2020, 28(2):202-212.
[3] Gwinn M, MacCannell D, Armstrong GL. Next-generation sequencing of infectious pathogens. JAMA, 2019, 321(9):893-894.
[4] Pereira R, Oliveira J, Sousa M. Bioinformatics and computational tools for next-generation sequencing analysis in clinical genetics.
J Clin Med, 2020, 9(1):132.
[5] Rothberg JM, Hinz W, Rearick TM, et al. An integrated semiconductor device enabling non-optical genome sequencing. Nature, 2011, 475(7356):348-352.
[6] Patch AM, Nones K, Kazakoff SH, et al. Germline and somatic variant identification using BGISEQ-500 and HiSeq X Ten whole genome sequencing. PLoS One, 2018, 13(1):e0190264.
[7] Li H, Lei Y, Zhu H, et al. The application of NIPT using combinatorial probe-anchor synthesis to identify sex chromosomal aneuploidies (SCAs) in a cohort of 570 pregnancies. Mol Cytogenet, 2018, 11:59.
[8] Boland JF, Chung CC, Roberson D, et al. The new sequencer on the block: comparison of life technology's proton sequencer to an Illumina HiSeq for whole-exome sequencing. Hum Genet, 2013, 132(10):1153- 1163.
[9] Laehnemann D, Borkhardt A, McHardy AC. Denoising DNA deep sequencing data-high-throughput sequencing errors and their correction. Brief Bioinform, 2016, 17(1):154-179.
[10] Gupta N, Verma VK. Next-generation sequencing and its application: empowering in public health beyond reality. Microbial Technol Welfare Soc, 2019, 17:313-341.
[11] Fu Y, Gao XN, Huang J. Quality evaluation of in vitro diagnostic reagents and traceability of reference materials. J Mol Diagn Ther, 2017, 9(2):73-77, 87. (in Chinese)
付岳, 高旭年, 黄杰. 体外诊断试剂质量评价与标准物质溯源. 分子诊断与治疗杂志, 2017, 9(2):73-77, 87.
[12] Yang Z, Huang J, Yu T, et al. Status and countermeasures of national standard substances for in vitro diagnostic reagent in China. Chin
J Biol, 2015, 28(7):765-771. (in Chinese)
杨振, 黄杰, 于婷, 等. 我国体外诊断试剂国家标准物质现状及对策分析. 中国生物制品学杂志, 2015, 28(7):765-771.
[13] Huang J, Liang X, Xuan Y, et al. A reference human genome dataset of the BGISEQ-500 sequencer. Gigascience, 2017, 6(5):1-9.
Development of the DNA reference sequence control materials for sequencing performance evaluation
QU Shou-fang, HUANG Chuan-feng, SUN Nan, YU Ting, HUANG Jie
Author Affiliation: Division of Diagnostic for Non-infectious Disease (QU Shou-fang, SUN Nan, YU Ting, HUANG Jie), Division of Physical and Chemical Testing (HUANG Chuan-feng), National Institutes for Food and Drug Control, Beijing 100050, China
To establish the DNA reference sequence control materials for sequencing performance evaluation.Immortalized human cell line was amplified and the 108bacterias were captured, including plasmid transfected by HPV11,and Olsenella high GC strain. Then genomic DNA was extracted, and the concentration was determined. Human genomic DNA, HPV genomic DNA,genomic DNA and high GC genomic DNA were packaged respectively according to the final concentration requirements of 20, 5, 50 and 50 ng/μl as national reference materials, followed by detection on different next-generation sequencing platforms, including BGISEQ-500, NextSeq CN500, NovaSeq 6000, BioelectronSeq 4000, and so on. The standard variation set of human genome and consensus sequence of national reference materials were constructed. Different sequencing platforms were used for collaborative calibration to evaluate the applicability.The standard variation set of human genome and consensus sequence was established. After calibration by different sequencing platforms, the results meet the requirements of the standard set of human genome variation and the consensus sequence.The national reference materials can be used for the performance evaluation of high-throughput gene sequencer, which provides the basis for product registration, verification and post-marketing supervision and management.
high-throughput gene sequencer; throughput of gene sequencing; standard set; consensus sequence; single nucleotide polymorphism; insertion-deletion
s: YU Ting, Email: yuting@nifdc.org.cn; HUANG Jie, Email: jhuang5522@126.com
10.3969/j.issn.1673-713X.2021.04.002
国家重点研发计划(2017YFC0906500)
于婷,Email:yuting@nifdc.org.cn;黄杰,Email:jhuang5522@ 126.com
2021-01-20
*同为第一作者

