基于空洞卷积U-Net模型的胰腺自动分割方法研究
秦楠楠,薛旭东,石 军,郝晓宇,吴爱林,闫 冰,朱雅迪,吴爱东,
随着计算机图形处理和医疗装备技术的发展,计算机辅助诊断(computer aided diagnosis,CAD)已成为现实,对身体各器官精准分割算法成为当前研究的热点之一。目前对于肾脏、肺和心脏等的器官分割研究已经取得较好的结果,但对胰腺等器官的分割精度并不理想。因解剖位置个体差异性大,易发生偏移,灰度与周围组织相近等特点,使得胰腺的精准分割仍是具有挑战性的任务。早期对胰腺的分割主要有边缘分割算法和基于概率模型的分割算法等,该类方法对较为复杂的问题会出现学习能力不足的情况,而深度卷积神经网络在处理复杂问题、提取抽象和高维度特征方面有着巨大的优势,对胰腺的分割精确度要优于传统的分割方法,Yao et al在文献中运用空洞卷积网络模块对血肿精准分割,该模块可以聚合多尺度上下文信息,而不会丢失分辨率或分析重新缩放的图像。该研究对先期研究应用的标准2D U-Net模型进行改进得到一种多尺度空洞卷积U-Net模型,并利用实际病例对改进的模型进行训练、测试及比较分析。
1 材料与方法
1.1 病例资料
选取100例自2017年6月—2019年8月由安徽医科大学附属省立医院放疗科收治的宫颈癌和直肠癌患者的增强CT图像,病例图像均包含完整胰腺。患者定位均采取仰卧位行真空袋体位固定,飞利浦Brilliance大孔径CT定位扫描,扫描厚度为5 mm,图像重建分辨率为512×512,由主管医生在CT图像上完成100例患者胰腺的勾画并经上级医生审核确认。1.2 数据预处理
为尽可能的排除无关因素对模型训练的影响,需对图像裁剪像素值处理。首先通过阈值分割的方式提取患者身体外轮廓并计算轮廓的中心坐标,以坐标中心为原点,对图像进行裁剪,裁剪尺寸为256像素×256像素。其次对像素值处理通过比对胰腺区域像素值出现的频率,确定胰腺区域所对应像素值范围为950~1 180,并只保留此范围像素值。图像经预处理后对比度更好,且胰腺部分占图像的比例也有所增加。此外,由于胰腺是软组织,形状不规则且变化较大,运用随机弹性仿射变换来模仿胰腺在人体内的形变,实现模型对胰腺有效提取。经过仿射变换的图像前后对比情况见图1,经过预处理后的图像见图2。
图1 通过弹性仿射变换进行数据增广A:原始CT图像;B:经过仿射变换后的图像
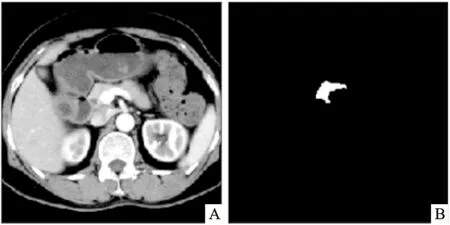
图2 经过预处理和裁剪后的图像A:CT图像;B:对应标签
1.3 模型构建及分割过程
1.3.1
模型构建 如图3所示,以Tensorflow为后端的Keras框架搭建模型。模型的主要模块是空洞卷积模块(dilated convolution module, DCM),它由两个四通道的卷积层组成。四通道的卷积对应3×3卷积核空洞率分别为1、2、4和6,用于提取不同尺寸的图像特征。图像输入首先经过四通道的卷积层卷积,在通道维度上进行特征融合,经过相同的卷积处理后经过BN层和池化层(最大池化)通道数增加一倍,图像尺寸减半。DCM和池化层共同组成编码模块,在U形底部是DCM模块。图像在底部经过上采样模块后进行1×1的卷积进行输出,与标准2D U-Net结构一致,在解码模块中,对应编码模块的图像与输出进行特征融合。在上采样过程中,路径收缩减半,图像尺寸增加一倍。解码模块由上采样、1×1卷积、特征拼接和DCM组成,最后采用Dropout用来防止过拟合。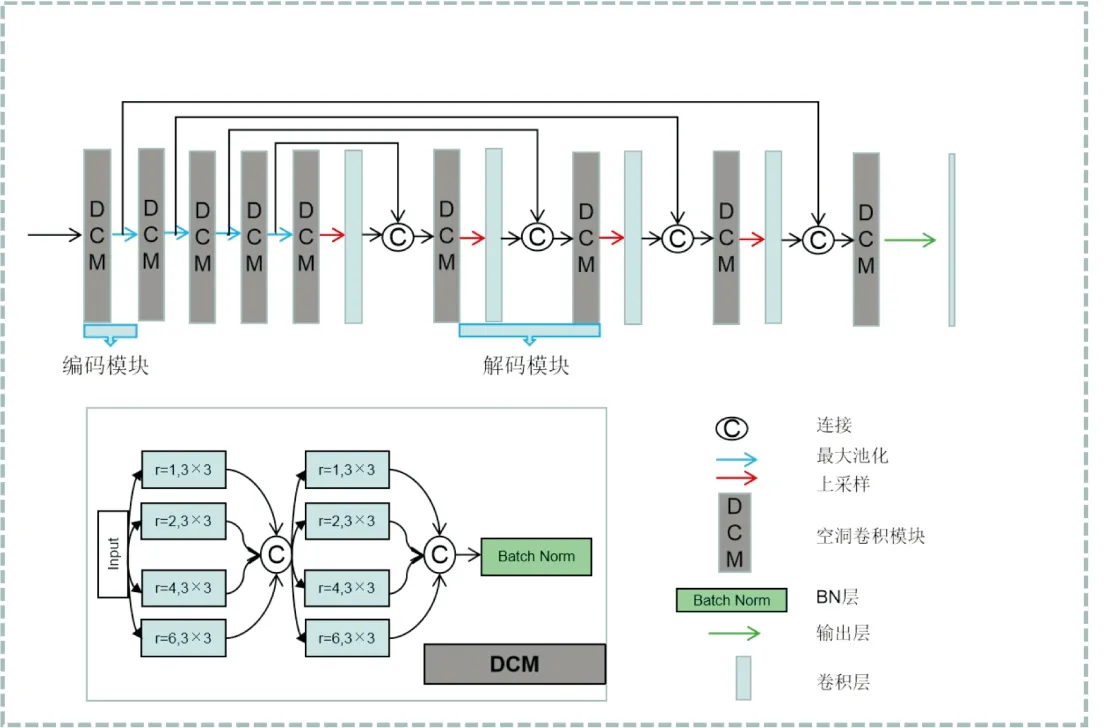
图3 基于标准U-Net改进的模型主体结构
1.3.2
分割流程及数据后处理 图4所示为胰腺勾画的流程,图像经过预处理后,输入尺寸为256像素×256像素的图像随机打乱送入网络进行训练。新的图像输入到训练完成的模型中,输出勾画的轮廓,计算轮廓大小,清除较小的噪声点。
图4 胰腺自动勾画流程图
1.3.3
评估标准 模型分割性能通过4个指标进行量化:戴斯相似系数(dice similarity coefficient,DSC)、豪斯多夫距离(hausdorff distance, HD)、杰卡德相似系数(jaccard similarity coefficient, JSC)和平均表面距离(average surface distance, ASD)来评估模型对测试集分割的精准度。DSC计算预测和手动勾画轮廓的符合程度:

其中A为手动勾画的轮廓,B为自动勾画(预测)轮廓,两者的交集部分表示手动勾画与自动勾画轮廓的重合部分。DSC越大,表明两种勾画结果的重合度越高,DSC介于0~1。
HD(A,B)是单向距离h(B,A)和h(A,B)中较大的一个,它表征了两个点集之间的最大失配距离,具体如下:
HD(A,B)=max(h(A,B),h(B,A))
h(A,B)=max(min‖a-b‖) a∈A,b∈B
其中,h(A,B)表示A集合中每一个点到B集合最小距离,HD越小,表示A,B重合度越高,分割的效果越好。
JSC是类似于DSC统计量,表示交叉区域的重合程度,其计算公式为:
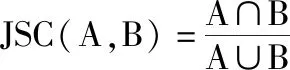
其中,0≤JSC(A,B)≤1,JSC值越大,勾画相似度越高。
ASD预测值和真实值的平均表面距离,ASD越大,表明预测值与真实值差别越大,勾画结果越差。
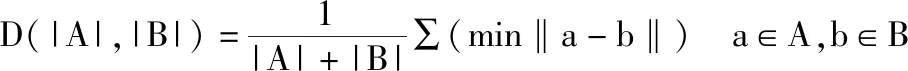
其中|A|和|B|分别表示A和B边界像素的集合, (min‖a-b‖)为A集合中每一个点到B集合各点的最小距离。最完美的分割是ASD的值为0。
1.4 统计学处理
使用基于python软件自编的程序分别对两个搭建的胰腺自动分割模型进行评估参数提取,并利用SPSS 22.2软件对结果进行统计分析和配对t
检验,P
<0.05为差异有统计学意义。2 结果
随机选择100例数据中的20例作为测试集,来评估所搭建的空洞卷积U-Net模型对胰腺自动分割的性能,其余80例数据并经过仿射变换处理的图像用作训练集,其中参数设置和相关配置:验证拆分(Validation)设置为0.2,训练轮次Epoch为200,单次放入样本量batch_size设置为8,优化器选择为Adam optimizer,学习率参数取0.000 1。训练模型使用的GPU型号为NVIDIA Tesla V100,训练时长为15 h。同时为评估改进的空洞卷积U-Net模型性能,本研究也利用标准2D U-Net模型对相同的病例数据对进行训练和测试,得到胰腺自动分割的数据,并对两模型结果进行比较分析。
两模型对胰腺自动分割性能的量化指标值及手动与自动分割分割情况见表1和图5。本研究搭建的空洞卷积U-Net模型的DSC均值达到0.87,JSC、HD和ASD均值也分别为0.78、9.85 mm和1.62 mm,且差异有统计学意义(P
<0.05)。其中标准U-Net模型HD指标明显偏高,与空洞卷积模型的HD值相差2.5倍之多,主要表现在当同一CT层面存在两个及以上连通区时两模型手动与自动分割结果差异大(见图6)。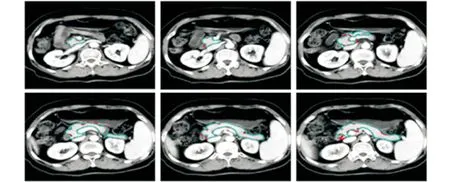
图5 胰腺手动与自动分割结果比较红线:手动勾画的轮廓;蓝线:自动分割的轮廓

图6 两模型在两个连通区手动和自动分割结果比较红线:手动勾画的轮廓;蓝线:自动分割的轮廓;上行:改进后的模型;下行:标准U-Net模型

表1 Dilated U-Net模型和标准U-Net模型分割性能对比
3 讨论
本研究基于标准2D U-Net改进的空洞卷积U-Net模型对胰腺组织进行分割,最终得到比较好结果。但由于数据集的差异,结果无法与其它相关研究进行横向比较,因此利用本课题组先前研究的标准U-Net模型,用相同的数据集进行训练和测试,并将二者的结果进行比较。在测试集中,改进的分割模型对胰腺自动分割DSC与HD分别达到0.87和9.85 mm,与标准U-Net比较,DSC提高了约4.5%,HD提高了2.5倍。
由于胰腺形状位置个体性差异大,胰腺背景的复杂性(其与周围器官灰度值相近,分割中容易错误识别),使得对胰腺分割难度大。对此情况,有研究对CT图像进行粗细分割:第一步先识别胰腺部分,并尽可能的裁剪出包含胰腺的图像尺寸,作为分割候选区,用来排除图像其他部分的干扰,尽可能提高胰腺在输入图像中所占的比例;第二步评估分割候选区的准确度,评估后裁剪出胰腺分布的图像作为模型输入,来提高分割准确率。Recall是用于评估分割候选区分割的准确度,即与原有手动分割的后选区的重合程度,确定是否包含完整胰腺。Roth et al使用整体空间聚集方法将胰腺分割性能提高到平均DSC为81.27%,Recall接近100%。 Fu et al使用分层组合深度学习架构获得胰腺分割的DSC为82.41%。Man et al在胰腺分割中使用深层卷积神经网络模型,DSC达到86.93%,Recall为86.91%。但这种方法在裁剪出候选区不完全包含胰腺时,将导致信息的丢失,使得分割结果出现差异。
本研究的空洞卷积模型使用分辨率为256×256的CT图像作为输入,以确保100%包括胰腺,具有不同感受野的四个通道用于多尺寸特征特的提取,这种结构补偿了由Maxpooling引起的信息丢失,并且有利于多联通区域特征识别。与标准U-Net的HD比较,空洞卷积U-Net模型的HD提高了2.5倍,这主要由于标准U-Net在两个相连区域的分割上表现不佳,不能很好的识别第二个连同区域,甚至会出现无法识别的情形,在空洞卷积模型中使用的大感受野有助于提取更远的连接区域形状特征。
综上所述,本研究利用改进的空洞卷积U-Net模型对胰腺进行分割,以四通道空洞卷积替代原有的卷积层,通过聚合多尺度特征信息完成分割。研究结果表明,改进后的分割模型评估指标均优于标准U-Net模型,可以更准确地完成胰腺分割。

