BRAF 基因突变对肝切除后的结直肠癌肝转移患者预后影响的meta分析
程 盛,赵 益,王永琛,黄 平
重庆医科大学附属第一医院肝胆外科,重庆400016
结直肠癌(colorectal cancer,CRC)是指发生在结肠和直肠的恶性肿瘤,又名大肠癌,是消化系统常见的恶性肿瘤。进入21世纪,CRC是世界范围内发病率排名第三的恶性肿瘤,也是导致癌症相关死亡的第四大主要原因[1]。CRC在全球有明显的地域分布差异,高发区为大洋洲(澳大利亚和新西兰),低发区为非洲、亚洲和南美洲。在我国表现为男性多见、发病年龄明显提前、直肠癌较结肠癌多见、经济发达地区发病率高、右半结肠癌比例呈上升趋势等[2]。在我国,男性CRC的发病率在全身恶性肿瘤中排第五位,女性排第四位;男性和女性CRC的死亡率均排在第五位[3]。
15%~25%的CRC 患者在确诊时已存在肝转移,而25%~50%的患者在3 年内发生肝转移[4]。目前,当技术上可行时,肝切除仍然是CRC 唯一的潜在治疗方法[5]。截至目前,由于新手术技术与更有效的治疗方法的结合,适合肝切除的患者数量急剧增加[6-7]。因此,肝切除患者5 年生存率从10%增加到50%[8]。在英国,尽管CRC 在手术、局部治疗(消融、栓塞和放射治疗)和系统治疗方面取得了进展,但处于美国癌症联合委员会(American Joint Committee on Cancer,AJCC)CRC TNM分期Ⅳ期的患者5年总生存率仍很低,仅为8%[9]。可见,晚期CRC 预后差,严重影响人类生命健康。评估结直肠癌肝转移(colorectal cancer liver metastasis,CRLM)患者预后的因素以往主要依据临床特征,但此方法不够敏感,无法准确评估CRLM患者的预后。在此背景下,生物标志物可以在筛选、诊断、治疗、预后、随访以及优化治疗方案的选择方面发挥关键作用。
在过去的数年,评估KRAS、NRAS和BRAF的基因状态对于转移性CRC的优化治疗越来越重要[10]。有10%的CRC 患者存在BRAFV600E 突变[11]。BRAFV600E 突变明确定义了预后不良、具有特定临床生物学特征、淋巴结及腹膜转移率高的患者亚群[12-15]。然而,V600E 以外的BRAF突变患者(594 或596 密码子)在分子特征和病理特征上是不同的,并没有表现出不良的临床结局[16]。近年来,有关BRAF突变对肝切除后的CRLM患者预后影响的报道很少,BRAF在CRLM 患者中突变率较低,因此不能得出明确的预后结论。因此,本研究对有BRAF突变的肝切除后CRLM 患者预后的文献进行meta 分析,以期为临床决策提供循证医学依据。
1 资料与方法
1.1 结局的定义
为了评估BRAF突变对肝切除后的CRLM 患者预后的影响,meta分析的主要终点为总生存期(overall survival,OS),次要终点为无病生存率(disease-free survival,DFS)。
1.2 检索策略
检索PubMed 上10 年间的临床研究,起止时间为2010年1月—2020年6月。仅检索用英语发表的文献,检索方式为"BRAF" and("colon" or "colorectal" or "rectal" or"rectum")and("metastasis" or "metastatic" or "metastases" or"mets"or"metastasectomy")and("hepatic"or"liver")。
1.3 文献的纳入及排除标准
纳入标准:①研究对象被确诊为CRLM。②CRLM患者行肝转移瘤全切除术。③有BRAF基因状态的遗传检测结果。④有根据BRAF基因状态进行分层分析的OS 和/或DFS 结果。⑤在多变量分析基础上,根据基因状态提供患者OS 和/或DFS 的危险比(hazard ratio,HR)。上述标准都必须符合。对于符合纳入标准的重复文献或基于相同患者队列的文献,纳入患者人数较多的一篇。
排除标准:①评论、书信、案例、综述、系统评价或meta 分析。②研究对象存在CRC 以外的原发性癌症。③研究对象初诊时已存在肝外转移(如CRC 肝、肺转移)。④研究只有单变量分析。⑤BRAF与其他基因或预后因素合并的分析。
1.4 文献筛选
2位研究者根据上述纳入及排除标准独立地进行文献筛选。若2 位研究者未能达成共识,则征询第3 人的意见,并经过讨论达成共识后做出最终决定。
1.5 数据提取
根据系统评价和meta 分析首选报告的条目(Preferred Reporting Items for Systematic Reviews and Meta-Analyses,PRISMA)进行数据提取。提取的数据内容包括第一作者、发表年份、研究数量、检测BRAF基因状态的组织来源、治疗方案、结局指标(OS和/或DFS)、根据BRAF突变状态的多变量分析的95%CI和HR值。整理结局指标数据时,将纳入患者根据BRAF基因类型分为2组:BRAF突变型以及BRAF野生型。
1.6 统计学分析
采用RevMan 5.4 进行统计学分析。二分类变量采用HR为效应值,区间估计使用95%CI。异质性检验使用Q检验和I²统计值检验法。当P<0.1、I²>50%时,显示纳入各研究间存在统计学异质性,选用随机效应模型估计合并效应值。当P≥0.1、I²≤50%时,显示纳入各研究间存在统计学同质性,选用固定效应模型估计合并效应值。使用漏斗图评估OS 和DFS 分析的发表偏倚。P<0.05 表示差异有统计学意义。
2 结果
2.1 文献检索
初步检索了159 篇文献。经阅读题目及摘要初筛后,剩余16 篇文献。精读全文后,根据纳入和排除标准,排除了9 篇文献[17-25]。再次2 人同时进行反复评估,最终有7 篇文献[26-32]符合纳入标准。具体筛选过程见图1。
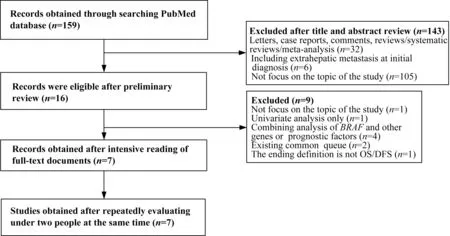
图1 文献筛选流程图Fig 1 Flow chart of literature inclusion
2.2 纳入研究的基本特征
共2 722 例患者被纳入meta 分析,其中BRAF突变占5.77%(157例)(表1)。纳入研究的患者均接受了化学治疗(化疗)、靶向治疗。多数研究化疗方案均含有奥沙利铂、氟尿嘧啶。1 项研究[31]中,肝切除后患者还接受了肝动脉灌注化疗。
2.3 纳入文献的质量评价
通过RevMan 5.4 分析系统中的偏倚风险评估工具进行文献评价。7 个评价条目包括随机序列产生、分配隐藏、研究者与被研究者盲法、结果评估盲法、不完全结局资料、选择性结局报告和其他偏倚。选项中绿色为低风险,黄色为风险不明确,红色为高风险,结果见图2。
2.4 统计结果分析
2.4.1BRAF突变与肝切除后CRLM 患者的OS 7 篇研究[26-32]报道了CRLM 患者行肝切除后,按BRAF基因状态进行分层分析的OS,总计2 722 例患者(表1),BRAF突变率为5.77%。异质性检验结果显示,I2=0、P=0.72,说明各研究间存在统计学同质性,使用固定效应模型。meta 分析结果(图3)显示,合并效应量HR=3.04,95%CI2.30~4.01,P=0.000,有统计学意义,说明有BRAF突变的CRLM 患者,行肝切除后OS 缩短。图4 报告了根据BRAF突变进行分层分析的OS的HR漏斗图,漏斗图基本对称,未发现明显的发表偏倚。
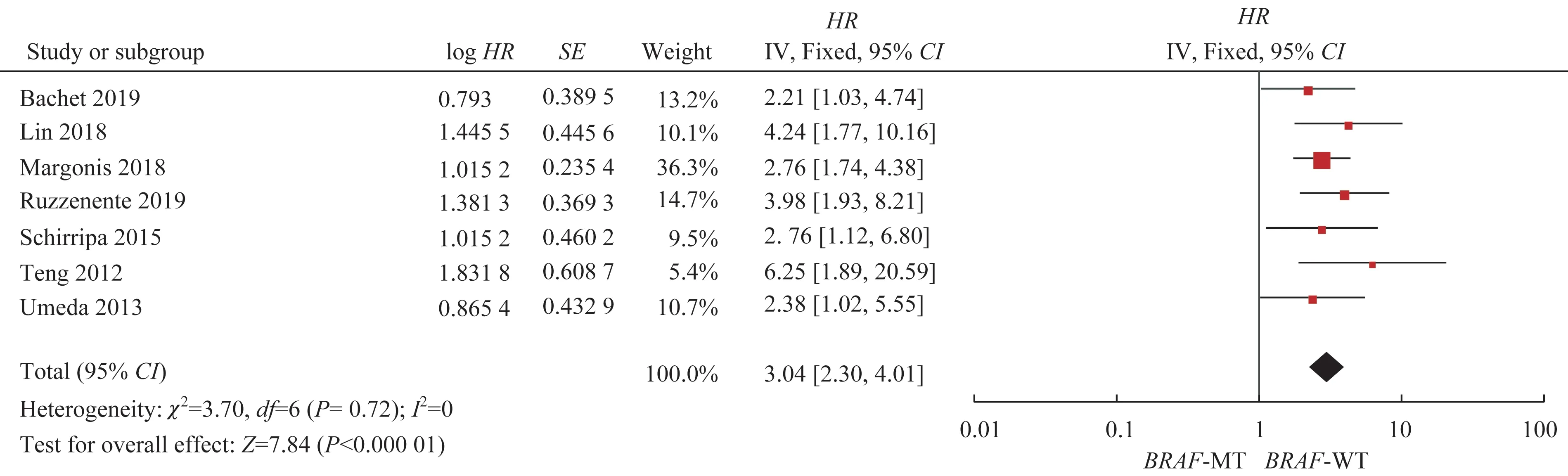
图3 BRAF突变与肝切除后CRLM患者OS的meta分析Fig 3 Meta-analysis of BRAF mutation and OS of CRLM patients after hepatectomy
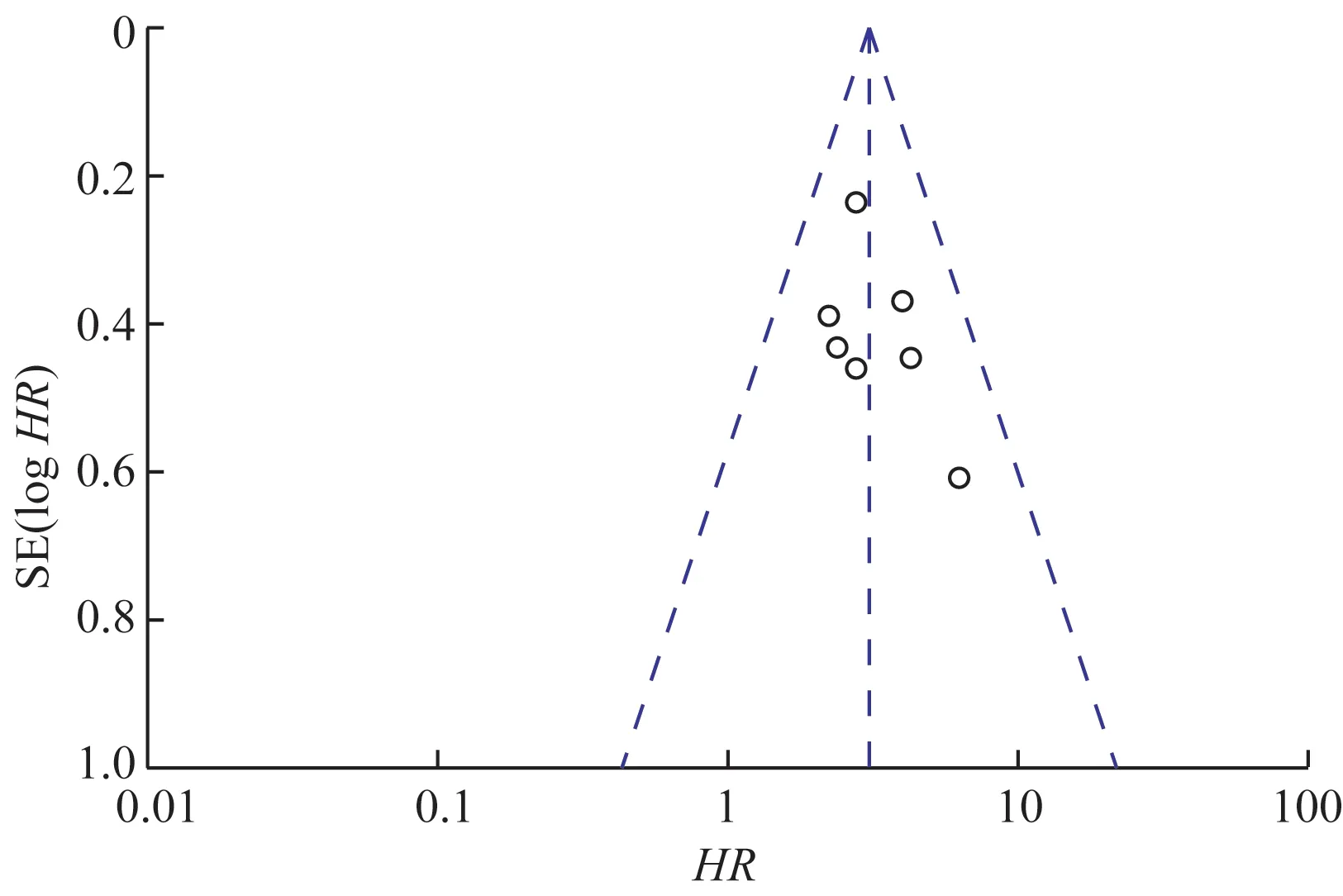
图4 BRAF突变的CRLM患者行肝切除后OS的HR漏斗图Fig 4 HR funnel plot of CRLM patients' OS with BRAF mutation after hepatectomy
2.4.2BRAF突变与肝切除后CRLM 患者的DFS 3 篇文献[29-31]报道了CRLM 患者行肝切除后,按BRAF基因状态进行分层分析的DFS,共1 237 例患者(表1),BRAF的突变率为9.62%。异质性检验结果显示,I2=77%、P=0.01,说明各研究间存在统计学异质性,使用随机效应模型。meta 分析结果(图5)显示,合并效应量HR=2.07,95%CI1.06~4.03,P=0.03,有统计学意义,说明有BRAF突变的CRLM 患者,行肝切除术后DFS 缩短。图6 报告了根据BRAF突变分层分析的DFS 的HR漏斗图,可见漏斗图不对称,不能确定是否存在发表偏倚,可能与纳入的研究较少有关。
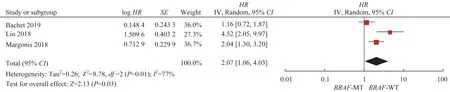
图5 BRAF突变与肝切除后CRLM患者DFS的meta分析Fig 5 Meta-analysis of BRAF mutation associated with DFS of CRLM patients after hepatectomy
图6 BRAF突变CRLM患者行肝切除后DFS的HR漏斗图Fig 6 HR funnel plot of CRLM patients' DFS with BRAF mutation after hepatectomy
3 讨论
本研究共纳入2 722 例患者,其中BRAF突变占5.77% (157 例)。据文献[33]报道,在转移性CRC 中BRAF突变的发生率低于10%,与本研究结果相仿。Bachet 等[31]研究中提供的26.5%的数据,并非BRAF突变率;因其采用了匹配病例对照研究的设计方法,26.5%是匹配后的BRAF突变患者占所纳入总患者的比例,作者提供的BRAF突变率为1%~9%。本研究结果发现,存在BRAF突变的CRLM 患者,肝切除后具有较差的OS,漏斗图基本对称,未发现明显的发表偏倚;存在BRAF突变的CRLM 患者,肝切除后的具有较差的DFS,但漏斗图出现明显的不对称,不能排除样本量越小的研究产生越大影响的可能性,即所涉及的研究样本量较小也可能导致漏斗图的不对称[34]。Bachet 等[31]研究结果表明,CRLM 患者是否存在BRAF突变,与肝切除后较差的DFS无关,原因可能是存在统计学异质性和偏倚。因此,关于BRAF突变对DFS 的影响,只有3 篇文献被纳入,可能存在潜在的发表偏倚,需更多研究来证实。
Tosi 等[35]研究的是KRAS突变对CRLM 患者肝切除后OS 和无复发生存率(relapse-free survival,RFS)的影响,其中纳入的3 篇文献涉及BRAF突变。本研究在其基础上增加4 篇文献[29-32],扩大了meta 分析的样本量,汇总后得出了更可靠的结论,并且还分析了BRAF突变对DFS 的影响。Passiglia 等[36]研究的仍是KRAS突变对CRLM 患者肝切除后OS 和RFS 的影响,纳入的BRAF突变对OS 影响的文献也只有3 篇,且未研究BRAF突变对RFS 或DFS 的影响。本分析中,没有纳入Karagkounis等[17]的研究,因为他们认为BRAF突变率低,对生存的影响不显著,并未给出BRAF突变对OS 或RFS 影响的结论,故我们认为与主题无关。此外,本研究主要分析BRAF突变对OS 的影响,与Passiglia 等[36]的研究相比,增加5 篇文献[27,29-32],并且分析了BRAF突变对DFS 的影响,得出更加全面的结论。
目前,肝切除是治疗CRLM 患者的主要手段,但肝切除手术程序复杂且费用昂贵,许多患者仍有可能复发,无法长期获益。因此,筛选适宜的CRLM 患者行肝切除手术有着重要的意义。以往的筛选标准主要根据临床特征,而不是肿瘤的生物标志物水平。术前的系统治疗现已作为多学科治疗方法的一部分,用于检测肿瘤的生物侵袭性和选择最适合手术的患者[37]。据报道[38-40],术前化疗的放射学反应和术后病理反应是CRLM 患者长期预后的重要预测因素。一些预后评分系统(包括临床表现、分期、肝转移程度等)已被开发出来,亦可用于支持治疗的决策[41-43]。然而,这些评分系统不够敏感,不能排除不必要手术的患者。此外,评估CRLM 是否可切除的标准存在争议,仍缺乏支持治疗决策的生物标志物。因此,本研究结果提示,BRAF突变可以与其他已知的临床特征结合,从而考虑患者是否有必要施行肝切除术。
本研究具有以下局限性:①纳入的研究在术前或术后的治疗方案中存在差异。大多数研究的化疗方案中含有奥沙利铂、氟尿嘧啶,也用到了靶向治疗,但其中4篇文献[27,29,31-32]未提及这些内容。对于手术后无法留下足够体积肝脏的患者,1项研究[31]采取了局部消融的方法。②检测BRAF基因状态的组织来源不同。多项研究检测的组织来源于肝转移瘤,部分研究[28-29,31-32]来源于原发灶。研究[44-45]表明,尽管不常见,但这2 个位点之间存在差异。为解决这一问题,有研究者[46]提出使用液体活检进行循环肿瘤DNA 检测,可能可以克服原发灶或肝转移瘤组织检测引起的空间异质性和肝活检固有的采样偏差,还有药物选择压力造成的时间异质性。③未将BRAF基因分为V600E 和非V600E 突变进行亚组分析。研究[16]表明,V600E 以外的BRAF突变(594 或596 密码子)并没有表现出不良的临床结局。本研究纳入的3 篇文献[27-28,30]中,仅检测了BRAF基因V600E 突变;1 篇文献[29]检测了V600E 和非V600E 突变,并进行了亚组分析;2 篇文献[26,31]检测了V600E 和非V600E 突变,但未进一步进行亚组分析,作者认为非V600E突变在BRAF基因中发生率较小,无法准确判断其对预后的影响;1篇文献[32]中未提及BRAF基因分型,也未给出具体的说明。我们认为,非V600E突变在BRAF基因中占比很小,可能对最终的分析结果影响不大。
综上,BRAF突变是肝切除后的CRLM 患者预后不良的生物标志物,与较差的OS 相关;而与较差的DFS 的关系,仍需更多研究来证实。为预测肝切除后CRLM 患者的预后,并制定个性化全身和局部治疗策略,可综合考虑BRAF基因以及其他临床病理因素。
参·考·文·献
[1] Bahrami A, Amerizadeh F, ShahidSales S, et al. Therapeutic potential of targeting wnt/β-catenin pathway in treatment of colorectal cancer: rational and progress[J]. J Cell Biochem,2017,118(8):1979-1983.
[2] Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin,2016,66(2):115-132.
[3] 王锡山. 中美结直肠癌流行病学特征及防诊治策略的对比分析[J]. 中华结直肠疾病电子杂志,2017,6(6):447-453.
[4] van Cutsem E, Cervantes A, Nordlinger B, et al. Metastatic colorectal cancer: ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up[J]. Ann Oncol,2014,25(Suppl 3):iii1-iii9.
[5] Bouviez N, Lakkis Z, Lubrano J, et al. Liver resection for colorectal metastases: results and prognostic factors with 10-year follow-up[J].Langenbeck's Arch Surg,2014,399(8):1031-1038.
[6] Primrose JN. Surgery for colorectal liver metastases[J]. Br J Cancer, 2010,102(9):1313-1318.
[7] Kopetz S, Chang GJ, Overman MJ, et al. Improved survival in metastatic colorectal cancer is associated with adoption of hepatic resection and improved chemotherapy[J]. J Clin Oncol,2009,27(22):3677-3683.
[8] Hayashi M, Inoue Y, Komeda K, et al. Clinicopathological analysis of recurrence patterns and prognostic factors for survival after hepatectomy for colorectal liver metastasis[J]. BMC Surg,2010,10:27.
[9] Kayhanian H,Goode E,Sclafani F,et al. Treatment and survival outcome ofBRAF-mutated metastatic colorectal cancer: a retrospective matched casecontrol study[J]. Clin Colorectal Cancer,2018,17(1):e69-e76.
[10] Schmoll HJ, van Cutsem E, Stein A, et al. ESMO Consensus Guidelines for management of patients with colon and rectal cancer. A personalized approach to clinical decision making[J]. Ann Oncol, 2012, 23(10): 2479-2516.
[11] Cancer Genome Atlas Network. Comprehensive molecular characterization of human colon and rectal cancer[J]. Nature,2012,487(7407):330-337.
[12] Richman SD, Seymour MT, Chambers P, et al.KRASandBRAFmutations in advanced colorectal cancer are associated with poor prognosis but do not preclude benefit from oxaliplatin or irinotecan: results from the MRC FOCUS trial[J]. J Clin Oncol,2009,27(35):5931-5937.
[13] Kadowaki S, Kakuta M, Takahashi S, et al. Prognostic value ofKRASandBRAFmutations in curatively resected colorectal cancer[J]. World J Gastroenterol,2015,21(4):1275-1283.
[14] Tran B, Kopetz S, Tie J, et al. Impact ofBRAFmutation and microsatellite instability on the pattern of metastatic spread and prognosis in metastatic colorectal cancer[J]. Cancer,2011,117(20):4623-4632.
[15] Yokota T,Ura T,Shibata N,et al.BRAFmutation is a powerful prognostic factor in advanced and recurrent colorectal cancer[J]. Br J Cancer, 2011, 104(5):856-862.
[16] Cremolini C, di Bartolomeo M,Amatu A, et al.BRAFcodons 594 and 596 mutations identify a new molecular subtype of metastatic colorectal cancer at favorable prognosis[J]. Ann Oncol,2015,26(10):2092-2097.
[17] Karagkounis G, Torbenson MS, Daniel HD, et al. Incidence and prognostic impact ofKRASandBRAFmutation in patients undergoing liver surgery for colorectal metastases[J]. Cancer,2013,119(23):4137-4144.
[18] Price TJ, Beeke C, Townsend AR, et al.BRAFmutation testing and metastatic colorectal cancer in the community setting: is there an urgent need for more education?[J]. Mol Diagn Ther,2016,20(1):75-82.
[19] Huang CJ,Teng HW, Chien CC, et al. Prognostic significance of C-reactive protein polymorphism andKRAS/BRAFin synchronous liver metastasis from colorectal cancer[J]. PLoS One,2014,8(6):e65117.
[20] Deshwar A, Margonis GA, Andreatos N, et al. DoubleKRASandBRAFmutations in surgically treated colorectal cancer liver metastases: an international, multi-institutional case series[J]. Anticancer Res, 2018, 38(5):2891-2895.
[21] Lang H, Baumgart J, Heinrich S, et al. Extended molecular profiling improves stratification and prediction of survival after resection of colorectal liver metastases[J]. Ann Surg,2019,270(5):799-805.
[22] Brunsell TH, Sveen A, Bjørnbeth BA, et al. High concordance and negative prognostic impact ofRAS/BRAF/PIK3CAmutations in multiple resected colorectal liver metastases[J]. Clin Colorectal Cancer,2020,19(1):e26-e47.
[23] Lin Q, Ye QH, Zhu DX, et al. Determinants of long-term outcome in patients undergoing simultaneous resection of synchronous colorectal liver metastases[J]. PLoS One,2014,9(8):e105747.
[24] Margonis GA,Buettner S,Andreatos N,et al. Prognostic factors change over time after hepatectomy for colorectal liver metastases: a multi-institutional,international analysis of 1 099 patients[J]. Ann Surg,2019,269(6):1129-1137.
[25] Løes IM,Immervoll H,Sorbye H,et al. Impact ofKRAS,BRAF,PIK3CA,TP53status and intraindividual mutation heterogeneity on outcome after liver resection for colorectal cancer metastases[J]. Int J Cancer,2016,139(3):647-656.
[26] Teng HW,Huang YC,Lin JK,et al.BRAFmutation is a prognostic biomarker for colorectal liver metastasectomy[J]. J Surg Oncol,2012,106(2):123-129.
[27] Umeda Y, Nagasaka T, Mori Y, et al. Poor prognosis ofKRASorBRAFmutant colorectal liver metastasis without microsatellite instability[J].J Hepatobiliary Pancreat Sci,2013,20(2):223-233.
[28] Schirripa M, Bergamo F, Cremolini C, et al.BRAFandRASmutations as prognostic factors in metastatic colorectal cancer patients undergoing liver resection[J]. Br J Cancer,2015,112(12):1921-1928.
[29] Margonis GA, Buettner S, Andreatos N, et al. Association ofBRAFmutations with survival and recurrence in surgically treated patients with metastatic colorectal liver cancer[J]. JAMA Surg,2018,153(7):e180996.
[30] Lin Q,Jian M,Niu ZC,et al. Prognostic impact ofKRASandBRAFmutations in patients who underwent simultaneous resection for initially resectable colorectal liver metastases[J]. Int J Clin Exp Pathol,2018,11(12):5981-5991.
[31] Bachet JB, Moreno-Lopez N, Vigano L, et al.BRAFmutation is not associated with an increased risk of recurrence in patients undergoing resection of colorectal liver metastases[J]. Br J Surg, 2019, 106(9): 1237-1247.
[32] Ruzzenente A, Bagante F, Ratti F, et al. Response to preoperative chemotherapy: impact of change in total burden score and mutational tumor status on prognosis of patients undergoing resection for colorectal liver metastases[J]. HPB(Oxford),2019,21(9):1230-1239.
[33] Garcia-Carbonero N, Martinez-Useros J, Li WY, et al.KRASandBRAFmutations as prognostic and predictive biomarkers for standard chemotherapy response in metastatic colorectal cancer: a single institutional study[J]. Cells,2020,9(1):219.
[34] Sterne JA, Sutton AJ, Ioannidis JP, et al. Recommendations for examining and interpreting funnel plot asymmetry in meta-analyses of randomised controlled trials[J]. BMJ,2011,343:d4002.
[35] Tosi F, Magni E, Amatu A, et al. Effect ofKRASandBRAFmutations on survival of metastatic colorectal cancer after liver resection: a systematic review and meta-analysis[J]. Clin Colorectal Cancer, 2017, 16(3): e153-e163.
[36] Passiglia F, Bronte G, Bazan V, et al. CanKRASandBRAFmutations limit the benefit of liver resection in metastatic colorectal cancer patients? A systematic review and meta-analysis[J]. Crit Rev Oncol Hematol, 2016, 99:150-157.
[37] Kopetz S, Vauthey JN. Perioperative chemotherapy for resectable hepatic metastases[J]. Lancet,2008,371(9617):963-965.
[38] Blazer DG, Kishi Y, Maru DM, et al. Pathologic response to preoperative chemotherapy: a new outcome end point after resection of hepatic colorectal metastases[J]. J Clin Oncol,2008,26(33):5344-5351.
[39] Chun YS, Vauthey JN, Boonsirikamchai P, et al. Association of computed tomography morphologic criteria with pathologic response and survival in patients treated with bevacizumab for colorectal liver metastases[J]. JAMA,2009,302(21):2338-2344.
[40] Shindoh J, Loyer EM, Kopetz S, et al. Optimal morphologic response to preoperative chemotherapy: an alternate outcome end point before resection of hepatic colorectal metastases[J]. J Clin Oncol,2012,30(36):4566-4572.
[41] Nordlinger B, Guiguet M,Vaillant JC, et al. Surgical resection of colorectal carcinoma metastases to the liver. A prognostic scoring system to improve case selection, based on 1 568 patients. Association Française de Chirurgie[J].Cancer,1996,77(7):1254-1262.
[42] Fong Y, Fortner J, Sun RL, et al. Clinical score for predicting recurrence after hepatic resection for metastatic colorectal cancer: analysis of 1 001 consecutive cases[J]. Ann Surg,1999,230(3):309-318.
[43] Rees M, Tekkis PP, Welsh FK, et al. Evaluation of long-term survival after hepatic resection for metastatic colorectal cancer: a multifactorial model of 929 patients[J]. Ann Surg,2008,247(1):125-135.
[44] Kopetz S, Overman MJ, Chen K, et al. Mutation and copy number discordance in primaryversusmetastatic colorectal cancer (mCRC) [J].J Clin Oncol,2014,32(15_suppl):3509.
[45] Artale S, Sartore-Bianchi A, Veronese SM, et al. Mutations ofKRASandBRAFin primary and matched metastatic sites of colorectal cancer[J]. J Clin Oncol,2008,26(25):4217-4219.
[46] Sartore-Bianchi A, Loupakis F,Argilés G, et al. Challenging chemoresistant metastatic colorectal cancer: therapeutic strategies from the clinic and from the laboratory[J]. Ann Oncol,2016,27(8):1456-1466.

