基于TCGA数据库分析胃癌可变剪接与肿瘤免疫的关系
顾琦晟,张米粒,曹 灿,李继坤
上海交通大学附属第一人民医院普外科,上海201620
胃癌是常见的恶性肿瘤之一。根据2018 年全球癌症统计数据,胃癌新发病例超过100 万例,估计死亡病例783 000 例;在所有癌症类型中,胃癌的发病率(5.7%)和病死率(8.2%)分别居第五位和第三位[1]。虽然胃癌的早期诊断和整体治疗水平不断提高,但是进展期胃癌的疗效仍不尽如人意。近年来,除了传统的手术治疗、放射治疗、化学治疗,肿瘤免疫治疗也展现出了无限潜力。以程序性死亡蛋白1(programmed death-1,PD-1)单抗为首的肿瘤免疫检查点抑制剂对黑色素瘤、血液系统肿瘤等非实体肿瘤显示出了极高的临床治疗活性[2-3];但是,对进展期胃癌的治疗效果较差,这与胃癌免疫逃避的潜在机制有着密切关系[4]。因此,研究胃癌的免疫逃避机制,找到增强胃癌免疫原性的方法,是现今进展期胃癌免疫治疗方面亟待解决的一个问题。
可变剪接又称为选择性剪接,是一项精密、复杂的转录后调控机制,是基因的mRNA 前体转录所产生的RNA 外显子以多种方式通过RNA 剪切进行重连的过程;从而,单个mRNA 可产生不同的蛋白质构体,是增加mRNA 复杂性和蛋白质多样性的原因之一[5]。研究[6-7]发现,在肿瘤的发生、发展过程中,伴随着基因的可变剪接异常产生,其中不乏许多已经证实的癌基因和抑癌基因的变化,并进一步推进肿瘤的发生、发展。
目前,有关胃癌可变剪接和肿瘤免疫浸润的关系尚待深入研究。本研究旨在利用生物信息学方法,探讨胃癌中的可变剪接情况,及其与胃癌表型、临床病理学因素以及肿瘤免疫浸润的关系,为胃癌的免疫治疗提供新的思路。
1 资料与方法
1.1 数据采集
在TCGA(The Cancer Genome Atlas)数据库中下载375 例胃癌患者及32 例配对癌旁正常组织的转录组、基因组数据和所有纳入研究的胃癌患者的临床信息。在SpliceSeq 数据库中下载与TCGA 的胃癌样本相对应的样本剪接百分比(percent-spliced in,PSI)矩阵,纳入具有PSI 值的样本占比<75%的剪接事件,共包含48 141 个剪接事件以及415 例胃癌组织和37 例配对癌旁正常组织样本, 以 备 后 续 分 析[8]。 在GEO (Gene Expression Omnibus)数据库中下载包含192 例胃癌患者转录组芯片数据的数据集GSE15459[9]。
1.2 数据分析及统计学处理
1.2.1 胃癌样本可变剪接数据的主成分分析 利用missMDA 包,基 于EM-PCA (Expectation Maximization Algorithm for Principal Component Analysis)算法对参数求极大似然估计(Maximum Likelihood Estimate,MLE),对缺失数据进行插补[10]。利用FactoMineR 包对数据进行PCA 降维分析,利用ggbiplot 和factoextra 包对PCA 数据进行可视化[11]。
1.2.2 利用一致性聚类方法对胃癌样本的可变剪接进行无监督聚类 利用ConsensusClusterPlus包的一致性聚类方法对PSI矩阵进行k-中心点聚类(k-medoid),聚类值k取2~10,根据聚类效果选择具有较高聚类稳定性的k值[12]。
1.2.3 基因集通路富集分析 使用limma 包的竞争性基因集测试算法(Competitive Gene Set Test Accounting for Inter-gene Correlation,CAMERA),以分子特征数据库(Molecular Signatures Database,MsigDB)的Hallmark 基因集为功能基因集,分析相关样本集与其他样本的差异,分析与该样本集密切相关的通路[13-14]。错误发现率(false discovery rate,FDR)<0.05 表示差异有统计学意义。利用GSVA 包的基因集变异分析(Gene Set Variation Analysis,GSVA)方法,将基因在不同样品间的表达水平矩阵转化为基因集在样品间的表达水平矩阵,从而分析该基因集表达水平在各个样本间的差异[15]。
1.2.4 可变剪接相关基因的筛选 对404 个经实验确认的剪接相关因子与胃癌可变剪接事件变化的关系进行研究[16]。根据基因的表达水平,以中位数为界,分为高表达组和低表达组,利用limma 包进行剪接事件的差异分析。以logFC>0.3 且BH 法修正后P 值<0.05 的差异可变剪接事件作为潜在受剪接因子表达水平影响的事件,筛选影响超过20%总剪接事件且相对mRNA 平均表达水平>4的基因,作为胃癌中潜在调控可变剪接的剪接因子。
1.2.5 与胃癌免疫微环境浸润的关系分析 利用CIBERSORT 方法和LM22 免疫细胞特征矩阵,对胃癌免疫细胞浸润丰度情况进行反卷积分析[17]。从美国国家肿瘤研究所基因数据库(National Cancer Institute Genomic Data Commons)获取TCGA 胃癌样本的肿瘤免疫相关特征数据[18]。
1.3 统计学分析
采用R 3.6.2 软件进行统计学分析。定性资料的比较采用Kruskal-Wallis H 检验;定量资料以±s 表示,采用ANOVA检验进行比较。P<0.05表示差异有统计学意义。
2 结果
2.1 胃癌可变剪接的整体情况
提取SpliceSeq 的初始矩阵,内含48 141 个剪接事件和452 例样本。制定严格的过滤标准,将有效值占比>90%且PSI 标准差>0.1 的事件纳入后续研究,同时排除有效值占比<80%的样本7 例(图1A、1B)。经过滤,获得8 649 个剪接事件和445 例样本,其中肿瘤样本409 例,癌旁正常组织样本36例。
通过观察4 051 个基因和相关8 649 个剪接事件的大体分布情况,发现平均每个mRNA 具有超过2 种不同形式的剪接变化,其中外显子跳跃(exon skipping,ES)、可变初始外显子(alternate promoter,AP)和可变终末外显子(alternate terminator,AT)是胃癌中最常见的剪接类型,5′端和3′端可变剪接、内含子保留事件相对较少,互斥外显子事件最少(图1C、1D)。
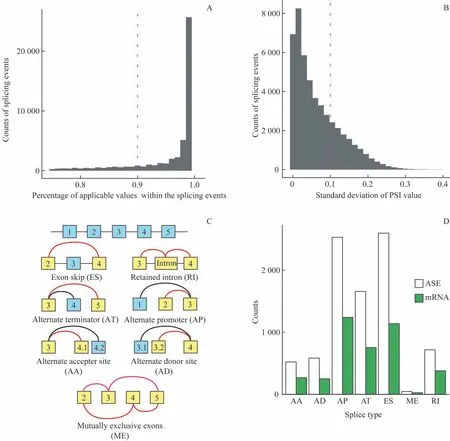
图1 TCGA中胃癌可变剪切概况Fig 1 Overview of alternative splicing of gastric cancer in TCGA
2.2 胃癌的可变剪接和主成分分析及聚类分析
使用EM-PCA 算法对剪接事件进行缺失值插补后,通过主成分分析降维。利用碎石图展示剪接事件的主成分对剪接全景的贡献度,其中PC1(主成分1)作为最重要的主成分,解释了剪接全景10.7%的方差值(图2A)。通过PCA 图发现PC1 可以明显区别肿瘤组织与癌旁正常组织(Wilcox test,P<0.01),提示肿瘤与癌旁正常组织中存在许多有显著差异的剪接事件(图2B、2C)。
利用ConsensusClusterPlus 包对剪接事件矩阵进行无监督聚类(图3A、3B)。基于一致性矩阵和Delta 区域结果,认为k=4时稳定性较佳,且能够减少过拟合效应。聚类簇C3、C1、C2+C4 在PC1 水平明显区分,而C2 和C4在PC2 水平区分(图3C)。C3 与癌旁正常组织的分布距离较大,提示该聚类簇包含的样本具有较为显著的肿瘤相关异常剪接发生。
如表1所示,通过比较4个亚组之间患者年龄、TMN分期、病理分期、组织学分期、Lauren 分型、性别、种族等临床特征,发现可变剪接的亚型与淋巴结转移分期、性别无关,而与其他临床特征有较大的关系。其中C3 型患者平均患病年龄偏大、远处转移可能更高、美国癌症联合委员会(American Joint Committee on Cancer,AJCC)分期相对较差;而C4 型患者患病年龄较小,肿瘤浸润相对更深,多表现为弥漫性胃癌,组织学多表现为G3低分化。
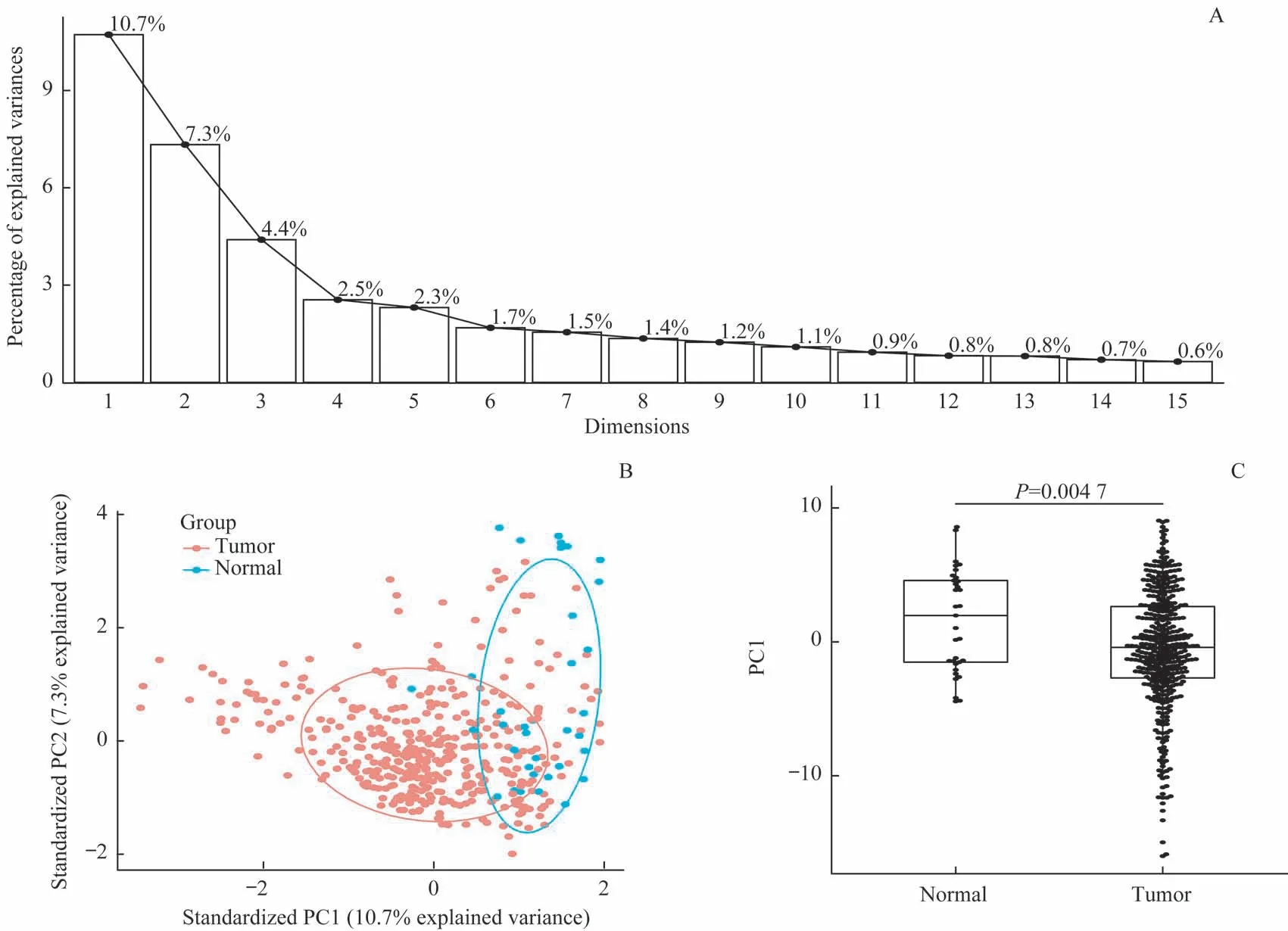
图2 通过PCA对可变剪接事件进行降维分析Fig 2 Dimensional reduction analysis of alternative splicing events by using PCA
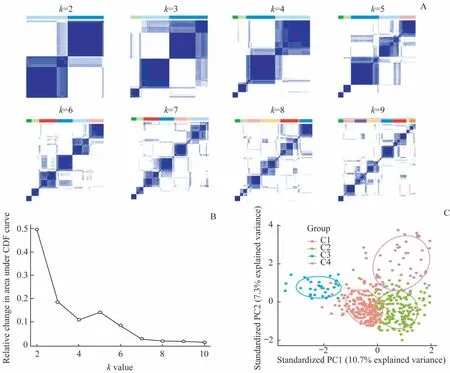
图3 根据可变剪接事件对胃癌样本进行一致性聚类分析Fig 3 Consensus clustering of gastric cancer samples based on alternative splicing events
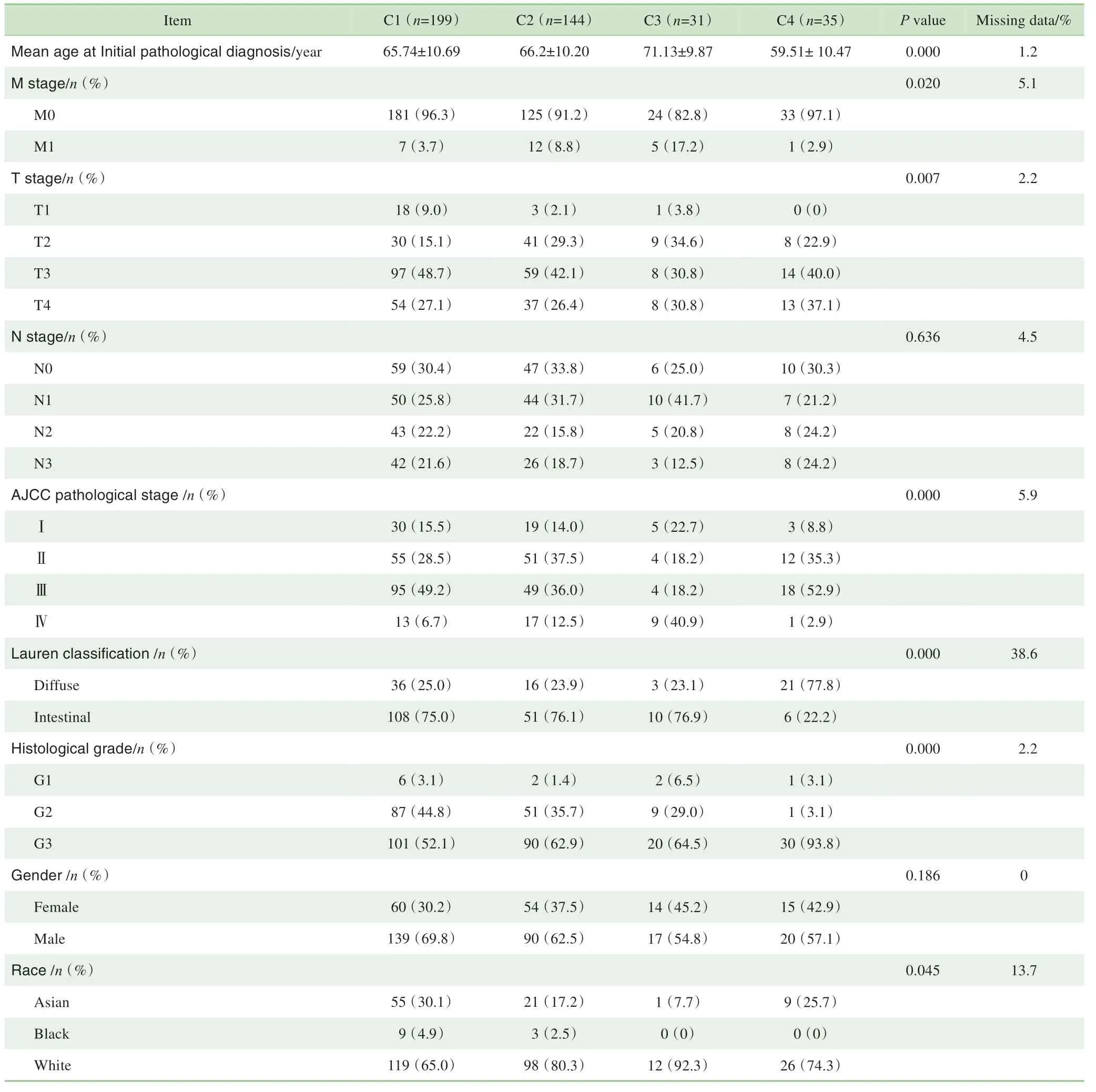
表1 基于可变剪接分类的4种亚型胃癌患者的临床资料比较Tab1 Comparison of clinical information of patients among the 4 subtypes of gastric cancer based on alternative splicing classification
2.3 胃癌的可变剪接亚型与肿瘤标志特征和免疫微环境的关系
对4种可变剪接亚型基于包含50个主要生物活动类型的MSigDB 的标志基因集(Hallmark Gene Set)分别进行CAMERA分析,结果显示C1型的上皮-间质转化(epithelialmesenchymal transition,EMT)通路显著下调;C2型的EMT通路相对上调;C3型蛋白分泌通路相对上调;C4型的细胞周期相关E2F目标基因显著下调。该结果提示4种亚型的肿瘤生物学特性各不相同,C1型肿瘤的侵袭转移能力相对较弱,而C4型肿瘤的增殖速度较其他类型更慢(图4)。
如图5所示,结合TCGA泛癌免疫全景研究结果,探索了可变剪接亚型和一些肿瘤特征的关系。结果显示,C2 型与C4 型的基质比率、淋巴细胞比率和转化生长因子-β(transforming growth factor-β,TGF-β)应答的特征较强;C1型的巨噬细胞调节水平较低;C3型的肿瘤浸润性淋巴细胞(tumor infiltrating leukocyte,TIL)区域比例较低;C4 型的增殖、创伤修复、突变水平较低,而淋巴细胞浸润等相对较高。
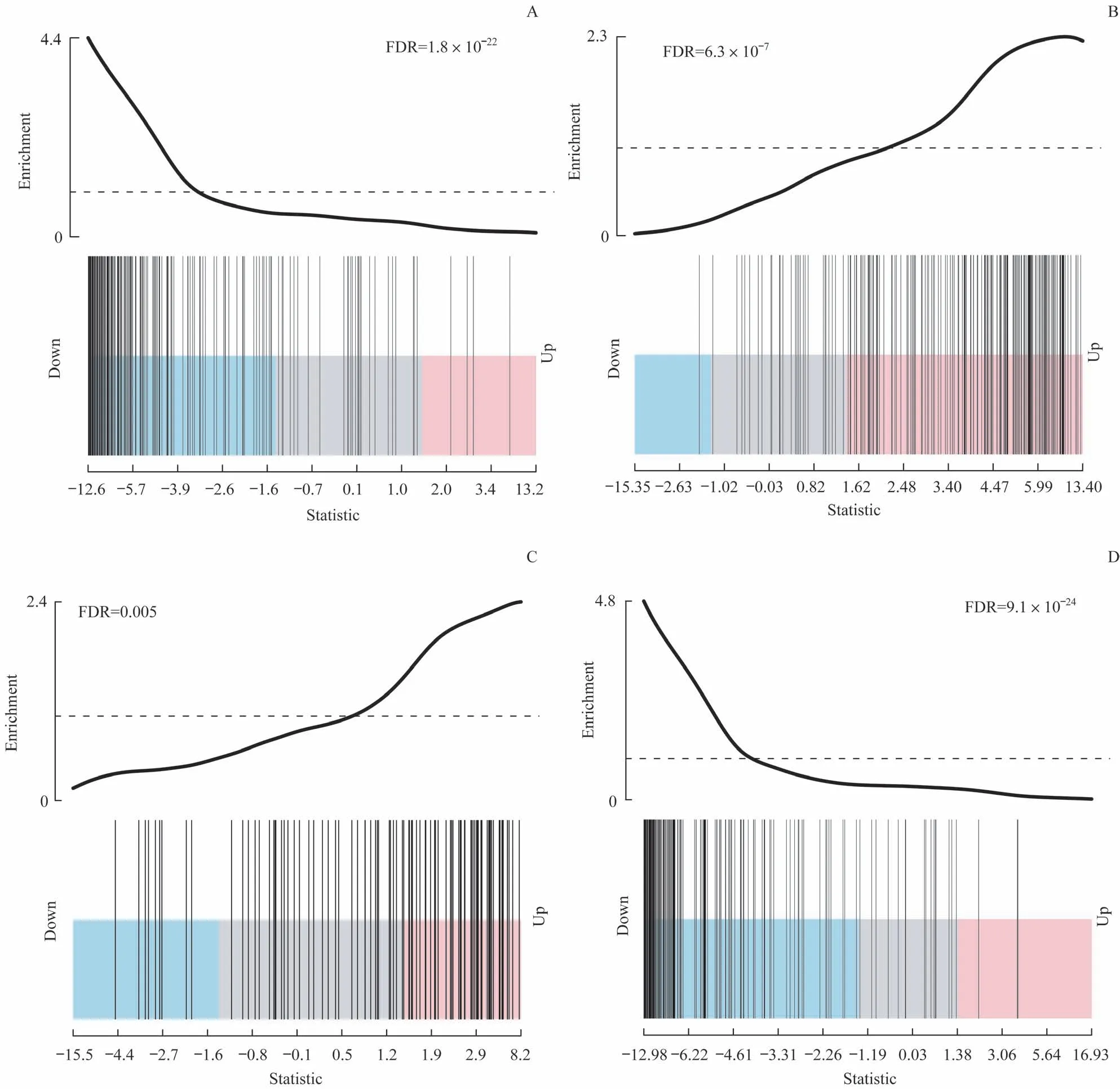
图4 标志基因集在4个可变剪接亚型中的最显著相关通路Fig.The most significantly related pathways of hallmark gene set in the 4 subtypes based on splicing events
基于22 种免疫细胞的签名矩阵,利用CIBERSORT对TCGA 的转录组数据进行反卷积,得到胃癌组织各免疫细胞组分的比例,并通过归一化去除肿瘤纯度带来的影响,比较各个亚型间免疫细胞组分的差异。如图6 所示,可变剪接亚型间的肿瘤微环境组分差异明显。C1 型的M2 型巨噬细胞丰度较低;C3 型的自然杀伤(natural killer,NK)细胞几乎完全处于静息状态,调节性T 细胞丰度极低,T 细胞受体(T cell receptor,TCR)香农指数较低,而有相对高水平的活化记忆性T 细胞和M2 型巨噬细胞;C4 型活化NK 细胞、肥大细胞丰度较高,TCR 香农指数相对最高。
2.4 胃癌的可变剪接情况与剪接因子表达水平的关联
如图7A 所示,对胃癌的剪接因子mRNA 表达数据进行了PCA 降维,发现胃癌的4 个亚型聚集效果良好,提示剪接因子的表达水平变化是影响可变剪接事件发生的主要因素。如图7B 所示,绝大多数剪接因子的高表达与PC1水平的降低相关,而CELF2、QKI、RBMS1等基因的表达模式和其他剪接因子相反,一定程度上起到拮抗异常可变剪接事件发生的作用。
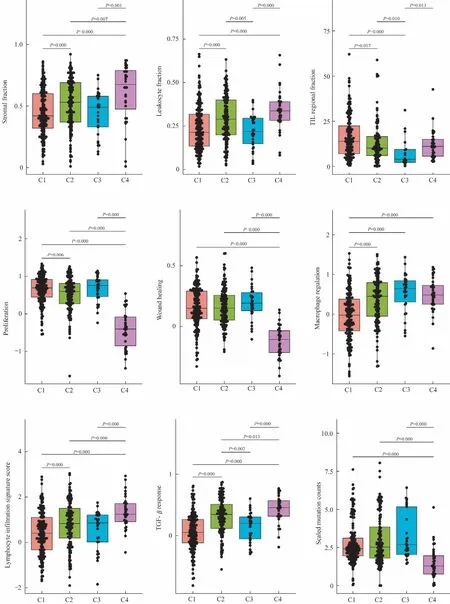
图5 可变剪接亚型与肿瘤特征的关系Fig.Association of subtypes based on splicing events and tumor characteristics
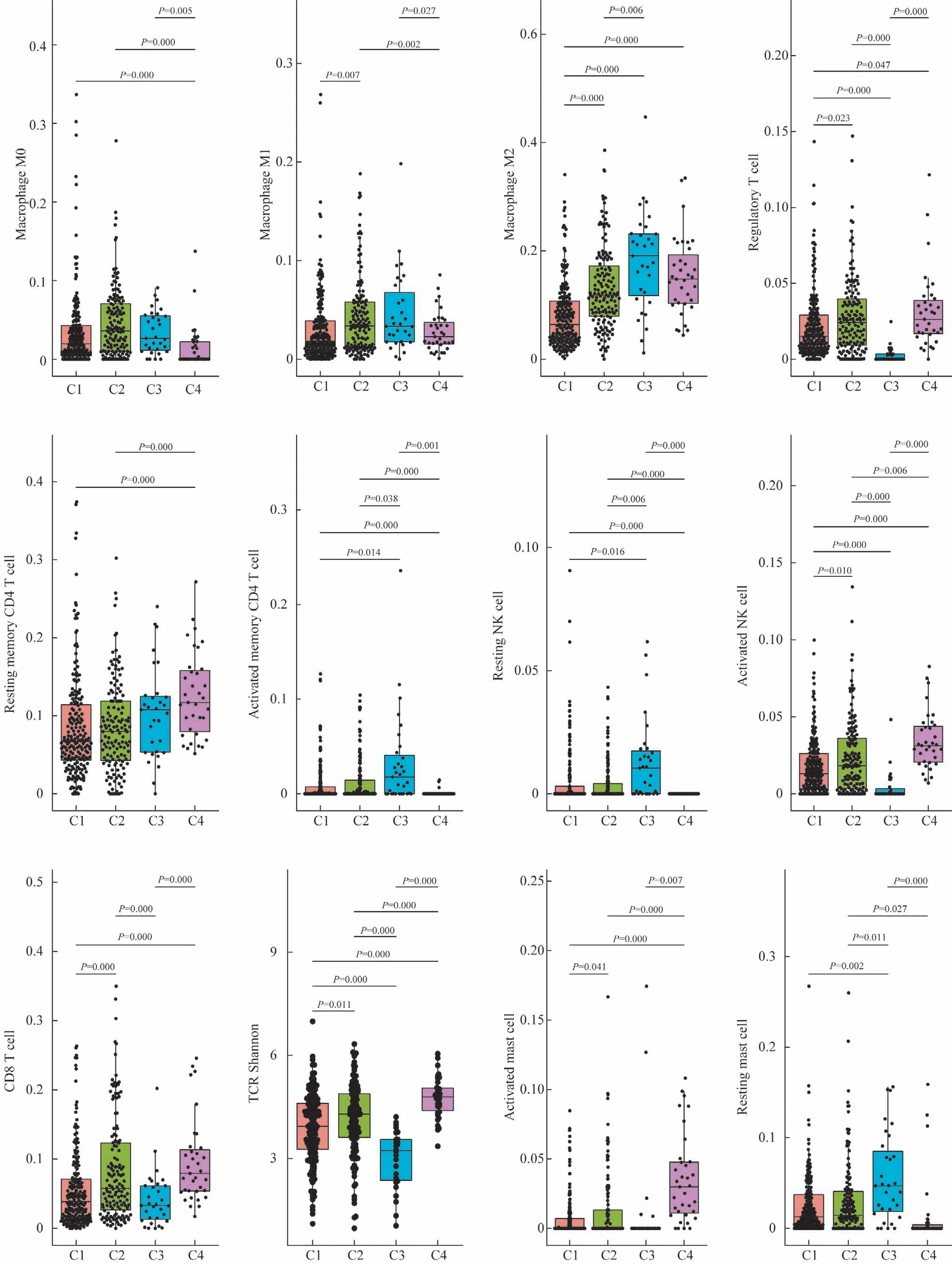
图6 可变剪接亚型与肿瘤微环境的关系Fig.Association of subtypes based on splicing events and tumor microenvironment
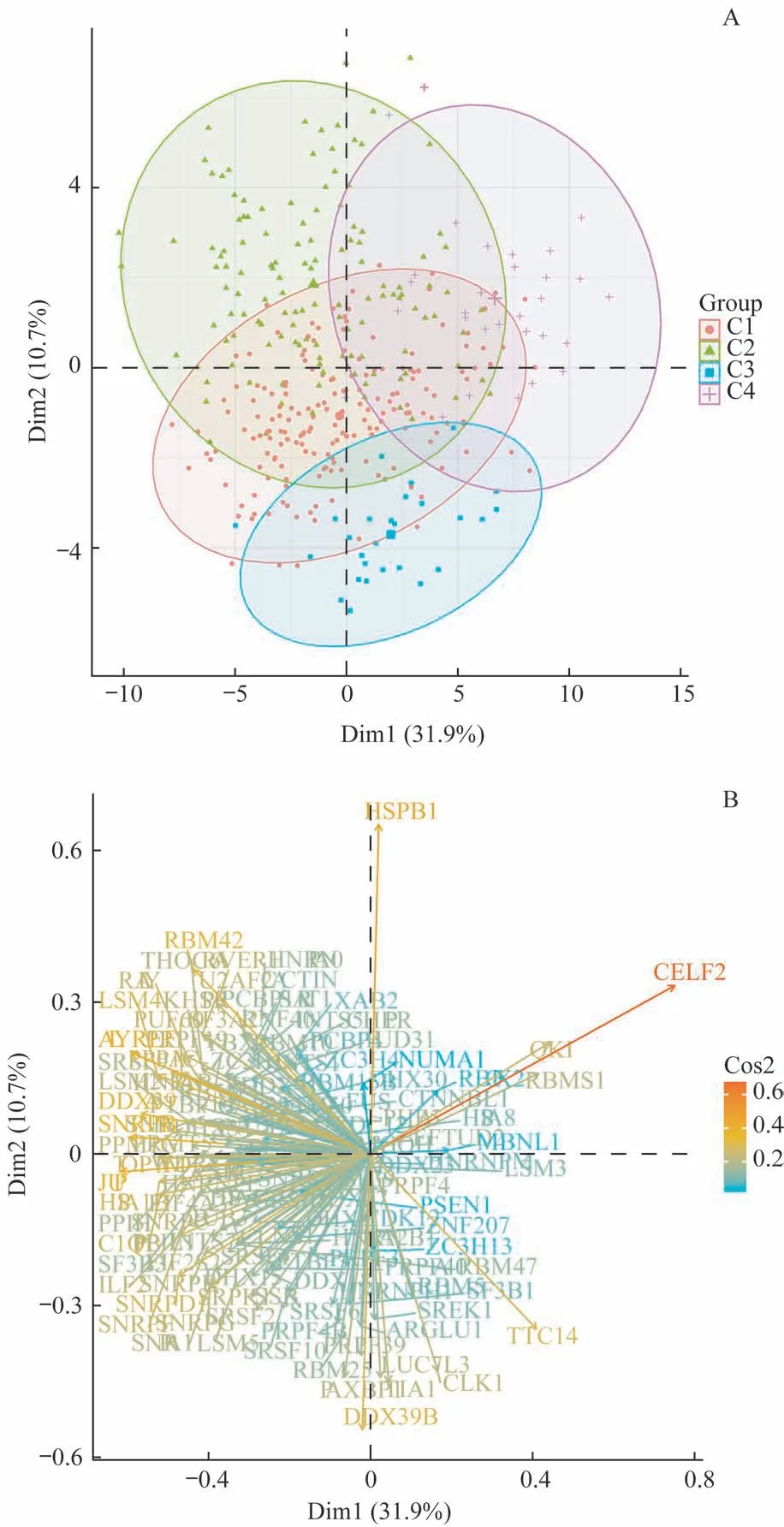
图7 通过PCA分析可变剪接亚型与剪接因子的关系Fig 7 Association of subtypes based on splicing events and splicing factors using PCA
通过差异基因分析对剪接因子进行研究,筛选log2FC>1 且FDR<0.05 的差异表达剪接因子。结果显示C1 型与RBM25、SREK1 的上调关系密切,且受到众多剪接因子的共同影响;C2 型与RAVER1、RBM42、ALYREF等基因上调相关;C3 型与TIA1、DDX39B、PAXBP1 等基因高表达相关;C4 型与QKI、CELF2、RBMS1 等基因的高表达相关(图8)。
2.5 胃癌的剪接因子表达水平与肿瘤抗原提呈水平的关系
选择图8中陈列的所有剪接因子基因作为样本的剪接因子签名。此外,PSMB5、PSMB6、PSMB7、PSMB8、PSMB9、 PSMB10、 TAP1、 TAP2、 ERAP1、 ERAP2、CANX、CALR、PDIA3、TAPBP、B2M、HLA-A、HLA-B、HLA-C 共18 个基因是明确的抗原提呈细胞相关基因,采用其作为基因集分析胃癌样本的抗原提呈水平[19]。
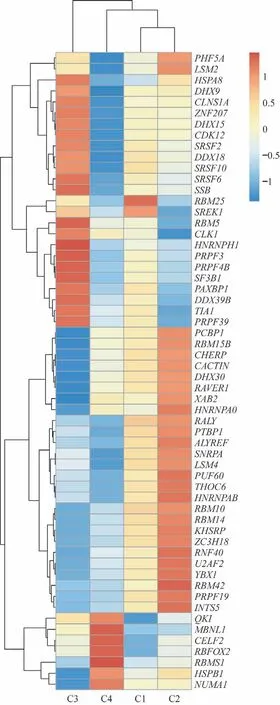
图8 可变剪接亚型与主要剪接因子中位值表达水平的归一化后热图Fig 8 Median normalized expression level of splicing factors in the 4 subtypes based on splicing events
利用上述剪接因子基因集和抗原提呈相关基因集,采用GSVA 分别对样本进行评分,随后进行相关性分析,以Pearson 相关系数表示其相关性。结果显示(图9):剪接因子基因集和抗原提呈相关基因具有明显的相关性(Pearson R=0.44,P = 0.000)。利用GEO 数据库中包含192 例胃癌转录组芯片数据的队列GSE15459 进行验证,结果与TCGA 胃癌数据一致,提示剪接因子家族的表达对肿瘤的抗原提呈过程有重要的潜在作用。
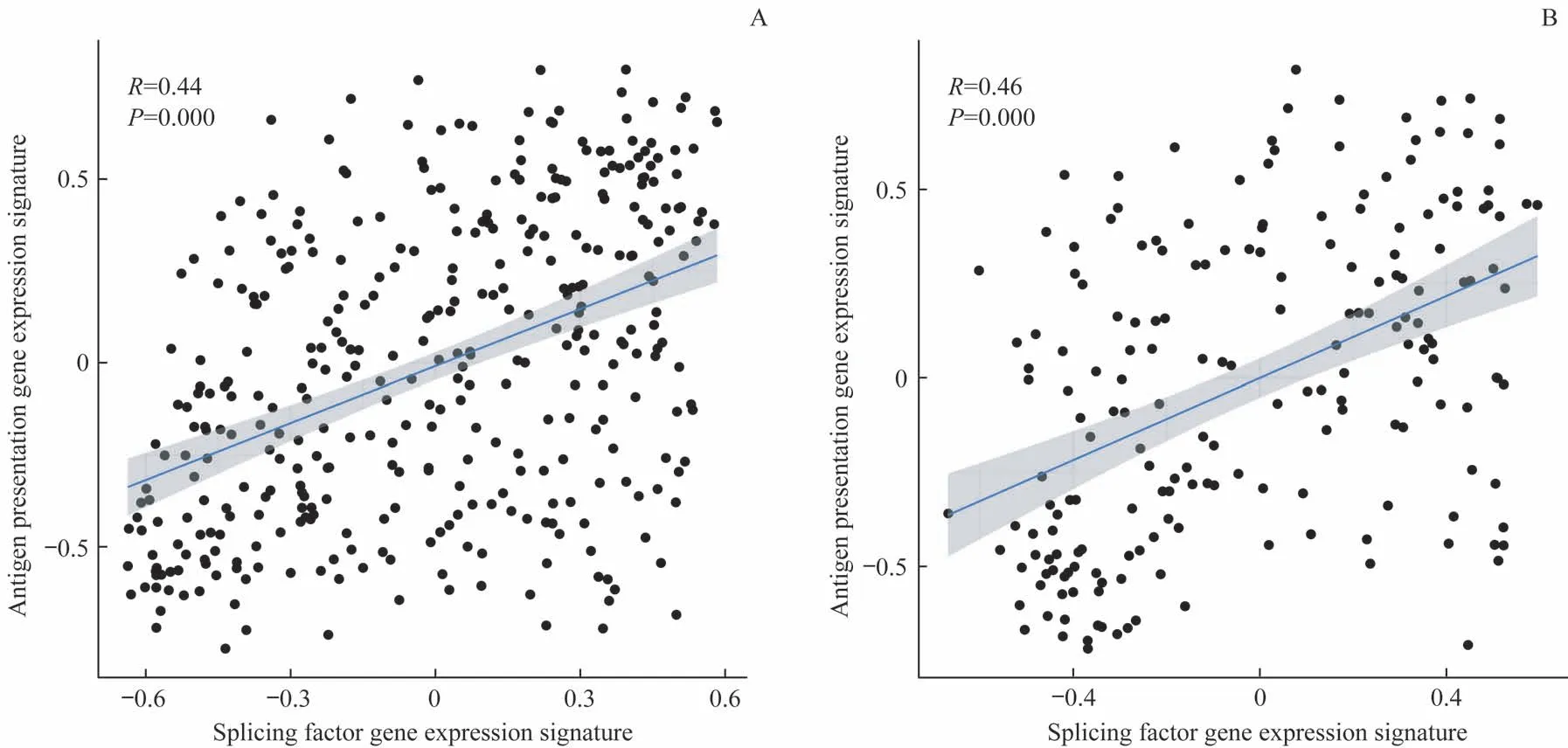
图9 胃癌剪接因子基因表达特征与抗原提呈基因表达特征的关系Fig 9 Scatter plots of gene expression signatures between antigen presentation and splicing factors
3 讨论
可变剪接过程在正常机体中受到严密的调控,剪接网络在机体中有可靠的鲁棒性。而在肿瘤发生、发展的过程中,随着肿瘤的基因组不稳定等内在环境压力和免疫微环境等改变带来的外在环境压力的产生,肿瘤细胞利用可变剪接这一重要的转录后调节水平变化调整自身的活性和功能,从而起到增强自身增殖信号、促进侵袭转移、逃避免疫监视等作用[20]。然而,目前有关可变剪接与胃癌肿瘤免疫相关的研究较为有限,值得进一步深入研究。
本研究利用剪接事件矩阵对TCGA 数据库的胃癌可变剪接事件全景进行了分析。结果显示,肿瘤与癌旁正常组织有显著的剪接差异。利用ConsensusClusterPlus 包对剪接事件矩阵进行无监督聚类,将所有样本分为4个亚型,可以认为C3 型在剪接事件水平上的异型性最强,C1型次之,C2型和C4型与癌旁正常组织最为相近。通过分析4 个亚型的临床特征,发现C3 型和C4 型的特征最明显:C3 型的病理分期较差,相对容易出现远处转移;C4型组织学表现为低分化,多为弥漫性胃癌。
基于Dunn 等的免疫编辑(immunoediting)学说,本研究中的肿瘤组织多处于免疫平衡(immune equilibrium)或免疫逃避(immune escape)阶段[21]。胃癌的发生、发展与慢性炎症密切相关,肿瘤多已能够免疫耐受。本研究发现4 个亚型的免疫逃避机制较为不同,C1 型与C2 型的抗原提呈水平较高,但C1 型的髓系细胞和淋巴细胞浸润程度都较低,而C2 型中M2 型巨噬细胞和调节性T 细胞等免疫抑制细胞的高丰度可能诱导了T 细胞无能,使得高丰度的CD8+T 细胞无法完成肿瘤杀伤的功能。C3 型和C4 型的抗原提呈水平较低,在C3 型中与较低的TCR多样性相关,而C4 型与基因组稳定、体细胞突变较少有关。总体上,4 个亚型潜在的免疫逃避机制分别为:C1型与低免疫浸润水平相关;C2型与高免疫抑制环境相关;C3 型表现为低抗原提呈水平;C4 型的肿瘤免疫原性较低。
此外,剪接因子对于可变剪接的分型起到了主导作用。C2 型和C3 型肿瘤的主要剪接因子簇的表达明显互斥。Mayeda等[22]证明了C3型剪接因子SRSF2和C2型剪接因子hnRNPA1 的拮抗关系,与本研究的结论相符。C1型的上述2 个剪接因子簇表达较为均衡,而C4 型主要以QKI、MBNL1、CELF2、RBFOX2 等 基 因 为 主。有 研究[23]发现MBNL1 和RBFOX2 的下调影响了体细胞重编程为多能干细胞过程中的剪接变化。另有研究[24]表明CELF2 通过在T 细胞激活过程中减少内含子保留事件,促进T 细胞分化和细胞因子产生。QKI 是重要的调控circRNA 生成的剪接因子[25]。有研究[26]显示,QKI因子的下调与胃癌的预后有显著关联。根据剪接因子表达和抗原提呈的关系,我们推测C2 主导型剪接因子簇更容易引起异常剪接体的产生,而C3和C4主导型剪接因子簇通过无义介导的mRNA 降解等方式起到抑制异常剪接体通过翻译发挥蛋白功能的作用。
综上所述,本研究发现可变剪接事件的全局变化与胃癌的肿瘤生物学特性密切相关,且与肿瘤微环境的变化有潜在关联。主要剪接因子的表达模式决定了可变剪接事件全局模式,且对于肿瘤的抗原提呈过程有着潜在的重要作用,对患者免疫治疗的适用性起到了提示作用。同时,剪接因子网络在胃癌中通过可变剪接调控适应和影响肿瘤免疫微环境的具体机制尚不明确,有待进一步研究。
参·考·文·献
[1] Siegel RL,Miller KD,Jemal A. Cancer statistics,2019[J]. CA Cancer J Clin,2019,69(1):7-34.
[2] Mahoney KM, Freeman GJ, McDermott DF. The next immune-checkpoint inhibitors: pd-1/PD-L1 blockade in melanoma[J]. Clin Ther, 2015, 37(4):764-782.
[3] Bryan LJ, Gordon LI. Blocking tumor escape in hematologic malignancies:the anti-PD-1 strategy[J]. Blood Rev,2015,29(1):25-32.
[4] Kang YK, Boku N, Satoh T, et al. Nivolumab in patients with advanced gastric or gastro-oesophageal junction cancer refractory to, or intolerant of,at least two previous chemotherapy regimens (ONO-4538-12,ATTRACTION-2): a randomised, double-blind, placebo-controlled, phase 3 trial[J]. Lancet,2017,390(10111):2461-2471.
[5] Stamm S, Ben-Ari S, Rafalska I, et al. Function of alternative splicing [J].Gene,2005,344:1-20.
[6] Biamonti G, Catillo M, Pignataro D, et al. The alternative splicing side of cancer[J]. Semin Cell Dev Biol,2014,32:30-36.
[7] Kozlovski I, Siegfried Z, Amar-Schwartz A, et al. The role of RNA alternative splicing in regulating cancer metabolism[J]. Hum Genet, 2017,136(9):1113-1127.
[8] Ryan M, Wong WC, Brown R, et al. TCGASpliceSeq a compendium of alternative mRNA splicing in cancer[J]. Nucleic Acids Res, 2016, 44(d1):D1018-D1022.
[9] Ooi CH, Ivanova T, Wu J, et al. Oncogenic pathway combinations predict clinical prognosis in gastric cancer[J]. PLoS Genet,2009,5(10):e1000676.
[10] Josse J, Husson F. missMDA: a package for handling missing values in multivariate data analysis[J]. J Stat Softw,2016,70(1):1-31.
[11] Lê S, Josse J, Husson F. FactoMineR: an R package for multivariate analysis[J]. J Stat Softw,2008,25(1):1-18.
[12] Wilkerson MD,Hayes DN. ConsensusClusterPlus:a class discovery tool with confidence assessments and item tracking[J]. Bioinformatics, 2010, 26(12):1572-1573.
[13] Wu D, Smyth GK. Camera: a competitive gene set test accounting for intergene correlation[J]. Nucleic Acids Res,2012,40(17):e133.
[14] Liberzon A, Birger C, Thorvaldsdóttir H, et al. The molecular signatures database hallmark gene set collection [J]. Cell systems, 2015, 1(6): 417-425.
[15] Hänzelmann S,Castelo R,Guinney J. GSVA:gene set variation analysis for microarray and RNA-seq data[J]. BMC Bioinformatics,2013,14(1):7.
[16] Kahles A, Lehmann KV, Toussaint NC, et al. Comprehensive analysis of alternative splicing across tumors from 8,705 patients [J]. Cancer Cell,2018,34(2):211-224. e216.
[17] Newman AM, Liu CL, Green MR, et al. Robust enumeration of cell subsets from tissue expression profiles[J]. NatMethods,2015,12(5):453-457.
[18] Thorsson V,Gibbs DL,Brown SD,et al. The immune landscape of cancer[J].Immunity,2018,48(4):812-830.e14.
[19] Leone P, Shin EC, Perosa F, et al. MHC class I antigen processing and presenting machinery: organization, function, and defects in tumor cells[J].J Natl Cancer Inst,2013,105(16):1172-1187.
[20] Oltean S, Bates DO. Hallmarks of alternative splicing in cancer[J].Oncogene,2014,33(46):5311-5318.
[21] Dunn GP, Bruce AT, Ikeda H, et al. Cancer immunoediting: from immunosurveillance to tumor escape[J]. Nat Immunol, 2002, 3(11): 991-998.
[22] Mayeda A, Krainer AR. Regulation of alternative pre-mRNA splicing by hnRNP A1 and splicing factor SF2[J]. Cell,1992,68(2):365-375.
[23] Venables JP,Lapasset L,Gadea G,et al. MBNL1 and RBFOX2 cooperate to establish a splicing programme involved in pluripotent stem cell differentiation[J]. Nat Commun,2013,4:2480.
[24] Mallory MJ,Allon SJ,Qiu J,et al. Induced transcription and stability of CELF2 mRNA drives widespread alternative splicing during T-cell signaling[J]. PNAS,2015,112(17):E2139-E2148.
[25] Conn SJ, Pillman KA, Toubia J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell,2015,160(6):1125-1134.
[26] Bian Y, Wang L, Lu H, et al. Downregulation of tumor suppressor QKI in gastric cancer and its implication in cancer prognosis [J]. Biochem Biophys Res Commun,2012,422(1):187-193.

