基于GEO数据库筛选并分析胃癌标志物及其作用
陆汉凌,任旻,唐玉宝,朱云祥
(扬州大学附属医院,江苏 扬州 225000)
0 引言
胃癌是全球常见恶性肿瘤之一,在中国,胃癌的发病率和死亡率分别在第三位和第二位[1]。现阶段的胃癌诊断方法中,主要依靠CT、胃镜检查及肿瘤标志物等[2-4]。但胃镜检查属于侵入性检查,费用相对较高,普通患者接受度不高,因此对于寻找有效胃癌基因标志物成为新的热门选择。本文从基因表达数据库中筛选胃癌相关基因芯片并分析其生物学功能及信号通路,同时筛选了胃癌相关核心差异表达基因,并探讨了胃癌相关核心差异表达基因与预后的关系。
1 材料与方法
1.1 GC相关数据的获取
登录GEO数据库(https://www.ncbi.nlm.nih.gov/geo),以“gastric cancer”为检索词,获取到与GC相关的两组数据集:GSE33651、GSE54129。GSE33651共有样本数52个,其中GC组织组40个,正常黏膜组12个。GSE54129共有样本数132个,其中GC组织组111个,正常黏膜组21个。
1.2 差异表达基因的筛选
利用在线平台GEO2R分别对两组芯片的胃癌组织组和正常黏膜组差异表达基因进行筛选,以LogFC>1或者LogFC<-1且校正后P值(adj.Pvalue)<0.01为筛选标准分别对两组数据集进行差异表达基因筛选。筛选得到的数据利用DrawVenn Diagram (http://bioinformatics.psb.ugent.be/webtools/Venn/),绘制出两组芯片差异表达基因的韦恩图,得到共有的差异表达基因。
1.3 GC相关差异基因信号通路及生物学功能分析
登录基因功能数据库DAVID(https://david.ncifcrf.gov/),将差异基因导入,对其进行基因本体(Gene Ontology,GO)分析,通过GO分析了解到差异基因的细胞组成(CC)、生物学过程(BP)及分子功能(MF)。接着进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路分析,得到相关作用的信号通路。
1.4 GC相关核心基因的获取
将上述筛选出的差异基因利用STRING数据库(https://string-db.org/)进行分析,得出蛋白互作网络(PPI),以R-软件对PPI进行核心基因筛选,以节点联系进行排序,筛选出12个核心基因(FN1、CXCL8、CD44、COL1A1、MMP2、COL1A2、ITGB2、PECAM1、THBS1、ACTB、CXCR4、ITGAM),对核心基因进一步分析其表达及生存预期。
2 结果
2.1 GC相关差异表达基因结果
两组GC相关芯片,以LogFC>1或者LogFC<-1且校正后P值(adj.Pvalue)<0.01为筛选标准。GSE33651共有样本数52个,其中GC组织组40个,正常黏膜组12个,共筛选出差异表达基因1821个。GSE54129共有样本数132个,其中GC组织组111个,正常黏膜组21个,共筛选出差异表达基因2092个。将两个芯片组基因用DrawVenn Diagram取交集得到韦恩图,得到差异共表达基因330个,其中下调基因有123个,上调基因207个。具体可见图1。
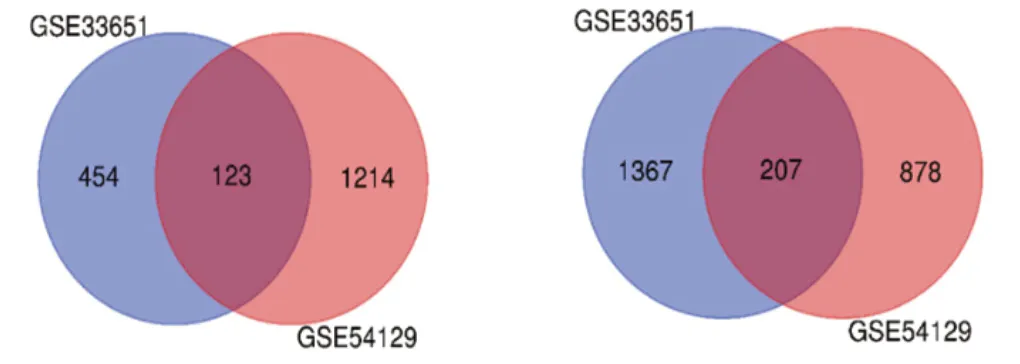
图1 两组芯片数据差异表达基因韦恩图
2.2 GC相关差异表达基因的生物学功能及信号通路
在GO分析中可以发现(表1),330个差异共表达基因的分子功能(MF)主要集中在胶原结合、蛋白质结合、整联蛋白结合、纤连蛋白结合等过程中。细胞组成(CC)主要参与在细胞外泌体、细胞外基质、胞外区域、细胞表面等。同时,在生物学过程(BP)中主要在胶原分解代谢过程、血管生成、细胞粘附、白细胞迁移等发挥着作用。KEGG分析中(表2),差异表达基因主要集中于百日咳、阿米巴病、白细胞跨内皮迁移及ECM-受体相互作用等通路。

表1 GC相关差异表达基因GO分析

表2 GC相关差异表达基因KEGG分析
2.3 蛋白互作网络构建(protein-protein interaction network,PPI)及核心基因获取
登录STRING数据库(https://string-db.org/),将差异表达基因导入数据库中,构建出PPI,得到下图2。使用STRING数据库对差异基因进行蛋白互作网络分析,以R-软件对PPI进行核心基因筛选,筛选后以节点联系排序,得到下图3。

图2 GC差异表达基因PPI图
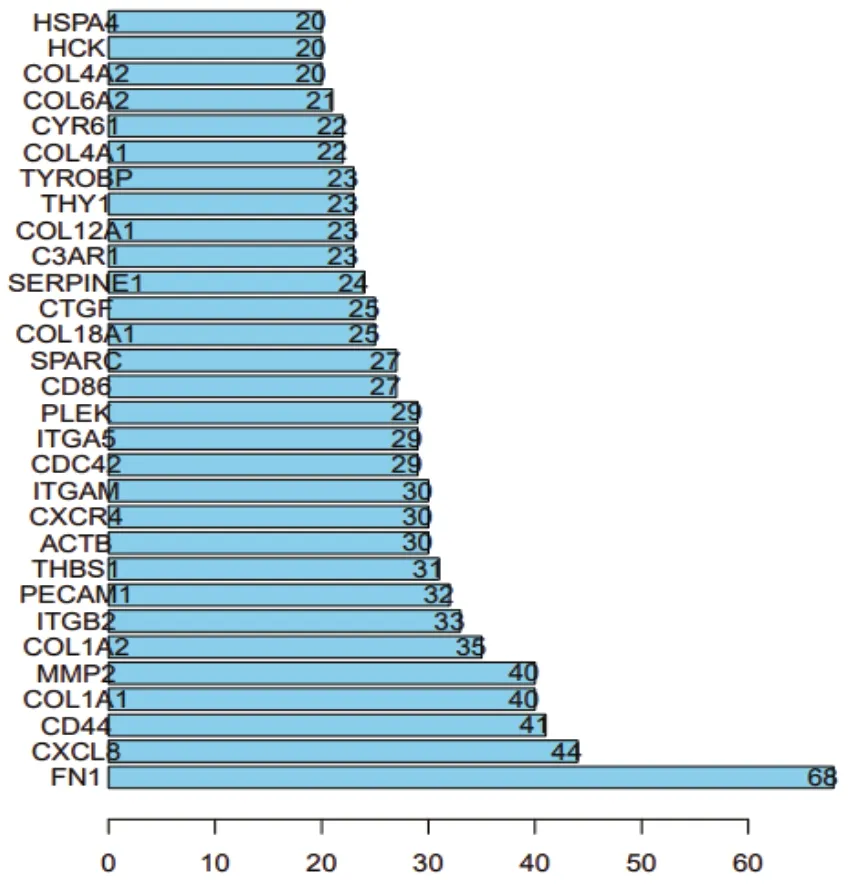
图3 GC相关核心基因
2.4 核心差异基因的表达水平分析及生存分析情况
登录在线分析网站GEPIA(http://gepia.cancer-pku.cn/),比较所获取的12个核心差异表达基因在胃腺癌(stomach adenocarcinoma,STAD)病人与正常人组织的表达水平情况(见图4)。我们发现,除了PECAM1外,其余基因在STAD病人中均表达上调。在生存分析图5中,可以找到基因FN1、THBS1、CXCR4、COL1A1是有益于胃癌患者的生存。
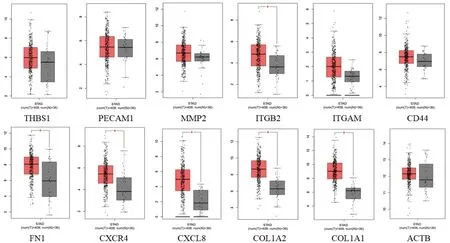
图4 GC核心差异表达基因的表达分析
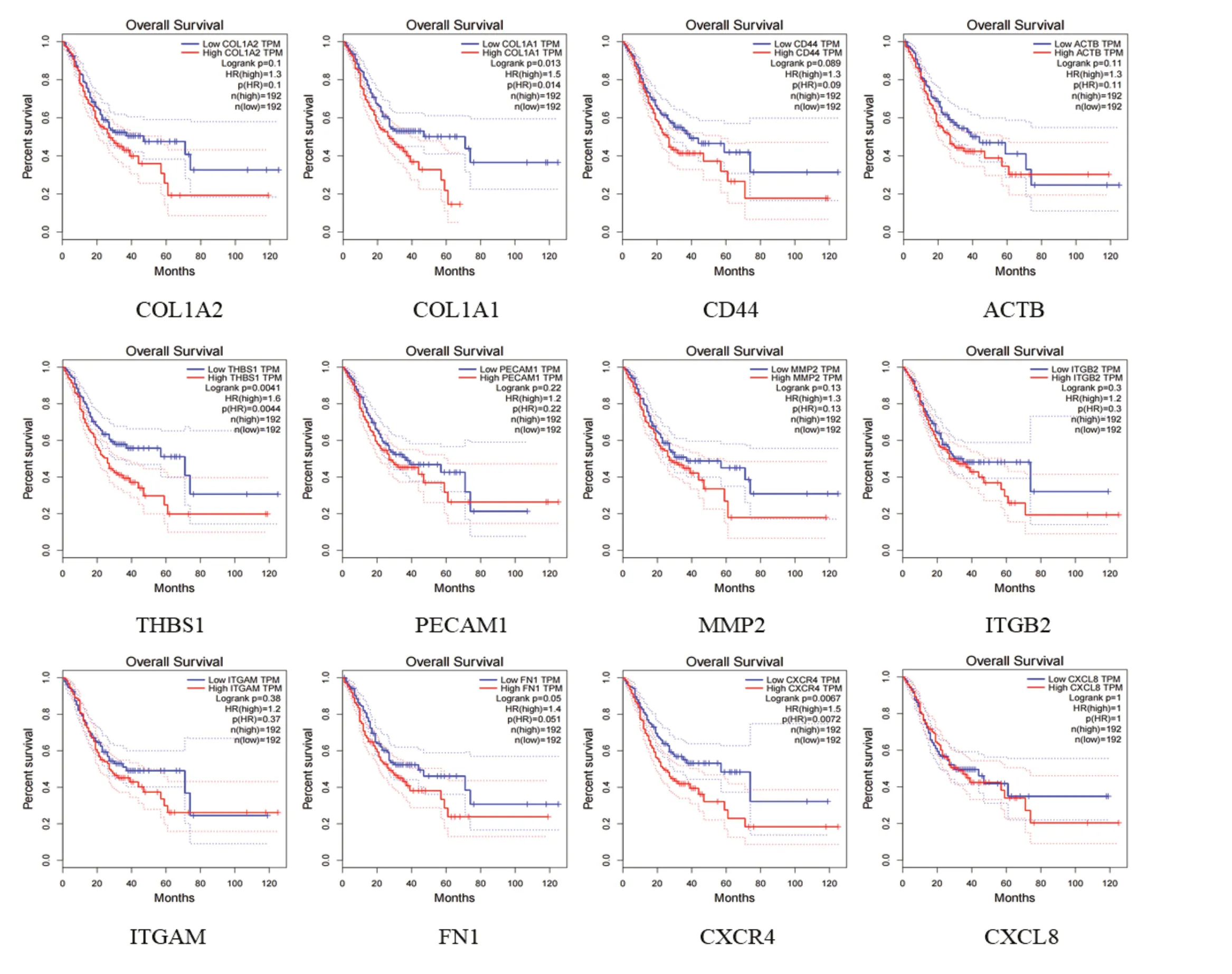
图5 核心差异表达基因生存分析图
3 讨论
在2018年全国最新癌症报告中指出,胃癌在我国的发病率为第二位[5],它的发生发展与多种因素可能相关,例如性别、年龄、种族、生活饮食方式等[6]。为了更好、更方便地去了解胃癌相关发病机制,从而达到理想的诊断和治疗效果,我们可以从基因芯片方面去入手。本研究通过GEO数据库进行检索,得到两组基因芯片GSE33651、GSE54129,通过韦恩图筛选出差异共表达基因330个,其中上调基因有207个,下调基因123个。结合GO及KEGG分析我们发现,这些差异共表达基因主要集中在胶原结合、蛋白质结合、整联蛋白结合、纤连蛋白结合等过程中。主要参与在细胞外泌体、细胞外基质、胞外区域、细胞表面等。同时,在胶原分解代谢过程、血管生成、细胞粘附、白细胞迁移等发挥着作用。KEGG分析中,差异表达基因主要集中于百日咳、阿米巴病、白细胞跨内皮迁移及ECM-受体相互作用等通路。
为了进一步筛选合适基因,我们利用STRING数据库,构建出PPI,并由R软件利用节点联系得出12个核心基因。为评估这些核心基因的表达情况及预后分析,我们利用GEPIA,比较所获取的30个核心差异表达基因在胃腺癌病人与正常人组织的表达水平情况。根据所得图表发现除了PECAM1外,其余基因在STAD病人中均表达上调。结合生存分析图,可以找到基因FN1、CXCR4、COL1A1的低表达是有益于胃癌患者的生存。
纤维连接蛋白(FN1)是一种糖蛋白分子,它参与各种细胞的细胞粘附,迁移和运动[7]。先前有研究表明其与卵巢癌、乳腺癌的发生发展有关[8,9]。在非小细胞肺癌(NSCLC)中,FN1可能与血管内皮生长因子A相互作用,以此起到重要作用,且其相应的蛋白质可作为NSCLC患者诊断或治疗的靶点[10]。经过进一步的分析验证后,发现FN1的低表达对胃癌细胞的侵袭有抑制作用[11,12]。趋化因子受体4(CXCR4)是CXCL12的主要受体[13]。相对于正常人而言,胃癌患者体内的CXCR4和CXCL12呈现高表达状态[14],实验发现CXCL12与CXCR4的结合上调了磷脂酰肌醇3激酶(PI3K)/雷帕霉素(mTOR)和c-MET途径,从而使胃癌的侵袭性增加[15,16]。此外,CXCR4在乳腺癌、胰腺癌、结直肠癌中,也有报道说明其高表达[17,18]。胶原蛋白是ECM的重要组成部分,能够增强癌细胞的迁移、侵袭和粘附性[19,20]。I型胶原蛋白α1(COL1A1)是胶原蛋白家族的成员之一,有报道说,在乳腺癌、肺癌、肾癌中,COL1A1与肿瘤细胞的增殖及侵袭性有关[21-23]。在肝细胞癌变和转移的潜在靶标里,COL1A1也被当做可靠的生物标志物[24]。在胃癌中,尚未有明确的实验证明COL1A1的作用和分子调控机制。但最新的一项研究猜测,COL1A1通过激活TGF-β信号通路来促进了GC细胞的迁移和侵袭[25]。
综上所述,我们通过GEO数据库进行筛选分析,确定核心差异表达基因,发现了FN1、CXCR4、COL1A1这三个基因,通过富集分析和蛋白互作、生存分析发现,这些基因可能作为胃癌的潜在标志物来帮助胃癌的早期诊断、治疗及预后判断。在基因诊断技术愈发成熟的现在,更加简便、快捷、有效的方法一定可以得到进一步的发掘。

