曲霉来源的α-L-鼠李糖苷酶结构特征规律分析
蔡雨晨,李孟泽,李利君,2,倪辉,2
(1.集美大学食品与生物工程学院,福建厦门 361021)
(2.福建省食品微生物与酶工程重点实验室,福建厦门 361021)
关键字:α-L-鼠李糖苷酶;进化树;代表性序列;结构特征
α-L-鼠李糖苷酶(α-L-rhamnosidase,EC3.2.1.40)是一种专一性水解鼠李糖苷键的酶[1],近年来,在食品加工特别是饮料加工中被广泛应用,如水解柚子中的苦味物质制作柚子汁,改善酿造酒及果汁的风味等[2],此外,α-L-鼠李糖苷酶还可作为食品添加剂改变流体性质[3]。在自然界中,α-L-鼠李糖苷酶主要来源于细菌[4]和霉菌[5],最初是从青霉菌和曲霉菌代谢生产的酶制剂中纯化得到,国外关于真菌中α-L-鼠李糖苷酶基因的克隆研究也主要集中于曲霉来源的α-L-鼠李糖苷酶[6];在碳水化合物活性酶数据库(CAZy)[7]中,α-L-鼠李糖苷酶主要存在于糖苷水解酶第 78家族(glucoside hydrolase family 78,GH78),少量存在于GH28和GH106家族中,且来源于真菌的晶体结构有且仅有一个,即Aspergillus terreus来源的α-L-鼠李糖苷酶(PDB:6gsz)[8],因此对曲霉来源的α-L-鼠李糖苷酶在蛋白结构方面上缺乏系统性研究。
序列比对是解决进化树构建、保守区和保守位点分析等众多问题的开端和基础步骤[9]。系统进化树能够展示蛋白的系统进化关系,描述发生或进化顺序,是系统性分析蛋白或基因序列的重要手段[10]。同源建模是利用蛋白质的三级结构比一级结构更保守的原理,使用已经确定结构的模板蛋白对未知结构的蛋白进行三级结构构建[11]。穿线法建模则利用自然界中蛋白质折叠类型数目是一定的,且相似性比较低的氨基酸序列可能对应着一致的折叠类型这一原理,弥补同源建模中必须有相似度较高的模板的这一不足[12,13]。
通过三级结构建模对蛋白结构进行直观的观察[14],并结合结构叠合的方法进一步对蛋白结构进行分类,可以分析得到结构进化的规律。因此,本文通过序列比对方法及进化树构建技术对曲霉来源的α-L-鼠李糖苷酶进行研究,并利用生物信息学手段对α-L-鼠李糖苷酶蛋白序列的一级序列和二级结构进行分析,再采用同源建模与穿线法建模的方法进行三级结构建模,结合结构叠合的结果,综合分析探索曲霉来源α-L-鼠李糖苷酶的蛋白结构特征。
1 材料与方法
1.1 实验数据来源
进入美国国家生物信息中心 NCBI网站(http://www.ncbi.nlm.nih.gov/),输入检索词“(α-L-rhamnosidase)AND "Aspergillus"”,下载NCBI核苷酸数据库中曲霉来源α-L-鼠李糖苷酶核酸序列的FASTA文件和GenBank文档,并使用NCBI Blast+2.10.0[15]筛选出非重复序列。
1.2 分析方法
1.2.1 系统进化树构建
利用 ClustalX 2.0[16]软件进行核酸和蛋白质的多序列比对;运用 MEGAX 6.0[17]软件对得到的多序列比对结果分别构建核酸和蛋白质序列进化树。
1.2.2 蛋白质一级结构和二级结构分析
(1)通过 ProtParam[18](https://web.expasy.org/protparam/)工具预测蛋白的理化性质;
(2)利用 ProtScale[19](http://www.expasy.org/cgi-bin /protscale.pl)工具进行蛋白质疏水性分析;
(3)利用 TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0)工具寻找蛋白质的跨膜区域并进行分析;
(4)使用 SignalP 5.0[20](http://www.cbs.dtu.dk/services/SignalP/)工具进行蛋白的信号肽预测分析。
1.2.3 蛋白质三级结构分析
利用同源建模在线服务器 Swiss-Model[11](https://swissmodel.expasy.org/),穿针引线法建模在线服务器 I-TASSER[12](https://zhanglab.ccmb.med.umich. edu/ I-TASSER/)和 Phyre2[13](http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)进行蛋白的三级结构建模,并使用工具 Verify3D[21](https://servicesn. mbi.ucla.edu/Verify3D/)对模型的质量进行评价分析;运用UCSF Chimera 1.14[22]软件进行蛋白质的三维结构结合叠合和比对。
1.3 数据处理
所有数据都根据实验方法中各个软件及网站收集得来,最多保留两位小数;序列比对结果使用Espript 3.0[23](http://espript.ibcp.fr/ESPript/cgi-bin/ ESPript.cgi)进行美化;281条核酸序列进化树使用 ITOL(https://itol.embl.de/)进行美化;21条蛋白序列进化树使用MEGAX 6.0软件自动生成的图片结果;蛋白三维结构模型及结构叠合结果使用 UCSF Chimera 1.14软件对进行渲染。
2 结果与讨论
2.1 曲霉来源α-L-鼠李糖苷酶的核酸序列分析
2.1.1 核酸序列数据收集

表1 NCBI中291条曲霉来源α-L-鼠李糖苷酶核酸序列统计信息Table 1 Sequence statistics of α-L-rhamnosidase from 310 Aspergillus species in NCBI
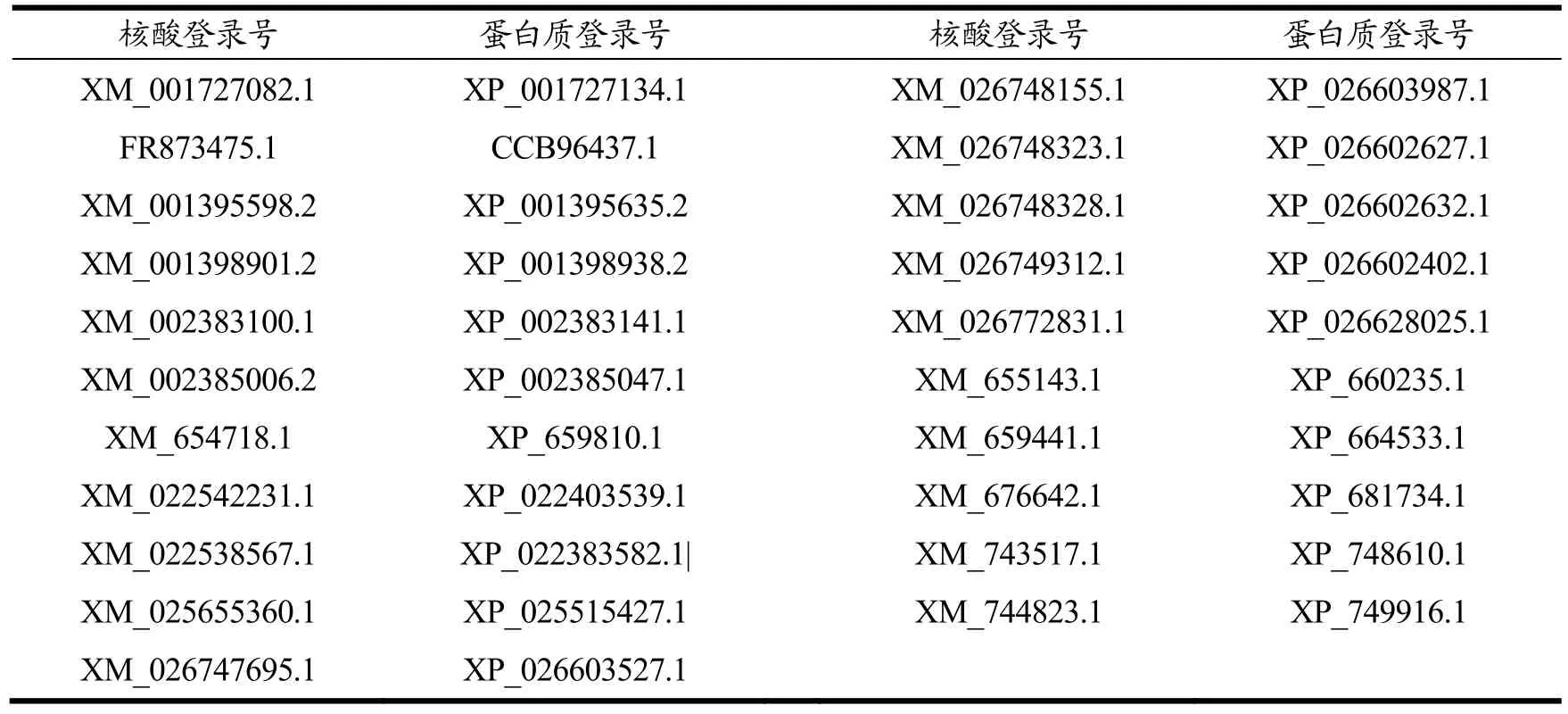
表2 21条代表性核酸序列对应蛋白质登录号Table 2 The corresponding protein registration numbers of 21 representative nucleic acid sequences
在NCBI核苷酸数据库中,检索到291条曲霉来源的α-L-鼠李糖苷酶核酸序列,经过统计将它们分成了37个种类(表1)。发现几乎所有种类的曲霉都有分离纯化得到α-L-鼠李糖苷酶的记录。
2.1.2 核酸系统进化树构建及代表性序列筛选
筛除掉10条完全重复序列,最终得到281条未重复曲霉来源的α-L-鼠李糖苷酶核酸序列,然后对281条核酸序列建立进化树(图1),并根据系统进化树筛选出21条代表性的核酸序列(图1中的橙色标识),并得到了对应的蛋白质序列(表2)。
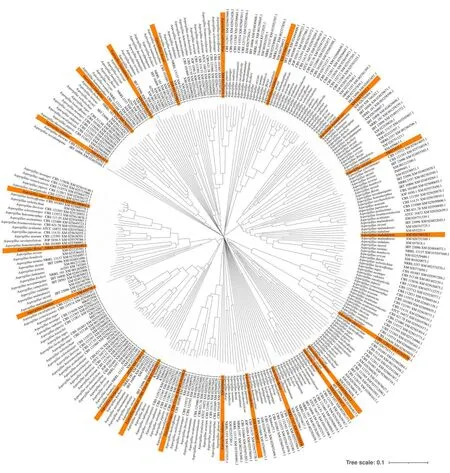
图1 281条曲霉来源α-L-鼠李糖苷酶核酸序列构建的系统进化树Fig.1 Phylogenetic tree of 281 Aspergillus derived α-L-rhamnosidase nucleic acid sequences
2.2 曲霉来源α-L-鼠李糖苷酶的蛋白序列分析
2.2.1 多序列比对分析
多序列比对结果显示了α-L-鼠李糖苷酶蛋白质序列的保守氨基酸位点(图2),发现这21条α-L-鼠李糖苷酶代表序列的氨基酸保守位点较为分散且仅有22个较保守位点,分别位于285、359、392、507、510、574、595、596、626、631、634、648、644、651、678、681、684、685、825、831、844和852位,推测这些氨基酸是与保持结构或催化功能密切相关的极其重要关键性氨基酸,且并没有发现有非常保守的位点和保守区存在,说明选择的这21条序列相互独立,可以通过对这 21条代表性序列的分析可以基本概括出所有曲霉来源α-L-鼠李糖苷酶的相关规律。
2.2.2 理化性质分析
对21条代表性蛋白质序列进行理化性质分析(表3)发现它们氨基酸数量范围为556~1032个,分子量(Mr)的极差为37606 u,原子总数的极差为5225个,在氨基酸数量、分子量、原子总数上波动较大。对负电荷残基总数(Asp+Glu)和正电荷残基总数(Arg+lys)进行比较,发现负电荷残基总数略多,大多带负电荷。理论等电点(pI)的范围是 4.66~7.17,除了XP_660235.1的pI为7.13、XP_664533.1为7.17,是弱碱性蛋白质;其余α-L-鼠李糖苷酶蛋白质的理论等电点都小于7,属于酸性蛋白质,与张霞[6]总结的关于真菌来源的α-L-鼠李糖苷酶的pI实验值一致;α螺旋指数的范围是71.36~86.19;亲水性平均系数的范围是-0.355到-0.051,说明曲霉来源的α-L-鼠李糖苷酶为亲水性蛋白[24]。

图2 曲霉来源21条α-L-鼠李糖苷酶序列保守位点Fig.2 Conserved sites of 21 α-L-rhamnosidase sequences from Aspergillus species
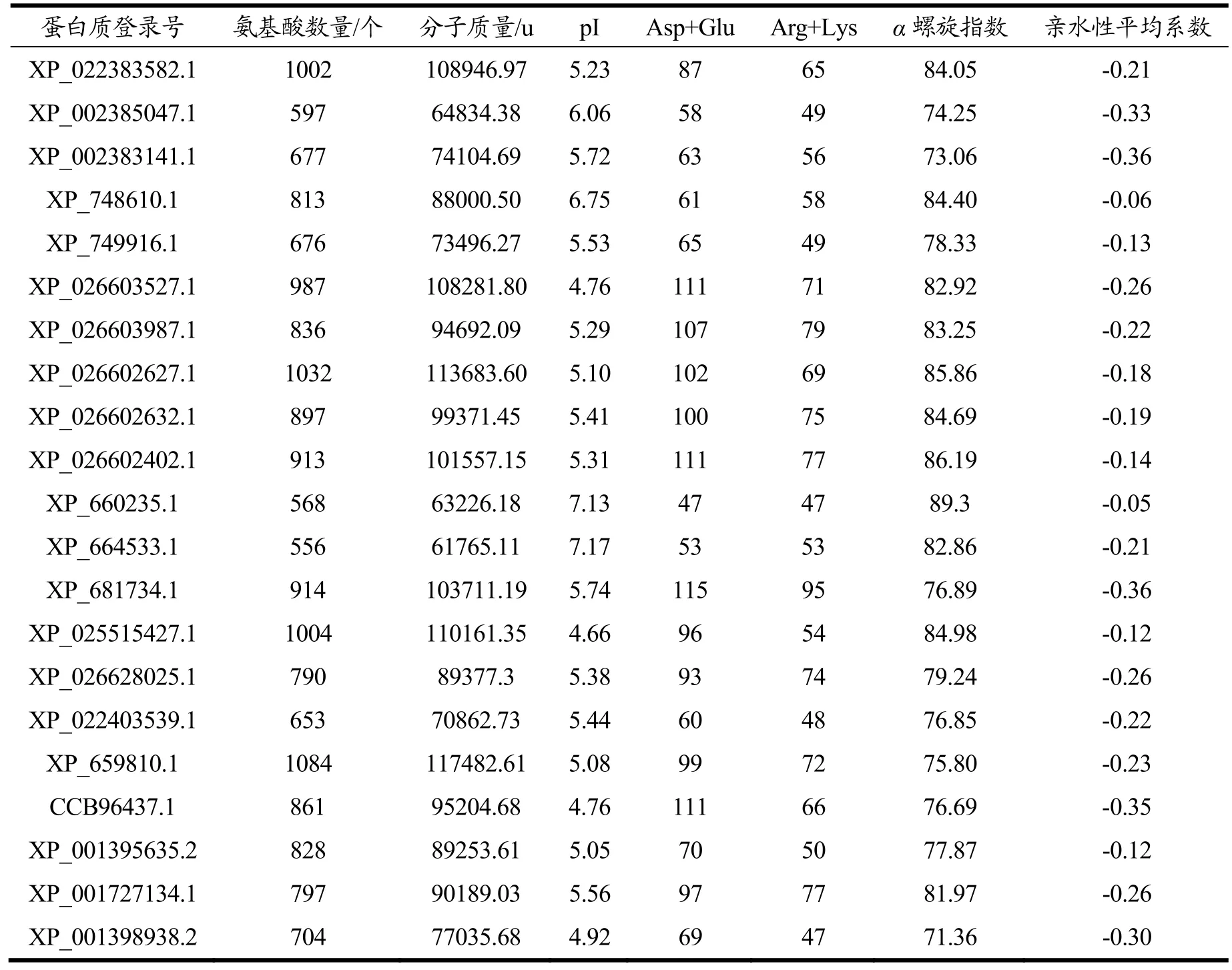
表3 21条曲霉来源α-L-鼠李糖苷酶理化性质预测Table 3 Prediction of physicochemical properties of α-L-rhamnosidase from 21 Aspergillus species
2.2.3 疏水性和跨膜区分析
氨基酸的疏水性反映α-L-鼠李糖苷酶的折叠情况,在潜在的跨膜区域会出现疏水区。对亲水性平均系数比较小的XP_660235.1蛋白进行疏水性分析和跨膜区预测。使用ProtScale得到了对XP_660235.1蛋白的分析结果(图3)。发现XP_660235.1在100~200之间有明显的两个疏水峰,是潜在的跨膜区域[25]。

图3 XP_660235.1蛋白的疏水性预测图Fig.3 Hydrophobicity prediction map of XP_660235.1 protein
使用TMHMM工具预测XP_660235.1蛋白的跨膜区,发现136到158位和171到193位拥有跨膜螺旋区的可能性接近1(图4),说明XP_660235.1蛋白有两部分位于细胞膜表面,分别是氨基酸序列的 1到135位和194到568位,XP_660235.1为两次跨膜的蛋白质,这与 ProtScale预测结果一致。对剩余的20条蛋白质序列进行预测,结果显示无跨膜区存在。
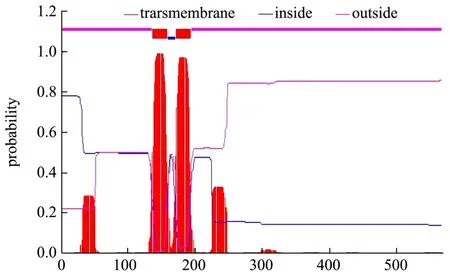
图4 XP_660235.1蛋白的跨膜区预测图Fig.4 Prediction map of transmembrane region of XP_660235.1 protein
2.2.4 蛋白进化树分析
对这21条曲霉来源α-L-鼠李糖苷酶蛋白质序列进行系统进化树构建分析(图 5),根据进化树结果可以将21条代表性序列分为两组,第一组包含17个序列为 XP_660235.1、XP_664533.1、XP_748610.1、XP_749916.1、XP_001395635.2、XP_001398938.2、XP_002383141.1、XP_002385047.1、XP_022403539.1、XP_001727134.1、CCB96437.1、XP_681734.1、XP_659810.1、XP_026602402.1、XP_026602632.1、XP_026603987.1、XP_026628025.1,第二组包含 4个序列为 XP_022383582.1、XP_025515427.1、XP_026602627.1、XP_026603527.1。

图5 21条曲霉来源α-L-鼠李糖苷酶蛋白质序列构建的邻接法进化树Fig.5 Phylogenetic tree of 21 Aspergillus derived α-L-rhamnosidase protein sequences
2.2.5 信号肽预测分析

表4 信号肽预测结果Table 4 Prediction results of signal peptide
在 SignalP 5.0的预测结果中(表 4),XP_002383141.1、XP_022403539.1、XP_659810.1、XP_001395635.2、XP_022383582.1、XP_748610.1、XP_001398938.2、XP_025515427.1、XP_026602627.1和XP_026603527.1共10条α-L-鼠李糖苷酶蛋白序列含有信号肽。与进化树分类结果结合发现,含有信号肽的序列均匀的分布在第一组与第二大组中,说明曲霉来源的α-L-鼠李糖苷酶存在胞外酶与胞内酶两种,且信号肽的有无不能反映出曲霉来源α-L-鼠李糖苷酶的进化规律。
2.3 曲霉来源 α-L-鼠李糖苷酶的蛋白三级结构分析
2.3.1 三级结构建模分析
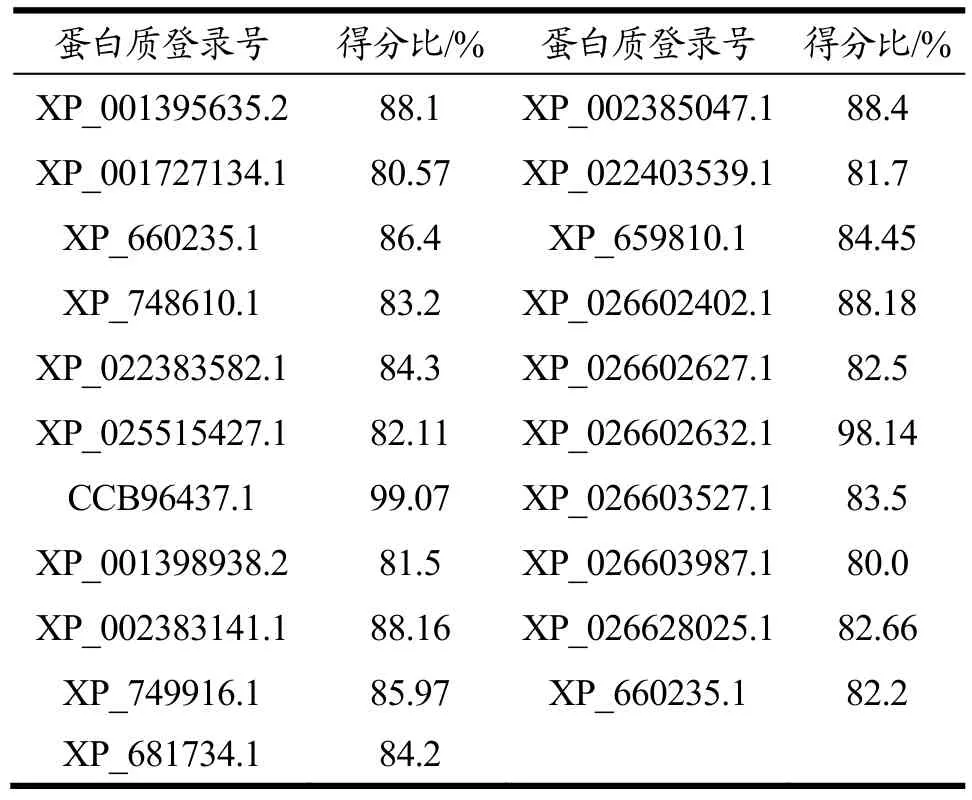
表5 Verify3D建模评分表Table 5 Verify3D modeling scoring table
对21条曲霉来源α-L-鼠李糖苷酶的蛋白质序列进行三维建模,其中 5条采用同源建模法,另外 16条采用穿针引线法进行建模。对所有蛋白质三级结构建模结果进行Verify3D评价。从结果(表5)中可以看到蛋白质序列的得分比最小值为80.0%,说明α-L-鼠李糖苷酶的蛋白质序列的建模结果良好。
2.3.2 结构叠合分析
结构叠合的分类结果显示(表6),这21个曲霉来源α-L-鼠李糖苷酶可分为两大类(图 6),XP_681743.1、XP_664533.1、XP_660235.1、XP_026602402.1、XP_022403539.1、XP_659810.1、XP_001727134.1、XP_026602632.1、XP_026603987.1、XP_026628025.1、XP_748610.1、XP_749916.1、XP_001395635.2、XP_001398938.2、XP_002383141.1、XP_002385047.1与CCB96437.1共17条序列组成第一大类,每条序列都拥有一个(α/α)6桶状结构和桶底的一个β折叠结构这一基本结构,根据除基本结构外的β片层结构的数量,又可以将第一大类分为4个小类(图7),其中XP_664533.1与XP_660235.1组成无额外β片层的第一个小类;XP_022383582.1、XP_749916.1、XP_001395635.2、XP_001398938.2 与XP_002383141.1共5条序列组成有1个额外β片层的第二小类;XP_659810.1、XP_001727134.1、XP_026603987.1、XP_026628025.1、XP_748610.1 与XP_002385047.1共6条序列组成有2个额外β折叠的第三小类;XP_681743.1、XP_026602402.1、XP_026602632.1与CCB96437.1共4条序列组成有3个额外β折叠的第四小类,目前已报道的曲霉来源的晶体结构(PDB:6gsz)[8]就属于这一类;XP_022383582.1、XP_026602627.1、XP_025515427.1与XP_026603527.1共4条序列组成第二大类,与第一大类不同,第二大类4个序列都属于GH106家族,因此拥有GH106的基本结构(α/β)8结构和环绕在桶装结构域周围的β折叠结构[26]。第一大类与第二大类的分类结果与进化树分类相符,说明蛋白的进化规律会一定程度的体现在其三级结构上;第一大类的四个小类与进化树分类不符,说明β折叠结构的数量并不能反映曲霉来源α-L-鼠李糖苷酶的进化规律。

表6 结构分类表Table 6 Structure classification table

图6 α-L-鼠李糖苷酶的两个大类叠合图Fig.6 Two kinds of superposition graphs of α-L-rhamnosidase
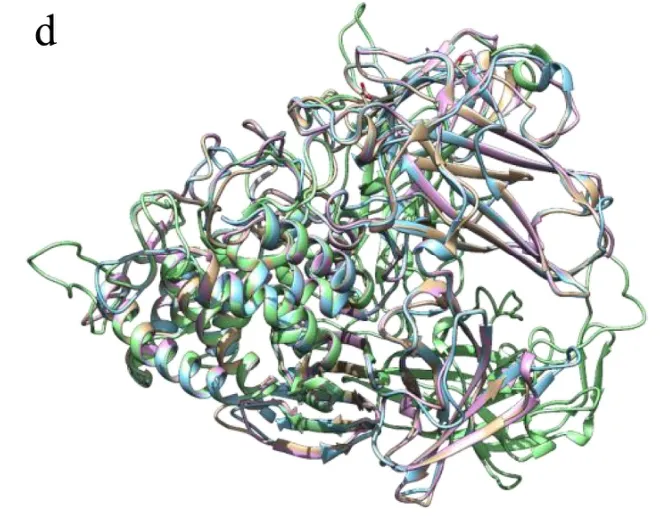
图7 α-L-鼠李糖苷酶的第一大类的四个小类叠合图Fig.7 Four subclasses of the first class of α-L-rhamnosidase
3 结语
本文通过NCBI数据库,收集了所有非重复的曲霉来源α-L-鼠李糖苷酶的核酸数据共291条,并通过进化树筛选得到具有代表性的 21条序列并预测了蛋白理化性质。通过对这21条代表性序列进行序列比对和进化树构建发现,虽然这21条序列具有非常少的保守位点,但是它们之间依然存在进化规律,且根据这一进化规律,可以将这些序列分为两组;信号肽分析结果显示有 10条序列含有信号肽,说明曲霉来源的α-L-鼠李糖苷酶有胞外酶与胞内酶两种;跨膜区分析发现1条来源于Aspergillus nidulans的α-L-鼠李糖苷酶为二次跨膜蛋白;通过蛋白的三级结构建模及叠合,发现21条α-L-鼠李糖苷酶主要存在于GH78与GH106家族中,将21条α-L-鼠李糖苷酶分为两个类型,第一大类都含有一个(α/α)6桶状结构和桶底的一个β片层结构,并根据额外含有的β片层结构的数量进一步分成4个小类;第二大类则含有1个(α/β)8结构和环绕在桶装结构域周围的β片层结构,且结构叠合分类与进化树的分类一致,说明蛋白的进化规律会一定程度的体现在其三级结构上,而小类的分类结果说明β折叠结构的数量并不能作为说明曲霉来源α-L-鼠李糖苷酶进化规律的依据。本文通过筛选出21条代表性序列阐明曲霉来源的α-L-鼠李糖苷酶蛋白序列性质及结构特征,系统性的分析了其结构规律,为该酶的定向进化和分子改造提供了强有力的参考。

