骨髓红粒细胞自动识别的深度学习模型
吴汾奇, 吕丽丽, 吕 迪, 冯辰彬, 施 恬, 王 维, 崔红花, 周 柚,c
(吉林大学 a. 计算机科学与技术学院; b. 第二医院; c. 符号计算与知识工程教育部重点实验室, 长春 130012)
0 引 言
骨髓是人体重要的造血器官和免疫器官, 骨髓血细胞来源于造血干细胞,造血干细胞具有长期自我更新和分化成各类成熟血细胞的能力[1]。基于细胞形态学对骨髓血细胞进行分类和统计是临床上对白血病、 各种贫血和恶性组织细胞病等多种重大血液疾病诊断的依据, 是疾病治疗方案选择的决定性因素[2]。骨髓血细胞形态极为复杂, 包括髓系和淋系, 髓系包含红系、 粒单系、 巨核系三大类共20余种子类; 淋系包含4种子类, 根据细胞核与胞浆的颜色、 形态和大小等特征共同决定血细胞的类型[3-4]。目前, 在国内外大型医疗机构中, 一些重大血液疾病的临床诊断仍然采用医师人工观察显微镜下血细胞涂片, 以细胞形态学为理论基础, 对骨髓血细胞进行识别和计数统计, 该项工作十分耗时耗力, 而且医师需要多年的培训才能胜任该项工作[5]。因此, 将人工智能领域中主流的深度学习技术与图像处理技术相结合[6], 研究骨髓血细胞自动识别的新技术, 部分替代专家繁琐劳动, 辅助医生诊断, 是一项有意义的工作。
近年来随着人工智能, 特别是深度学习理论和技术的发展[7], 为医学影像处理领域提供了更高效的解决方案。在细胞自动识别领域, 主要人工智能技术包括图像分割技术[8]、 目标检测技术[9]和图像语义分割技术[10]。图像分割是将数字图像细分为多个图像子区域的过程, 实现定位图像中的物体和边界, 在细胞识别中将细胞从整体图像中分割出来, 送入分类器进行识别。目标检测是一种基于目标几何和统计特征的图像分割技术, 从图像中定位所需识别的目标, 准确判断它们的位置和类别, 并给出每个目标的边界框。目标检测技术根据有无候选框生成阶段分为两类: 一类是基于候选区域的双步骤算法[11], 即先对细胞进行定位, 然后再对细胞做分类; 另一类是使用卷积神经网络将定位和分类两阶段步骤融合的目标检测算法[12], 直接给出定位和分类结果。而图像语义分割技术是通过构建端到端的深度学习网络对输入图像中的每个像素点根据其所属类别进行分类,并为图像中像素点分配表示其语义类别的标签。
在血细胞自动识别研究领域, 学者们在血细胞自动分割[13]、 定位[14]、 分类[15]和语义分割[16]等领域开展了广泛研究, 提出了一系列算法和可用数据集。这些方法中包括Ramoser等[17]利用K均值聚类方法将血细胞与其他细胞分离, 然后利用支持向量机对血细胞进行分类。Ghosh等[18]使用分水岭算法分割细胞, 将分割后的血细胞图像送入朴素贝叶斯分类器中进行分类。这类方法对细胞分割的精确度要求较高, 而细胞分割方法对形状不规则细胞以及多粘连细胞分割效果不稳定, 易对实验结果产生影响。Prayag等[14]建立基于CNN(Convolutional Neural Networks)框架使用双阶段目标检测方法实现外周血细胞图像的自动分类, 这种方法准确度较高, 但检测速度较慢。Alam等[15]使用基于YOLO(You Only Look Once)框架的目标检测算法识别3种类型的血细胞, 这种单阶段目标检测方法检测速度较快, 但分类准确性较低。图像语义分割技术是图像处理领域的新技术, Long等[16]提出了全卷积网络(FCN: Fully Convolutional Networks)将图像分类问题从图像级别的分类延伸到像素级别的分类, 推动了语义分割技术领域的发展。这种像素级别的分割方法保证了速度和分割结果的准确度, 是目前图像处理领域主流的技术。近年来, 学者们提出了多种图像语义分割模型, 如Badrinarayanan等[19]改进FCN网络模型, 提出了具有编码器-解码器结构的SegNet模型。Chen等[20]提出DeepLab模型对图像进行语义分割。虽然语义分割模型实验效果较好, 但需要大量的数据图像进行训练, 而医学影像的获取, 以及对图像进行准确的标记对大多数任务是一项耗时耗力的工作。Ronneberger等[21]提出同样具有编码器-解码器结构的U-Net模型, 该模型在训练数据不足的情况下仍能表现出较好的分割效果, 并在ISBI(IEEE International Symposium on Biomedical Imaging)细胞跟踪挑战赛中获得冠军。Zeng等[22]使用基于U-Net的神经网络模型在计算精度医学细胞核分割挑战中获得第3名。目前很多理论和实验表明加深网络的层次有利于获得更好的训练结果[23]。然而, 随着网络模型深度的增加, 模型的训练变得更加困难。He等[24]提出了ResNet网络结构, 利用残差模块使网络随深度增加而不退化, 有效解决了网络深度的训练问题, 同时能更好地结合图像语义信息。
虽然深度学习技术在血细胞分割、 定位和识别研究方面有了长足的进步, 但骨髓血细胞形态复杂、 种类繁多, 部分细胞间类别差异达到细粒度级别, 使骨髓血细胞自动识别技术成为一项极具挑战性的工作, 在国内外文献中未见相关报道。笔者构建了骨髓红系细胞和粒系细胞数据集, 提出了一种基于U-Net网络结构和ResNet残差网络结构的CellNet网络模型, 并通过实验验证了CellNet在骨髓红系细胞和粒系细胞自动识别上的有效性, 在骨髓血细胞自动识别领域开展了探索性研究。
1 数据采集与处理
笔者构建了骨髓血细胞数据集BMCD(Bone Marrow Cells Dataset)(https:∥pan.baidu.com/s/1EPAJ_AzAe AWFt9th4mP2_g. 提取码: nf08), 该数据集由吉林大学第二医院血液科采集, 血液科医师使用IDA-2000数学医学图像分析仪系统-200139和日本OLYMPUS显微镜, 首先镜下锁定目标图像, 其次将图像上传至医学图像分析系统, 再次导出图像并根据专家经验标记各类不同细胞, 构建骨髓血细胞数据集。骨髓血细胞数据集中包含图像共298张, 包含红系和粒系细胞共计2 682个, 其中红系细胞819个, 粒系细胞1 863个, 图像尺寸为512×512像素, 采用手工方式对所有血细胞进行了标记。基于图像语义分割技术本质上是对原始图像中像素进行分类, 因此笔者将图像中红系细胞与粒系细胞作为待识别的两类, 其余细胞及背景区域作为第3类, 标记方式如图1a所示, 用RGB(Red Green Blue)值(255,0,0)标记红系细胞像素、 (255,255,0)标记粒系细胞像素、 (0,0,0)标记背景像素, 得到掩膜(Mask)图像。另外, 笔者在训练深度学习模型时, 采用了数据增强技术[25]为模型训练过程提供更多的训练数据, 数据增强包括旋转、 翻转、 随机放大裁剪等方法, 如图1b所示。

图1 骨髓血细胞图像数据处理过程
2 CellNet模型
CellNet是一种基于U-Net[21]和ResNet50[24]的图像语义分割模型, CellNet网络结构与U-Net相似, 为编码器-解码器结构, 如图2所示。
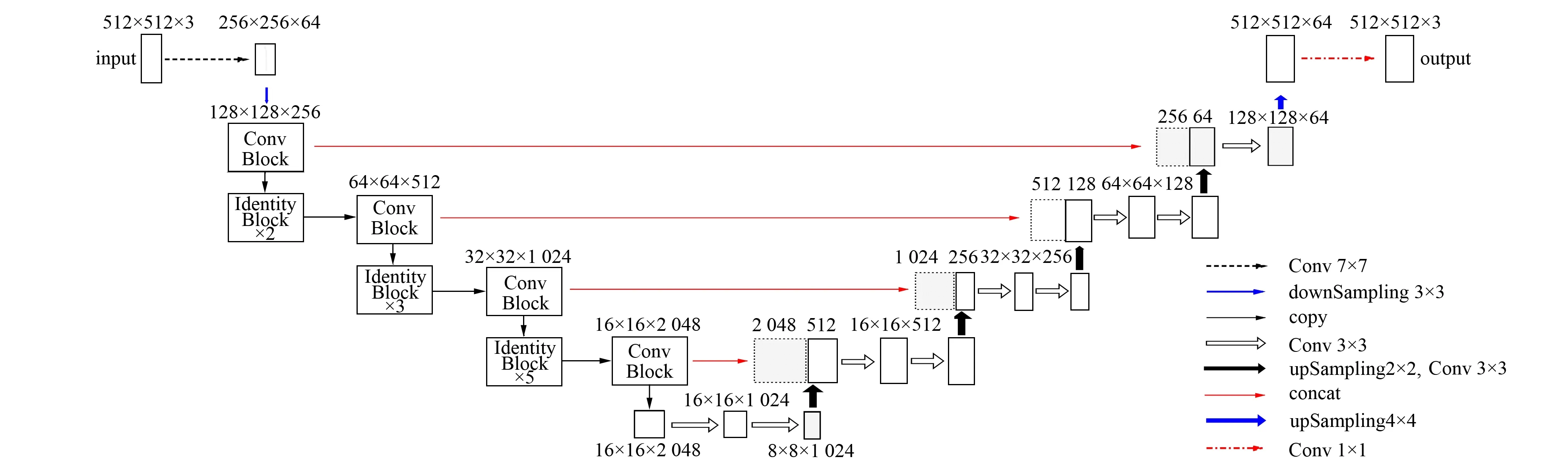
图2 CellNet模型网络架构
CellNet网络的输入图像尺寸为512×512×3像素, 经过3次卷积、 1次下采样操作和14个3层残差模块逐步缩小图像尺寸, 得到红系、 粒系细胞在骨髓血细胞图像中上下文语义信息(深层信息)。在解码器中经过上采样操作逐步恢复图像大小, 并融合同等高度编码器中的信息(浅层信息), 最后经过softmax得到输出图像。CellNet与U-Net的区别在于将ResNet50中的残差模块加入到网络中, 增加了网络的深度; 另外, CellNet网络采用卷积代替U-Net网络中用于特征提取的最大池化操作, 并移除U-Net网络中的裁剪操作, 为分割提供更精细的特征。
CellNet网络的左侧编码器部分采用ResNet50中的3层残差模块结构, 3层残差模块包括恒等残差块(Identity Block)和卷积残差块(Convolutional Block), 结构如图3所示。恒等残差块是ResNet网络中使用的标准残差块, 对应于输入激活与输出激活具有相同的维度。卷积残差块是另一种类型的残差块, 与恒等残差块不同之处是卷积残差块的shortcut路径中为一个CONV2D层。Batch Norm即批标准化(Batch Normalization), 可以在训练过程中使每层神经网络的输入保持相同分布。这种结构有效克服了由于加深网络层数造成的参数过多和梯度弥散问题, 而且使网络模型更容易训练, 提升了模型的训练速度。
CellNet网络模型第1个卷积层定义卷积核大小为7×7, 步长为2, 下采样操作定义卷积核大小为3×3, 步长为2, 输出图像尺寸为128×128×64像素, 之后经过14个3层残差模块和两次步长分别为1和2的卷积, 输出图像的尺寸变为8×8×1 024, 编码器结构参数如表1所示。
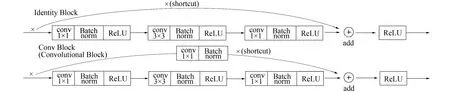
图3 3层残差模块

表1 CellNet模型编码器结构参数
CellNet网络模型右侧扩展路径基于U-Net的解码器结构, 由5次上采样和12次卷积组成, 并融合由同等高度的编码器传递的浅层信息, 将图像尺寸逐步恢复为512×512×3, 具体结构参数如表2所示。

表2 CellNet模型解码器结构参数

(续表2)
3 实验设计与结果分析
3.1 实验设计
为了验证笔者提出的CellNet模型在骨髓血细胞上识别的效果, 在BMCD数据集上设计了实验, 并与U-Net在相同实验条件下进行了对比。实验是在BMCD数据集中随机选取40张图像作为测试集, 利用Python的Pillow库对其余258张骨髓血细胞图像做数据增强处理, 扩充到1 200张图像, 并将其划分为训练集和验证集(9 ∶1), 其中训练集1 080张, 验证集120张, 采用十折交叉验证的方法对网络进行训练。
实验基于Keras2.2.4开源框架, 在型号为NVIDIA GeForce GTX 1080 Ti的GPU上进行训练。CellNet网络的输入图像尺寸为512×512×3, 批大小设置为12, 学习率设置为0.000 1, 训练轮次设置为100, 采用Adam优化器和交叉熵损失函数(Categorical Crossentropy)。
3.2 评价方法
为了评价CellNet模型效果, 笔者使用4个性能指标对网络模型进行测试, 分别为均交并比(MIoU: Mean Intersection over Union)、 精确率(Precision)、 召回率(Recall)和算法执行时间。
交并比(IoU: Intersection over Union)为计算机视觉图像分割领域中经常使用的一个概念, 是产生的候选框与原标记框的交叠率, 即它们的交集与并集的比值, 是衡量定位精确度的一种方式。均交并比作为衡量图像分割精度的标准度量, 其原理为在每个类上计算IoU值并取平均值, 定义为

(1)
其中k为类别数量,nij为类别i预测为类别j的像素点个数。
精确率(Precision,PPrecision)即在被所有预测为正的样本中实际为正样本的概率。召回率(Recall,RRecall)即在实际为正的样本中被预测为正样本的概率。精确率与召回率定义如下

(2)

(3)
其中NTP为在原图像与模型预测图中均为正例的细胞数量;NFP为在原图像中为反例且在模型预测图中为正例的细胞数量;NFN为在原图像中为正例且在模型预测图中为反例的细胞数量。
3.3 结果与分析
实验结果给出的是十折交叉验证训练结果的均值, CellNet和U-Net对骨髓血细胞图像语义分割的交并比和运算时间的结果如表3所示, 较U-net相比, CellNet模型将MIoU从87.63% 提高到91.21%。在训练时间和测试时间上, 笔者设计的网络都优于U-Net网络。

表3 实验结果1
通过统计两种网络模型在预测图中红系、 粒系细胞数量的识别情况, 计算出精确率和召回率如表4所示。CellNet相比U-Net网络模型, 精确率和召回率有更优秀的表现, 笔者方法在红系、 粒系细胞识别召回率上分别提高了3.97%、 4.74%。

表4 实验结果2
如图4所示, 笔者在测试集上选用4幅骨髓血细胞图像进行对比分析, 其中图4a为原图像, 图4b为对应的掩膜(Mask)图, 图4c和图4d分别为CellNet、 U-Net网络分割的结果。

a 原图 b Mask c CellNet d U-Net
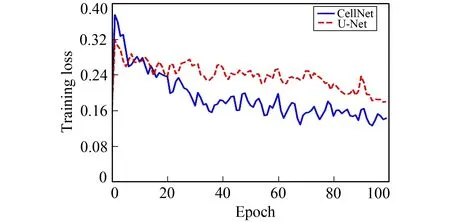
图5 训练过程曲线
根据分割结果对比可以看出, CellNet网络在分割精度上有较大提高, 拟合效果较强, 对不同形态分布的骨髓血细胞图像都能获得较好的分割结果, 对U-Net未能识别的边缘处细胞也能达到较为准确的识别效果, 甚至在复杂并且细胞密度较大的图像上也能有出色的表现。CellNet和U-Net网络模型的训练过程如图5所示, 与U-Net相比CellNet网络在训练过程中收敛速度更快, 同时能获得较低的损失值。
4 结 语
骨髓血细胞自动识别技术对于白血病、 各种贫血、 恶性组织细胞病等多种重大血液疾病诊断和治疗有重要的研究意义, 可以部分替代专家繁琐重复工作, 是一项有意义的研究工作。骨髓血细胞因其形态学的复杂特点, 是该领域研究的难点问题。笔者探索性研究了骨髓血细胞中红系细胞和粒系细胞自动识别技术, 构建了骨髓血细胞数据集(BMCD: Bone Marrow Cells Dataset), 提出了一种基于U-Net网络结构和ResNet残差网络结构的CellNet网络模型, 通过实验验证了CellNet在骨髓红系细胞和粒系细胞自动识别上的有效性。

