酿酒酵母基因组进化的研究进展
夏思杨,江丽红,蔡谨,黄磊,徐志南,连佳长
(1浙江大学化学工程与生物工程学院,生物质化工教育部重点实验室,浙江杭州310027;2浙江大学化学工程与生物工程学院,合成生物学研究中心,浙江杭州310027)
由于对能源安全、可持续性发展和全球变暖的日益担忧,利用微生物细胞工厂将可再生资源转化为燃料和化学品是目前国内外研究的一大热点。通过自下而上或自上而下[1]的工程策略,深入了解生物网络对于构建有实际应用价值的“微生物细胞工厂”至关重要。此外,系统生物学和合成生物学的最新进展使人们对生物系统有了更深的理解,能够做出更具预测性的工程设计。然而,由于生物系统的复杂性,诸如胁迫耐受性等受到多基因调控的复杂表型,单基因理性改造方法很难实现预定的目标。为了克服这一主要限制,越来越多的研究人员将目标转向一种强大的多功能工具——基因组进化,以构建满足工业生产需求的微生物细胞工厂。
酿酒酵母是第一个被完全测序的真核细胞[2],其分子生物学和细胞生物学研究最为透彻。与原核生物不同,酿酒酵母含多种细胞器,能够为生物合成提供不同的环境。此外,酿酒酵母是公认安全的模式生物,遗传操作简单,并且对恶劣的工业条件表现出很高的耐受性[3],适合大规模生产,因此在生物技术领域得到了广泛应用。
定向进化已经在蛋白、途径和基因组水平上取得了里程碑式的进展[4]。本文作者将重点介绍基于酿酒酵母基因组水平定向进化的重要技术进展及其在构建高效酵母细胞工厂方面的应用,并简要介绍酿酒酵母基因组进化面临的挑战和发展趋势。
1 酿酒酵母基因组进化的技术策略
基因组进化起源于选择性育种和驯化,如今已发展成为一种强大的多功能工具,可用于与各种工业过程相关的蛋白质和全细胞生物催化剂的工程设计。基因组进化在试管中模拟达尔文进化,涉及遗传多样性和文库筛选的迭代过程。与理性设计不同,基因组进化工程较少依赖于基因型-表型关系的先验知识,通过非定点或定点诱变有效地创造遗传多样性,然后在选择性生长条件下筛选优异表型。本节将从随机进化(非理性)以及可示踪基因组进化(半理性)两个方向总结酿酒酵母全基因组水平进化的技术发展(图1)。
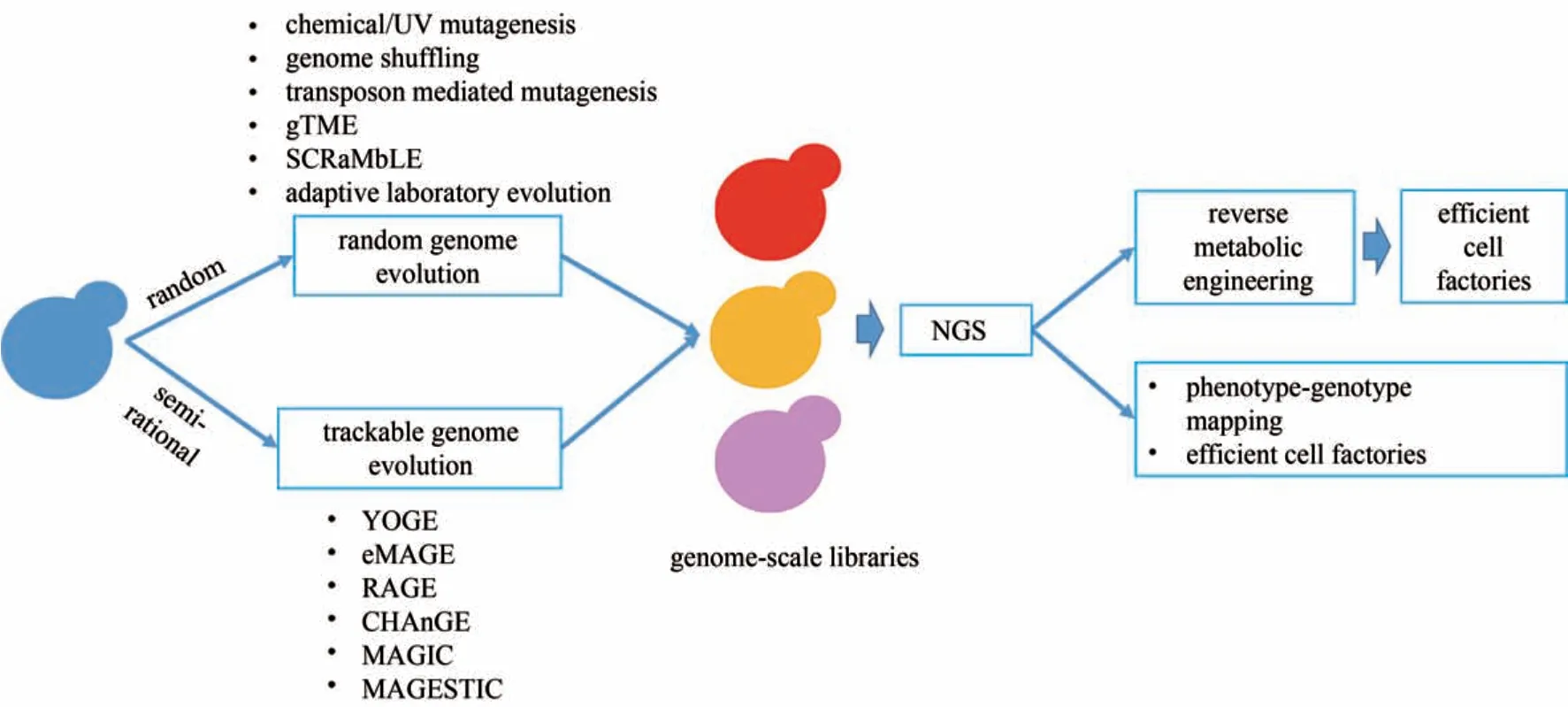
图1 酿酒酵母基因组进化的主要技术策略(基因组进化主要包括基于随机突变的基因组进化技术和基于高效基因组编辑技术的可示踪基因组进化技术)gTME—全局转录机制工程;SCRaMbLE—LoxP介导的合成染色体重组和修饰进化系统;YOGE—酵母寡核苷酸介导的基因组工程;eMAGE—真核多重自动化基因组工程;RAGE—RNAi辅助基因组进化;CHAnGE—CRISPR/Cas9和同源定向修复辅助的全基因组进化;MAGIC—多功能全基因组CRISPR系统;MAGESTIC—基于短、可示踪、整合的细胞条形码的多重精准基因编辑技术;NGS—二代测序Fig.1 Major technologies for genome evolution in Saccharomyces cerevisiae,including random genome evolution and trackable genome evolutiongTME—global transcription machinery engineering;SCRaMbLE—synthetic chromosome recombination and modification by LoxP-mediated evolution;YOGE—yeast oligo-mediated genome engineering;eMAGE—eukaryotic multiplex automated genome engineering;RAGE—RNAi-assisted genome evolution;CHAnGE—CRISPR/Cas9-and homology-directed-repair-assisted genome-scale engineering;MAGIC—multi-functional genome-wide CRISPR system;MAGESTIC—multiplexed accurate genome editing with short,trackable,integrated cellular barcodes;NGS—next-generation sequencing
1.1 基于随机突变的基因组进化
传统的基因组进化方法依赖于随机突变,可通过化学诱变剂/紫外线辐射、适应性进化、转座子插入和基因组改组等实现。基于随机突变的基因组进化方法因操作简单并且能有效产生改良的表型,在工业上被广泛采用,尤其是对于遗传学定义不明确且遗传工具有限的宿主。
1.1.1 化学/物理诱变
化学/物理诱变[5]是一种经典的菌株改良的方法。该技术将微生物细胞暴露在适量的诱变剂下,通过链断裂以及碱基的添加、删除或替换造成DNA随机损伤,再结合有效的筛选策略,获得性状优良的目标菌株。但其分子机制在很大程度上是未知的。常用的化学诱变剂包括亚硝基胍(nitrosoguanidine,NTG)和甲基磺酸乙酯(ethyl methane-sulfonate,EMS),而紫外线也被广泛用于改变微生物的遗传物质。Hashimoto等[6]利用紫外诱变成功选育出营养缺陷型的二倍体工业酵母突变体。Rous等[7]利用EMS诱变技术选育的酵母细胞,降低了异戊醇等杂醇的生产,使葡萄酒的香气更为浓郁。
1.1.2 基因组改组
基因组改组主要是利用原生质体融合和有性重组实现基因组间的同源重组[8],能够显著增加菌株的进化概率,大幅度缩短菌株的选育周期。与传统原生质体融合不同,基因组改组可以将重组库中的多个最佳突变体制备成原生质体并对其进行递归重组,产生多亲本杂交后代[9]。有性重组利用减数分裂机制,创造一个用于结合有利突变的遗传多样性文库。Shi等[10]利用原生质体融合介导的基因组改组成功提高了酿酒酵母的耐热性。Zheng等[11]利用G418和吉欧霉素双抗性标记作为筛选标签,在紫外诱变和两轮有性重组后,分离得到在乙酸胁迫下具有更好生长和乙醇发酵性能的突变菌株。
1.1.3 转座子插入诱变
基因缺失分析是确定基因功能的重要技术。与传统的基因删除策略不同,转座子插入诱变利用一个可移动的基因元件——转座子,在基因组规模上创建一个突变文库。Kumar等[12]利用细菌来源的Tn7系统,通过大规模穿梭诱变在酵母中生成Tn7插入突变文库。证明了转座子不仅可以被用作标签来快速鉴定目标突变体,还能在全基因组范围内研究酵母的基因功能。Ni等[13]利用转座子插入诱变技术,成功构建了木糖利用效率提升的重组酿酒酵母菌株,并且证明了PHO13和TAL1两个基因的表达与木糖特异性生长相关。
1.1.4 全局转录机制工程gTME
细胞在面对内部和外界的刺激时,通过转录因子可以迅速调控成百上千个基因的表达水平,即转录因子对基因表达具有全局性影响,能够在全局范围内引发基因组的转录重排。基于此,全局转录机制工程(global transcription machinery engineering,gTME)通过易错PCR对全局转录组的关键蛋白(如TFIID组分)进行定向进化,使细胞能在转录水平上产生全基因组规模的多样性。Alper等[14]将TATA结合蛋白SPT15和TATA结合蛋白相关因子TAF25进行突变,最佳突变体(SPT15含有3个氨基酸突变)的乙醇生产率提高了70%。此外,gTME结合转录组分析(如RNAseq)可以深入了解基因型与表型的相关性,对菌株的进一步改良具有很强的指导价值。
1.1.5 重组酶介导的基因组进化
重组酶催化短同源区域(30~40 bp)的DNA片段交换,可以实现DNA片段的缺失、重复、移位和反转等基因组结构变异,是基因组进化的重要驱动力之一。Cre(来自噬菌体P1)和FLP(来自酿酒酵母的2μ质粒)分别识别loxP位点和FRT位点,是目前普遍使用的两种重组酶[15-16]。Dymond等[17]利用Cre介导的同源重组建立了一种基于染色体重排和修饰的进化方法——SCRaMbLE(synthetic chromosome recombination and modification by LoxP-mediated evolution)。首先合成酵母染色体时在每个非必需基因后添加loxP位点,通过表达Cre重组酶实现多个loxP位点之间的随机重组,以创建随机的遗传多样性文库。利用SCRaMbLE技术,经过5个迭代循环和筛选,类胡萝卜素的产量提高了38.8倍[18]。
1.1.6 实验室适应性进化与逆向代谢工程
实验室适应性进化是指在实验室中通过施加人为干扰及控制微生物生长环境推动生物体生长和繁殖的进化过程。适应性进化可以在分批培养或连续培养中进行,已广泛应用于野生型和工程酵母菌株的系统性改造。例如,适应性进化可以提高纤维素中的纤维二糖[19]和木糖[20]以及褐藻中的甘露醇和4-脱氧-L-赤式-5-己酮糖糖醛酸(DEHU)等可再生糖的利用率[21];也可用于提高酿酒酵母对高浓度乙醇[22]、有机酸[23-24]、松柏醛[25]和高温[26]的耐受性。
由于高通量测序和基因组编辑技术的发展,基于全基因组突变分析的逆向代谢工程成为研究热点[27]。Nielsen课题组[26]通 过 适应性进化筛选得到在培养温度≥40℃时细胞生长和乙醇产量提高的酵母菌株,并通过全基因组测序、全基因组基因表达和代谢流分析发现,甾醇组成变化(ERG3基因突变)是提高酵母耐热性的分子机制。Jin课题组[28]通过构建全基因组突变文库,在高浓度乙醇条件下筛选得到与乙醇耐受性 相 关 的4个 基 因MSN2、DOG1、HAL1和INO1,过表达这4个基因均可显著提高酿酒酵母对乙醇的耐受性。
1.2 可示踪的基因组进化
与随机突变不同,靶向基因组编辑可以通过可追踪的方式进行全面的大规模基因组修饰。结合CRISPR/Cas诱导的双链断裂和同源重组修复(HDR),可以实现酵母的高效基因组编辑。另外,寡核苷酸芯片和二代测序技术的发展,不仅能够快速创建大规模基因组突变文库,还能够快速和精确地定位引入的遗传修饰,确定基因型-表型关系及其分子机制。本节将介绍近年来基于酿酒酵母基因组水平的半理性进化技术的发展,包括酵母寡核苷酸介导的基因组工程(YOGE)、更加精确高效的寡核苷酸整合技术eMAGE、RNAi辅助基因组进化方法(RAGE)及基于CRISPR/Cas的可追踪的基因组进化方法CHAnGE、MAGIC和MAGESTIC技术(表1)。
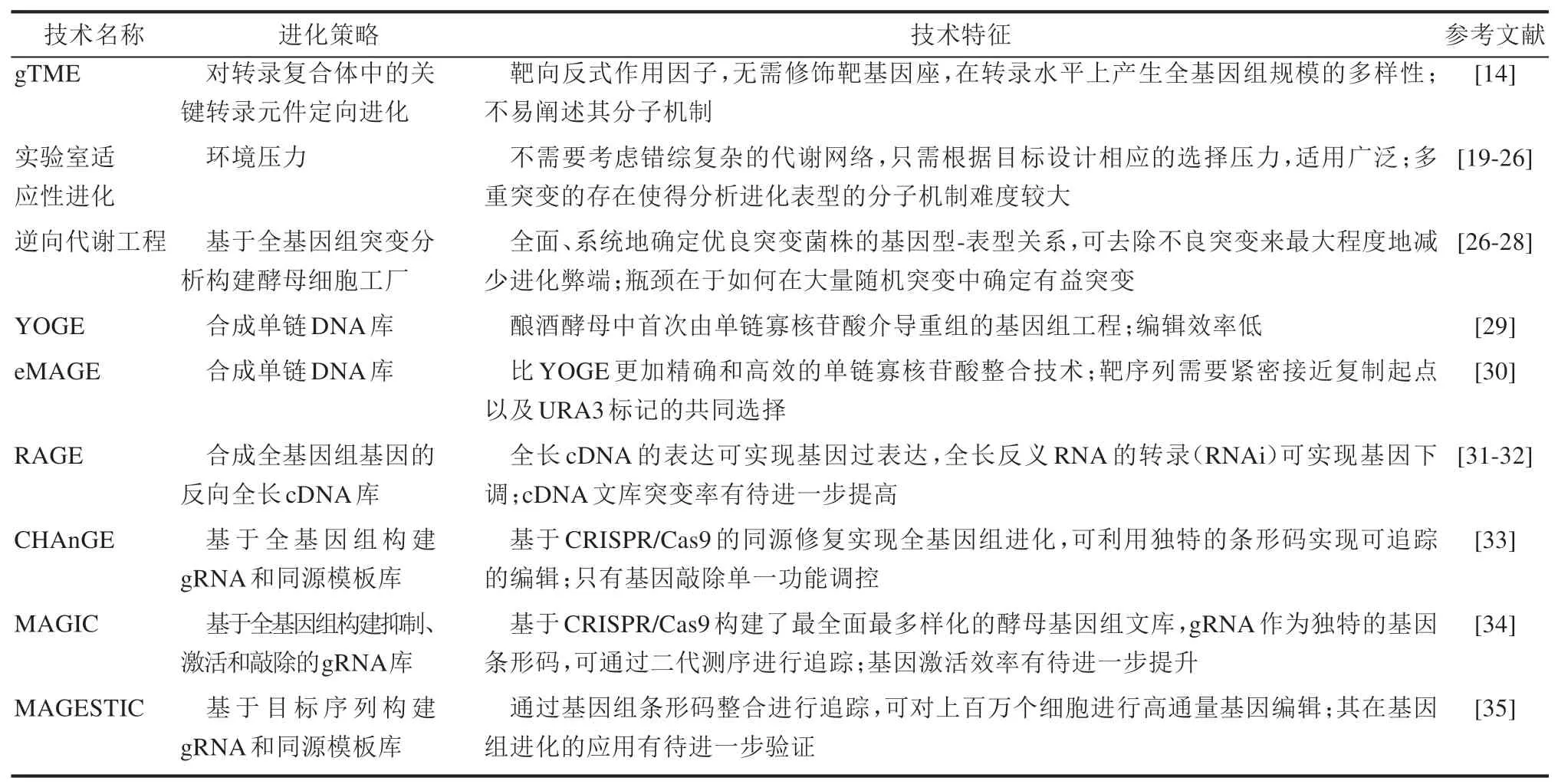
表1 酿酒酵母基因组进化策略Tab.1 Genome evolution strategies for S.cerevisiae
1.2.1 YOGE(yeast oligo-mediated genome engineering)
酿酒酵母因其高效的同源重组机制而具有强大的基因编辑能力,但通常需要较长的同源片段,随着同源臂长度的降低,编辑效率也会显著下降。因此,利用较短的单链寡核苷酸片段在酵母中实现高效的基因编辑成为研究热点。尽管在一些微生物中已能利用寡核苷酸介导的基因重组对基因组进行定点突变[36-37],但在酵母中的低重组效率限制其在酵母基因组进化中的应用。Church课题组[29]成功开发了一种在酿酒酵母中由单链寡核苷酸介导重组的方法,称为酵母寡核苷酸介导的基因组工程(YOGE)。通过敲除酵母中错配修复相关基因MLH1和MSH2,过表达DNA重组酶突变体Rad51(K342E)和Rad54蛋白,并通过对寡核苷酸长度、寡核苷酸加入量以及转化效率优化后,在3种不同的酿酒酵母(VL6-48、CEN.PK113-7D和VTT C-68059)中成功对基因组进行了修饰。并且YOGE可以进行迭代操作,每轮可产生105个细胞的基因组文库,对基因组进行多重修复。但是与大肠杆菌(超过30%)相比,YOGE的等位基因替换频率只提高到约0.2%~2%,寡核苷酸片段整合效率仍然较低,在酿酒酵母基因组进化中的应用比较有限。
1.2.2 eMAGE(eukaryotic multiplex automated genome engineering)
鉴于YOGE的等位基因替换频率较低,Isaacs教授[30]构建了一种更加精确和高效的寡核苷酸整合技术eMAGE。与YOGE不同的是,eMAGE并不依赖DNA重组酶介导的同源重组,而是在DNA复制起始阶段形成复制叉后,由ssDNA退火蛋白(SSAP)介导在后随链上退火合成寡核苷酸,并且利用羟基脲(HU)减慢复制叉速度进一步提高等位基因替换频率。研究表明,靶序列与复制起点的紧密接近以及URA3标记的共选择可以有效提高eMAGE的编辑效率。最后,通过寡核苷酸长度或浓度等参数优化,一次转化后能整合12个寡核苷酸片段,实现60个位点靶向突变。将一个复杂的寡核苷酸池进行迭代转化,可以快速产生>105的基因组多样性组合。与YOGE相比,eMAGE提高了编辑效率,但这需要依赖于靶序列与复制起点的紧密接近以及URA3标记的共同选择,在全基因组规模进化的应用还有待进一步研究。
1.2.3 RAGE(RNAi-assisted genome evolution)
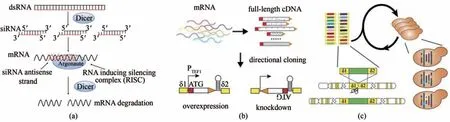
图2 基于RNAi的酿酒酵母自动化多重基因组进化技术[31-32](a)酵母RNAi工作原理,核酸内切酶Dicer切割双链RNA(dsRNA)生成短干扰RNAs(siRNA),siRNA与效应蛋白Argonaute形成RNA诱导的沉默复合体RISC,siRNA反义链与靶标mRNA结合,指导Dicer干扰靶标基因的转录;(b)基于RNAi机制构建全基因组规模调控文库,在组成型启动子下定向克隆全长的cDNA文库,有义和反义构型分别引起目标基因的过表达或敲降;(c)基于RNAi的多重基因组突变方法,编码各种遗传修饰的基因调节部分的侧翼是同源δ序列,用于迭代和多重整合到重复的基因组序列。为了使CRISPR/Cas能进行高效且无需选择的δ整合,将Cas9表达盒整合到含有RNAi机制的酵母菌株中Fig.2 Scheme of automated RNAi-assisted genome evolution in yeast(a)RNAi mechanism in yeast.The double-stranded RNA(dsRNA)is digested by the endonuclease Dicer into short interference RNA(siRNA),which binds to the effector protein Argonaute to form the RNA-induced silencing complex(RISC).The non-sense strand of siRNA binds to the target mRNA,leading to the degradation and interference of the transcription of the target gene.(b)Construction of a genome-wide modulation part library in the yeast strain with the reconstituted RNAi machinery.Full-length cDNA library was directionally cloned under the control of a constitutive promoter.The sense and anti-sense configurations resulted in genetic overexpression and knockdown,respectively.(c)RNAi-assisted multiplex genomic mutations in yeast.Gene modulation parts were flanked by homologous arms for iterative and multiplexδintegration into the repetitive genomic sequences.To enable efficient and selection-freeδintegration,a Cas9 expression cassette was integrated into the RNAi harboring yeast strain
RNA干 扰(RNA interference,RNAi)是 一种由双链RNA引发的基因沉默途径,存在于多种真核生物中,但酿酒酵母缺乏天然的RNAi机制。有研究报道,通过异源表达芽殖酵母的RNAi相关蛋白Ago1和Dcr1,在酿酒酵母中成功重构了RNAi途径[38],这使得能够利用RNAi筛选来快速了解和改造工程酵母中的复杂表型[图2(a)]。随后,Si等[31]开发了酿酒酵母中RNAi辅助的基因组进化方法RAGE。在CEN.PK2-1c中引入RNAi途径后,根据酵母基因组DNA片段构建了双链RNA库,并利用该RNA库筛选得到YKU70(与端粒酶功能缺失相关的基因)突变的两个已知和三个未知的抑制基因。同时经过3轮迭代的RNAi筛选,对3个基因PTC6、YPRP84W和tRNAVal(AAC)同 时 敲 降 后,显著改善了酿酒酵母对乙酸的耐受性。Xiao等[39]利用RNAi全基因组进化,发现通过下调SIZ1基因的表达水平,可以显著提高酿酒酵母的糠醛耐受性。在RNAi基因组进化的基础上,Si等[32]又开发了酿酒酵母自动化多重基因组进化技术。根据酵母基因组构建包括基因过表达和基因下调的全长cDNA文库,其中基因过表达调控通过全长cDNA的表达实现,基因下调调控通过全长反义RNA的转录(RNAi)实现[图2(b)]。在CRISPR/Cas的帮助下,通过δ整合的方式将同源供体(donor)整合到酵母基因组上[图2(c)],并利用自动化平台进行多轮迭代基因组进化提高酿酒酵母乙酸耐受性,最后筛选到的菌株在1.1%乙酸浓度下,4 d内可将20 g/L葡萄糖转化为5.9 g/L乙醇,且整个筛选过程在一个月内就能完成。
1.2.4 CHAnGE(CRISPR/Cas9-and homology-directedrepair-assisted genome-scale engineering)
2014年,Bao等[40]构建了HI-CRISPR(homologyintegrated CRISPR-Cas)系统。该系统在质粒pCRCT上同时表达酿脓链球菌Cas9突变体(iCas9),整合同源臂的crRNA和tracrRNA,其中同源臂删去了靶标序列8 bp,造成靶标基因移码而丧失功能。利用HI-CRISPR,在酿酒酵母中同时敲除3个基因,最高效率可达100%。在此基础上,他们又提出一种CRISPR/Cas9和同源定向修复(HDR)辅助的全基因组进化方法CHAnGE[33],可以实现对酿酒酵母基因组精确且可追踪的编辑。首先他们在单个寡核苷酸上同时编码guide RNA(gRNA)和同源重组模板(删除靶标序列8 bp),靶向酵母基因组6459个开放阅读框(ORFs)构建不同的CHAnGE cassette,其中每个ORF又设计了4个不同的gRNA。将这些寡核苷酸在芯片上合成,并组装到质粒pCRCT上构建CHAnGE质粒库,再转入酿酒酵母细胞后可进行全基因组进化。而且每个CHAnGE cassette上带有独特的DNA条形码,利用二代测序可以对进化后的突变体进行追踪。该方法能使超过98%的靶序列被有效编辑,平均效率为82%。利用CHAnGE方法对酿酒酵母全基因组一轮进化后,在5 mmol/L糠醛浓度下筛选出与糠醛耐受性相关的3个靶点SIZ1、SAP30和UBC。在SIZ1敲除菌基础上进行第2轮进化,在10 mmol/L糠醛浓度下筛选到能提高糠醛耐受性的靶点LCB3,但该靶点需要SIZ1的协同作用,单独敲除对提高糠醛耐受性没有明显效果。同时,CHAnGE也增强了酵母的乙酸耐受性,与野生型相比提高了20倍。
1.2.5 MAGIC(multi-functional genome-wide CRISPR system)
利用CRISPR/Cas技术可以实现基因敲除,而在核酸酶失活的Cas蛋白(dCas)上融合表达激活域或抑制域可以实现转录激活和抑制[41]。2017年,Lian等[42]开发了三功能CRISPR体系(CRISPRAID),可以在一个细胞内同时进行转录激活、转录抑制以及基因敲除。其中dLbCpf1-VP用于转录激活(CRISPRa),dSpCas9-RD1152用于转录抑制(CRISPRi),SaCas9用于基因敲除(CRISPRd)。利用CRISPR-AID和寡核苷酸芯片技术,Lian等[34]又发展了多功能全基因组进化技术MAGIC(图3),构建了最全面最多样化的酵母基因组文库。根据酵母基因组对基因敲除、转录激活和转录抑制分别设计不同的gRNA,构建基因敲除、转录激活和转录抑制3个质粒库,将这3个质粒库同时转入酿酒酵母中,通过高通量筛选实现基因组进化,而gRNA可以作为独特的基因条形码,可通过二代测序进行追踪。利用MAGIC技术,他们鉴定了复杂表型(糠醛耐受性和蛋白表面展示)一些之前未知的遗传决定因素。比如在5 mmol/L糠醛浓度下除了筛选到和糠醛耐受性相关的已知靶点SIZ1和SAP30,还筛选到了之前未发现的新的靶点SLX5、NUP133、GPI17和UME1。在SIZ1基因表达水平下调的菌株基础上进行第2轮进化,一些与线粒体功能相关的靶点MRPL32、NAT1等被发现可以提高糠醛耐受性,但这些靶点依赖SIZ1基因的下调。第3轮进化在SIZ1基因表达水平下调及NAT1基因表达水平激活的菌株上进行,发现PDR1基因表达水平的下调可以进一步提高糠醛耐受性。最后经过3轮进化的酵母菌能在17.5 mmol/L糠醛浓度下,两天内消耗掉大部分的葡萄糖(初始浓度20 g/L),且乙醇的最终浓度与对照菌株相当,说明该工程酵母菌株的中心代谢并没有明显改变。
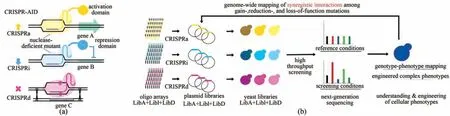
图3 MAGIC用于全基因组基因型-表型关系研究[34,42](a)使用三种正交的CRISPR蛋白开发CRISPR-AID体系,其中CRISPRa由核酸酶缺陷的CRISPR蛋白与激活域融合(dLbCpf1-VP)实现转录激活,CRISPRi由核酸酶缺陷的突变体与抑制域融合(dSpCas9-RD1152)实现转录抑制,CRISPRd由具有催化活性的CRISPR蛋白(SaCas9)实现基因敲除;(b)通过DNA芯片寡核苷酸阵列的形式合成用于基因组规模激活(橙色)、干扰(浅蓝色)和缺失(品红色)的gRNA文库,并克隆到相应的gRNA表达质粒中。通过将质粒文库转入CRISPR-AID整合的酵母菌株中构建MAGIC文库,通过高通量筛选或者生长富集,并结合二代测序分析富集的gRNA序列。MAGIC可用于更好地理解、设计和改造复杂表型Fig.3 MAGIC for genome-wide mapping genotype-phenotype relationships[34,42](a)Development of CRISPR-AID using three orthogonal CRISPR proteins,a nuclease-deficient CRISPR protein fused with an activation domain(dLbCpf1-VP)for CRISPRa,a nuclease-deficient mutant fused with a repression domain(dSpCas9-RD1152)for CRISPRi,and a catalytically active CRISPR protein(SaCas9)for CRISPRd.(b)Guide sequences for genome-wide activation(orange),interference(light blue),and deletion(magenta)were synthesized as arrayed oligos on DNA chip and cloned into the corresponding gRNA expression plasmids.The transformation of the pooled plasmid libraries into the CRISPR-AID integrated yeast strain resulted in the construction of the MAGIC library.The MAGIC library was subject to growth enrichment or high throughput screening,and the corresponding enrichment or depletion of guide sequences were profiled using next-generation sequencing.MAGIC can be employed to better understand and engineer complex phenotypes
1.2.6 MAGESTIC(multiplexed accurate genome editing with short,trackable,integrated cellular barcodes)
Steinmetz课题组[35]在CRISPR/Cas9技术的基础上开发了MAGESTIC技术,利用该技术可以在酿酒酵母体内实现多重精确的、可追踪的、基因组整合条形码的基因组编辑技术。利用寡核苷酸芯片技术合成设计含gRNA和同源片段的寡核苷酸,质粒组装后携带有一个短的、独特的31mer DNA条形码。同时,质粒上表达另一个gRNA(guide X),可以同时靶向该质粒和酵母基因组基因FCY1两侧,使质粒线性化并将条形码整合到FCY1位点。通过基因组条形码整合,可防止质粒条形码不稳定而丢失的现象,并实现稳定的表型分型。而设计的gRNA可以指导靶标位点的准确编辑,可以对整个基因组进行编辑。研究表明,LexA-Fkh1p蛋白融合表达能招募更多供体DNA至DNA双链断裂位点,编辑效率可提高5倍以上。利用MAGESTIC技术对必需基因SEC14进行了饱和编辑并鉴定出对抑制脂质信号传导至关重要的氨基酸Y111和Y122,生成了Sec14p 102~137区域的氨基酸与抑制剂相互作用的完整功能图。通过gRNA及同源模板的设计,MAGESTIC技术可以对上百万个细胞进行高通量基因编辑。
2 酿酒酵母基因组进化工程的实际应用
基因组进化已在不同领域得到广泛应用,比如拓宽底物的利用范围、增强细胞的耐受性、提高生产速率和产物浓度等等。本节将讨论近年来在基因组水平上使用定向进化方法改善酿酒酵母细胞工厂的示例。
2.1 拓宽底物利用范围
出于环境和能源安全的考虑,人们对利用可再生原料(如木质纤维素和大型藻类)生产燃料和化学品的工程微生物越来越感兴趣。由于底物的利用可以很容易地与细胞生长相耦合,便于建立高通量筛选方法,因此基因组进化已被广泛地用于提高酿酒酵母对不同底物的利用能力和利用效率。
作为地球上最丰富的生物材料,木质纤维素生物质是可再生燃料和化学品生产中最有希望的原料之一。目前己糖(如葡萄糖)可以被大多数微生物有效发酵,而戊糖(主要是木糖和阿拉伯糖,占总碳水化合物的30%以上)的利用存在很多技术瓶颈,其利用效率较六碳糖低得多。基因组进化使构建高效的木糖发酵酵母菌株成为可能,Kim等[20]在含木糖的培养基中进行了适应性进化,进化菌株的全基因组测序结果以及后续研究表明,PHO13的功能缺失在有效利用木糖方面起着主导作用。
尽管利用基因组进化策略已经成功构建高效利用木糖的酵母菌株,但葡萄糖抑制现象的存在导致发酵生产率较低,因此希望设计一种能够同时高效利用所有碳源以提高发酵生产率的平台菌株。在之前的研究中,通过引入纤维糊精转运蛋白和β-葡萄糖苷酶实现纤维二糖的胞内水解被证明是缓解葡萄糖阻抑的有效策略[43],因为逐渐释放的葡萄糖被迅速消耗,使得酵母菌株可以同时消耗纤维二糖和木糖。同时,纤维素水解产物中普遍存在高浓度乙酸,由于其对酿酒酵母有细胞毒性而大大降低了糖化合物的生物转化率。有研究发现引入乙酸盐还原途径能平衡细胞氧化还原电位,实现原位解毒,使得木糖和乙酸盐可以同时转化为乙醇,获得更高的产率[44]。最后,Wei等[45]将这两种策略结合起来,在适应性进化得到的木糖发酵酵母基础上,成功构建了一株可以同时高效利用纤维二糖、木糖和乙酸盐的酿酒酵母菌株(图4)。
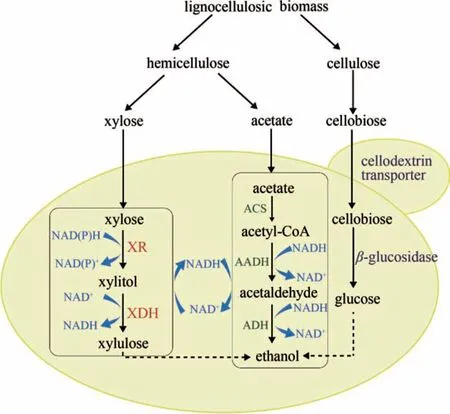
图4 同时利用木质纤维素碳源,如纤维二糖、木糖和乙酸的酵母菌株的构建XR—木糖还原酶;XDH—木糖醇脱氢酶;ACS—乙酰辅酶A合成酶;AADH—乙酰化乙醛脱氢酶;ADH—醇脱氢酶Fig.4 Construction of a recombinant yeast strain for simultaneous utilization of lignocellulosic carbons,such as cellobiose,xylose,and acetateXR—xylose reductase;XDH—xylitol dehydrogenase;ACS—acetyl-CoA synthetase;AADH—acetylating acetaldehyde dehydrogenase;ADH—alcohol dehydrogenase
2.2 提高产物合成水平
以高浓度和高产率形成所需产物是大多数生物技术过程的最终目标。但是在许多情况下,产物的形成不易与细胞生长耦联,甚至损害细胞生长。这意味着普遍适用于提高产量的高通量筛选方法并不容易获得,因此可以将基因组进化和代谢工程结合以提高产量和生产率。
在某些情况下,可以利用产物的特性来开发与细胞生长耦联相关的基因组进化策略。例如,利用类胡萝卜素的抗氧化特性,以过氧化氢作为选择压力,通过基因组进化最终使类胡萝卜素的产量增加了3倍(从6 mg/g干细胞重量增加到18 mg/g干细胞重量)[46]。与此类似,采用丙烯醛作为选择压力,通过基因组进化可以提高进化菌株中谷胱甘肽的积累水平,与出发菌株相比提高了3.3倍,达到了接近6%细胞干重的谷胱甘肽含量[47]。
由于酿酒酵母存在葡萄糖固有的发酵代谢,阻碍了除乙醇以外的其他化学品的生产。近年来,微生物脂肪酸生物合成因其在生产油性化学品和生物燃料中的潜在用途而备受关注。其中,游离脂肪酸(FFA)是制造洗涤剂、润滑剂、化妆品和药物成分的理想原料。Yu等[48]通过模拟产油酵母的代谢途径对酿酒酵母进行了大规模的代谢重编程,最佳菌株的FFA浓度为33.4 g/L,是微生物发酵中目前报道的最高水平。又通过实验室进化进一步消除了FFA高产菌株中的乙醇发酵,从而成功将酿酒酵母进化为产油酵母(图5)。基因组测序和代谢特征表明丙酮酸激酶突变对于平衡糖酵解和细胞生长至关重要。最近,Zhu等[49]通过在蛋白质/途径/基因组水平上对酵母进行多维工程设计,大大提高了中链脂肪酸(MCFA)的产量,是最经典的代谢工程与进化工程相结合的实例之一。他们首先对FAS酶进行工程改造,然后通过定向进化得到了Tpo1 M49突变体和ZWE03抗性菌株,二者分别赋予了细胞对C10和C8脂肪酸的耐受性,最后,选用高耐受性菌株ZWE03进行一系列的代谢途径改造,最终进化菌株ZWE37的MCFA产量为1.39 g/L,与出发菌株相比,其浓度提高了250倍以上。
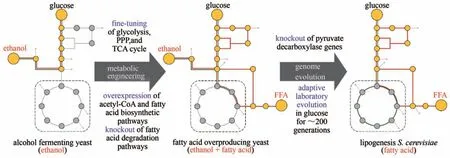
图5 高效生产FFA酵母菌株的构建[48](野生型酿酒酵母中,中心碳代谢是“葡萄糖转化为乙醇”;高产脂肪酸酿酒酵母中,为了实现“葡萄糖转化为油”的代谢模型,建立了FFA生产的有效途径;产油酿酒酵母中,通过适应性进化,酒精发酵成功地重新编程为纯脂肪酸合成)PPP—磷酸戊糖途径;TCA—三羧酸循环;FFA—游离脂肪酸Fig.5 Construction of yeast cell factories for an efficient production of FFA[48](In wild-type yeast,the major carbon metabolism is to drive ethanol fermentation from glucose.In the fatty acid-overproducing yeast,metabolic engineering strategies were employed to establish efficient biosynthetic pathway from glucose to fatty acid.In the lipogenesis yeast,genome evolution was performed to completely reprogram the cellular metabolism from alcohol fermentation to fatty acid biosynthesis)PPP—pentose phosphate pathway;TCA—tricarboxylic acid;FFA—free fatty acid
2.3 改善细胞属性
由于工业生物技术过程中生产条件非常苛刻,所以需要创建能够抵抗多种压力的微生物细胞工厂。抗逆性的分子机制十分复杂,难以通过纯理性设计来构建抗逆性菌株,因而基因组进化是个更为有效的策略。目前,基因组进化已经广泛用于提高工业酿酒酵母菌株的乙醇[50-51]、长链烷烃[52]以及喷气燃料的耐受性[53]。
基于CRISPR的基因组进化技术可以在不同菌株背景下构建全基因组突变体,与传统的基因组规模工程策略相比,其针对性更强、突变范围更广。以糠醛耐受性为例,RAGE通过敲降或缺失SIZ1基因可显著提高糠醛耐受性[39],但是用5 mmol/L糠醛进行一轮筛选后未能鉴定出其他靶标;CHAnGE将SIZ1、SAP30、UBC和LCB3鉴定为糠醛耐受性的重要基因[33],但是在10 mmol/L糠醛中进行两轮筛选后无法获得其他更多的靶标;而MAGIC在15 mmol/L糠醛中进行第3轮筛选后仍能继续鉴定其他与糠醛耐受性相关的遗传决定因素[34]。同样地,在10 mmol/L糠醛中进行两轮进化筛选,MAGIC工程菌株(SIZ1i-NAT1a)的性能要优于CHAnGE工程菌株(SIZ1d-LCB3d)。这也表明,MAGIC不仅确定了更多的糠醛耐受性遗传决定因素,而且能构建更多的糠醛耐受性菌株。
除了在工业生物技术过程中耐受性的应用外,基因组进化还可用于细胞营养需求相关的改造。生物素在所有生物中都起着重要作用,但并非所有生物都能合成这种维生素。因此,通常将这种昂贵的维生素添加到培养基中,这极大增加了生产成本。有研究证明在酿酒酵母CEN.PK113-7D中,通过实验室适应性进化实现了快速的非生物素依赖性生长[54]。原始的酿酒酵母菌株在没有生物素添加的情况下,其生长非常缓慢(µ<0.01 h-1),通过实验室适应性进化在无生物素的培养基中获得了生长速率提高了32倍的突变酵母菌株。全基因组重测序结果表明,BIO1基因的扩增(40倍)以及膜转运蛋白基因TPO1和PDR12的突变是提高非生物素依赖生长速率的决定性因素。进化菌株可以在廉价的培养基上快速生长,有利于降低生产成本、工艺复杂性和发酵过程中的污染风险。
3 总结与展望
基于CRISPR系统等最新基因组编辑技术的全基因组规模进化工程(如CHAnGE和MAGIC)克服了许多传统菌株改良技术的局限,大大提高了对生物系统进行重新编程的能力。由于具有高通量、高覆盖率和低成本等特点,这些新型基因组编辑技术能够快速生成庞大的组合突变体文库。因此,目前在大多数基因组进化实验中,最大的难点在于如何开发高通量筛选方法,从数百万种可能性中寻找最佳突变体。
当前,基因组进化的成功实例主要限于生长相关的表型,如底物利用以及对有毒化合物的耐受性。为了拓展全基因组进化的应用范围,基于转 录 因 子(TFs)[55-56]、G蛋 白 偶 联 受 体(GPCRs)[57]、核糖体开关[58]等的生物传感器已被整合到全基因组进化流程中。例如,基于FapR[55]的丙二酰辅酶A生物传感器能简单、快速地监测细胞内丙二酰辅酶A的浓度,将其与全基因组cDNA过表达文库结合使用,获得了细胞内丙二酰辅酶A水平显著提高的突变菌株;基于XyIR[56]的生物传感器用于筛选木糖转运蛋白突变体,获得了木糖转运能力提高6.5倍的HXT14突变体。此外,通过将产物形成与基本的细胞过程相结合,生物传感器可以进一步发展为基因组规模的代谢工程和进化工程的合成选择电路。例如,利用芳香族氨基酸响应性启动子驱动遗传霉素抗性基因的表达,可用于筛选高产黏糠酸的酵母突变菌株[59]。
近年来,微流控系统[60-62]和机器人平台[51,63]已经成为高通量筛选目标表型的有效手段。使用微流控液滴(microfluidic droplets)[60]进行超高通量筛选可大大提高基因组进化的效率。最新的µSCALE[61](微毛细管单细胞分析和荧光提取)利用荧光测定法在微毛细管阵列内快速回收活的阳性细胞,从而实现大规模的并行筛选。此外,实验室自动化可以极大加速微生物基因组规模的工程设计,通过消除人工干预,有望提高生产率和可靠性。
总之,基因组进化是生物系统工程化改造的强大工具,高通量筛选和自动化平台的开发将加快基因组进化过程。另一方面,基因组进化与先进计算机设计和人工智能的集成将大大推动基因组规模进化工程的发展,进而促进基础生物学和生物技术的系统性研究。

