转录组学分析意大利蜜蜂脑部哺育行为相关基因
高艳,朱雅楠,李秋方,苏松坤,聂红毅
转录组学分析意大利蜜蜂脑部哺育行为相关基因
高艳,朱雅楠,李秋方,苏松坤,聂红毅
(福建农林大学动物科学学院(蜂学学院),福州 350002)
【目的】意大利蜜蜂()哺育行为在维护蜂群稳定和生产蜂王浆方面发挥重要作用。本研究通过人工组建蜂群获得相同日龄的哺育蜂和采集蜂,筛选出排除日龄因素干扰后哺育蜂脑部与哺育行为密切相关的差异表达基因,揭示哺育蜂脑部调控哺育行为的分子网络。【方法】通过人工组建蜂群的方法获得3日龄工蜂、10日龄哺育蜂和采集蜂、21日龄哺育蜂和采集蜂,解剖各组工蜂头部获得脑组织样本,应用RNA-seq技术对5组脑部样本(3日龄工蜂、10日龄哺育蜂、10日龄采集蜂、21日龄哺育蜂、21日龄采集蜂)中基因表达量进行转录组测序的全面分析,筛选出哺育蜂脑部哺育行为密切相关的差异表达基因,并对这些差异表达基因进行GO和KEGG富集分析。同时利用实时荧光定量PCR(qPCR)对随机选取的4个差异表达基因的表达模式进行验证。【结果】RNA-seq分析筛选得到32个与哺育蜂哺育行为密切相关的差异表达基因,这些基因在10日龄哺育蜂脑中表达量均显著高于3日龄工蜂、10日龄采集蜂、21日龄采集蜂,且在21日龄哺育蜂脑中的表达量也显著高于21日龄采集蜂。GO富集分析发现上调差异表达基因主要参与氧化还原酶活性、气味结合、跨膜运输等功能。KEGG富集结果显示,上调的差异表达基因主要参与蛋白质代谢(核糖体)、能量代谢(氧化磷酸化、碳代谢、三羧酸循环、淀粉和蔗糖代谢、氮代谢、其他聚糖降解通路)、信号转导(Toll和Imd信号通路、光传导、鞘脂代谢)、消化作用(溶酶体),其中显著性富集在鞘脂代谢和其他聚糖降解通路。qPCR结果显示3个上调差异表达基因(、、)和1个下调差异表达基因()表达模式的检测结果与测序数据一致。【结论】通过对3日龄工蜂、10日龄哺育蜂、10日龄采集蜂、21日龄哺育蜂、21日龄采集蜂5组样本脑部进行全面分析,获得相同日龄哺育蜂和采集蜂脑部基因的转录组图谱,分析得到了脑部哺育行为相关的32个上调差异表达基因。哺育蜂脑部的差异表达基因主要通过信号转导和能量代谢等途径调控哺育蜂的哺育行为。
意大利蜜蜂;哺育蜂;脑部;哺育行为;蜂王浆;RNA-seq
0 引言
【研究意义】蜜蜂脑是调节其生物学各项行为的中心器官,脑部的神经系统能够调控蜜蜂表现出的各种行为响应,其中包括从哺育行为到采集行为的生命过渡过程,如分泌蜂王浆哺育幼虫、采集、识别颜色和嗅觉线索、舞蹈交流、群体防御等[1-3]。工蜂的哺育行为在蜜蜂幼虫生长发育、维护蜂群稳定等方面发挥重要作用,同时工蜂的哺育行为也受到幼虫信息素的调节[4]。蜂王浆的分泌由哺育蜂哺育行为所介导,这一哺育行为已被人工用于生产蜂王浆供人类食用。一般认为,蜂王浆由适龄工蜂的咽下腺、上颚腺共同分泌,同时哺育行为介导的蜂王浆分泌过程离不开脑部复杂的网络机制调控[5-6]。因此,研究哺育蜂脑部调控哺育行为相关的分子网络机制可为深入解析意大利蜜蜂()蜂王浆分泌的分子机理提供新的思路和线索。【前人研究进展】随着日龄的发育,工蜂从巢内工作向巢外采集活动转变,包括环境调节的行为变化[7-9],这一过程涉及大脑的结构、基因表达和蛋白质合成的变化[10];同样也涉及到脑中激素和神经化学物质的生理过程以及成千上万基因表达的变化[11],例如,其在功能和形态方面的转变会受保幼激素的控制[12]。此外,脑部中枢神经系统通过调控咽下腺和上颚腺相关基因的表达,促进这些腺体的发育并且增强它们分泌蜂王浆的功能[13]。研究表明,哺育蜂的大脑对蜂王浆分泌产生了一种独特的神经肽,哺育蜂分泌蜂王浆能力的增强与调节行为过程中高度增强的神经肽有关,并通过调节水稳态、对幼虫信息素的识别、采集能力和收集花粉来增加蜂群群体的营养供应进而提高蜂王浆的分泌,从而在提高蜂王浆产量方面发挥了作用[13]。哺育蜂脑中高量表达的基因可能参与轴突形成和细胞黏附,这些基因可能涉及到哺育蜂在转向采集活动之前的脑结构变化[7]。哺育蜂脑部磷脂酰肌醇信号转导和花生四烯酸代谢水平的提高有助于增强嗅觉对幼虫信息素刺激的反应[14]。【本研究切入点】关于脑部调控蜂王浆分泌机制的研究主要集中于不同日龄工蜂脑部磷酸化蛋白质组学和膜蛋白质组学方面,以及不同日龄普通意大利蜜蜂与高产王浆意大利蜜蜂之间的脑部蛋白质组学[2,6,10,13]。此外,借助微阵列分析技术展开不同日龄工蜂脑部基因表达变化也有相关的报道[15]。目前,尚未有排除日龄因素影响而全面开展的脑部调控蜂王浆分泌网络机制相关的研究。【拟解决的关键问题】为了排除日龄因素的干扰,人工组建蜂群,收集3日龄工蜂、10日龄哺育蜂、10日龄采集蜂、21日龄哺育蜂、21日龄采集蜂,利用RNA-seq技术全面筛选哺育蜂脑部哺育行为相关的关键差异表达基因(differentially expressed gene,DEG),为深入研究哺育蜂脑部调控蜂王浆分泌的分子机制提供参考。
1 材料与方法
1.1 供试蜂种
供试蜂群为‘蜂强1号’意大利蜜蜂蜂种,样品于2017年10月至2018年8月采自福建农林大学动物科学学院(蜂学学院)教学蜂场。
1.2 主要仪器及试剂
体视显微镜1台、冷光源仪器1台、DEPC水(上海生工生物工程股份有限公司)、SYBR® Premix Ex TaqTMII(Tli RNaseH Plus)(TaKaRa公司)、75%酒精、干冰、碎冰。
1.3 标记蜜蜂及收集样品
试验所用蜂群为正常健康的5群强群,每群蜂中至少含有2—3张即将出房的封盖子脾。在组建新的蜂群之前,去除封盖子脾上的蜜蜂,放于恒温恒湿培养箱(34.5℃,相对湿度60%)中,每隔24 h用不同颜色的记号笔在刚出房的蜜蜂胸部或腹部做好标记,连续标记5 d,每天标记刚出房工蜂数目约为2 000—3 000头。标记完后投入由一只蜂王、一张蜜粉脾、一张幼虫脾组建的人工蜂群。
标记的工蜂发育到第3天时,直接收取工蜂作为3日龄工蜂;第10、21天时,收集头部伸到有幼虫巢房且持续时间超过10 s的工蜂作为哺育蜂;第10、21天时,在巢门口收集后足花粉筐中载有花粉的外勤蜂,将其作为采集蜂。按照这个要求,收集3日龄工蜂(3 d)、10日龄哺育蜂(10 dN)、10日龄采集蜂(10 dF)、21日龄哺育蜂(21 dN)、21日龄采集蜂(21 dF)。
1.4 样品测序
参考赵元洪等[16]解剖蜜蜂大脑的方法,解剖5组样品(3 d、10 dN、10 dF、21 dN、21 dF)的脑部,每组解剖10—15只,每组均设置3个生物学重复。3 d的3个生物学重复分别为3 d_1、3 d_2、3 d_3;10 dN的3个生物学重复分别为10 dN_1、10 dN_2、10 dN_3;10 dF的3个生物学重复分别为10 dF_1、10 dF_2、10 dF_3;21 dN的3个生物学重复分别为21 dN_1、21 dN_2、21 dN_3;21 dF的3个生物学重复分别为21 dF_1、21 dF_2、21 dF_3。委托北京诺禾致源生物有限公司开展总RNA质量控制、cDNA文库构建和Illumina测序。
1.5 哺育蜂脑部哺育行为相关DEG的筛选及表达水平分析
根据FPKM(Fragments Per Kilobase of exon model per Million mapped reads)值法计算每个基因在5组样本中的表达量。利用DESeq2软件分析差异表达基因,将校正后的-adjust<0.05作为筛选DEG的标准[17],其中log2fold change (FC)>0和log2FC<0分别作为筛选上调和下调DEG的标准。
筛选与哺育行为密切相关的DEG的标准如下:DEG在10 dN脑中的表达量显著高于3 d和21 dF;DEG在10 dN脑中的表达量显著高于10 dF;DEG在21 dN脑中的表达量显著高于21 dF;DEG在10 dN与21 dN脑中的表达量无显著差异。将10 dN与3 d之间产生的DEG记为10 dN3 d组,10 dN与21 dF之间产生的DEG记为10 dN21 dF组,10 dN与10 dF之间产生的DEG记为10 dN10 dF组,21 dN与21 dF之间产生的DEG记为21 dN21dF组,10 dN与21 dN产生的DEG为10 dN21 dN组。
通过韦恩图分析,10 dN3 d与10 dN21 dF、10 dN10 dF、21 dN21 dF做交集会产生共有的DEG,这一共有DEG包括调控哺育行为及日龄发育等相关,最后为减少日龄发育对筛选的影响,从共有DEG中去除10 dN21 dN与其交集产生的DEG,最后得到的即为严格筛选条件下与哺育行为密切相关的DEG。
通过clusterProfiler R软件包和基因本体(GO)将关键DEG映射到GO数据库中。通过BLAST软件将筛选得到的DEG与KEGG数据库比对。利用诺禾Novogene售后工具平台(https://magic.novogene.com/ public/customer/main#/tool_rna/add_tool)对DEG进行KEGG pathway富集分析。
1.6 DEG的实时荧光定量PCR(qPCR)验证
在DEG中随机选取4个基因。利用Primer Premier 6设计特异性引物(表1),其中以(NM_001185146.1)作为内参基因,委托上海生工生物工程有限公司进行引物合成。反应体系:SYBR Premix Ex Taq Ⅱ(TaKaRa公司,日本)5 μL,引物(2 μmol·L-1)2 μL,模板(500 ng·μL-1)2 μL,ddH2O补充至10 μL。PCR程序分为3步:第一步为95℃预变性30 s;第二步为95℃变性5 s,60℃退火30 s,共 40个循环;最后一步为熔解曲线分析:65℃开始,每5 s上升0.5℃,直至上升到95℃。整个反应程序在荧光定量PCR仪(Bio-Rad公司,美国)上进行,按照说明书进行操作。以2-ΔΔCt法[18]计算哺育蜂脑部DEG的相对表达量。
2 结果
2.1 不同发育时期工蜂脑部测序文库的测序数据质控与评估
15个工蜂脑样品经建库和测序,有效读段数介于56 348 822—82 964 416,样本的单一匹配率均在72%以上,Q30值也都在88%以上,说明RNA-Seq数据质量较好,测序数据可靠性高(表2)。
2.2 哺育蜂哺育行为相关DEG的筛选及趋势分析
韦恩图分析发现,10 dN3 d有1 596个DEG;10 dN21 dF有578个DEG;10 dN10 dF有166个DEG;21 dN21 dF有1 315个DEG,将这4组进行交集分析得到42个DEG(图1-A),这42个DEG包括调控哺育行为及日龄发育等相关。为减少日龄对筛选与哺育行为相关DEG的干扰,去除10 dN21 dN与42个DEG交集产生的1个DEG,最后得到41个DEG(图1-B)。

表1 qPCR引物信息
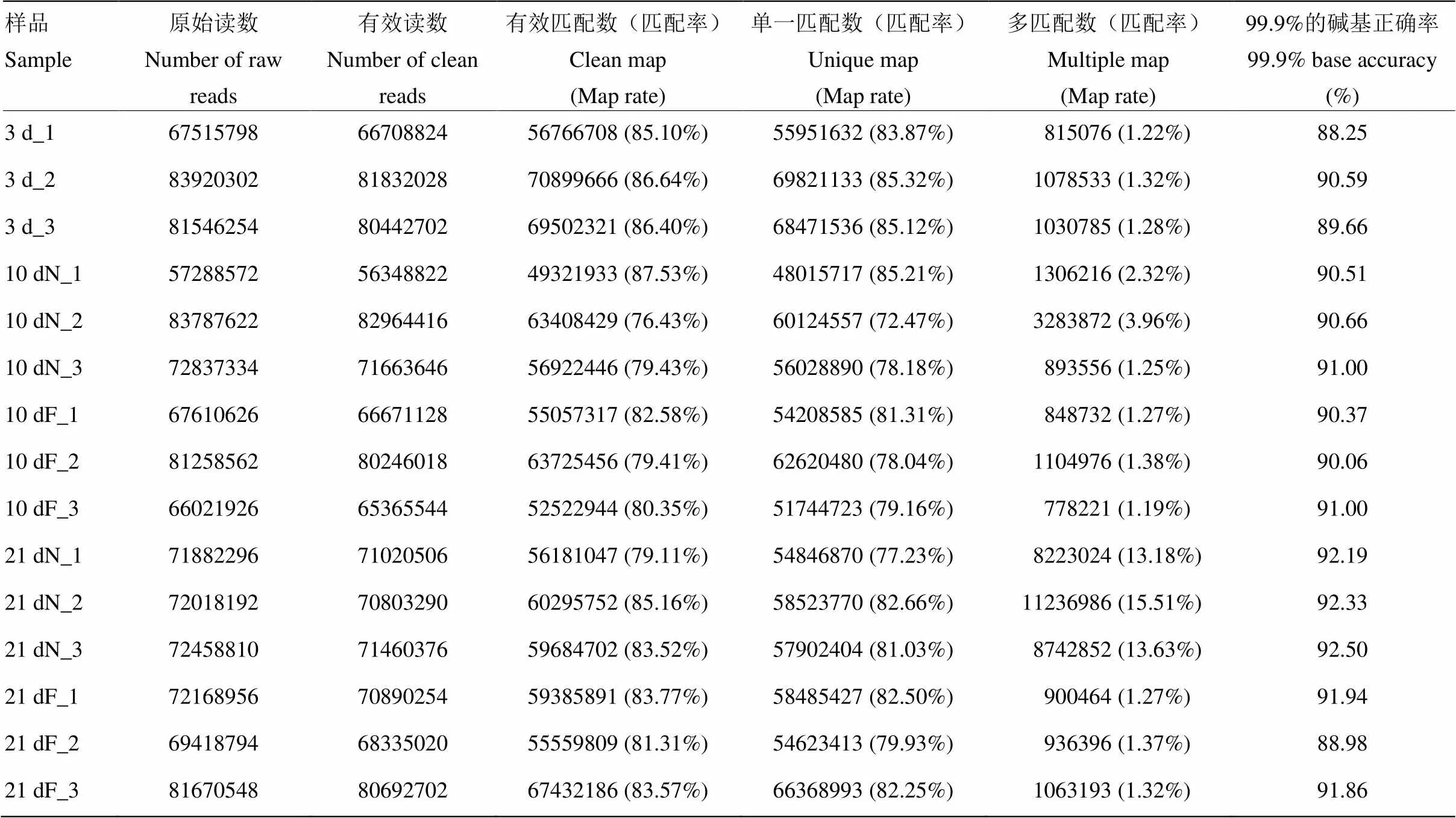
表2 RNA-seq数据总览
41个DEG的表达量热图聚类分析发现,这些DEG主要在21 dN和10 dN组表达量较高(图1-C);其中,在哺育蜂阶段均上调表达的DEG有32个,均下调表达的有6个,剩余的3个DEG除在10 dN3 d中呈上调表达外,其余都是在哺育蜂阶段呈下调表达(表3)。
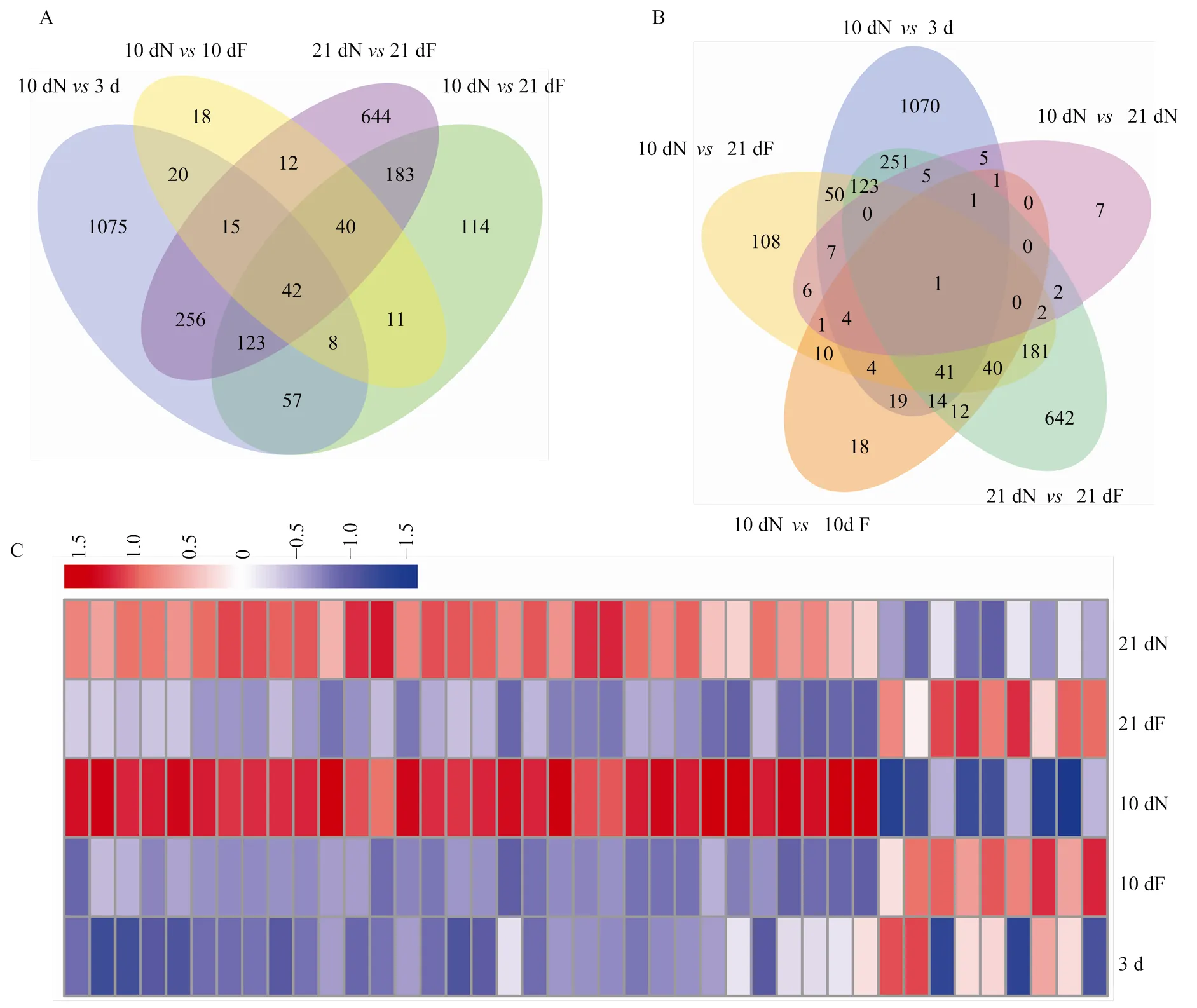
A、B:筛选哺育蜂脑部哺育行为相关DEG韦恩图 Venn diagram of selected-DEGs related to nursing behavior of nurses;C:41个DEG表达量聚类热图 41 DEGs expression clustering heat map
32个在哺育蜂阶段均上调表达的DEG为脑中与哺育行为密切相关的DEG。将32个DEG在10 dN10 dF和21 dN21 dF中按照log2FC值从高到低的顺序排列,其中差异倍数最大的为编码王浆主蛋白1(MRJP1)的基因(log2FC (10 dN10 dF)=9.44,log2FC (21 dN21 dF)=5.71),该基因在哺育蜂阶段高量表达(10 dN、21 dN脑中FPKM值分别为1 564.78和510.71),在3日龄工蜂和采集蜂阶段呈低量表达(3 d、10 dF和21 dF脑中FPKM值分别为0.40、2.27和9.64);其次为(编码毒酸磷酸酶Acph-1样)、(编码突触小泡糖蛋白2C)、(编码羧肽酶Q样)、(编码葡萄糖基神经酰胺酶4),其中在哺育蜂阶段呈高量表达(10 dN和21 dN脑中FPKM值分别为75.03、50.90),在3日龄工蜂和采集蜂阶段呈低量表达(3 d、10 dF和21 dF脑中FPKM值分别为2.83、7.81和8.63);还在上调DEG中发现编码气味结合蛋白(odorant binding protein,OBP)的3个基因、、,以及编码细胞色素P450家族之一的基因(表4)。

表3 脑部41个DEG基于日龄依赖性在5组样本中的显著性趋势
↑:显著性上调significantly up-regulated;↓:显著性下调significantly down-regulated;ns:两组样本之间无显著性差异No significant difference
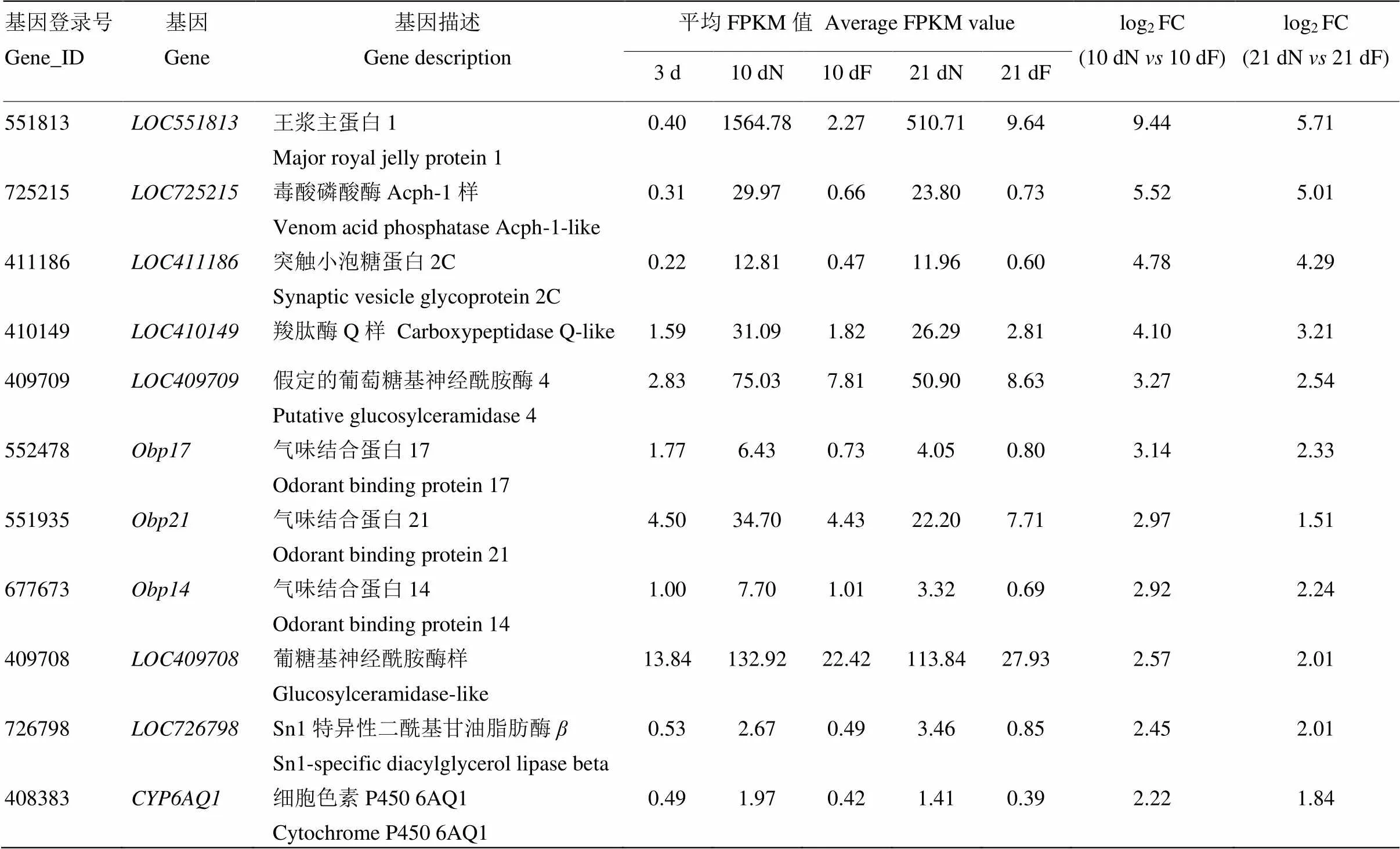
表4 脑部前11个上调DEG在5组样本中的表达信息
2.3 哺育蜂哺育行为相关DEG的调控网络分析
GO富集分析(表5)发现32个上调DEG主要参与氧化还原酶活性(4个DEG)、气味结合(3个DEG)、跨膜运输(2个DEG),GO富集中未发现有显著性富集的基因。
通过KEGG富集分析,发现上调表达的DEG(图2)主要参与蛋白质代谢(核糖体)、能量代谢(氧化磷酸化、碳代谢、三羧酸循环、淀粉和蔗糖代谢、氮代谢、其他聚糖降解通路)、信号转导(Toll和Imd信号通路、光传导、鞘脂代谢)、消化作用(溶酶体),其中显著性富集在鞘脂代谢和其他聚糖降解通路上。
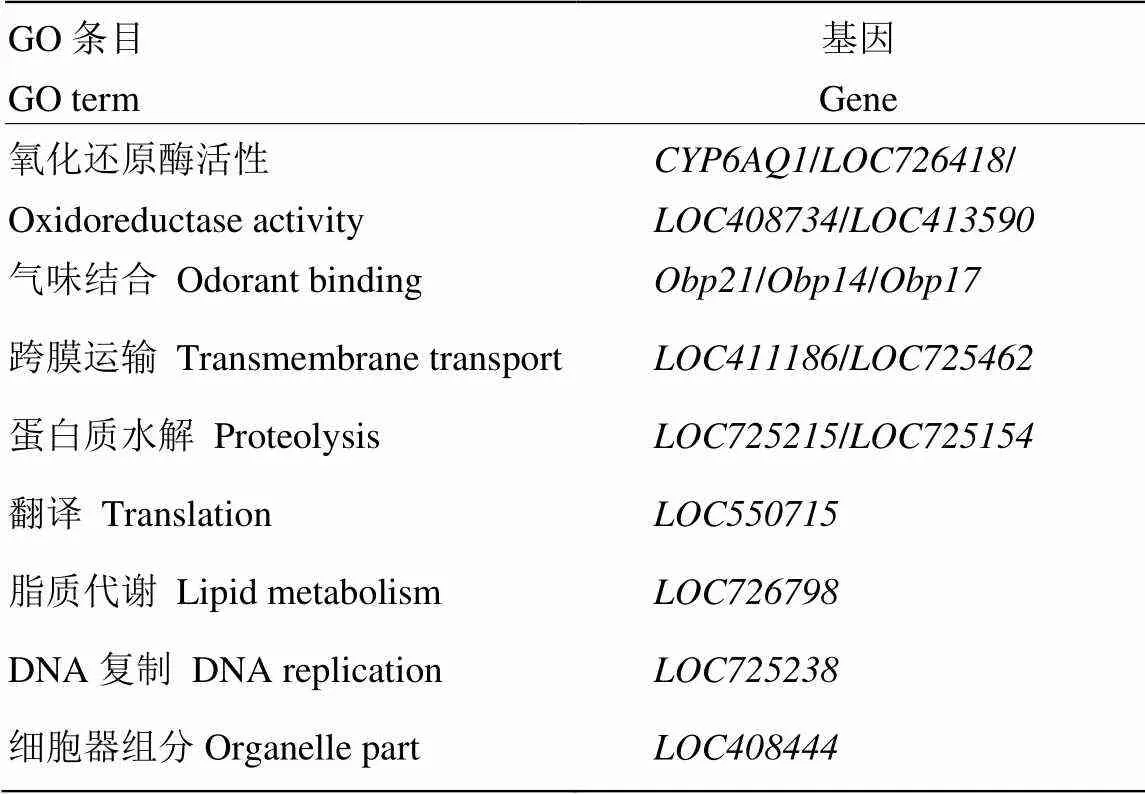
表5 上调DEG富集的GO条目
2.4 转录组中DEG的qPCR验证
为验证测序数据的准确性,从41个DEG中选取3个上调基因(、和)和1个下调基因(),结果显示这些基因表达水平的趋势变化与RNA-seq数据中的变化趋势基本一致(图3),证实了测序结果的可信性。
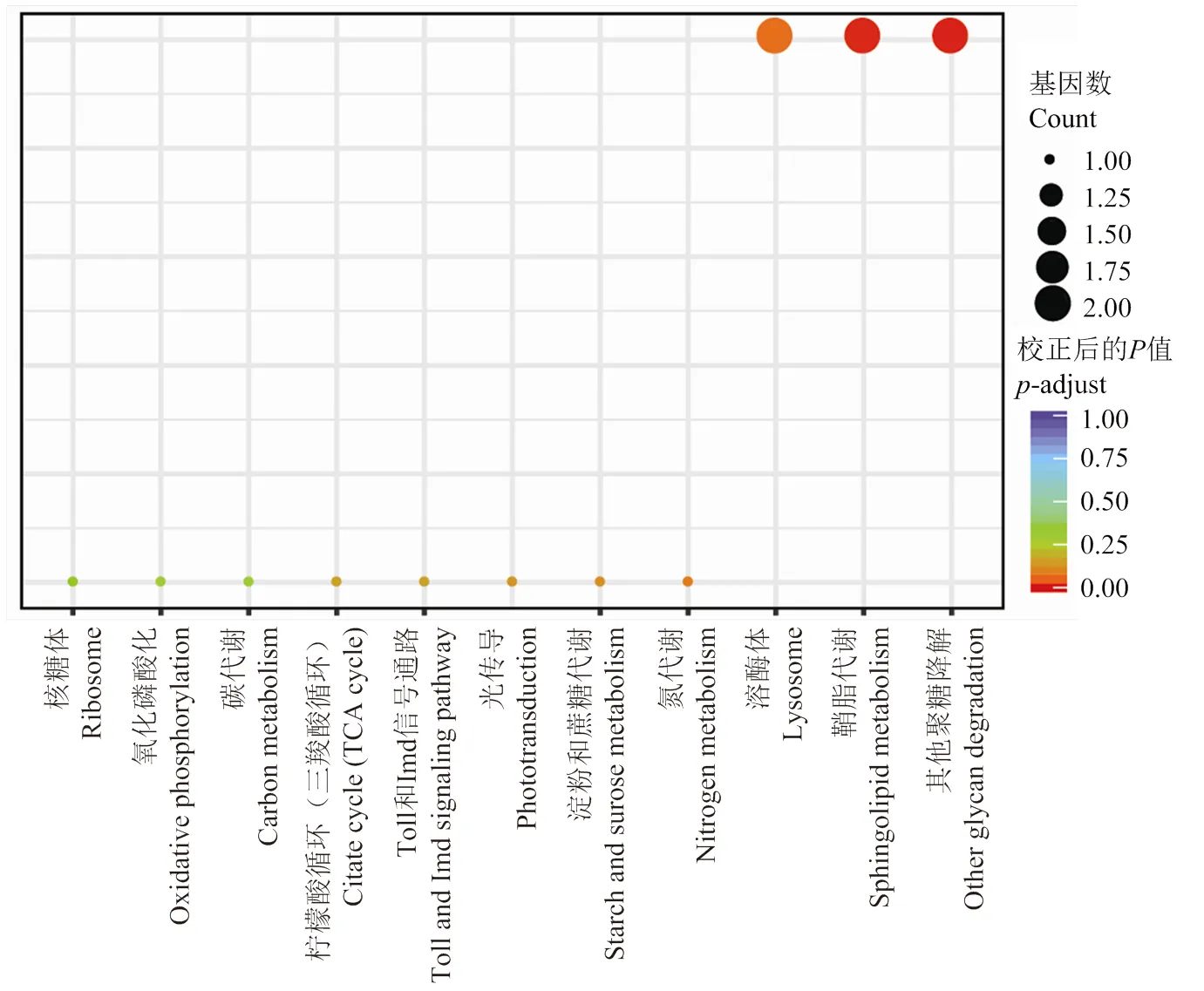
图2 哺育蜂哺育行为相关DEG的KEGG功能富集分析

黑色柱状图表示RNA-seq数据中基因的FPKM值,灰色表示qPCR数据中基因的相对表达量,纵坐标均为相应取过以2为底的对数值The black bar graph represents the FPKM value of the gene in the RNA-seq data, and the gray represents the relative expression of genes in the qPCR data; the ordinates are the corresponding logarithmic values with a base of 2。图中数据为平均值±标准误,采用单因素方差分析 Data in the figure are the mean±SE, One-way Anova. *P<0.05; **P<0.01; ***P<0.001
3 讨论
蜜蜂脑部的研究大多基于脑细胞的化学功能、结构、内分泌活动以及脑部基因和蛋白质表达的时态变化,如保幼激素、生物胺、多巴胺、5-羟色胺和章鱼胺在脑中对蜜蜂行为发育的调节起着关键作用[19]。此外,神经分子(例如神经肽)被称为中枢神经系统回路中的神经递质和神经调节剂[20],对蜜蜂脑中神经功能的肽能调节产生重大影响[21],也有研究表明脑部神经肽组的重构用以适应蜂王浆高产的需要[14]。哺育蜂对幼虫的哺育行为是建立在其大脑对幼虫信息素信号的接收和反馈基础上的[4,14]。哺育蜂哺育行为介导的蜂王浆分泌机制的研究大多集中在咽下腺和上颚腺[6,22-23],有关脑部调控哺育行为介导蜂王浆分泌的分子机制方面的研究相对较少。
本研究人工组建蜂群收集3日龄工蜂,10日龄哺育蜂、采集蜂,21日龄哺育蜂、采集蜂,利用RNA-seq技术,在减小日龄的干扰下严格筛选出41个DEG,其中32个与哺育蜂哺育行为密切相关。WHITFIELD等通过基因芯片技术检测到哺育蜂和采集蜂脑之间约有1 800个DEG[7],而本研究只筛选出41个DEG,主要有以下原因:首先本研究的试验样本来自人工构建相同日龄的哺育蜂和采集蜂;构建两个时间点(10日龄和21日龄)的同日龄哺育蜂和采集蜂,然后取共有的DEG。这两个严格条件可以排除日龄的影响,导致筛选的DEG数目较少,进一步保证结果更加可靠。GO富集分析显示上调DEG主要参与氧化还原酶活性、气味结合、跨膜运输;KEGG富集通路显示这些上调DEG主要参与蛋白质代谢、能量代谢、信号转导等(图2),其中显著性富集在鞘脂代谢和其他聚糖降解通路上。
3.1 哺育蜂脑中信号转导相关的上调DEG
突触囊泡糖蛋白2(SV2)具有许多功能,包括囊泡运输、稳定神经递质囊泡负载、锚定囊泡蛋白、协助囊泡运输、调节钙敏感性以及与细胞外基质相互作用;在脊椎动物中,SV2家族由3个副产物组成,包括SV2A、SV2B和SV2C,其中SV2C的表达是最受限制的,仅局限于进化古老的大脑区域,在纹状体、中脑和腹侧苍白球均有较强的表达,而在新皮层的表达很少[24]。SV2C参与中枢系统中多巴胺的释放,其缺失可以引起小鼠运动障碍、增加-突触核蛋白单体的聚集和降低纹状体多巴胺的释放[25]。本研究在上调DEG中发现编码SV2C的基因,该基因在3日龄工蜂和采集蜂阶段基本不表达(3 d、10 dF和21 dF中该基因的FPKM值分别为0.22、0.47和0.60),而在哺育蜂阶段的表达量相对较高(10 dN和21 dN脑中FPKM值分别为12.81和11.96)。化学神经传递对神经元之间的交流至关重要,在这个交流过程中,Ca2+涌入突触前末端,触发神经递质释放进入突触,作用于突触后受体[24]。此外,GO富集结果也显示参与跨膜运输功能,推测工蜂脑部的在调节Ca2+敏感性,促进神经递质作用于突触后受体,从而引发哺育行为方面发挥重要作用。
葡萄糖神经酰胺是300多种结构不同的鞘糖脂(包括神经节苷脂和硫苷脂)的骨架,对于哺乳动物的发育至关重要。葡萄糖神经酰胺是一种膜鞘磷脂,是许多糖脂的前体[26]。葡糖苷神经酰胺合成酶催化神经酰胺产生葡萄糖神经酰胺,它是神经节苷脂类的前体物质[27]。在上调DEG中发现编码葡萄糖基神经酰胺酶的基因和,KEGG富集结果显示这两个基因显著性富集在鞘脂代谢路径上。鞘脂及其代谢产物不仅是构成细胞膜的重要结构分子,而且参与调节细胞的生长、分化、衰老和细胞程序性死亡等许多重要的信号转导过程,使细胞产生各种不同的生物学功能[28]。
药理学研究表明,在发育的大脑中,sn-1特异性二酰基甘油脂肪酶活性是轴突生长和引导所必需的[29-30]。在哺育蜂阶段富集的光传导途径可能表明该通路在哺育蜂从事巢外工作接受光信号中发挥重要作用[31]。本研究在上调DEG中发现编码sn-1特异性二酰基甘油脂肪酶的基因富集在光传导途径中。
在昆虫中,气味结合蛋白(OBP)和化学感受蛋白(chemosensory protein,CSP)在运载疏水性气味分子和信息素穿过淋巴液的信号转导中发挥重要作用[32-33]。OBP14在蜂群的上颚腺中含量丰富,与单萜类结构有较好的亲和力;OBP21在老蜂体内含量丰富,并与法尼素结合,而法尼素是一种吸引蜂群的信息素[34]。OBP和CSP可以与多种配体结合,但是与蜜蜂的幼虫信息素有更高的结合力[33,35]。本研究在上调DEG中发现了、、;Nie等在意蜂的触角转录组中发现、、,其中、在触角中的表达量从刚出房工蜂到哺育蜂阶段呈增加趋势,而在哺育蜂阶段到采集蜂阶段呈减小趋势[36],这与本研究在上调DEG中发现的这两个基因的表达趋势基本一致,推测这两个基因可能在脑部化学信息的传递和调控方面发挥重要作用。
哺育蜂通过调节对幼虫信息素的识别和采集蜂的采集能力等来增加蜂群群体的营养供应进而提高蜂王浆的分泌。细胞的一切行为都需要信号分子和受体结合,通过信号转导实现。这些与信号转导相关的基因(和)在哺育蜂脑中呈上调表达,暗示哺育蜂可能通过这些基因增强信号转导,进而在感知挥发物信息素后启动高效蛋白合成的机制。
3.2 哺育蜂脑中与能量代谢相关的上调DEG
氧化磷酸化是真核生物体内有机物包括糖、脂、氨基酸等在分解过程中发生氧化并驱动ATP合成的过程,生物体内95%的ATP来自这种方式[37]。脑部为满足其较高的代谢速率需要消耗很多的ATP,因此有大量的线粒体位于大脑细胞中。除氧化磷酸化外,三羧酸循环(TCA循环)和碳代谢也是生物体能量代谢产生ATP的重要途径。在细胞和器官中,三羧酸循环(TCA循环)是产生能量的关键过程,本研究在上调DEG中发现编码琥珀酸脱氢酶[泛醌]黄素蛋白亚基的基因,表明哺育蜂脑部可能通过有氧氧化(TCA循环)葡萄糖产生大量ATP。此外,该基因还显示富集在氧化磷酸化、碳代谢、三羧酸循环通路,这些结果表明哺育蜂脑部可能通过多种途径为哺育行为提供充足的能量。
昆虫细胞色素P450酶参与多种代谢活动,包括外源性物质降解、保幼激素和蜕皮类固醇生物合成、信息素代谢[9]。在相同日龄哺育蜂和采集蜂脑中,发现在10日龄哺育蜂和21日龄哺育蜂脑中的表达量均显著高于相同日龄采集蜂中表达量;GO富集分析表明该基因参与氧化还原酶活性。唐晓伟根据细胞色素P450与血红素结合区域设计的简并引物克隆出一段蜜蜂细胞色素P450基因片段,进而克隆得到该基因全长,并由细胞色素P450命名委员会命名为[38]。该基因属于CYP6家族成员,但是关于该基因的功能尚未有深入研究。在果蝇中,CYP6家族的成员羟化了月桂酸的ω-1位置,这表明CYP6家族的成员参与了昆虫的脂肪酸羟化反应[23,39]。BOUTIN等通过在清理行为和非清理行为工蜂脑部的差异表达基因中发现在清理行为工蜂脑部高表达的细胞色素P450基因,推测可能是清理行为工蜂对外源性物质(如幼虫发出的信息素等气味)的高敏感性引发细胞色素P450基因的高量表达[40]。在哺育蜂脑中发现上调表达,推测该基因可能在哺育蜂脑部调控脂肪酸羟基化或降解幼虫释放的请求饲喂的信息素过程中发挥作用。哺育蜂能够感知幼虫信息素并通过外围化学感受器官传输信号到大脑;通过高级中枢神经器官-大脑调控上颚腺和咽下腺合成蛋白质和脂肪酸等重要大分子物质,引发脑部产生哺育行为,进而促进咽下腺和上颚腺等腺体产生蜂王浆分泌的行为。上述富集的通路说明大脑在这一时期的基础能量供应和蛋白、脂肪酸合成都得到加强,这样更有利于大脑进行信息处理和行为调控,从而促进哺育蜂的哺育行为。
值得注意的是,在上调DEG中发现编码王浆主蛋白1(MRJP1)的,目前该基因尚未有相关功能的报道。该基因在NCBI意大利蜜蜂基因组新版本Amel_HAv3.1中尚未注释,但以前版本Amel_4.5中有注释。针对该基因特异序列设计引物,通过荧光定量检测该基因在脑中表达,且在哺育蜂脑中显著高量表达(图3),该基因有可能是蜂王浆主蛋白家族的新成员,后续可以通过克隆基因全长进行验证。前人在蜜蜂脑中也检测到蜂王浆主蛋白家族基因的表达,但在脑中发挥的作用尚不明确。RNA-seq测序将具有卫生行为和不具有卫生行为工蜂脑进行比较[40],也发现在这两种行为工蜂脑部高量表达。蛋白质组学发现MRJP1、MRJP2和MRJP7蛋白在哺育蜂脑中的含量显著高于采集蜂脑中含量,推测这些基因可能为脑中蛋白质的合成过程储存氨基酸[41]。在本研究构建的相同日龄哺育蜂和采集蜂脑的转录组数据中,检测到()在哺育蜂脑中显著高量表达,暗示该基因可能在哺育行为调控蜂王浆合成和分泌过程中发挥重要作用。
4 结论
在mRNA组学水平对3日龄工蜂、10日龄哺育蜂、10日龄采集蜂、21日龄哺育蜂、21日龄采集蜂5组样本脑组织进行全面表征分析,获得了相同日龄哺育蜂和采集蜂脑部基因的转录组图谱,分析得到了哺育行为密切相关的32个上调差异表达基因。哺育蜂脑部的差异表达基因主要通过信号转导和能量代谢等途径调控哺育蜂的哺育行为,研究结果可为阐明哺育蜂哺育行为的分子机制提供理论参考。
[1] BEZABIH G, CHENG H, HAN B, FENG M, XUE Y, HU H, LI J K. Phosphoproteome analysis reveals phosphorylation underpinnings in the brains of nurse and forager honeybees ()., 2017, 7: 1973.
[2] HAN B, FANG Y, FENG M, HU H, HAO Y, MA C, HUO X, MENG L, ZHANG X, WU F, LI J K. Brain membrane proteome and phosphoproteome reveal molecular basis associating with nursing and foraging behaviors of honeybee workers., 2017, 16(10): 3646-3663.
[3] SIALANA F J, MENEGASSO A R S, SMIDAK R, HUSSEIN A M, ZAVADIL M, RATTEI T, LUBEC G, PALMA M S, LUBEC J. Proteome changes paralleling the olfactory conditioning in the forager honey bee and provision of a brain proteomicsdataset., 2019, 19(13): e1900094.
[4] CONTE Y L, MOHAMMEDI A, ROBINSON G E. Primer effects of a brood pheromone on honeybee behavioural development., 2001, 268(1463): 163-168.
[5] FUJITA T, KOZUKA-HATA H, AO-KONDO H, KUNIEDA T, OYAMA M, KUBO T. Proteomic analysis of the royal jelly and characterization of the functions of its derivation glands in the honeybee., 2012, 12(1): 404-411.
[6] ALTAYE S Z, MENG L F, LI J K. Molecular insights into the enhanced performance of royal jelly secretion by a stock of honeybee () selected for increasing royal jelly production., 2019, 50(4): 436-453.
[7] WHITFIELD C W, CZIKO A M, ROBINSON G E. Gene expression profiles in the brain predict behavior in individual honey bees.,2003, 302(5643): 296-299.
[8] KUCHARSKI R, MALESZKA R. Evaluation of differential gene expression during behavioral development in the honeybee using microarrays and northern blots., 2002, 3(2): RESEARCH0007.
[9] RODRIGUEZ-ZAS S L, SOUTHEY B R, SHEMESH Y, RUBIN E B, COHEN M, ROBINSON G E, BLOCH G. Microarray analysis of natural socially regulated plasticity in circadian rhythms of honey bees., 2012, 27(1): 12-24.
[10] HERNÁNDEZ L G, LU B, DA CRUZ G C, CALÁBRIA L K, MARTINS N F, TOGAWA R, ESPINDOLA F S, YATES J R, CUNHA R B, DE SOUSA M V. Worker honeybee brain proteome., 2012, 11(3): 1485-1493.
[11] PAERHATI Y, ISHIGURO S, UEDA-MATSUO R, YANG P, YAMASHITA T, ITO K, MAEKAWA H, TANI H, SUZUKI K. Expression ofof the gustatory receptor family in honey bee is correlated with nursing behavior., 2015, 10(11): e0142917.
[12] KNECHT D, KAATZ H H. Patterns of larval food production by hypopharyngeal glands in adult worker honey bees., 1990, 21(5): 457-468.
[13] HAN B, FANG Y, FENG M, HU H, QI Y P, HUO X M, MENG L F, WU B, LI J K. Quantitative neuropeptidome analysis reveals neuropeptides are correlated with social behavior regulation of the honeybee workers., 2015, 14(10): 4382-4393.
[14] 韩宾. 工蜂劳动分工与蜂王浆高产机理的大脑神经肽组、膜蛋白质组和膜磷酸化蛋白质组研究[D]. 北京: 中国农业科学院, 2017.
HAN B. Investigation of molecular basis associating with division of labor and high royal jelly yields by analyzing brain neuropeptidome, membrane proteome and membrane phosphoproteome[D]. Beijing: Chinese Academy of Agricultural Sciences, 2017. (in Chinese)
[15] TSUCHIMOTO M, AOKI M, TAKADA M, KANOU Y, SASAGAWA H, KITAGAWA Y, KADOWAKI T. The changes of gene expression in honeybee () brains associated with ages., 2004, 21(1): 23-28.
[16] 赵元洪, 赵晓蒙, 苏松坤. 蜜蜂全脑解剖新方法的研究. 中国蜂业, 2014, 65(Z1): 4-7.
ZHAO Y H, ZHAO X M, SU S K. New method on dissection of whole brain of honeybee., 2014, 65(Z1): 4-7. (in Chinese)
[17] ANDERS S, HUBER W. Differential expression analysis for sequence count data., 2010, 11(10): R106.
[18] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCTmethod., 2001, 25(4): 402-408.
[19] SCHULZ D J, BARRON A B, ROBINSON G E. A role for octopamine in honey bee division of labor., 2002, 60(6): 350-359.
[20] XU G, TENG Z W, GU G X, QI Y X, GUO L, XIAO S, WANG F, FANG Q, WANG F, SONG Q S, STANLEY D, YE G Y. Genome- wide characterization and transcriptomic analyses of neuropeptides and their receptors in an endoparasitoid wasp,., 2019, 103(2): e21625.
[21] PRATAVIEIRA M, DA SLIVA MENEGASSO A R, ESTEVES F G, SATO K U, MALASPINa O, PALMA M S. MALDI imaging analysis of neuropeptides in africanized honeybee () brain: effect of aggressiveness., 2018, 17(7): 2358-2369.
[22] LIU Z, JI T, YIN L, SHEN J, SHEN F, CHEN G. Transcriptome sequencing analysis reveals the regulation of the hypopharyngeal glands in the honey bee,Pollmann., 2013, 8(12): e81001.
[23] WU Y Q, ZHENG H Q, CORONA M, PIRK C, MENG F, ZHENG Y F, HU F L. Comparative transcriptome analysis on the synthesis pathway of honey bee () mandibular gland secretions., 2017, 7(1): 4530.
[24] STOUT K A, DUNN A R, HOFFMAN C, MILLER G W. The synaptic vesicle glycoprotein 2: structure, function, and disease relevance., 2019, 10(9): 3927-3938.
[25] MAZZUCCHELLI C, BRAMBILLA R. Ras-related and MAPK signalling in neuronal plasticity and memory formation., 2000, 57(4): 604-611.
[26] ASTUDILLO L, THERVILLE N, COLACIOS C, SÉGUI B, ANDRIEU-ABADIE N, LEVADE T. Glucosylceramidases and malignancies in mammals., 2016, 125: 267-280.
[27] 孙九丽, 林慧珍, 苟萍. 鞘脂代谢及其相关疾病研究进展. 生物技术, 2011, 21(5): 93-97.
SUN J L, LIN H Z, GOU P. Research progress of sphingolipid metabolism and related diseases., 2011, 21(5): 93-97. (in Chinese)
[28] SHI J, ANDERSON D, LYNCH B A, CASTAIGNE J G, FOERCH P, LEBON F. Combining modelling and mutagenesis studies of synaptic vesicle protein 2A to identify a series of residues involved in racetam binding., 2011, 39(5): 1341-1347.
[29] BISOGNO T, HOWELL F, WILLIAMS G, MINASSI A, CASCIO M, LIGRESTI A, MATIAS I, SCHIANO-MORIELLO A, PAUL P, WILLIAMS E J, GANGADHARAN U, HOBBS C, DI MARZO V, DOHERTY P. Cloning of the first sn1-DAG lipases points to the spatial and temporal regulation of endocannabinoid signaling in the brain., 2003, 163(3): 463-468.
[30] BRITTIS P A, SILVER J, WALSH F S, DOHERTY P. Fibroblast growth factor receptor function is required for the orderly projection of ganglion cell axons in the developing mammalian retina., 1996, 8(2/3): 120-128.
[31] RIDGE K D, ABDULAEV N G, SOUSA M, PALCZEWSKI K. Phototransduction: crystal clear., 2003, 28(9): 479-487.
[32] STEINBRECHT R A. Odorant-binding proteins: expression and function., 1998, 855(1): 323-332.
[33] BRIAND L, SWASDIPAN N, NESPOULOUS C, BÉZIRARD V, BLON F, HUET J C, EBERT P, PERNOLLET J C. Characterization of a chemosensory protein (ASP3c) from honeybee (L.) as a brood pheromone carrier., 2002, 269(18): 4586-4596.
[34] IOVINELLA I, DANI F, NICCOLINI A, SIMONA S, MICHELUCCI E, GAZZANO A, TURILLAZZI S, FELICIOLI A, PELOSI P. Differential expression of odorant-binding proteins in the mandibular glands of the honey bee according to caste and age., 2011, 10(8): 3439-3449.
[35] WU F, FENG Y L, HAN B, HU H, FENG M, MENG L F, MA C, YU L S, LI J K. Mechanistic insight into binding interaction between chemosensory protein 4 and volatile larval pheromones in honeybees ()., 2019, 141: 553-563.
[36] NIE H Y, XU S P, XIE C Q, GENG H Y, ZHAO Y Z, LI J H, HUANG W F, LIN Y, Li Z G, SU S K. Comparative transcriptome analysis ofantennae of workers performing different tasks., 2017, 293(1): 237-248.
[37] LESNEFSKY E J, HOPPEL C L. Oxidative phosphorylation and aging., 2006, 5(4): 402-433.
[38] 唐晓伟. 西方蜜蜂细胞色素P450单加氧酶特性初步研究[D]. 北京: 中国农业科学院, 2011.
TANG X W. Preliminary study on cytochrome P450 monooxygenase of[D]. Beijing: Chinese Academy of Agricultural Sciences, 2011. (in Chinese)
[39] HELVIG C, TIJET N, FEYEREISEN R, WALKER F A, RESTIFO L L.CYP6A8, an insect P450 that catalyzes lauric acid (omega-1)-hydroxylation., 2004, 325(4): 1495-1502.
[40] BOUTIN S, ALBURAK M, MERCIER P L, GIOVENAZZO P, DEROME N. Differential gene expression between hygienic and non-hygienic honeybee (L.) hives., 2015, 16(1): 500.
[41] GARCIA L, GARCIA C H, CALÁBRIA L K, DA CRUZ G C, Puentes A S, BÁO S N, FONTES W, RICART C A, ESPINDOLA F S, DE SOUSA M V. Proteomic analysis of honey bee brain upon ontogenetic and behavioral development., 2009, 8(3): 1464-1473.
Transcriptomic analysis of genes related to nursing behavior in the brains of
GAO Yan, ZHU YaNan, LI QiuFang, SU SongKun, NIE HongYi
(College of Animal Sciences (College of Bee science), Fujian Agriculture and Forestry University, Fuzhou 350002)
【Objective】The nursing behavior ofplays an important role in maintaining colony stability and production of royal jelly (RJ). In this study, samples (3-day-old worker bees, 10-day-old nurses/foragers, 21-day-old nurses/foragers) were obtained by constructing artificial colony, which can eliminate the influence of age on nursing behavior. Moreover, genes closely related to the nursing behavior were strictly screened out in the brain of these samples, which could reveal the molecular network of brain regulation of nursing behavior.【Method】The 3-day-old worker bees, 10-day-old nurses and foragers, 21-day-old nurses and foragers were obtained by constructing artificial bee colony. And then the head of different group’s worker bees was dissected to obtain brain tissues of these samples. RNA-seq was used to analyze the transcriptome sequencing of the 5 groups (3-day-old worker bees, 10-day-old nurses, 10-day-old foragers, 21-day-old nurses, 21-day-old foragers) of brain samples. Differentially expressed genes (DEGs) which are closely related to the nursing behavior in the nurses’ brain were screened out. GO and KEGG enrichment analysis were carried out for these genes. qPCR was used to verify the expression patterns of 4 randomly selected DEGs.【Result】RNA-seq analysis screened out 32 DEGs that were closely related to the nursing behavior of nurses. These genes were significantly up-regulated in the brain of 10-day-old nurses than 3-day-old worker bees, 10-day-old foragers and 21-day-old foragers, and the expression level in the brain of 21-day-old nurses was significantly higher than that in 21-day-old foragers. Go enrichment analysis showed that the up-regulated DEGs were mainly involved in oxidoreductase activity, odor binding, transmembrane transport and other functional items. KEGG enrichment results showed that the up-regulated DEGs were mainly involved in protein metabolism (ribosome), energy metabolism (oxidative phosphorylation, carbon metabolism, the citrate cycle (TCA cycle), starch and sucrose metabolism, nitrogen metabolism and other glycan degradation pathway), signal transduction (Toll and Imd signaling pathways, phototransduction, sphingolipid metabolism), digestive function (lysosome). Of them, only sphingolipid metabolism and other glycan degradation pathway were notably enriched. qPCR results showed that the expression patterns of 3 up-regulated DEGs (,,) and 1 down-regulated DEG () were consistent with the sequencing data.【Conclusion】To obtain gene expression profiles of brains from age-matched nurses and foragers, the brains of 5 groups (3-day-old worker bees, 10-day-old nurses, 10-day-old foragers, 21-day-old nurses, and 21-day-old foragers) were comprehensively analyzed using transcriptome sequencing, revealing that the 32 up-regulated DEGs were associated with brain nursing behavior. These genes were mainly involved in signal transduction and energy metabolism, which can affect the nursing behavior.
; nurses; brain; nursing behavior; royal jelly; RNA-seq
2020-01-17;
2020-03-10
国家现代农业产业技术体系(蜜蜂)(CARS-44-KXJ4)、福建省自然科学基金(2018J05043)、福建省省属高校科研项目(JK2017014)
高艳,E-mail:2726820378@qq.com。通信作者苏松坤,E-mail:susongkun@zju.edu.cn。通信作者聂红毅,E-mail:hnhynie@126.com
(责任编辑 岳梅)

