盐胁迫培养下季也蒙毕赤酵母的转录组学差异分析
曹璇,郑晓冬
(浙江大学生物系统工程与食品科学学院,杭州310058)
季也蒙毕赤酵母(Meyerozyma guilliermondii)是一类好氧菌,在25~30 ℃条件下可以快速生长。YAN等发现,从果园土壤中分离出的季也蒙毕赤酵母菌株(Y1)可以直接抑制梨果实表面扩展青霉(Penicillium expansum)的生长[1];高云慨等发现,从海南杧果园取样分离到的季也蒙毕赤酵母菌株(LZ5)对杧果采后炭疽病菌有明显的抑制效果[2];丁玥等的试验证明,季也蒙毕赤酵母可以有效控制番茄贮藏期灰霉病的发生[3]。同时,RANGARAJAN等[4]、SIM等[5]、SHARMA等[6]的研究证明,部分生防菌种经外界逆境胁迫培养后,可以明显增强其对农作物或果蔬采后病害的防治效果。由于季也蒙毕赤酵母对果蔬采后具有较好的生物防治效果,近年来受到国内外研究者的广泛关注。笔者所在实验室曾于海南省三沙市西沙群岛海沙中分离得到1株季也蒙毕赤酵母(保藏号:CGMCC 17240),试验证明此酵母菌株对樱桃番茄果实灰霉病有一定的抑制作用,并且试验还证明该酵母菌株有一定的耐盐性,在12%NaCl胁迫培养条件下仍可生长,且经盐逆境胁迫培养后其生防效果有所提升。
转录组学是了解基因组遗传信息与生物功能的桥梁[7],使用Illumina公司高通量测序平台的转录组测序技术,可以在不需要知道物种基因组详细信息的条件下获得某一物种在特定状态下几乎所有转录本的序列信息[8-9]。且转录组数据会随着生物体的生长发育情况及所处环境状态发生变化,因此,对转录组进行分析可以帮助我们有效研究生物在逆境胁迫下应激生理的作用机制[10]。本研究利用转录组测序技术对经盐胁迫与未经盐胁迫培养后的M.guilliermondii 进行转录组测序,对筛选得到的差异表达基因进行基因本体(gene ontology,GO)功能注释及京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路富集分析,旨为M.guilliermondii的耐盐机制及其进一步研究提供理论支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
季也蒙毕赤酵母(Meyerozyma guilliermondii)为笔者所在实验室于海南省西沙群岛海沙中分离得到,保藏于中国微生物菌种保藏管理委员会普通微生物中心,保藏号:CGMCC 17240。
1.1.2 样品制备
从保藏斜面上挑取少许季也蒙毕赤酵母接种于营养酵母葡萄糖琼脂(nutrient yeast dextrose agar,NYDA)固体培养基(牛肉浸膏8 g,酵母浸膏5 g,葡萄糖10 g,琼脂20 g,用水定容至1 000 mL,于121 ℃条件下高压蒸汽灭菌20 min)上,25 ℃静置培养48 h以进行活化;再用接种环将活化好的菌株接种到50 mL 营养酵母葡萄糖液体(nutrient yeast dextrose broth,NYDB)培养基中,置于250 mL锥形瓶内,于28 ℃、200 r/min条件下振荡培养24 h;随后吸取1 mL上述菌液分别加入到50 mL NYDB培养基和50 mL 12%NaCl-NYDB培养基(牛肉浸膏8 g,酵母浸膏5 g,葡萄糖10 g,氯化钠120 g,用水定容至1 000 mL,于121 ℃条件下高压蒸汽灭菌20 min)中,再于28 ℃、200 r/min条件下传代培养24 h。收集一定体积的上述2 种培养液,在4 ℃、4 000 r/min 条件下离心10 min,对离心收集所得菌体用无菌水洗涤2次,弃上清液,转移至冻存管中,立即放入液氮进行速冻,并且于-80 ℃冰箱中保存,备用。
1.2 仪器与设备
MLS-3750 全自动灭菌锅(日本Sanyo 公司);SW-CJ-1F 超净工作台(苏州安泰空气技术有限公司);SIGMA 3K-15 离心机(德国Sigma 公司);QYC-211全温空气摇床(上海福玛实验设备有限公司);LRH-250 生化培养箱(上海一恒科技有限公司);移液枪(德国Eppendorf公司);光学显微镜(日本奥林巴斯有限公司);涡旋振荡器(海门市其林贝尔仪器制造有限公司);PTC-225热循环聚合酶链式反应(polymerase chain reaction, PCR)仪(美国Bio-Rad公司);一步法实时荧光PCR仪(美国ABI公司);Oligo(dT)磁珠试剂盒(武汉哇哇噻纳技术开发有限公司);PrimeScriptTM单链cDNA 合成反转录酶试剂盒、PrimeScriptTM双链cDNA 合成试剂盒、RNAiso Plus 动物/植物/微生物/培养细胞总RNA 提取试剂盒、PrimeScriptTM除去基因组DNA 反转录试剂盒、TB GreenTM嵌合Ex TaqTM荧光定量PCR 试剂盒,均由日本TaKaRa公司提供。
1.3 试验方法
1.3.1 RNA 的提取与文库的构建
采用Invitrogen Trizol 试剂(美国Thermo Fisher科技公司)[11]提取2 种培养条件(盐胁迫和非盐胁迫)下M. guilliermondii 的总RNA,利用Nanodrop(美国Thermo Fisher 科技公司)检测RNA 纯度,然后利用Qubit 2.0 对RNA 浓度进行精确定量,使用Aglient 2100 检测RNA 样品的完整性。样品检测合格后,首先用带有Oligo(dT)的磁珠富集真核生物的mRNA,随后加入缓冲碎片将mRNA打断成短片段,以mRNA 为模板,用六碱基随机引物(random hexamers)合成第1链cDNA,然后加入缓冲液、dNTPs 和DNA 聚合酶Ⅰ和RNaseⅡ合成第2 链cDNA,再用AMPure XP磁珠纯化双链cDNA。纯化的双链cDNA 先进行末端修复、加A 尾并连接测序接头,再用AMPure XP磁珠进行片段大小选择。最后进行PCR扩增,得到最终的DNA扩增文库。文库构建完成后,分别使用Qubit 2.0和Agilent 2100对文库的浓度和插入片段大小进行检测,使用定量PCR(quantitative PCR,qPCR)对文库浓度进行准确定量(文库有效浓度>2 nmol/L),以保证文库质量。
1.3.2 测序及数据过滤
库检合格后,把不同文库按照有效浓度及目标上机数据量的需求合并后进行Illumina HiSeqTM高通量测序,得到原始测序序列(raw reads)。本试验所有测序工作在北京诺禾致源科技股份有限公司完成。为了保证信息分析质量,数据分析之前需先对原始测序序列进行过滤:1)去除带接头的测序序列;2)去除未知碱基N 比例大于10%的测序序列;3)去除过大过小等低质量的测序序列,以得到高质量序列(clean reads)。
1.3.3 测序数据的分析
本试验研究的M.guilliermondii为非模式生物,目前无相关全基因组测定结果。对于无参考基因组的转录组分析,采用Trinity 法[12]将测序所得的高质量序列拼接成转录本(mRNA 序列),用Corset 程序对转录本进行层次聚类,以聚类后的序列为参考,在此基础上进行基因结构注释、基因表达水平分析、差异基因功能注释和富集分析等。
1.3.4 实时荧光定量反转录PCR(real time quantitative reverse polymerase chain reaction, RT-qPCR)对转录组结果的验证
cDNA 模板合成步骤参照去基因组DNA PrimeScriptTM反转录试剂盒[RT reagent Kit with gDNA Eraser(Perfect Real Time)]说明书,用无菌水对反转录样品进行梯度(2、4、6、8、10倍)稀释,并以稀释后的cDNA 为模板进行qPCR 反应(每个浓度设置3 个重复),选取CT值在15~25 间的cDNA 的合适稀释倍数进行后续试验,整个试验重复2 次及以上,直至2次试验结果一致,RT-qPCR试验详细步骤参照TB GreenTMPremix Ex TaqTM试剂盒说明书。两步法PCR 扩增反应条件如下:95 ℃预变性30 s;95 ℃变性5 s,60 ℃复性30 s,40个循环。溶解曲线条件如下:95 ℃变性15 s,以0.3 ℃为步阶从60 ℃升温至95 ℃,95 ℃保温15 s。利用一步法实时荧光定量PCR 仪(美国Thermo Fisher 科技公司)检测反应体系荧光变化,相对定量计算采用2-△△CT方法[13]。目的基因的引物设计和合成均由生工生物工程(上海)有限公司完成,引物序列如表1所示。

表1 实时荧光定量反转录PCR扩增目的基因的引物序列Table 1 Primer sequences used for amplification of target genes by RT-qPCR
2 结果与分析
2.1 测序数据的统计和质控报告
采用Illumina HiSeqTM高通量测序技术对在盐胁迫(12%NaCl)与非盐胁迫(0%NaCl)条件下培养24 h的M.guilliermondii进行转录组测序,结果如表2 所示。经测序质量控制,共获得55.39 Gb 高质量碱基,各样品碱基错误率接近于0,Q20 和Q30 碱基百分比均不小于93%,样品各项检测参数达到质量要求,适合之后的深度分析。
2.2 季也蒙毕赤酵母响应盐胁迫的基因差异表达分析
为了探究M. guilliermondii 在盐胁迫(12%NaCl)与非盐胁迫(0%NaCl)培养条件下的差异表达基因,对样品数据进行差异表达分析,筛选条件为:校正后的P值<0.05且|log2(差异倍数)|>1。结果如图1所示:盐胁迫组与非盐胁迫组之间有1 027个显著性差异表达基因,其中458个基因表达上调,569个基因表达下调。
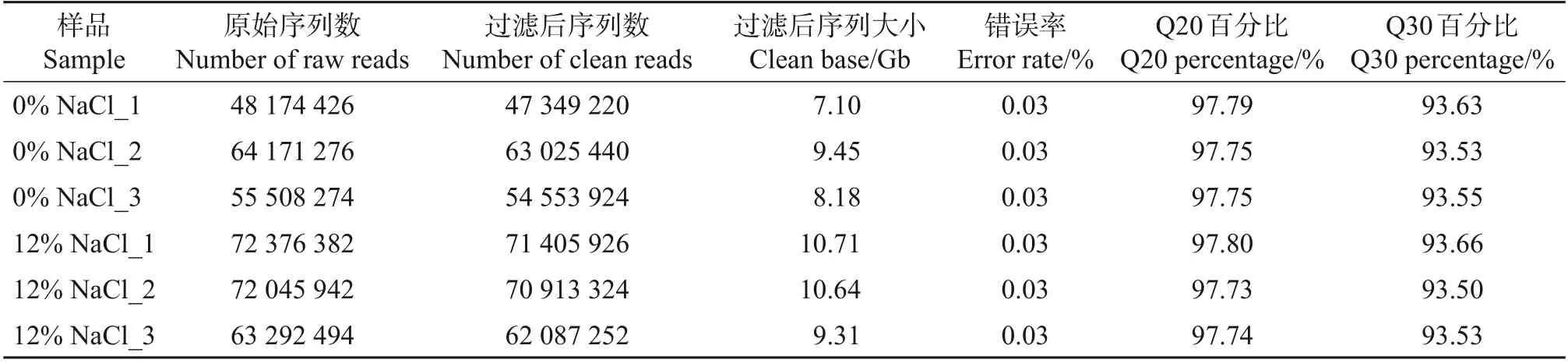
表2 数据产出质量情况统计Table 2 Statistics of data output quality
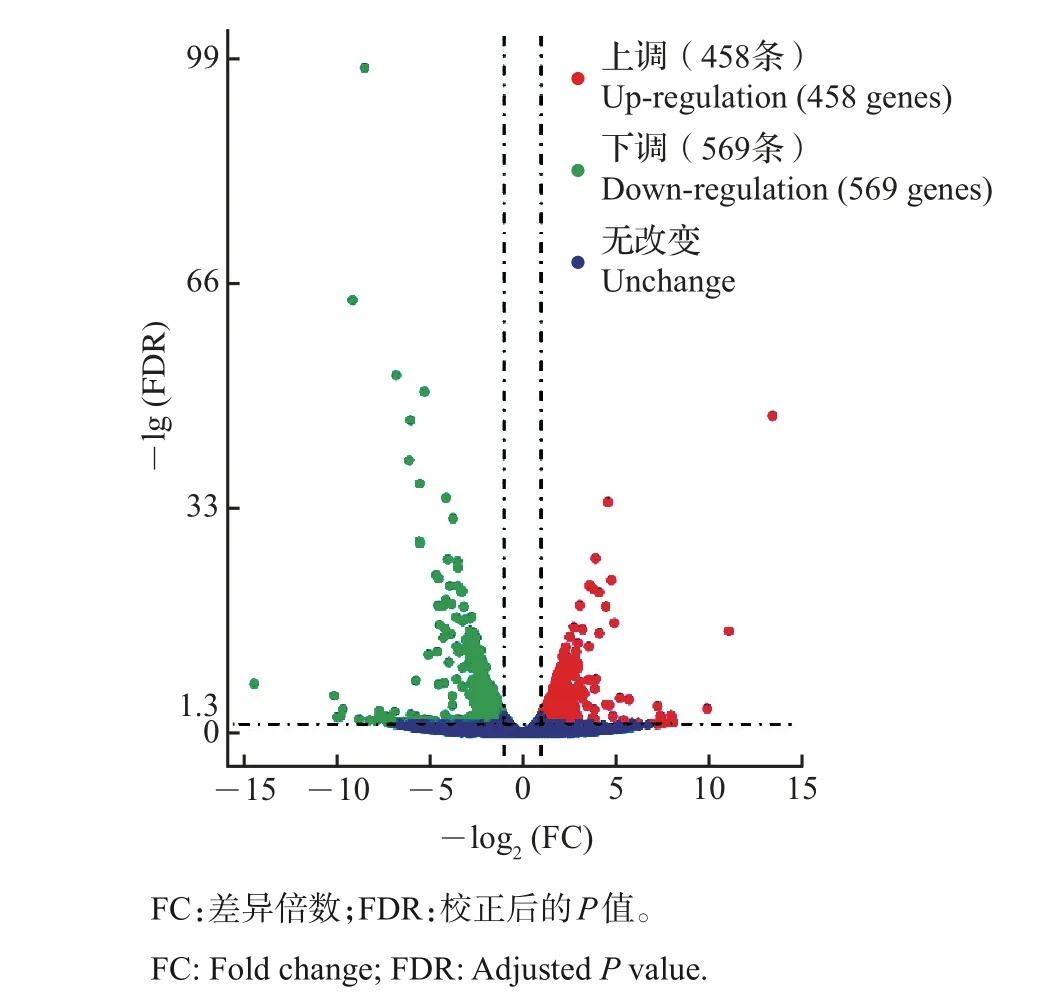
图1 试验组差异表达基因火山图Fig.1 Volcano plot of the differential expression genes among test groups
2.3 差异表达基因的GO 功能注释和富集分析
GO富集分析能够了解差异表达基因与哪些生物学功能显著相关,共涵盖基因的分子功能(molecular function)、所在的细胞组分(cellular component)和参与的生物过程(biological process)等3 方面的内容。本研究将M. guilliermondii 在盐胁迫(12%NaCl)与非盐胁迫(0%NaCl)培养条件下筛选得到的差异基因采用GOseq方法[14]进行GO富集分析,并挑选富集最显著的30 个GO 项绘制差异基因GO富集柱状图,结果如图2所示:经盐胁迫后M.guilliermondii的差异表达基因主要富集在生物过程分类中,其中,代谢过程类别所占比例最大,其次为生物合成过程类别及单组织过程类别;在细胞组分分类中,细胞核部分所占比例最大;在分子功能分类中,离子结合及催化活性功能类别占比最大。由此可以得出,由于盐胁迫培养,导致M.guilliermondii的核组装、核苷酸代谢、糖代谢及辅酶代谢基因产生了较大差异,进而影响了细胞的分裂及代谢过程。
2.4 差异基因KEGG 富集分析
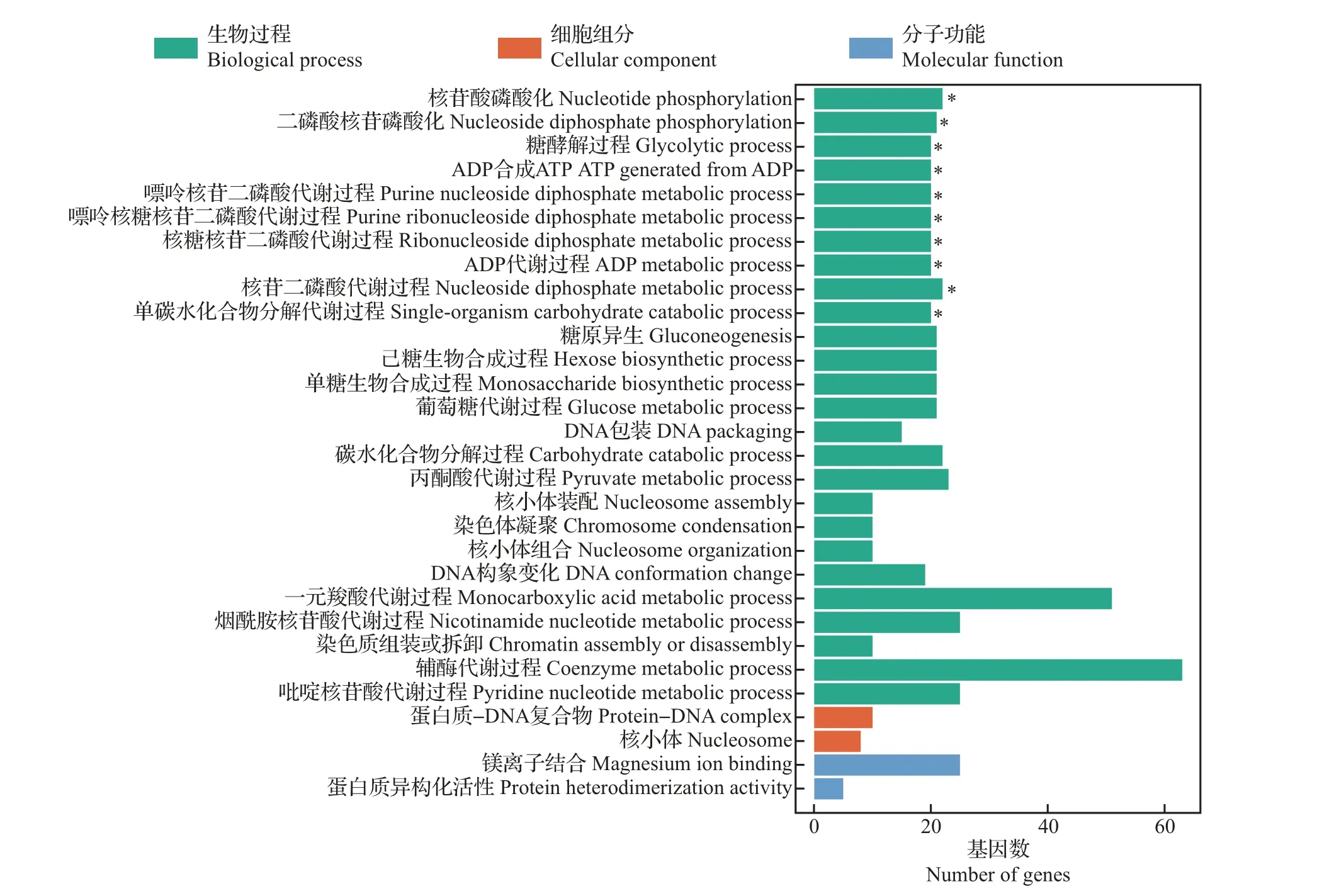
图2 差异表达基因GO富集柱状图Fig.2 GO enrichment histogram of differential expression genes
KEGG 是有关基因通路的主要公共数据库[15]。在生物体内,不同基因相互协调行使其生物学功能,通过通路(pathway)显著性富集能确定差异表达基因参与的最主要生化代谢途径和信号转导途径。本试验差异基因的通路富集分析结果表明:差异表达基因分布在90个通路中,其中将富集最显著的20条通路绘制差异基因KEGG富集散点图,并通过富集因子、Q 值和富集到此通路上的基因个数来衡量富集程度,结果如图3 所示。季也蒙毕赤酵母在盐胁迫(12% NaCl)与非盐胁迫(0% NaCl)培养条件下,KEGG代谢通路中显著富集的有甘氨酸、丝氨酸和苏氨酸代谢,乙醛酸和二羧酸代谢,DNA复制,脂肪酸生物合成,减数分裂-酵母调控,半乳糖代谢,过氧化物酶体,甲烷代谢,氨基糖和核苷酸糖代谢,丙氨酸、天冬氨酸和谷氨酸代谢,MAPK信号通路-酵母,苯丙氨酸代谢,氰基氨基酸代谢,细胞周期-酵母等信号通路。其中大部分富集通路都与细胞的分裂、代谢有关,且也有文献证明MAPK 信号通路与生物受到逆境胁迫的应激反应相关[16-17]。
2.5 差异表达基因的RT-qPCR 验证
本试验随机选取12个分别在MAPK信号通路-酵母中显著变化的基因(STE12、RHOA、PBS2、GPD1、GRE2、FKS2、HOG1、BMH1, 2-YWHAE、SHO1、STE20-PAK1、WSC2、CSNKI)进行RT-qPCR分析以验证转录组测序结果的可靠性,以Actin作为内参基因,验证结果如图4 所示。经过盐胁迫培养后,M.guilliermondii上述12个基因中有7个基因表达显著上调,5 个基因表达显著下调。RT-qPCR 定量分析显示,RT-qPCR验证结果与转录组测序结果中差异表达基因的变化趋势一致,说明此次转录组测序的可信度很高。
3 讨论与结论
本研究将M. guilliermondii 在盐胁迫(12%NaCl)与非盐胁迫(0%NaCl)条件下进行培养,并通过Illumina HiSeqTM高通量测序技术进行转录组测定。结果表明:盐胁迫(12%NaCl)与非盐胁迫(0%NaCl)下培养的M.guilliermondii 相比共有1 027 个显著性差异表达基因,其中458 个基因表达上调,569个基因表达下调。通过GO富集发现,经盐胁迫处理后M.guilliermondii 的差异表达基因主要富集在生物过程分类中,其中核苷酸代谢、糖代谢及辅酶代谢基因产生较大差异。对差异基因进行KEGG富集分析,结果大部分富集通路都与细胞的分裂、代谢有关,与GO 富集结果相对应。推测可能是因为盐胁迫促进了M.guilliermondii的生物代谢过程,提升了自身的复制与代谢效率,从而增强了M.guilliermondii 的抗逆性。MAPK 信号通路-酵母与酵母受到逆境胁迫的应激反应相关[18-20],对随机选取的MAPK 信号通路-酵母的12 个显著上/下调表达基因进行RT-qPCR验证,其结果与转录组测序结果趋于一致。以上转录组数据可以为盐胁迫培养对M.guilliermondii 的生长影响及进一步的生物学探究提供科学依据。
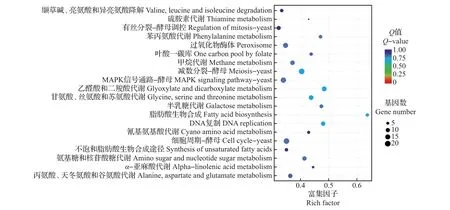
图3 差异表达基因的KEGG通路富集散点图Fig.3 Enrichment scatter plot of differential expression genes’ KEGG pathway
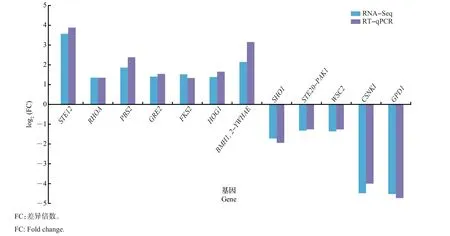
图4 基于实时荧光定量反转录PCR(RT-qPCR)的季也蒙毕赤酵母基因转录表达分析Fig.4 Transcriptional expression analysis of differential expression genes on M. guilliermondii detected by real-time quantitative reverse polymerase chain reaction(RT-qPCR)

