基于CD169 表达水平的小鼠脾红髓巨噬细胞分型研究
杨施琪,李梦瑶,刘思明,刘智多
上海交通大学医学院,上海市免疫学研究所,上海 200025
脾是人体最大的周围淋巴样器官,实质由红髓、白髓及其边界(称为边缘区)组成[1-2],是重要的次级免疫器官。小鼠脾内的巨噬细胞主要分为位于红髓的F4/80+CD11b-红 髓 巨 噬 细 胞(red pulp macrophage,RpMΦ)、位于边缘区的F4/80-/lowMARCO+边缘区巨噬细胞(marginal zone macrophage,MZMΦ)、F4/80-CD169+嗜金属巨噬细胞(marginal metallophilic macrophage,MMMΦ),以及白髓内的F4/80-CD68+着色小体巨噬细胞(tingible body macrophage,TBMΦ)[3-4]。因此,利用小鼠含生长因子样模体黏液样激素样受体(mouse EGFlike module-containing mucin-like hormone receptor-like 1,F4/80)和CD11b 可以很好地将RpMΦ 与其他脾巨噬细胞种群区别开。RpMΦ 分布在脾红髓中,其寿命长,从卵黄囊巨噬细胞或胚胎造血干细胞发育而来,长期表达于成年组织中,在稳定状态下骨髓来源的单核细胞对其产生的贡献很小,被认为是组织驻留巨噬细胞[5-8]。
与其他组织驻留巨噬细胞相比,RpMΦ 具有独特的 转 录 谱,Spic、Axl、F4/80、Cd68、Vcam1、Pecam1和Ccr3 等基因在RpMΦ 中高表达[9-12]。更重要的是,RpMΦ 对于机体十分关键,其主要功能包括:①吞噬流经红髓的衰老或受损红细胞[13-14],回收血红蛋白中的铁[6]。②产生Ⅰ型干扰素对抗寄生虫感染[15]。③通过分泌抑炎因子白介素-10(interleukin-10,IL-10)和转化生长因子-β(transforming growth factor-β,TGF-β),诱导调节性T 细胞(regulatory T cell,Treg)产生,从而调控过度的免疫反应[16]。由于RpMΦ 拥有诸多功能,对其展开的研究可加深人们对于脾内血源性感染以及某些自身免疫病及免疫缺陷病的理解。因此,对于RpMΦ 的功能及分型值得进一步研究。
CD169 分子最初发现于巨噬细胞中[17]。然而,CD169的功能至今仍未被完全阐明。一方面,CD169 可直接或间接地识别病原体结构,将病原体从血液中移除并递呈给其他免疫细胞[18]。另一方面,CD169 可与多种淋巴细胞进行唾液酸依赖性结合,介导细胞间的相互作用,调节免疫反应[19]。目前的研究[20]发现,CD169 除了在MMMΦ 上表达,也能够在部分RpMΦ 上表达,但该现象尚未被明确解释。因此,本研究利用流式细胞术和免疫荧光法2 种手段证实CD169 能够在RpMΦ 上表达,并通过对CD169+和CD169-RpMΦ 亚群进行RNA 测序,研究这2 个亚群在基因表达水平之间的差异,从而探索不同亚群可能具有的免疫学功能。
1 对象与方法
1.1 研究对象、试剂及仪器
1.1.1 实验动物 8 ~12 周龄、雌性、近交系C57BL/6J小鼠购于上海灵畅实验动物有限责任公司[生产许可证号为SCXK(沪)2018-0003,使用许可证号为SYXK(沪)2018-0027];向C57BL/6J 小鼠敲入CD169 Dtr (diphtheria toxin receptor)获得CD169 Dtr+/+小鼠[该纯合子小鼠无法表达CD169,被用来作为CD169 敲除(CD169 KO)小鼠,C57BL/6J 背景]购自日本 Riken 实验室(实验动物伦理批件号为A-2017-003),饲养于上海交通大学医学院实验动物中心屏障环境内。实验操作符合上海交通大学医学院动物伦理相关规范。
1.1.2 主 要 试 剂 小 鼠CD11b-APC/Cy7 抗 体(M1/70,BioLegend,美国),小鼠F4/80-FITC(fluorescein isothiocyanate)抗 体(BM8,BioLegend, 美 国), 小 鼠CD169-PE 抗体(SER-4,eBioscience,美国),Ⅳ型胶原酶(Sigma,美 国), 脱 氧 核 糖 核 酸 酶Ⅰ(Dnase Ⅰ,Roche, 美国),甲醇(上海大合化学品有限公司),4%多聚甲醛(paraformaldehyde,PFA,BBI 生命科学有限公司),蔗糖[生工生物工程(上海)股份有限公司],Mouse FITC 阳性结合试剂盒(Stemcell,美国),总RNA 抽提试剂TRIzol(Thermo Scientific,美国),胎牛血清(fetal bovine serum,FBS)、RPMI 1640 无血清基本培养基(Gibco,美国),SYBR Green Master(Toyobo,日本)。
1.1.3 主要仪器 超净工作台、细胞培养箱(Thermo Scientific,美国),磁力架(Stemcell,美国),LSRFortessa 分析型流式细胞仪、FACSAria Ⅱ流式细胞仪(BD,美国),PCR 仪(Eppendorf,德国),实时荧光定量PCR仪(ABI,日本),SP8 激光扫描共聚焦显微镜、Leica CM1950 冰冻切片机(Leica,德国),Illumina Hiseq X Ten测序仪(Illumina,美国)。
1.2 研究方法
1.2.1 小鼠RpMΦ 的获取及CD169 的检测 将小鼠的脾剪碎,用含0.1% Ⅳ型胶原酶和0.005% Dnase Ⅰ的RPMI 1640 培养基于37 ℃培养箱进行培养。随后,经45 min消化后,过70 μm 细胞滤网,于400×g 离心后弃上清液。加入5 mL 红细胞裂解液,室温静置5 min,加入15 mL 磷酸盐缓冲溶液(phosphate buffered saline,PBS)终止裂红。于400×g 离心弃上清液后,用 1 mL FACS buffer[1×PBS,2%牛血清白蛋白(bovine serum albumin,BSA)和 5 mmol/L 乙二胺四乙酸二钠]重悬,计数单细胞悬液。取1×106个细胞于每个染色组,加入F4/80、CD11b 及CD169 抗体,4 ℃孵育 30 min,FACS buffer 洗涤2 遍后,利用LSRFortessa 分析型流式细胞仪进行检测。
1.2.2 CD169+RpMΦ 的免疫荧光检测 颈椎脱臼法处死小鼠。取脾,先后放入1% PFA 和30%蔗糖溶液中固定及脱水,用OCT 包埋剂(optimal cutting temperature compound)包埋脾,-80 ℃保存。使用冰冻切片机制备脾薄片,切片厚度为20 μm。将带有组织切片的载玻片用PBS 清洗并于-20 ℃甲醇中通透30 min,经PBS 清洗 3 遍后用封闭液(0.3% Triton X-100,1% BSA,1% FBS 和 0.1 mol/L Tris-HCl)室温封闭1 h,加入F4/80 和CD169 抗体室温染色3 h,PBS 清洗后封片,在避光处晾干。组织切片用Leica SP8 激光扫描共聚焦显微镜在20 倍物镜下进行拍摄,以LIF 格式保存,用Imaris 软件进行图像处理。
1.2.3 小鼠脾F4/80+细胞分选 由于F4/80+CD11b-RpMΦ 在脾细胞中占比约为1%,为收集到足够数量的CD169+和CD169-亚群,本研究利用FITC+磁珠对红髓巨噬细胞进行富集。获得单细胞悬液后调整细胞浓度至2×108个/mL,加入Fc 封闭抗体于室温封闭10 min 后,加入F4/80-FITC 于4 ℃避光染色30 min。每2×108个细胞加入11.6 μL 分选液,室温孵育15 min,再加入6 μL磁珠室温孵育10 min。将装有细胞的圆底管置于磁力架上,通过磁力吸附收集获得F4/80+细胞。加入CD11b-APC/cy7、CD169-PE 抗体于4 ℃避光孵育30 min,利用FACSAria Ⅱ流式细胞仪得到小鼠脾CD169+/CD169-红髓巨噬细胞。
1.2.4 RNA 测序及数据处理 向CD169+/CD169-细胞中加入TRIzol 裂解液提取总RNA,每个亚型做2 次重复。RNA 测序由中国科学院上海生命科学研究院计算生物学研究所多维组学数据平台的Illumina Hiseq X Ten 测序仪完成,使用Qubit Fluorometer 和Agilent Bioanalyzer 2100 RNA 6000 Chip 2 种方法评估RNA 的完整性,RNA 完整值均大于7.1,说明完整性较好。利用TopHat2 软件将RNA 测 序 数 据 与GRCm38(genome reference consorium m38)(http://asia.ensembl.org/Mus_musculus/Info/Index)小鼠基因组进行比对,通过Cufflinks 软件对Mapped Reads进行拼接,并与原有的基因组注释信息进行比较。表达定量软件RSEM 用于对基因和转录本的表达水平进行定量分析,定量指标为FPKM (fragments per kilobase of transcript per million fragments mapped)。CD169+/CD169-亚群的差异基因由DESeq2 软件对原始计数进行分析。当P<0.05且|log2FC|≥1 时认为该基因表达差异显著。所有分析均基于美吉生物云平台(https://www.i-sanger.com/)进行。
1.2.5 数据分析 对CD169+、CD169-2 个亚群细胞基因集进行分析,韦恩图显示亚群间共有或特有的表达基因数目。主成分分析(principal component analysis,PCA)体现出样本间的相似性;各个样本点的距离代表了样本的距离,距离越近表明样本间相似性越高。热图是对2 个亚群细胞差异表达的基因进行表达模式聚类分析。京都基因与基因组百科全书(Kyoto Encyclopedia of Genes,KEGG)功能分析用于系统分析基因功能,可将基因集中的基因按照参与的通路或行使的功能分类[21]。
1.2.6 qPCR 验证差异表达基因 将CD169+和CD169-红髓巨噬细胞的RNA 用HifairTMⅡ 1st Strand cDNA Synthesis SuperMix 即用型预混液反转录得到cDNA,选取8 个KEGG 通路分析中富集的差异表达基因进行qPCR 验证,结果分析用2-ΔΔCT法。所用的引物序列(表1)通过PrimerBank 搜索得到。
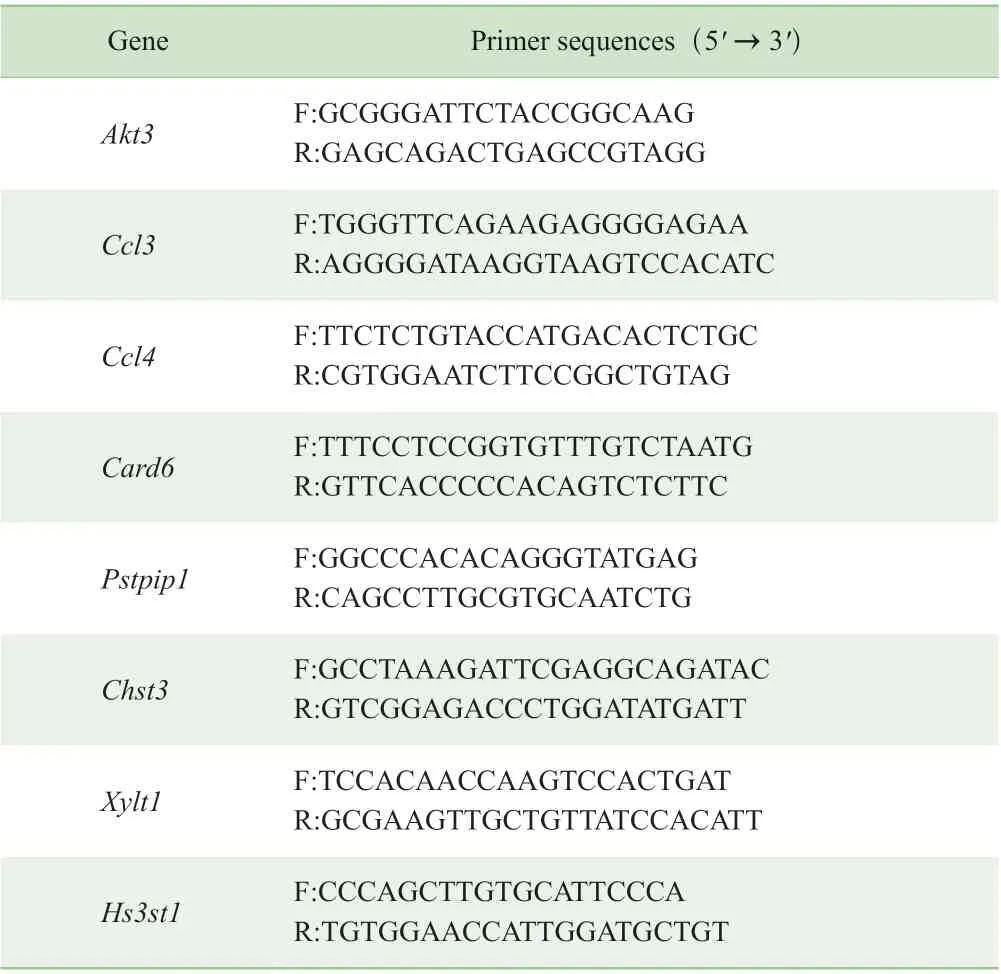
表1 qPCR 所用的引物序列Tab 1 Sequences of primers for qPCR
1.3 统计学方法
采用 GraphPad Prism 7.0 软件对研究数据进行统计学分析及作图。定量资料以±s 表示,采用t 检验或校正t 检验进行CD169+和CD169-亚群细胞比例的差异比较。P<0.05 表示差异具有统计学意义。
2 结果
2.1 部分RpMΦ 表达CD169
为了验证野生型(wild type,WT)小鼠部分RpMΦ表达CD169 分子,本研究先利用流式细胞术检测WT小鼠和CD169 KO 小鼠脾中RpMΦ 的比例,再比较RpMΦ 中CD169 分子的表达情况。根据已有的研究[22-23]表明,F4/80+CD11b-可作为RpMΦ 的表面标志物。因此,如流式细胞术策略(图1A)所示,在圈选了脾细胞并去黏连后,F4/80+CD11b-细胞群即为RpMΦ。结果显示,CD169 KO 小鼠脾内RpMΦ 的比例与WT 小鼠相似(图1B),2 组小鼠RpMΦ 占脾细胞的比例均在0.5%~1.5%,差异无统计学意义(图1C);但以CD169 KO 小鼠作为阴性对照,在WT 小鼠RpMΦ 中可明显地观察到部分RpMΦ 表达CD169 分子(图1B、D)。
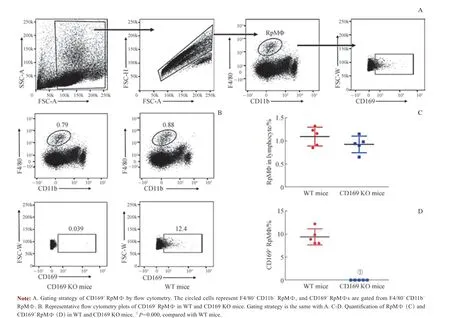
图1 WT 小鼠和CD169 KO 小鼠中CD169+ RpMΦ 比例Fig 1 Proportion of CD169+ RpMΦ in WT and CD169 KO mice
2.2 CD169+ RpMΦ 的空间分布
为进一步确认CD169 在小鼠脾RpMΦ 中的表达,本研究利用免疫荧光技术对脾RpMΦ 上CD169 表达情况进行检测,结果(图2)显示,在白色虚线内侧边缘(边缘区)存在一圈高表达CD169(红色)的MMMΦ;在脾红髓中CD169 呈散在分布,并与部分F4/80+的RpMΦ 有很好的共定位。由此再次证实,部分RpMΦ 可表达CD169分子。同时,结果还发现F4/80 主要分布于圆形区域以外的红髓区域,在边缘区有微量的表达,但其荧光强度明显低于红髓区域。
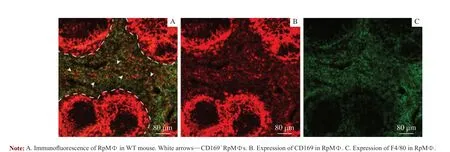
图2 免疫荧光法检测WT 小鼠脾内CD169+ RpMΦ 分布(×20)Fig 2 Locolization of CD169+ RpMΦ of spleen in WT mouse by immunofluorescence (×20)
2.3 CD169+和CD169-红髓巨噬细胞差异表达基因分析
为探究CD169+RpMΦ 与CD169-RpMΦ 在基因表达水平的差异,本研究通过富集RpMΦ,分选出CD169+和CD169-2 个亚群,提取RNA 并进行测序分析。在CD169+RpMΦ 群中检测到9 234 个基因,CD169-RpMΦ群中检测到9 812 个基因,其中共同表达的基因有8 866个(图3A)。主成分分析显示,2 群细胞可以被很好地区分开(图3B)。对差异基因进行聚类分析显示,共有485个差异表达基因,且相比于CD169+RpMΦ,在CD169-RpMΦ 中的上调基因为427 个,下调基因为58 个(图3C)。如表达量差异散点图所示,Siglec1(编码CD169的基因)在CD169+RpMΦ 群中高表达(图3D),认为RNA 测序结果与分选策略一致;同时,2 群细胞共同表达小鼠脾RpMΦ 的特征基因,如Vcam1、Axl、Spic、Cd68和Ccr3 等,表明这2 群细胞均属于RpMΦ。
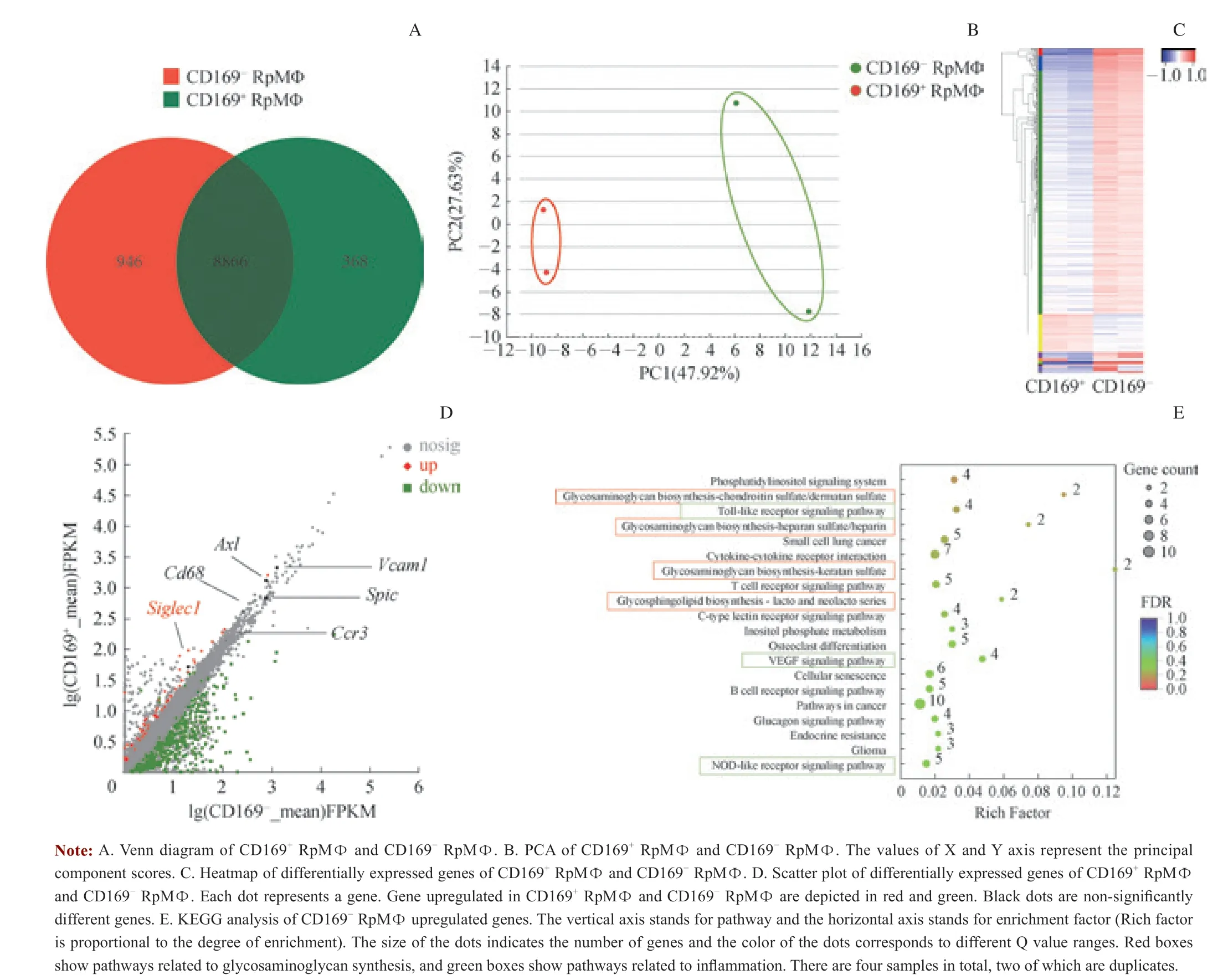
图3 小鼠CD169+ RpMΦ 和CD169- RpMΦ 差异表达基因分析Fig 3 Analysis of differentially expressed genes in CD169+ RpMΦ and CD169- RpMΦ
2.4 CD169-红髓巨噬细胞的促进炎症作用
通过对上述RNA 测序的数据分析可知,与CD169+RpMΦ 群相比,在CD169-RpMΦ 群中上调的基因数比下调的基因数多。因此,我们将重点放在CD169-RpMΦ 中上调的基因集上,并对该基因集进行KEGG 功能分析。结果(图3E)显示,差异表达基因在介导炎症(绿色方框显示)和糖胺聚糖(glycosaminoglycans,GAGs)生物合成(红色方框显示)的通路中显著富集,提示CD169-RpMΦ与CD169+RpMΦ 的功能有所不同。其中与介导炎症相关的通路包括Toll 样受体(Toll-like receptors,TLRs)信号通路、血管内皮生长因子(vascular endothelial growth factor,VEGF) 信 号 通 路 及NOD 样 受 体(nucleotide binding oligomerization domain-like receptors,NLRs)信号通路。与GAGs 生物合成相关的通路包括硫酸软骨素/硫酸 皮 肤 素(chondroitin sulfate/dermatan sulfate,CS/DS)、硫酸乙酰肝素/肝素(heparan sulfate/heparin,HS/H)、硫酸角质素(keratan sulfate,KS)以及乳酸。对GAGs 生物合成相关通路中高表达的基因进行分析,发现这些基因均用于编码糖基或磺基转移酶。而目前关于GAGs 的研究表明,这些GAGs 修饰酶的基因可以被促炎性细胞因子上调表达进而改变GAGs 的表面结构。这些结构的改变可以为不同种类的分子(包括炎症介质)提供大量的结合位点,从而促进炎症进程[24]。结合CD169-RpMΦ 还在炎症相关通路中富集,我们推测CD169-RpMΦ 可能与介导炎症 有关。
为了进一步验证RNA-Seq 的结果,我们挑选了8 个在上述多种通路中的富集基因(表2)。采用qPCR 分别检测了GAGs 生物合成通路中的 Chst3、Hs3st1 和Xylt1,以及TLR 信号通路、VEGF 信号通路及NLR 信号通路中的Card6、Pstpip1、Akt3、Ccl3 和Ccl4 的表达水平。结果(图4)显示,与CD169+RpMΦ 群相比,上述8 个基因在CD169-RpMΦ 群中显著上调。
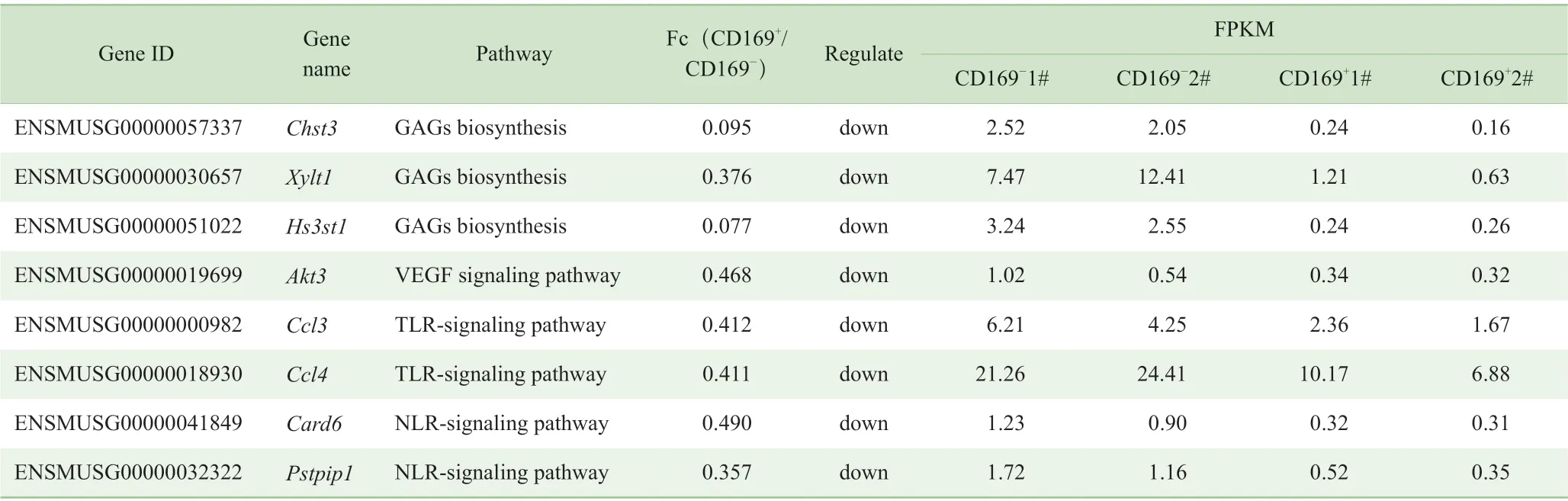
表2 CD169- RpMΦ 亚群中不同通路的差异表达基因Tab 2 Differentially expressed genes in different pathways of CD169- RpMΦ
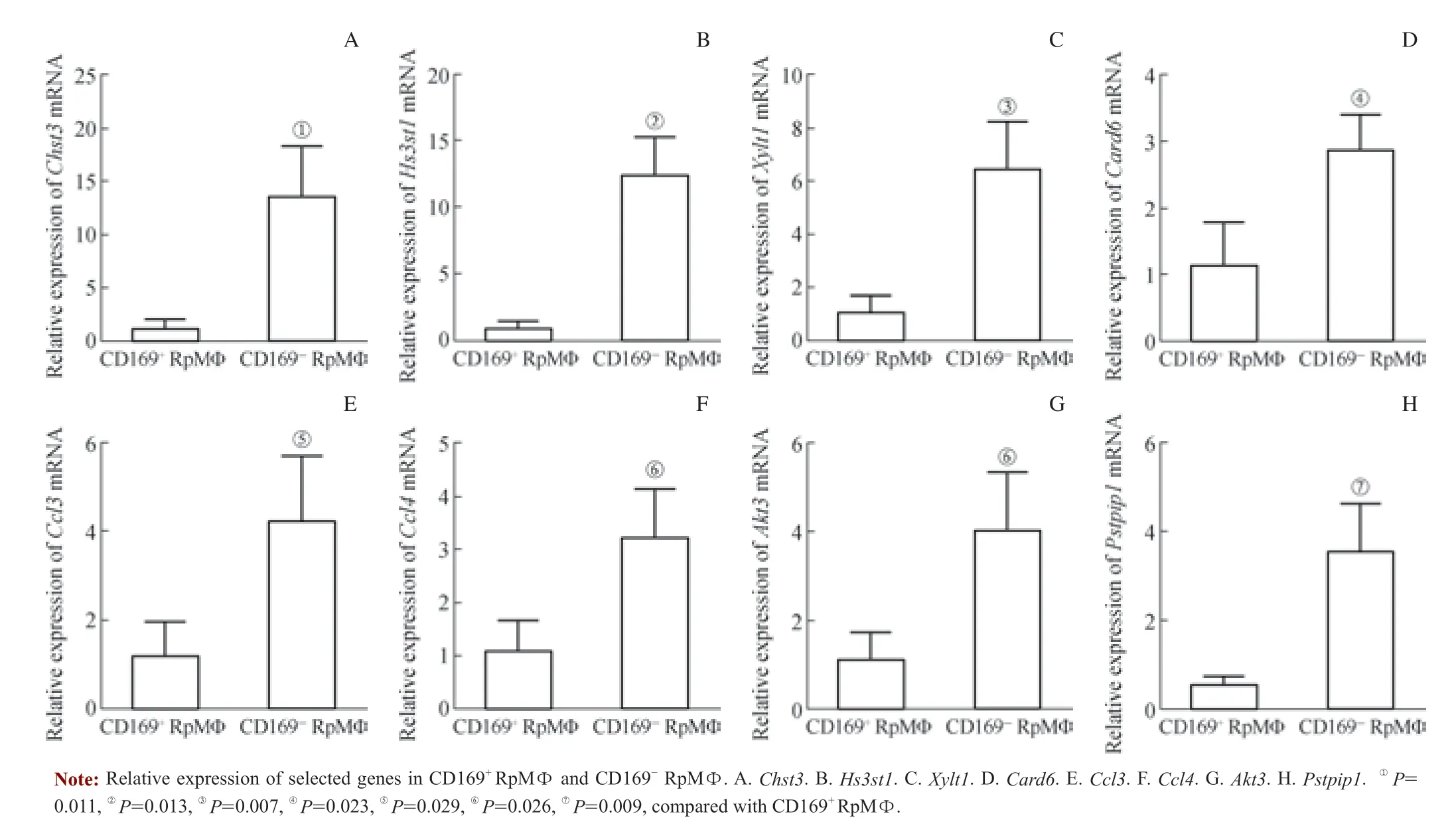
图4 qPCR 证实部分RNA 测序结果中的差异表达基因Fig 4 Validation of differentially expressed genes in RNA-sequencing by qPCR
3 讨论
组织驻留巨噬细胞的异质性一直是免疫学研究的热点问题。即使目前巨噬细胞已获得大致分类,由于组织驻留巨噬细胞在同一个组织内占据着多个局部微环境,在已知的巨噬细胞种群中仍不断发现具有微环境特异性表型和功能的亚群。例如近期有研究指出肺间质巨噬细胞(interstitial macrophages,IMs)仍存在异质性,它们可以分成2 群具有不同基因表达谱和表型的亚群,一组与伤口愈合有较高的关联性,另一组则具有更强的抗原递呈能力。在心脏、脂肪和真皮中也存在类似的亚群[25]。
鉴于RpMΦ 是一群拥有清除破损红细胞、调节铁循环、介导炎症反应、抵抗寄生虫感染及调节免疫反应等复杂功能的细胞群,我们猜测RpMΦ 中可能也存在不同分工的亚群。CD169 分子在限制病毒扩散[26]、介导T 细胞反应[27]和调节免疫反应[28]等方面有重要的作用。本研究利用流式细胞术和免疫荧光法证实了CD169 在部分RpMΦ 中表达。因此我们推测存在以CD169 表达量分型的RpMΦ 亚群,它们以互补的方式参与控制感染并诱导有效的免疫反应。
通过对于RNA 测序数据的进一步分析,本研究发现在CD169-RpMΦ 中高表达的且能够在介导炎症及GAGs生物合成相关通路中富集的差异表达基因,多与招募炎症细胞、推进免疫进程有关。例如,在GAGs 生物合成通路中富集的基因Chst3 是引起炎症和白细胞趋化的一种碳水化合物磺基转移酶。它可合成6-硫酸软骨素,而硫酸软骨素蛋白聚糖是重要的炎症介质。有研究[29]发现,利用小干扰RNA 使Chst3 沉默后,肺泡灌洗液和肺实质中巨噬细胞的数量减少,同时肿瘤坏死因子α(tumor necrosis factor-α, TNF-α)和基质金属蛋白酶-9(matrix metalloprotein-9,MMP-9)的表达被抑制。类似地,在TLRs 信号通路中富集的Ccl3 和Ccl4 基因能够编码免疫细胞招募的分子巨噬细胞炎症蛋白1α/β(macrophage inflammatory protein-1α/β,MIP-1α/β)。而MIP-1α/β 在 诱导Th1 型反应后会被大量释放[30-31],与T 细胞、树突状细胞、B 细胞和嗜酸性粒细胞上的CC 趋化因子受体1(CC chemokine receptors 1,CCR1)、CCR4 和CCR5 受 体 结合,招募免疫细胞浸润,是炎症反应期间募集细胞的重要组成部分[32]。这些分析结果均指向CD169-RpMΦ 拥有介导炎症、招募免疫细胞的能力。
巨噬细胞被广泛认为可以分为M1 MΦ 和M2 MΦ 2 种极化类型。巨噬细胞在受到脂多糖(lipopolysaccharide,LPS)、干 扰 素-γ(interferon-γ,IFN-γ)及 粒 细 胞- 巨噬细胞集落刺激因子(granulocyte-macrophage colony stimulating factor,GM-CSF)的刺激后可极化为M1 MΦ,产生炎性细胞因子,例如IL-1β、IL-6、IL-12、IL-23 和TNF-α 等,从而介导炎症,引起组织损伤。另一方面,巨噬细胞还可以被IL-4 和IL-13 转变为M2 MΦ,产生抗炎性细胞因子IL-10 和TGF-β,以限制炎症发展、促进组织修复[33]。通过与小鼠M1 和M2 巨噬细胞中存在的特征差异表达基因集[34]进行比对,我们发现CD169-RpMΦ 中无M2 型特征基因,但有6 个M1 型特征基因,它们分别是Gpr18、Itgal、Oasl1、H2-Q6、H2-T10 和Arhgap24。 结合上述对于单个通路内上调的基因的分析,本研究的结果提示CD169-RpMΦ 是一群拥有M1 倾向的RpMΦ。
关于RpMΦ 亚群的研究仍存在许多问题亟待解决。首先,现已知组织特异性转录因子和谱系特异性转录因子共同形成了控制网络,控制组织驻留巨噬细胞的基因表达,从而塑造了不同巨噬细胞亚群的独特表达谱[35],但对于影响RpMΦ 亚群表型至关重要的脾微环境因素仍不明确。通过将蛋白质组学、代谢组学、基因组学及表观基因组学与计算机模拟算法相结合,可以极大地帮助我们了解RpMΦ 生物功能的全貌。其次,关于CD169-RpMΦ 的功能目前只在基因表达水平上进行了检测,还需要更多体内和体外的功能实验证实本文关于CD169-RpMΦ 数据的分析结论。
结合RNA 测序结果,CD169-RpMΦ 中部分GAGs修饰酶的表达量上调。而目前已知M1/M2 型巨噬细胞中许多GAGs 修饰酶的表达发生改变,并伴随独特结构特征的GAGs 表达[36]。这说明GAGs 修饰酶可能通过改变GAGs 的结构诱导炎症因子的释放,或通过调节它们与靶膜上同源受体的结合,影响巨噬细胞的可塑性。GAGs 的结构重塑是炎症发生的原因还是后果仍不清楚。如果是后者,通过抗炎治疗可以恢复正常的聚糖结构和功能。相反,如果GAGs 的生物合成失调直接与疾病发生相关,则可以将GAGs 修饰酶作为疾病治疗的潜在靶点。
综上所述,通过流式细胞术和免疫荧光法2 种手段证实,在小鼠脾中有部分RpMΦ 表达CD169。随后,我们根据CD169 表达量将RpMΦ 分为2 个亚群后发现,与CD169+RpMΦ 相比,CD169-RpMΦ 参与介导炎症反应,具有M1 MΦ 倾向。RpMΦ 对于多种细菌或寄生虫感染、疾病的病理变化和组织重构过程有着重要的作用。因此,RpMΦ 异质性的研究结果将更好地揭示免疫系统的协同工作,从而为人工干预免疫反应提供临床指导依据。
参·考·文·献
[1] Mebius RE, Kraal G. Structure and function of the spleen[J]. Nat Rev Immunol, 2005, 5(8): 606-616.
[2] den Haan JM, Mebius RE, Kraal G. Stromal cells of the mouse spleen[J]. Front Immun, 2012, 3: 201.
[3] Kurotaki D, Uede T, Tamura T. Functions and development of red pulp macrophages[J]. Microbiol Immunol, 2015, 59(2): 55-62.
[4] dos Anjos Cassado A. F4/80 as a major macrophage marker: the case of the peritoneum and spleen[M]//Kloc M. Results and problems in cell differentiation. Cham: Springer International Publishing, 2017: 161-179.
[5] Davies LC, Jenkins SJ, Allen JE, et al. Tissue-resident macrophages[J]. Nat Immunol, 2013, 14(10): 986-995.
[6] Haldar M, Kohyama M, So AY, et al. Heme-mediated SPI-C induction promotes monocyte differentiation into iron-recycling macrophages[J]. Cell, 2014, 156(6): 1223-1234.
[7] Hashimoto D, Chow A, Noizat C, et al. Tissue-resident macrophages selfmaintain locally throughout adult life with minimal contribution from circulating monocytes[J]. Immunity, 2013, 38(4): 792-804.
[8] Murray PJ, Wynn TA. Protective and pathogenic functions of macrophage subsets[J]. Nat Rev Immunol, 2011, 11(11): 723-737.
[9] Lavin Y, Mortha A, Rahman A, et al. Regulation of macrophage development and function in peripheral tissues[J]. Nat Rev Immunol, 2015, 15(12): 731-744.
[10] Gautier EL, Shay T, Miller J, et al. Gene-expression profiles and transcriptional regulatory pathways that underlie the identity and diversity of mouse tissue macrophages[J]. Nat Immunol, 2012, 13(11): 1118-1128.
[11] Okabe Y, Medzhitov R. Tissue-specific signals control reversible program of localization and functional polarization of macrophages[J]. Cell, 2014, 157(4): 832-844.
[12] Perez OA, Yeung ST, Vera-Licona P, et al. CD169+macrophages orchestrate innate immune responses by regulating bacterial localization in the spleen[J]. Sci Immunol, 2017, 2(16): eaah5520.
[13] Kohyama M, Ise W, Edelson BT, et al. Role for Spi-C in the development of red pulp macrophages and splenic iron homeostasis[J]. Nature, 2009, 457(7227): 318-321.
[14] Taylor PR, Martinez-Pomares L, Stacey M, et al. Macrophage receptors and immune recognition[J]. Annu Rev Immunol, 2005, 23(1): 901-944.
[15] Kim CC, Nelson CS, Wilson EB, et al. Splenic red pulp macrophages produce type Ⅰ interferons as early sentinels of malaria infection but are dispensable for control[J]. PLoS One, 2012, 7(10): E48126.
[16] Kurotaki D, Kon S, Bae K, et al. CSF-1-dependent red pulp macrophages regulate CD4 T cell responses[J]. J Immunol, 2011, 186(4): 2229-2237.
[17] Crocker PR, Gordon S. Properties and distribution of a lectin-like hemagglutinin differentially expressed by murine stromal tissue macrophages[J]. J Exp Med, 1986, 164(6): 1862-1875.
[18] van Dinther D, Veninga H, Iborra S, et al. Functional CD169 on macrophages mediates interaction with dendritic cells for CD8+T cell cross-priming[J]. Cell Rep, 2018, 22(6): 1484-1495.
[19] Wu C, Rauch U, Korpos E, et al. Sialoadhesin-positive macrophages bind regulatory T cells, negatively controlling their expansion and autoimmune disease progression[J]. J Immunol, 2009, 182(10): 6508-6516.
[20] Klaas M, Oetke C, Lewis LE, et al. Sialoadhesin promotes rapid proinflammatory and type Ⅰ IFN responses to a sialylated pathogen, Campylobacter jejuni[J]. J Immunol, 2012, 189(5): 2414-2422.
[21] Kanehisa M, Goto S, Sato Y, et al. KEGG for integration and interpretation of large-scale molecular data sets[J]. Nucleic Acids Res, 2012, 40(Database issue): D109-D114.
[22] A-Gonzalez N, Castrillo A. Origin and specialization of splenic macrophages[J]. Cell Immunol, 2018, 330: 151-158.
[23] Taylor PR, Brown GD, Geldhof AB, et al. Pattern recognition receptors and differentiation antigens define murine myeloid cell heterogeneity ex vivo[J]. Eur J Immunol, 2003, 33(8): 2090-2097.
[24] Groux-Degroote S, Cavdarli S, Uchimura K, et al. Glycosylation changes in inflammatory diseases[J]. Adv Protein Chem Struct Biol, 2020, 119: 111-156.
[25] Chakarov S, Lim HY, Tan L, et al. Two distinct interstitial macrophage populations coexist across tissues in specific subtissular niches[J]. Science, 2019, 363(6432): eaau0964.
[26] Sewald X, Ladinsky MS, Uchil PD, et al. Retroviruses use CD169-mediated trans-infection of permissive lymphocytes to establish infection[J]. Science, 2015, 350(6260): 563-567.
[27] van Dinther D, Lopez Venegas M, Veninga H, et al. Activation of CD8+T cell responses after melanoma antigen targeting to CD169-antigen presenting cells in mice and humans[J]. Cancers (Basel), 2019, 11(2): E183.
[28] Wu Y, Lan C, Ren DR, et al. Induction of siglec-1 by endotoxin tolerance suppresses the innate immune response by promoting TGF-β1 production[J]. J Biol Chem, 2016, 291(23): 12370-12382.
[29] Kai Y, Tomoda K, Yoneyama H, et al. RNA interference targeting carbohydrate sulfotransferase 3 diminishes macrophage accumulation, inhibits MMP-9 expression and promotes lung recovery in murine pulmonary emphysema[J]. Respir Res, 2015, 16: 146.
[30] Canque B, Rosenzwajg M, Gey A, et al. Macrophage inflammatory protein-1alpha is induced by human immunodeficiency virus infection of monocytederived macrophages[J]. Blood, 1996, 87(5): 2011-2019.
[31] Lukacs NW, Strieter RM, Elner VM, et al. Intercellular adhesion molecule-1 mediates the expression of monocyte-derived MIP-1 α during monocyteendothelial cell interactions[J]. Blood, 1994, 83(5): 1174-1178.
[32] Annunziato F, Galli G, Nappi F, et al. Limited expression of R5-tropic HIV-1 in CCR5-positive type 1-polarized T cells explained by their ability to produce RANTES, MIP-1α, and MIP-1β[J]. Blood, 2000, 95(4): 1167-1174.
[33] Borges da Silva H, Fonseca R, Pereira RM, et al. Macrophage plasticity, polarization, and function in health and disease[J]. Front Immunol, 2015 (9), 22;6: 480.
[34] Jablonski KA, Amici SA, Webb LM, et al. Novel markers to delineate murine M1 and M2 macrophages[J]. PLoS One, 2015, 10(12): E0145342.
[35] Lavin Y, Winter D, Blecher-Gonen R, et al. Tissue-resident macrophage enhancer landscapes are shaped by the local microenvironment[J]. Cell, 2014, 159(6): 1312-1326.
[36] Martinez P, Denys A, Delos M, et al. Macrophage polarization alters the expression and sulfation pattern of glycosaminoglycans[J]. Glycobiology, 2015, 25(5): 502-513.

