近红外判别结合COI序列基因分析鉴定地龙物种基源
吴怡青 赵崇军 张文婷 赵霞 顾念念 孟雪丹 林瑞超 邹迪新


摘要 目的:建立地龙药材的真伪鉴别方法,为地龙药材优质用药提供新方法。方法:采用COI序列进行分子生物学层面的基因序列分析,近红外二级光谱判别建模分析进行光谱学层面的分析,两者共同鉴定地龙药材的真伪。结果:COI序列分析能够鉴别地龙种属,明确药材的基源,近红外建模分析结果与COI序列分析结果一致。结论:近红外判别与COI基因分析结合能够简便,全面的控制地龙药材真实性,明确药材基源。
关键词 地龙;COI序列;近红外建模;真伪鉴别;物种基源
Abstract Objective:To establish the authenticity identification method of Dilong medicinal materials,and to provide new methods for high-quality medication of Dilong medicinal materials.Methods:The COI sequence was used to analyze the gene sequence at the molecular biology level,and the near-infrared secondary spectrum discrimination modeling analysis was used to analyze the spectroscopy level.The 2 jointly identified the authenticity of the Dilong medicinal materials.Results:COI sequence analysis can identify the species of Dilong,identify the source of the medicinal materials,and the results of near infrared modeling analysis are consistent with the results of COI sequence analysis.Conclusion:Mutual verification shows that the combination of the 2 can be simple and comprehensively control the authenticity of Dilong medicinal materials,clarify the source of medicinal materials.
Keywords Dilong; COI sequence; Near infrared modeling; Authenticity identificat-ion; Species origin
中圖分类号:R282.5文献标识码:Adoi:10.3969/j.issn.1673-7202.2020.13.006
地龙为常见的平肝熄风类中药,《中华人民共和国药典》2015版规定:“蚓科动物参环毛蚓Pheretima aspergillum(E.Perrier)的干燥体称之为广地龙,通俗环毛蚓Pheretima vulgaris Chen、威廉环毛蚓Pheretima guillelmi(Michaelsen)、栉盲环毛蚓Pheretima pectinifera Michaelsen的干燥体称为沪地龙”。动物药分子生物学鉴定的研究对象一般选择线粒体基因[1],其可作为遗传信息的载体,用于研究动物种群起源以及进化。COI序列具有“变异少,易扩增,且不易发生碱基缺失和插入”的特性,国际上,一般将其作为研究动物类样品的通用序列。本实验基于COI引物稳定的特点,探究其对于地龙种属鉴别的优势。建立地龙物种鉴别的分子生物学方法。同时基于近红外光谱可应用广泛,可用于含氢基团的检测,同时具有简便、易操作的特性,根据其二维光谱分析结果建立的模型可用于药材真伪初步鉴别,可提高中药研究人员的工作效率,采用2种方法对地龙药材的真伪进行鉴别,互相验证,共同保证药材的基源真实性。
1 材料与方法
1.1 材料
实验试剂:琼脂糖凝胶(Sigma-Aldrich,批号:102012071);TAE buffer(北京兰博利德商贸有限公司,批号:15558026);Dneasy血液及组织核酸提取试剂盒(天根生化科技有限公司,批号:160024812);无水乙醇(北京化工有限公司,批号:bh100488);引物:HCO2198、LCO1490(北京议达成科技有限公司,批号:QPC2462-250);Taq扩增酶(美康仪器设备北京有限公司,批号:4398813);DNA maker(北京博迈德基因技术有限公司,批号:MD301-01);无菌双蒸水(上海源叶生物有限公司,批号:R21481);核酸染料GeneGreen(天根生化科技有限公司,批号:R6628);2号筛;40目筛(赛谱锐思北京科技有限公司,批号:YDS6)。电子分析天平(梅特勒-托利多仪器有限公司,型号:MS105DU);高速万能粉碎机(北京科伟永兴仪器有限公司,型号:FW-200型);高速离心机(Thermo Fisher,型号:STRATOS);电子恒温水浴锅(赛谱锐思北京科技有限公司,型号:SEP-1122-5);格兰仕微波炉(广州格兰仕微波生活电器制造公司,型号:G70D20CN1P-D2);涡旋混合仪(天根生化科技北京有限公司,型号:OSE-VX-02);高压蒸汽灭菌锅(Panasonic,型号:MLS-3751L-PC);PCR扩增仪(德国艾本德公司Eppendorf AG,型号:eppendorf);电泳槽、电泳仪(北京艾科仪器有限公司,型号:DYCP-31N型);凝胶成像仪(英国伯乐公司BIO-RAD,型号:GeL Doc XR+);近红外光谱仪(美国尼高力有限公司,型号:A020FT-IR);ContigExpress软件、DNAman软件、Editsep软件、MEGA 6.0软件、OMNIC软件、SMICA软件用于数据分析处理。本研究共收集地龙药材及其混伪品共27批,经北京中医药大学杨瑶君教授鉴定,其中S1-S12为市场上购买,S14-S28为陕西步长制药有限公司提供,具体样品信息见表1。
1.2 方法
1.2.1 COI序列分析
1.2.1.1 地龙组织中模板DNA的提取
取地龙药材组织、洗净、去除泥沙,挑选药材组织中肌肉组织较多者为优,采用DNA提取试剂盒上的规定方法进行操作。具体步骤如下:1)剪取地龙药材组织25 mg,以肌肉组织较多者为优,放于1.5 mL离心管中,加入180 μL的Buffer ATL,20 μL的蛋白酶K,涡旋均匀,56 ℃下孵育直到其完全裂解。2)加入200 μL的Buffer AL,涡旋混匀。3)加入200 μL的无水乙醇,涡旋混匀(沉淀DNA)。4)吸取混合液过吸附柱后放置于2 mL的收集管中,8 000 r/min转速,离心半径35 cm,离心时间为1 min,丢弃废液。5)将吸附柱放于2 mL的收集管中,加入500 μL Buffer AW1,8 000 r/min转速,离心半径35 cm,离心时间为1 min,丢弃废液。6)将吸附柱放置于2 mL的收集管中,加入500 μL Buffer AW2,14 000 r/min转速,离心半径10 cm,离心时间为3 min,丢弃废液。7)将吸附柱转移于新2 mL的离心管中。8)在吸附柱膜中心加入200 μL的Buffer AE洗脱DNA,室温下孵育1 min,8 000 r/min的转速,离心半径35 cm,离心时间为1 min。9)将步骤8重复3次获取地龙模板DNA。
1.2.1.2 PCR扩增
COI正向引物HC02198:5′-TAAACTTCAGGGTGACCAAAAAATCA-3′;反向引物LC01490:5′-GGTCAACAAATCATAAAGATATTGG-3′,將提取的地龙模板DNA经COI的正反引物序列进行PCR扩增处理,扩增体系为50 μL,反应体系见表2,PCR反应条件见表3。
提取出来的地龙模板DNA经过引物扩增后,扩增产物是否产生以及供测序分析处理,需要进行初步判定。将PCR扩增产物使用1%琼脂糖凝胶进行跑胶处理后,观察电泳条带进行分析,电泳仪的参数设置:电压为140 V,电泳时间为25 min,扩增产物的凝胶电泳图谱如图1所示,条带明亮清晰,可用于样品的测序分析。
1.2.1.3 切胶纯化测序
在紫外灯照射下观察,选择含有目的基因DNA片段中比较亮的条带进行切割,切割过程中要求尽量准确,提高其回收率,上测序仪进行双向测序。
1.2.1.4 地龙样品序列的数据处理
测序结果经ContigExpress软件拼接并更改错误碱基,剪除基因片段的引物区及低质量部分,获得长度为580个碱基的样品序列,修剪过的样品序列经BLAST检索对比分析后根据相似匹配度大小获得初步结论[2]。在BLAST上选取相似度最高、覆盖度较大、得分较高的完整序列,用DNAman软件将样品序列和完整序列进行多序列比对,选择样品序列中99%的序列,生成样品序列的反向互补序列,用Editsep软件将截取的样品序列与由其生成的反向互补序列进行合并,生成新的样品序列,再次进行BLAST检索对比,得到最终结果。以MEGA 6.0软件为分析平台,构建地龙样品的系统发育树,利用Bootstrap(1 000次重复)检验各分支的支持率。
1.2.2 近红外建模分析
取过40目筛的地龙粉末均匀分布于样品池中,将样品池放置于近红外光谱仪上,采集样品原始光谱,波数范围为12 000~4 000 cm-1,分辨率为4 cm-1,扫描次数为64次,每批样品采集3份光谱图,药材共收集60份正品光谱,15份伪品光谱。样品的近红外数据分析需要取3份光谱的平均光谱,随机抽取其中的48份正品光谱建立地龙近红外光谱的鉴别模型,剩余正品和伪品则用作验证集,目的是为了评价所建立模型的准确性和有效性。
2 结果
2.1 地龙COI分析
2.1.1 COI序列分析
收集市场上地龙药材27批,正品22批,混伪品为5批,经分子生物学鉴定后地龙种属鉴别结果见表4,其中,S2、S3、S5、S8、S9、S10为参状环毛蚓;S1、S6、S7、S14、S16、S17、S19、S20、S21、S22、S23、S24、S25、S26、S27、S28为通俗环毛蚓,其余均为伪品。观察物种鉴定分析结果,发现COI通用序列可以准确鉴别地龙的种属,为地龙的基源鉴别提供了科学的分子生物学鉴定方法。
2.1.2 COI序列多态性分析
地龙共有27条样品序列,参状环毛蚓6条样品序列,经DNAman软件进行多序列比对后发现共有6个单倍型,含有145个变异位点,一致性分析结果为73.49%,分析结果见图2;通俗环毛蚓有16条样品序列,每条样品序列都存在不相似位点[3],共有16个单倍型,经软件比对后发现有135个变异位点,一致性分析结果为75.36%,分析结果见图3;分析地龙样品不同物种的种间变异情况,将27批地龙样品序列导入DNAman软件进行多态性比对,种间变异一致性分析结果为66.87%,变异位点为192个,结果见图4。综上:地龙样品的种内变异一致性结果均大于种间变异,说明此COI序列作为地龙物种的条形码能够很好的鉴别不同种属的分布情况。
2.1.3 地龙遗传邻接系统聚类树的建立
一般系统发育用于描述物种的起源和进化情况,发育树作为载体可以将其过程生动形象的展现出来,进化树由结点和进化分支组成,每个结点表示一个分类学单位,分支表示亲代与后代的关系,分支长度表示进化过程的变化程度。通过邻接法(N-J法)构建的发育树具有分析准确,计算速度快,只得一棵树的显著优势。基于COI序列扩增的地龙样品序列,经N-J法构建的系统聚类树图。见图5。观察建立的发育树可得:通俗环毛蚓基本可成为独立一支,其支持率为99%,而参状环毛蚓与其他混伪品由同一结点,说明其来源于同一祖先,可用于地龙各种属之间的亲缘关系的推测。综上:COI序列可用于地龙正品、混伪品的鉴别,为该药材的物种鉴定提供了新的分子生物学方法。
2.2 近红外建模分析
2.2.1 正品与伪品近红外光谱差异图谱
以波数Wavenumber(cm-1)为横坐标,吸光度Absorbance为纵坐标,在10 000~4 000 cm-1波数范围内观察地龙正伪品的近红外光谱图,正品地龙的近红外原始光谱见图6,伪品见图7。用OMNIC软件对地龙正品和伪品药材的近红外原始光谱进行分析,发现在10 000~4 000 cm-1的近红外光谱图有明显差异,吸光度差异有统计学意义。
2.2.2 光谱预处理
近红外光谱包含了样品的绝大多数的化学信息,光谱区的信息重叠严重,需要借助一定的数据预处理方法和建模方法才可以进行定性分析。近红外光谱的原始光谱由样品的真实化学信息投射光谱和不确定的背景吸收组成,同时中药的成分众多、组成复杂,因此很难从药材的近红外原始光谱中寻找特征吸收谱带去进行差异性分析,故需要对光谱进行预处理,提取光谱中与待测量有关的信息,保证不同样品间的信息差异能更好的体现。近红外光谱的常见的预处理方式为:平滑处理和导数处理,平滑处理可以降低仪器的噪声,提高信噪比;导数处理可以减少原始光谱的基线偏移和光谱偏移,其中,一阶导数可以消除光谱中的扁平基线以减少光谱信息的不确定性,二阶导数消除光谱中的倾斜基线,帮助在复杂的峰型中找到峰的准确位置[4]。选择的光谱预处理方法[5-6]为SNV(标准正态变量校正)+Savitzky-Golay平滑模式(11点平滑)+二阶导数的方式进行光谱预处理,原始图谱经处理后得到的光谱。见图8。
2.2.3 主成分分析
主成分分析的原理:将样品中的影响变量进行降维处理后获得主要变量,然后进行数据的拟合或根据变量相似点进行聚合[7]。将地龙药材的原始近红外光谱图导入SMICA软件中,经光谱预处理后,进行主成分分析,选取Q值作为评价指标,共提取了2个主成分,主成分选择情况。见图9。主成分分析的3D得分图。见图10,其中:伪品S12单独为一类,其余正品为一类,但存在异常样本S15,其余样本聚类情况明显。主成分分析基本可以实现地龙药材产地的溯源。
2.2.4 偏最小二乘法辨别分析
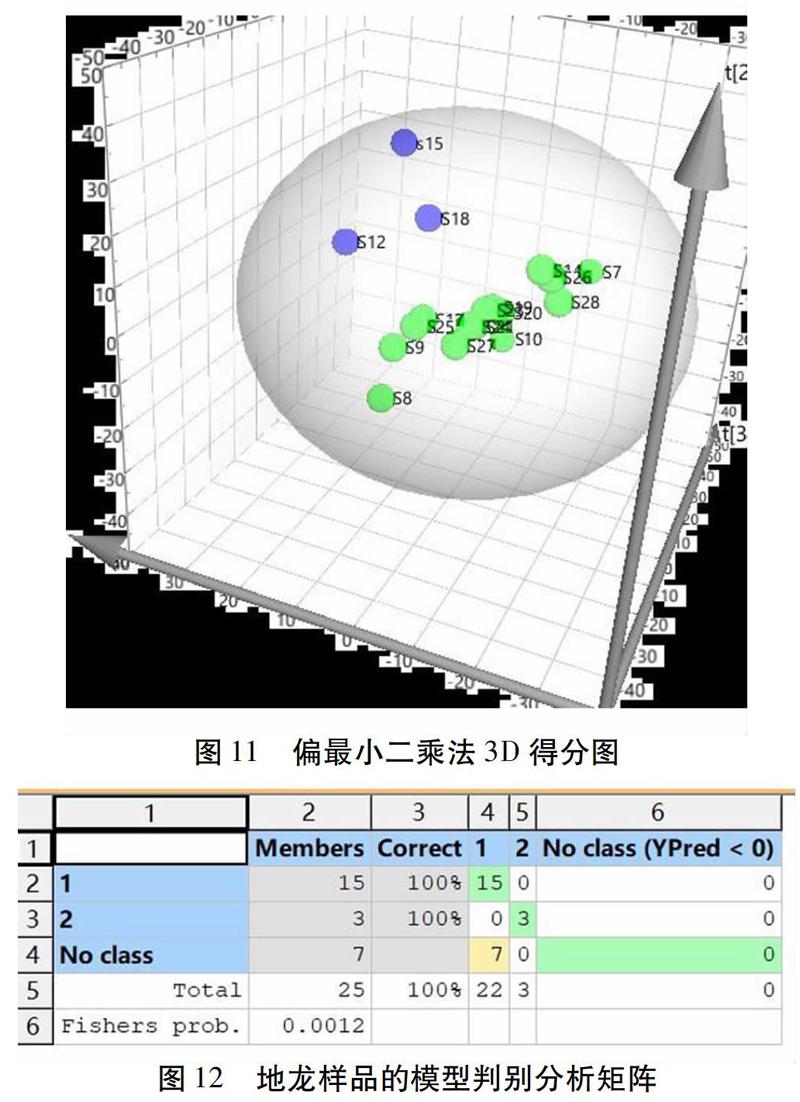
偏最小二乘法判别分析是通过测量传感器阵列和由正确分类矢量组成的类关系矩阵提取数据并建立2种矩阵关系模型的有监督模式识别[8]。多类判别分析法是一种集有效特征选择与识别于一体的分析方法,应用广泛,主要是判别未知样本应该归属于哪一个已知总体[9],首先根据已知样本的多种观测值建立判别函数,以此作为样本划归为某一总体的依据,最后将判别样品的有关数值代入判别函数,进行该样本的判别[10]。本实验对10 000~4 000 cm-1波段的地龙近红外光谱进行光谱预处理后进行偏最小二乘判别分析,提取前6个主成分,其累积贡献率达到99%,可代表大部分光谱信息,图11为偏最小二乘判别3D得分图。模型判别混合矩阵见图12,15批样品归1类为正品,3批样品归2类为伪品,剩余样品作为验证集,用于验证模型的准确性,验证集混合矩阵结果见图13,观察数据可以发现:S1、S2、S3为正品,S4为伪品,S11样本为异常样本,根据DNA条形码发育树分析,S11与正品药材亲缘关系较近。综上所述,此判别模型可应用于地龙药材的真伪判别,为药材的初步鉴别提供一种鉴别方法。
3 讨论
中药材组成复杂且其质量易受到产地,气候,采集等多方面的影响。本实验首先通过COI序列DNA条形码技术对各批次药材进行品种鉴定,以保证样品的准确性和代表性。近红外光谱作为一种二级分析方法,将近红外光谱技术用于药材鉴定方面常需要多种方法共同配合对原始近红外光谱数据进行光谱预处理后进行主成分分析,偏最小二乘辨别分析以及聚类分析3种建模方法,结果发现3种都可以用于评价地龙药材的真伪,但偏最小二乘辨别分析模型的准确度由于主成分分析。由此可知:地龙的辨别分析模型可为地龙的真伪初步筛选提供新思路,新方法。同时为动物药的近红外光谱分析建模提供相关的借鉴和参考。综上所述:近红外建模方法能够对地龙药材的真伪鉴别进行药材的初级鉴别,简便快捷,COI序列分析可以从基因层面准确的鉴定药材种属,二者相互配合,互相验证,能够很好的明确地龙药材的基源,为动物药材的真伪鉴别提供新思路,新方法。
参考文献
[1]韦健红.地龙类药材DNA条形码分子鉴定的研究[D].广州:广州中医药大学,2012.
[2]格小光,蒋超,田娜,等.基于DNA测序技术的市售地龙类药材基原调查与考证研究[J].中国现代中药,2019,21(9):1206-1214.
[3]袁伯川,李文东,马永生,等.柴胡属药用植物的分子鉴定及市售柴胡药材的质量调查[J].药学学报,2017,52(1):162-171.
[4]李希熙.基于近红外光谱技术的菜籽油品质快速评价方法的研究[D].武汉:华中农业大学,2015.
[5]李运,王元忠,杨维泽,等.红外光谱对三七及其野生近缘种亲缘关系研究[J].光谱学与光谱分析,2016,36(8):2420-2424.
[6]汪方舟.近红外光谱分析技术用于穿山龙鉴别的研究[D].泰安:山东农业大学,2019.
[7]张红,弓太生,赵国徽,等.基于红外光谱主成分分析的易混毛皮的鉴别研究[J].皮革科学与工程,2019,29(3):47-52.
[8]李静,王秀芬.基于红外光谱技术建立宁夏中宁产区枸杞子判别模型的應用研究[J].宁夏农林科技,2018,59(4):27-29.
[9]李四海,潘新波,任真,等.不同生长期当归红外光谱的偏最小二乘分析[J].中国实验方剂学杂志,2013,19(12):132-135.
[10]李运,徐福荣,张金渝,等.FTIR结合化学计量学对三七产地鉴别及皂苷含量预测研究[J].光谱学与光谱分析,2017,37(8):2418-2423.
(2020-06-10收稿 责任编辑:徐颖)

