DNA可编程纳米制造研究进展
陈 鑫, 王 方, 张新建, 石晓龙
(广州大学 计算科技研究院, 广东 广州 510006)
自Watson等[1]首次提出 DNA 碱基配对规则以来,DNA纳米技术不断发展,成为最受关注的科学技术前沿领域之一.以DNA为基础发展出了众多纳米技术.文章首先简要介绍了DNA纳米技术,然后阐述了DNA折纸、DNA tile和DNA纳米设备近几年的发展状况以及应用,并总结展望了未来的发展趋势.相信DNA纳米技术在未来一定会发挥出自身的优势,在各种领域发挥出重要的作用.
核酸分为脱氧核糖核酸(DNA)与核糖核酸(RNA)两种,二者作为生物体内储存和传递遗传信息的物质,承载着生命蓝图.DNA是一种由多个脱氧核苷酸线性构成的聚合物,携带腺嘌呤(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)四种不同碱基的核苷酸,按一定组成和顺序排列构成了决定DNA单链本身化学性质的一级结构,A与T、G与C之间按照Watson-Crick碱基配对原则严格互补,绝大多数情况下会形成清一色的右手手性双螺旋B-DNA,这种稳定性使得DNA具有作为纳米技术原材料的优势.同时,单链和双链DNA拥有截然不同的机械性质,局部交替使用代表灵活性的单链和代表稳定性的双链使得DNA可以编织出多样且稳定的纳米结构,刚柔并济的特点也是DNA作为新兴纳米材料的一大优势.
结构DNA纳米技术是将DNA作为原材料,使用稳定的分支DNA基序,如Holliday结,或单链DNA(single-stranded DNA, ssDNA)、双链DNA(double-stranded, DNA, dsDNA)等基序用来构建各类目标结构和功能器件的热点技术.从在显微镜下看到tile自组装构成的清晰二维网格[2],到DNA折纸的出现[3],它极大地扩大了可寻址DNA结构的规模,再到空间中DNA三维组装[4],研究者们一直致力于在分支连接的基础上,将这些DNA基序结合起来,辅以生物或化学分子,按需产生目标结构.这些结构不仅仅是DNA链基于Watson-Crick碱基配对规则粘合形成的复合物,亦是可编程化DNA产生的特定几何图案或立体结构.
自组装DNA的一大优势在于可以同时创造数以百万计的相同结构的拷贝,这样,就有可能以DNA自组装图形为基础,创造各种实用的纳米材料.同样,大量产生相同结构的特性赋予DNA基序算法自组装的潜力,DNA基序的动态行为使纳米机械设备能够模拟宏观设备,这些健壮的DNA序列可能在操作过程中改变其形状或位置,但它们不会分解或多聚化.DNA折纸技术在纳米制造领域的应用,使纳米结构拥有大小可控、更稳定、刚性更好等优势,同时,这项热点生物技术引入了可编程化组装的概念,基于其可寻址的特点,可以在特定位点做特定操作.这些想法与研究赋予了DNA除传统生物学意义外的其他价值,使其成为一种全新的可编程信息工具.这也是DNA纳米技术意义所在,除了研发新的DNA基底纳米结构、纳米工具外,纳米制造技术的发展也为分子计算领域提供了发展平台.本文按照DNA纳米技术领域较为热门的三个方向,包括DNA Tile自组装、DNA折纸和DNA纳米器械,对近三年DNA纳米制造领域的研究进展作出简单介绍.
1 DNA Tile自组装
自组装是指基本结构单元自发形成有序结构的一种现象,分子、纳米材料、微米或更大尺度的物质等均可以进行自组装.DNA由于其碱基互补配对规则,可以形成具有高度可编程性的自组装材料.在过去的三十年里,DNA被用来构建一系列由寡核苷酸组成的一维和二维阵列或三维晶体[2,5-6],这些结构被加以开发用来锚定和组织纳米颗粒或蛋白质.近年来,出现了许多DNA自组装结构,极大地丰富了可编程DNA纳米工具库.
1.1 结构DNA自组装
结构DNA纳米技术的最初目标是晶体物质的自组装,使用DNA粘性末端来引导分子间的结合.合理设计DNA晶格以支架分子物种,并用X射线结晶学解析其结构是结构DNA纳米技术的基本目标.DNA晶体基于Watson-Crick碱基配对原则,固定的Holliday结和粘性末端的内聚力促进了晶体在空间中的编程化自组装.通过使用核苷酸构建三维晶体,提出可以实现目标分子的特定取向,或者晶体可以用作分子筛和模块化器件[7].近期,Simmons等[8]报道了一种Holliday结介导的可寻址3D支架的组装,这源于第一个被成功设计的空间图案——张拉整体三角形[8],这种晶体包括三个独立的线性边,每个边都有两个螺旋形转角,每个轴线上包含三个或四个螺旋形转角,分辨率分别为6.5Å、4Å和10Å.
最新报道的结构被设计为四重对称的“张拉整体正方形”,然而实际的结构基序表现为包含了密集填充的、非周期性空穴阵列的三角空间群,且这个系统结晶良好,引入一个额外的碱基以诱导接头额外旋转,缓解了最初基序中表现出的扭转应力,扩大了空腔,使结构的分辨率达到3.05Å.更重要的一项工作中,引入新颖的Holliday结序列,能将分辨率显著提高至2.7Å,然后使用TA粘性末端结构进一步提高到2.6Å.这些晶格产生了高度紧凑的螺旋阵列,具有合适的允许结合更大分子的空腔大小和近原子分辨率,可用于潜在的蛋白质客体的特定位点定位.
1.2 DNA自组装纳米材料
自组装DNA结构已被用于构图各种材料.由于DNA纳米技术可以提供广泛的可编程化定制纳米尺度的二维和三维组装,许多为模拟生物复合材料制造过程而开发的方法可以制造出具有明确微观和宏观特征的合成材料.
DNA纳米结构制造需要克服DNA本身特性带来的问题.脱氧核糖核酸单链在溶液中以离子态存在,这意味着需要高离子强度的溶液来维持其结构,而这反过来会产生表面电荷,从而抑制材料沉积.近年,Liu等[9]报道了一种方法可以克服这种困难,该方法源于被广泛用于制备二氧化硅纳米结构的STöber法.然而,DNA折纸纳米结构需要高浓度的阳离子(如Mg2+)来维持其结构完整性,高离子强度也阻止了硅化所需的C9H24ClNO3Si(TMAPS)附着在DNA的带负电的磷酸盐骨架上.Liu等通过分子动力学模拟发现,含有3个以上TMAPS的水解簇Tab在高离子强度条件下可以克服静电势垒,吸附到DNA骨架上,其中,a和b分别为TMAPS和正硅酸乙酯(Tetraethyl orthosilicate, TEOS)的分子数,由此对合成方法进行了调整,并证明在合成溶液中TMAPS与TEOS的化学计量比设置为36.0 mM:90.0 mM时,水解步骤可以促进Tab簇的形成,这些Tab簇能够在DNA纳米结构周围形成诱导层,从而允许二氧化硅沉积.且这种覆盖有二氧化硅的杂化DNA材料被证明可以成功复原不同DNA折纸支架的几何信息,并比原模板更加坚韧.
单壁碳纳米管(Single-Walled Carbon Nanotubes,SWCNTs)由于其特殊的电子特性,可以用做纳米级的信息处理材料.寻找将单壁碳纳米管精确排列成复杂的纳米级几何形状的方法是前提与挑战.可编程DNA自组装在此提供了许多新思路.对DNA生物化学特性加以把握,有助于更好利用这一分子工具.单链DNA与单壁碳纳米管超声作用时,通过物理吸附DNA碱基的方式附着在单壁碳纳米管侧壁上[10].借此,Maune等[11]率先提出了利用DNA折纸可编程化制造纳米板的方法,即在折纸两侧伸出粘性末端作为“钩”序列,与DNA链与单壁碳纳米管超声复合物上垂直的单链DNA互补,有向地(使碳纳米管在溶液中相互垂直)网缚溶液中的碳纳米管,统计结果显示>50%的碳纳米管方向落在预期±15°范围内(图1a).随后,Han等[12]采用另一方法,使双链DNA-碳纳米管复合物在二价反离子存在的情况下沉积在带电底物上,再引入单价反离子使挂载DNA链的碳纳米管以2D方式沿沉积表面扩散.表面约束条件下,一个纳米管上的DNA连接体与相邻纳米管侧壁之间的弱分散相互作用,可以协同诱导均匀的平行单壁碳纳米管阵列的组装(图1b).
尽管有这些研究,但无约束和低约束表面旋转仍然限制了在DNA模板中实现纳米管的精确控制.近年,Pei等[13]在常见光学构件单壁碳纳米管连接DNA折纸构成复合材料方面作了深入研究.利用SWCNTs与单链DNA的非共价相互作用,让单链DNA探针部分的芳香族碱基借助π-π键包裹在单壁碳纳米管表面,在不破坏其电学/光学性质的前提下,得到了水溶性的SWCNTs.这种结构没有扰动SWCNTs侧面表面晶格,保持碳纳米管性质不变,同时解决了SWCNTs无法在DNA折纸表面特定的点以特定的方向组织这一重要问题.近期,Sun等[14]开发了一种空间受阻的纳米管电子学方法,来构建均匀间距的碳纳米管阵列.基于DNA砖晶体的纳米沟槽内的单链DNA柄与DNA-碳纳米管复合物上的反柄(与DNA柄互补的序列)之间的DNA杂交,可以补偿组装过程中的静电斥力.DNA纳米沟槽不允许未对准的碳纳米管接触到DNA柄,这成功限制了单个碳纳米管沿着其纵轴的精确取向.通过对DNA沟槽周期的编程,合理地将碳纳米管间距从24.1 nm调整到10.4 nm,组装得到角度偏差小于2°、组装产率大于95%的平行碳纳米管阵列(图1c).

图1 自组装DNA法组织碳纳米管技术的发展
1.3 DNA自组装其他应用
可编程DNA自组装结构可以基于其动态反应传感生物信号.Kwon等[15]注意到登革热病毒表面的包膜蛋白结构域Ⅲ(Envelope Protein Domain Ⅲ, ED3)病毒表位的分布具有特殊表面簇结构,ED3被组织成三价或五价位点.将这些ED3位点的簇连接起来,根据其分布特点设计了对应的五角星型DNA tile自组装的模型.该模型由21条DNA寡核苷酸链自主装而成,每条外部边含有42个碱基对,通过内部五个四臂分子顶点与内部链相连.五个内部边缘都带有一条DNA单链,与之对应的可以形成一个7 bp茎和6 bp环的发夹结构.笔者通过在该结构10个顶点上杂交一个特征良好的ED3结合适配体,在发卡结构的茎上杂交荧光剂和猝灭剂,得到了登革热病毒传感器(图2).实验证明,这种针对病毒表面表位特点“私人订制”的DNA自组装结构,不仅在病毒检测方面提高了检测效率和速度、降低了检测成本,而且在抑制病毒方面也取得了良好的效果.该方法的提出为病毒检测剂和疫苗的研究及制备提供了新的思路和方向.
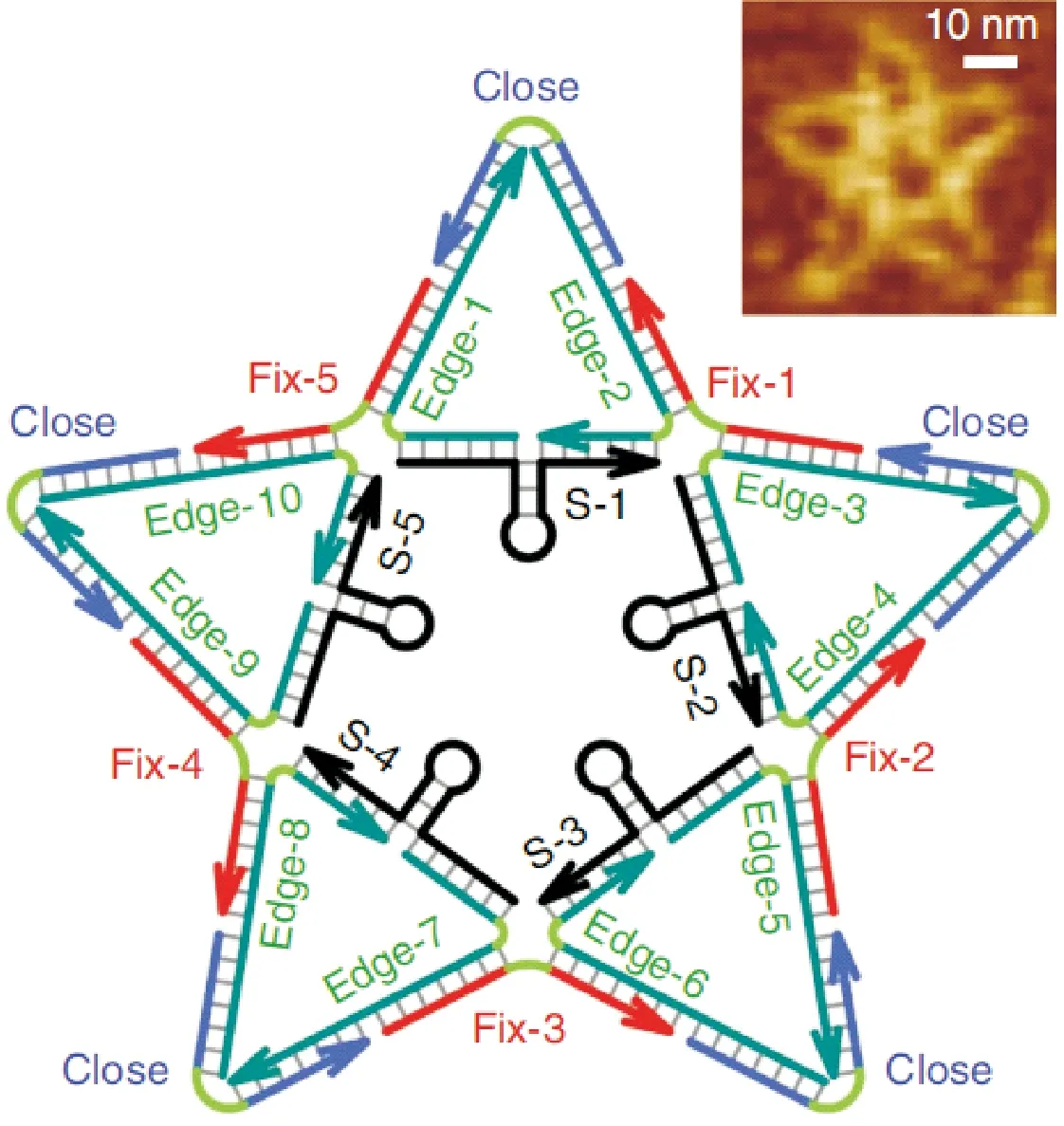
图2 自组装五角星结构识别登革热病毒表面
DNA分子的相互作用令其可以编程或执行算法,以生物系统中未曾见过的方式发挥作用.结合分子结晶的数学瓦片和统计力学模型的理论表明,算法行为可以嵌入到分子自组装过程中[16].近年,Chen等[17]报告了一个包含355个单链tile的DNA自组装集的设计和实验验证,以管状DNA折纸为基底,四个十字相邻的tile抽象为一个两输入两输出逻辑门,在6个并行输入tile控制下,通过简单的tile选择,DNA库可以被重新编程以实现各种6位算法.报告中该DNA集合被用来构建了21个电路,可执行的算法包括复制、排序、识别回文和3的倍数、随机行走、从有偏见的随机源中获得无偏见的选择、选举领导者、模拟元胞自动机、计数到63的计数器、生成确定性和随机化的模式,总的单位错误率小于1/3 000.这是一项自组装ssDNA基于DNA折纸基底进行生长的研究,结合了两种组装技术的优势,合成的管状结构同时拥有单链tile的高度灵活性和折纸作为基底和边框的稳定性.后文将对DNA折纸这一稳定纳米基底进行介绍.
2 DNA折纸
2006年,Winfree[2]通过一张灿烂的微笑首次提出了DNA折纸技术.DNA折纸技术的出现改变了结构DNA纳米技术的面貌,DNA的可编程性为纳米材料的精密控制提供了保障.Douglas等[18]设计的 410 nm 的六螺旋纳米管拉开了使用DNA折纸技术制造三维纳米结构的序幕.随后,DNA线框[19]、DNA网格[20]和多面体网格[21]、方块、螺母、桥架[22]以及带有弧线的立体 DNA 纳米结构(如圆球、椭圆球和花瓶)[23]等结构被提出.随着DNA折纸技术的不断发展,DNA折纸的可编程性和控制纳米形态性使得生物医学研究[24-25]、生物物理学[26]、生物传感[22]、纳米机器人[27]、药物传递[28]、信息安全[20]和生物计算[29-30]等领域应用广泛.
2.1 DNA折纸结构的应用
DNA折纸由于具有可编程性和精确的形状结构,近些年来被广泛用于探究抗原抗体和蛋白质相关性质的研究.Veneziano等[31]利用与eOD-60mer相似的直径为40 nm的三维二十面体折纸结构DNA-NP和刚性杆六螺旋束(6HB)(图3a),系统地研究了抗原空间排列和附加设计参数(包括抗原拷贝数、亲和力和支架呈现的刚性和维数),在触发B细胞和启动B细胞受体信号传导中的独立作用.DNA折纸相比较以往采用蛋白质、聚合物等作为支架,可以更好地探究单一参数在传导中的作用.这些参数的优化能够更高效地激活抗原特异性B细胞,为疫苗制备提供参考;Zhang等[32]在三角形DNA折纸上特异性地锚定了六对地高辛分子(780 Da),见图3b,形成了具有特异性锚定和空间组织的人工表位(DOSE).每个表位对的横向距离间隔为3~20 nm.利用DOSE模拟病毒表位的距离分布,以编程抗体结合能力,DOSE的定位能力和硬度使免疫球蛋白(IgGs)能够在室温下冷冻,从而在单分子水平上对IgGs结合构象进行瞬时的、功能性的高分辨率成像,达到室温下捕捉IgGs的瞬时构象的目的.DOSE表位在横向距离上精准的编程控制,能够灵活地确定表位距离相关的IgGs的亲和力.
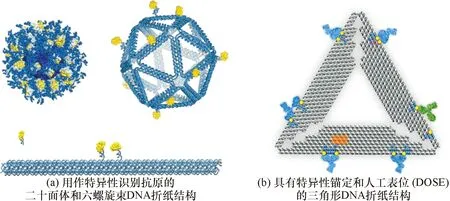
图3 用作特异性识别抗原的二十面体和三角形DNA折纸结构
为了探索多个适配体结合位点的几何排列或纳米机械性能等设计参数,在Tikhomirov等[33]设计的可扩展DNA折纸晶格的基础上,Rafat等[34]设计了带有中心腔的折纸结构,腔内排列有12个独特的可寻址的staple,可以被特定的DNA适配体识别(图4a、图4b).多个适配体可以以不同的几何关系、灵活可变地放置在多个位置上.实验中,将该结构进行2*2的排列形成含有4个空腔的结构,并引入非对称方式设计的条形码方案(图4c),便于在原子力显微镜下观察8种2*2阵列.实验证明,该DNA折纸模型在探究大量设计参数,甚至从头选择多价结合剂上效果明显.DNA折纸技术的可编程性使得设计者可以按照所需形成对应的二维、三维结构.这一突出优点为研究蛋白质相关性质或者模拟蛋白质之间以及蛋白质与抗原抗体之间的结合提供了良好的工具.相比于其他方法,DNA折纸能够控制单一变量研究其对蛋白质之间以及蛋白质与抗原抗体结合的影响.这些优点都将促使DNA折纸技术在蛋白质研究领域有更广泛的应用.
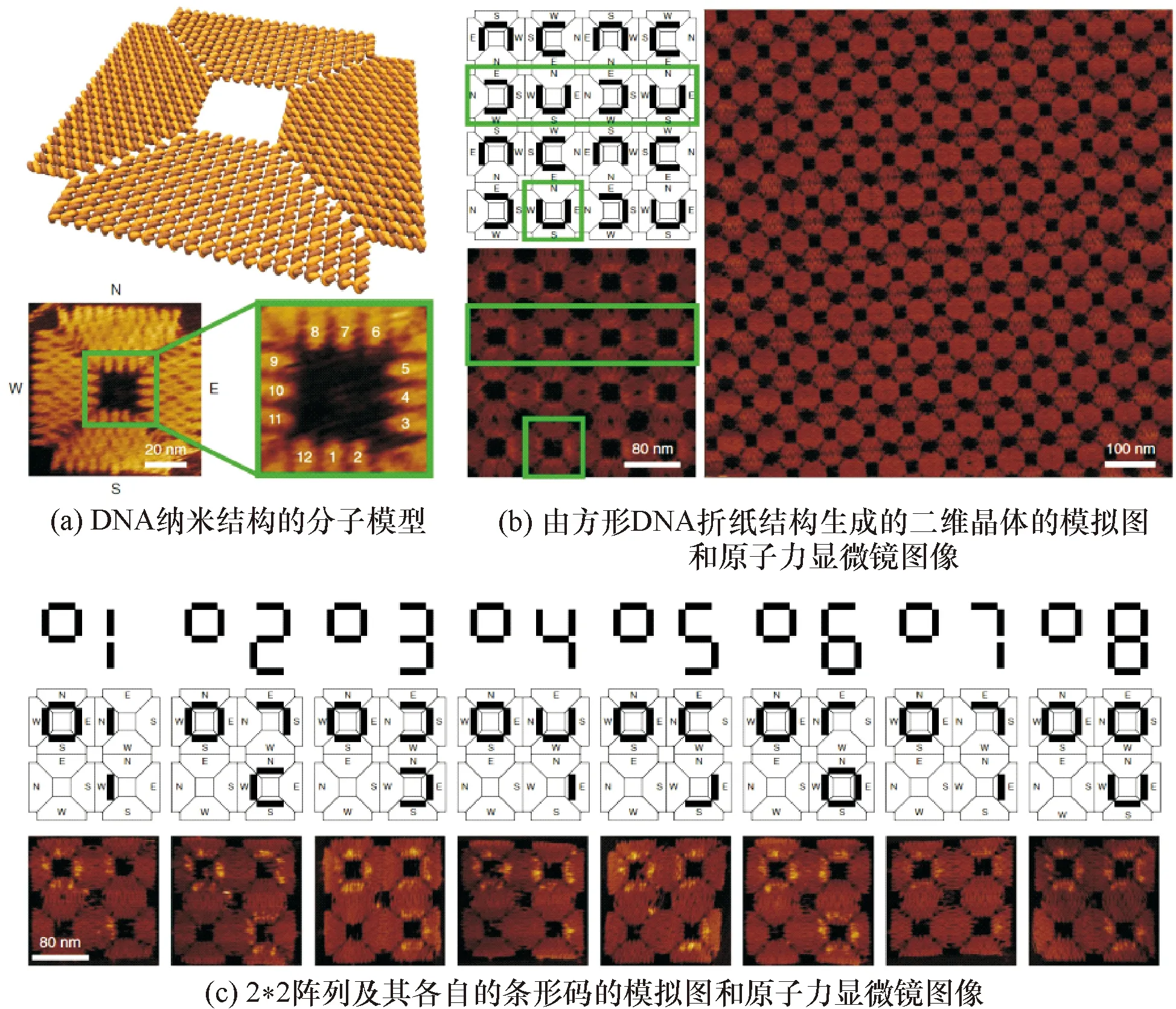
图4 带有空腔的DNA折纸模型
DNA折纸的稳定性与可寻址性使之具有作为结构基底的潜力.可以设计staple单链携带不同的基团实现特定目的.例如,近期Zhan等[35]通过DNA折纸成功构建了一种能够精确控制的具有大约5 nm间隙的金蝴蝶结状等离子体纳米结构,传统方法如电子束光刻,难以达到10 nm以下精度.已知两个金属三角尖端相隔纳米级距离时,缝隙中表现出高度的局域电磁场增强和超空间限制,研究借助DNA折纸的精密编程可视化证明了这一物理特性,开发出的这种金属-DNA纳米复合材料,其特性使之可用于单分子分析和光学信息处理.与DNA tile自组装相同,DNA折纸也可以和DNA以外的物质共同构造复合材料,并且是一种可编程的优良基底选择.
信息时代,信息安全的机密性、完整性和可用性成为人们关注的重点.1999年,Clelland等[36]首次提出了基于DNA的密码学方案,为信息安全提供了新的思路和方向,开创了DNA密码学的新时代.2019年,Zhang等[37]提出了DNA折纸加密技术.该技术通过将信息进行二进制编码,然后将二进制信息加密为顺序的点模式,将M13病毒支架折叠成纳米级的自组装盲文样图案以进行安全通信.接收人可以通过使用特定组的短链将支架链折叠成DNA折纸结构来解密消息.结合DNA折纸技术的纳米级可寻址的特性,还可进行基于蛋白质结合的隐写方式实验.实验证明该方法为消息、图片等安全通信提供了多层消息保护.
生物传感器是通过固定在生物传感表面上的生物受体实现对目标分子的特异性识别.分子识别层的性能不仅受到这些生物受体亲和力的限制,而且在很大程度上取决于生物受体在纳米水平上的结构.DNA折纸结构可以使生物传感器表面更有组织的功能化,提高生物传感器的性能.2020年Rutten等[38]利用3D DNA折纸设计,设计了一种检测特异性的天线状DNA折纸结构,并将其用于EvalutionTM平台通道中编码微粒的表面功能.与直接固定化的非DNA折纸系统相比,该方法使生物受体可获得性增加,结合动力增强了4倍,结合效率提高了6倍,大大提高了生物传感器的性能.高灵敏度光电探测器问世之后,探测单个分子的荧光成为世界各地许多实验室的常规任务.然而,直到今天,仍然需要成本高昂的光学仪器才能完成该项任务.Loretan等[39]设计了一种新的三维DNA折纸结构,能够实现高荧光信号放大功能.该结构类似玉米形状,用两根柱子连接纳米银颗粒,并在其间放置诱饵和核酸生物检测所需的结合靶标.该方法可以用传统的智能手机摄像头和廉价的物镜进行工作,并成功地对抗药性肺炎克雷伯氏菌特异性DNA进行了单分子检测.该方法的出现极大地降低了单分子诊断分析的费用,为DNA纳米折纸天线单分子检测的民用化做出了有益的探索.
2020年,Li等[40]构建了一种多路复用FDF放大器的拓扑工程方法,利用Cayley树状分形DNA框架来拓扑编码荧光状态,用于低丰度目标的多路检测.该放大器使用少量的16条DNA链,便可以最小的串扰编码多达36种不同的颜色,用于单分子识别和多重细胞识别,丰富了高通量细胞成像的工具箱,提高了人造荧光放大器的复用能力.
2.2 DNA折纸制备工艺改进
虽然DNA折纸已在诸多科研领域表现出了巨大潜力,但是因为受到制备工艺、保存技术和反应条件等诸多限制,还不能完全实现大规模大范围的量产.为了改善DNA折纸各个工艺流程当中出现的弊端,众多学者投入了研究.
Sobczak等[41]在2012年提出:大多数DNA折纸纳米结构保持适当的恒定退火温度时,可以在5~100 min内形成设计的折纸结构.2019年,Halley等[42]在这一发现的基础之上,提出一种低成本高效退火自组装的方法,即先65 ℃初始加热10 min,再51 ℃退火2 h,最后4 ℃短暂淬火,从而有效组装一系列的DNA折纸结构.这种方法可使用实验室常用的设备和用品来制造DNA折纸.例如,可以根据需要的量选择在合适的试管(0.05~50 mL)或者10~1 000 mL刻度的烧瓶中进行折叠,甚至是在更大的温度控制容器中也可以实现.此外,可以使用包括水浴、电炉和标准实验室燃烧器在内的广泛使用的设备进行加热.这使得DNA折纸实验可以在任何具有化学或生物学功能的实验室中,甚至在中学实验室也可以进行.与传统方法相比,这种方法有效地缩短了实验时间,降低了实验成本和试验门槛,扩大了生产规模.
DNA折纸纳米结构对pH值、温度、离子强度等环境因素十分敏感,如何储存成为实验室面对的一大难题.而在相关技术和医学应用中需要DNA折纸结构在适当的储存条件下长期保持其结构和形状完整性.冻干技术是室内长期保存DNA纳米结构的一种可行办法.Xin等[43]对悬浮DNA折纸结构在反复冻融的情况下的稳定性研究发现:二维、三维折纸结构分别经过32次和1 000次冻融之后会出现冻损现象.通过实验发现,这些损伤主要是在洗涤和干燥过程中造成的.反复冻融产生的冰晶导致DNA折纸结构出现划痕,这些划痕增加了在洗涤和干燥过程中的敏感性.笔者提出并通过实验验证证明,得出0.2×10-3M至200×10-3M的浓度下低温保护剂甘油和海藻糖可有效保护DNA折纸纳米结构免受冻害,并且对DNA折纸形状没有任何负面影响的结论.
在实际应用过程中,DNA折纸结构存在易溶于生物学相关的流体和纳米加工中使用的典型溶剂的问题.为解决此类问题,Nguyen等[44]提出了在DNA折纸表面两次涂布二氧化硅的方法.实验使用APTES或TMAPS作为偶联剂可促进二氧化硅从TEOS的沉积,从而获得分散良好的二氧化硅涂层结构.实验证明,TMAPS-TEOS涂层可在大范围的pH和DNA折纸浓度下获得成功.APTES-TEOS在低Mg2 +浓度下产生了更好的结果.并且在20%v/v IPA溶液中使用二次涂层可以提高二氧化硅壳的密度.
3 DNA纳米设备
3.1 DNA纳米装置
人们致力于开发各种工程方法,通过赋予微米或纳米粒子价键来制备“胶体原子”,这些“胶体原子”中存在短程互补粘合装置,使其可以自组装成具有低价和定向键合的复杂超胶体结构.近期,Yao等[45]设计了一种提供正交性、方向性、稳定性和选择性的金粒子单链DNA编码器.基于PolyA结构很容易吸附到金纳米颗粒表面的性质,留下键合区域延伸到溶液中,进行特定的可逆杂交反应.通过规划这两个结构域的顺序、长度和序列,合成了一系列S可编码的可编程原子当量(PAN),并且成功实现了单分散、线性或分支化的“胶体低聚物”的组装,实现了动态胶体反应,比如进行布尔逻辑运算.
DNA折纸提供了一种通过DNA序列直接设计其热力学稳定状态来构建纳米机器的方法.Chao等[46]设计了一个分子装置,命名为NanoMuscle,它是带有机械联锁的DNA折纸,由两个以双螺纹轮烷形式组装的单体组成.NanoMuscle实现了一维收缩和伸展,并可由通过添加特定的DNA链来控制.在DNA杂交和布朗运动中,通过将结合能转化为机械能的方式操纵分子运动,NanoMuscle可能成为未来机械的一种新的构建模块.
3.2 DNA 步行者
DNA计算的一个重要目标是实现类似传统计算机的可以根据输入信号执行一个任务并输出任务的功能.自Watson-Crick提出脱氧核糖核酸碱基配对及杂交所涉及的热力学理论后,DNA机器的构建得到了快速的发展.在使用DNA动态纳米技术构建的复合物中,具有处理信息功能的DNA 步行者领域受到了研究者们的关注.
DNA 步行者是一种DNA分子机器,可以在特定的刺激下执行准机械运动,并精准地在指定路径上行走实现各种功能(图5).2004年,Seeman等首次提出了一种使用两足沿着轨道行走的、可以精确控制的分子纳米机器[47].后来Shin等[48]设计了一种DNA 步行者,可以利用细胞内运动来激发DNA 步行者进行工作.目前,基于DNA 步行者的信号输入方式可分为以下4种:①使用保护性DNA探针,以特异性靶向的方式与DNA 步行者杂交;②一种可被靶点激活的结合诱导DNA 步行者;③DNA zyme驱动的可被金属离子触发的DNA Walker;④被特定酶激活的裂解诱导DNA 步行者.
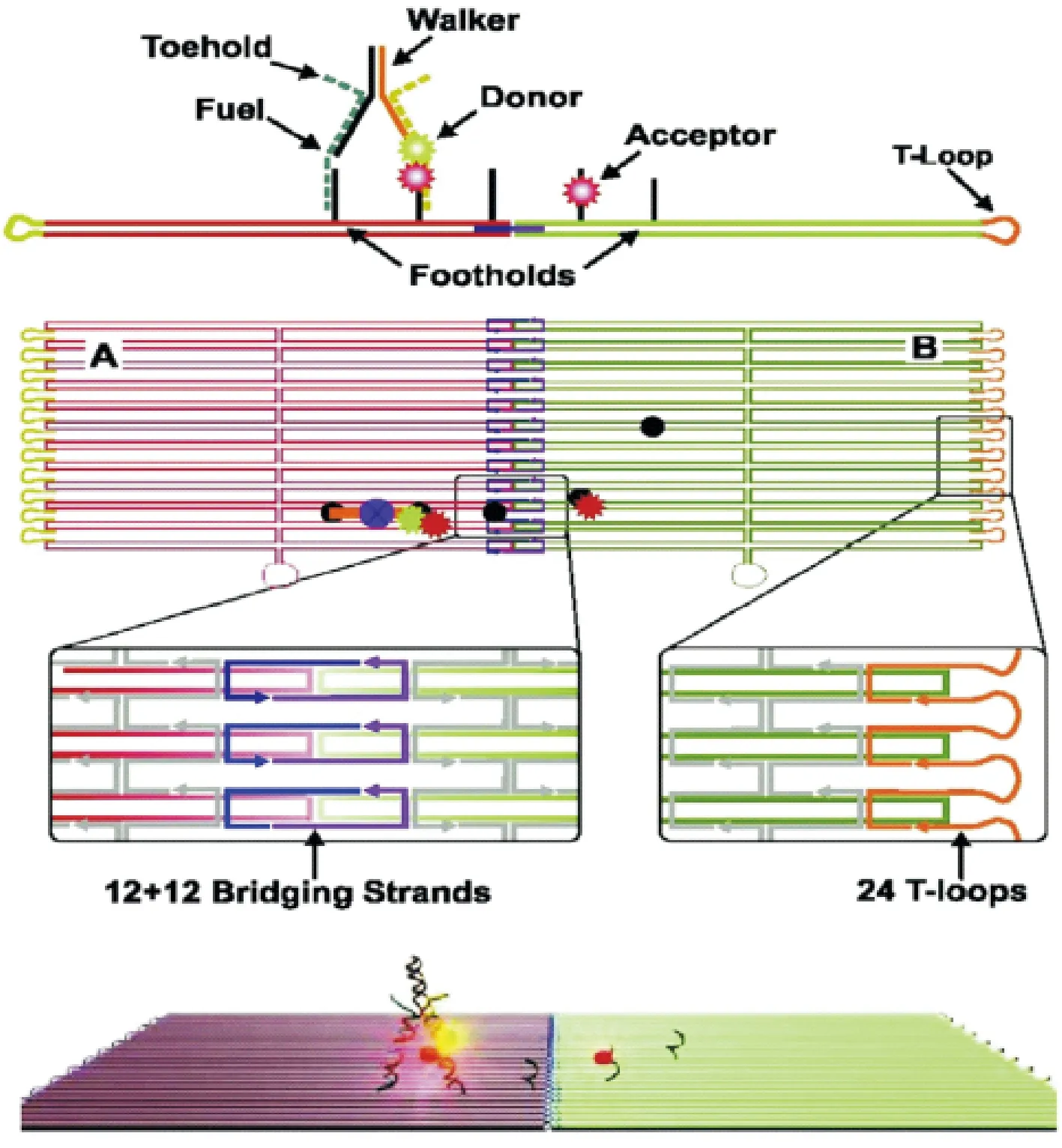
图5 使用3D DNA 折纸技术的 DNA步行者
在使用保护性DNA探针方面的研究中,Yang等[49]设计了一种新型的DNA纳米机器,它能够限制DNA 步行者在由DNA功能化的纳米金颗粒构建的三维轨道上的运动,并使用一种保护性的DNA探针来控制,该探针可以与DNA 步行者杂交,也可以以靶标特异性的方式与DNA 步行者杂交,成功实现对特定核酸的快速、恒温和均匀的信号放大.Li等[50]在可被靶点激活的结合诱导的DNA 步行者的研究中构建了一种无酶三维DNA步行器,其自主运动可以通过靶激活的纯DNA杂交来实现.该装置的纳米级运动可以产生DNA双链,并将荧光输出分子固定在MNB表面上,通过插入结构选择性荧光分子,获得荧光信号.在以天花基因为模型的分析中,显示出高灵敏度和特异性.因此,该步行装置为核酸和蛋白质分析提供了一种可靠、灵敏和选择性强的策略.
DNA zyme驱动的可被金属离子触发的DNA 步行者研究方面,Wang等[51]基于DNA zyme驱动的三维DNA 步行者的级联扩增和杂交链式反应,研制了一种无酶、无标记的荧光生物传感器,用于miRNA的检测.该DNA 步行者的高扩增效率和杂交链式反应保证了对LET-7a的良好敏感性,检测下限为7.9 fM.链置换反应保证了对let-7a极佳的特异性.此外,该传感器还可以用于实际生物样品中miRNA的分析,为定量miRNA提供了一种新思路.Qing等[52]将3D DNA 步行者与催化发夹组件相结合,开发了一种高选择性和超灵敏的电化学检测Cu2+的方法,为三维DNA 步行者在各种离子传感的应用中开辟了一条新途径.可被特定酶激活的、裂解诱导的DNA 步行者其原理是指通过酶使DNA 步行者自主运动.Feng等[53]设计了一种智能的3D DNA Walker,可与双链DNA核酸酶辅助切割反应相结合,应用于多核苷酸激酶活性的检测.因此,利用智能DNA步行者可以实现对T4多核苷酸激酶活性的灵敏、特异检测,具有操作方便、效率高和灵敏度高等优点.
这些模式都只能实现某种特定的功能,具有一定的局限性.因此,科学家们开始积极探索各种通用的信号输入方式.Xue等[54]利用化学选择性连接的方法设计了一种DNA步行器,并将其应用于新型生物传感器的开发.在设计中,将步行器的行走链分为作用链和支撑链,并用不同的化学选择性连接基对链进行修饰.作用链可通过与支撑链的化学选择性反应触发DNA步行器功能,并使用脂多糖、凝血酶和let-7a进行实验,验证了该步行器的高度灵敏度和准确性,证明了该生物传感器可成功应用于糖、蛋白质和核酸的分析,在检测多种生物分子方面具有广阔的应用前景.
4 结 语
DNA可编程纳米制造经历从基元的设计到一维、二维及三维结构的组装的发展.近三年来,DNA纳米结构自组装过程可以精准控制的优势突出明显,研究逐渐从结构上的控制向实际应用方向上过渡,涌现了众多交叉学科的科研成果.DNA纳米结构逐渐在更多的交叉领域发挥着重要作用,例如,合成生物学、免疫生物学等,为相关领域的难题提供新的思路和研究方向.随着DNA可编程纳米制造的结构控制研究和制备工艺的不断优化,将对DNA芯片、纳米元件与材料、疾病检测与治疗、疫苗研究与制备等领域的研究起到巨大的推进作用.同时人们对DNA可编程纳米制造研究的不断深入,学科之间不断融合,DNA自组装将会在更多的领域发挥作用,解决更多“卡脖子”的问题,推动相关学科的共同发展.
——求解可满足性问题

