DNA计算研究现状
阮扬名, 施倍宇, 洪尚伟, 强小利
(广州大学 计算科技研究院, 广东 广州 510006)
1946年第一台计算机诞生,其高速计算能力代替人类大脑完成了重复且复杂的计算工作.科技的发展速度越来越快,人们的生活日新月异,生活水平也不断提高,随之而来的是对运算性能有更大的需求.不同的领域对计算机提出了不同的需求,而传统计算机对层出不穷的新问题越来越难以应对,DNA计算在这种情形下应运而生,成为研究人员争相研究的热门技术方向.
DNA计算通常被归为数学、生物学和计算机等领域的交叉学科,该技术一开始被提出主要用于解决一类计算时间随问题规模呈指数增长的棘手问题,如NP问题,其原理已经在哈密顿路径问题的一个简单例子中得到证明.由于其强大的并行计算能力,科学家们寄望于该技术可以制作出新一代的分子计算机,因此,在DNA的逻辑电路上做了多方面的研究,并形成了基于酶和无酶的逻辑电路.本文主要阐述了基于酶、链置换的逻辑电路以及自组装算法的近况,并对近几年DNA计算的应用进行了论述和总结.
1 DNA逻辑电路
1.1 基于DNA聚合酶的逻辑电路
在DNA电路中使用酶有很长的历史.早期的论文有深刻的见解,主要集中在利用酶进行DNA计算的理论探索上[1-2].虽然这些设计有较好的理论应用,但由于使用了多种酶或复杂的操作,需要对实验论证进行简化.
最近引入的一种名为PENDNA的工具箱[3-6]被用来利用多种酶编写DNA电路,并且已经开发出几种基于它的多酶DNA电路.这为Song等[7]的结构设计提供了灵感.他们在最近设计出了一种基于链置换DNA聚合酶的单链门,这种设计仅用了一种酶——Bst2.0DNA聚合酶,来设计逻辑或门和与门,避免了额外的复杂设计,减缓了多酶DNA电路的催化反应造成的DNA裂解和泄漏.每个逻辑门最初只由单个DNA链组成,以减少以前体系构筑所造成的泄漏和“链的复杂性”[8-9](图1).
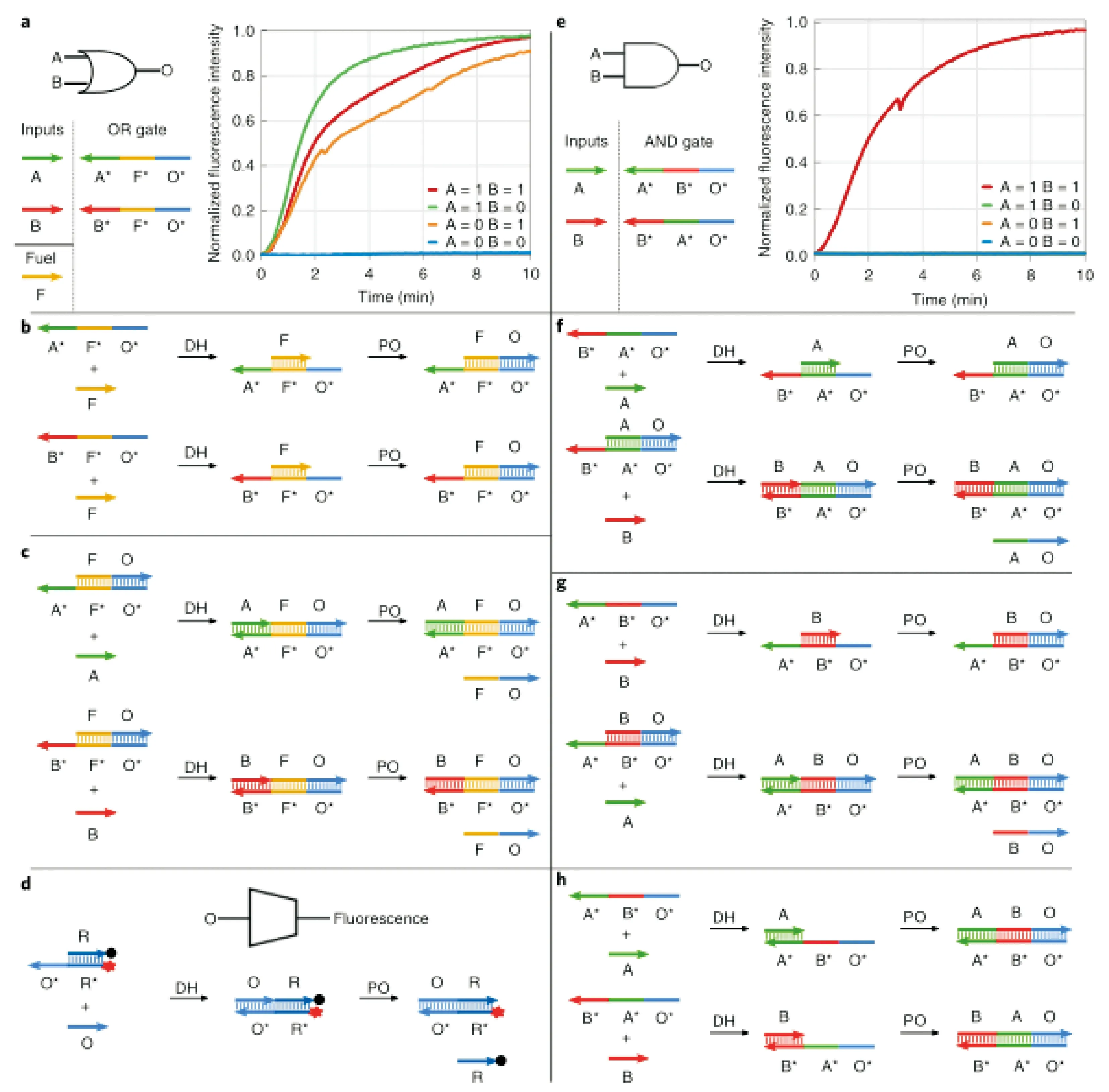
图1 Bst2.0聚合酶或门和与门的设计及性能
Song等[7]基于输入链和输出链有相同的域,以及逻辑门浓度确保上游为下游提供充足的输入链策略了构建了或门和与门.这样的构筑可以方便级联并形成更大的电路布局.他们用这种架构构建了一个用来计算4位数输入的平方根函数电路.该电路由37条DNA链构成,其完成时间仅需要25 min(半完成时间),而以前最好的结构[8],大约由100条DNA链组成,响应时间需要数小时.
Su等[10]用Bst聚合酶构建了另一种实现逻辑门的方法,该方法是利用上游引物和能够实现链置换的DNA聚合酶进行链置换DNA合成.随着引物的延伸,新的合成DNA链可以取代并释放下游的互补链.这一特性被称为链置换扩增(Strand Displacement Amplification,SDA)[11-12].基于这个特性,设计出了与门和或门(图2).用聚合酶链式反应(Polymerase Chain Reaction,PCR)和凝胶成像所得的结果表明,该电路能够很好地进行所需的逻辑运算,且计算时间较短(与和或运算分别为约6 min和3 min).为了实现逻辑非,他们将逻辑门开发成双轨逻辑门,通过构造一个单轨逻辑与门、一个单轨逻辑或门和定义一组错误的输入/输出来实现.
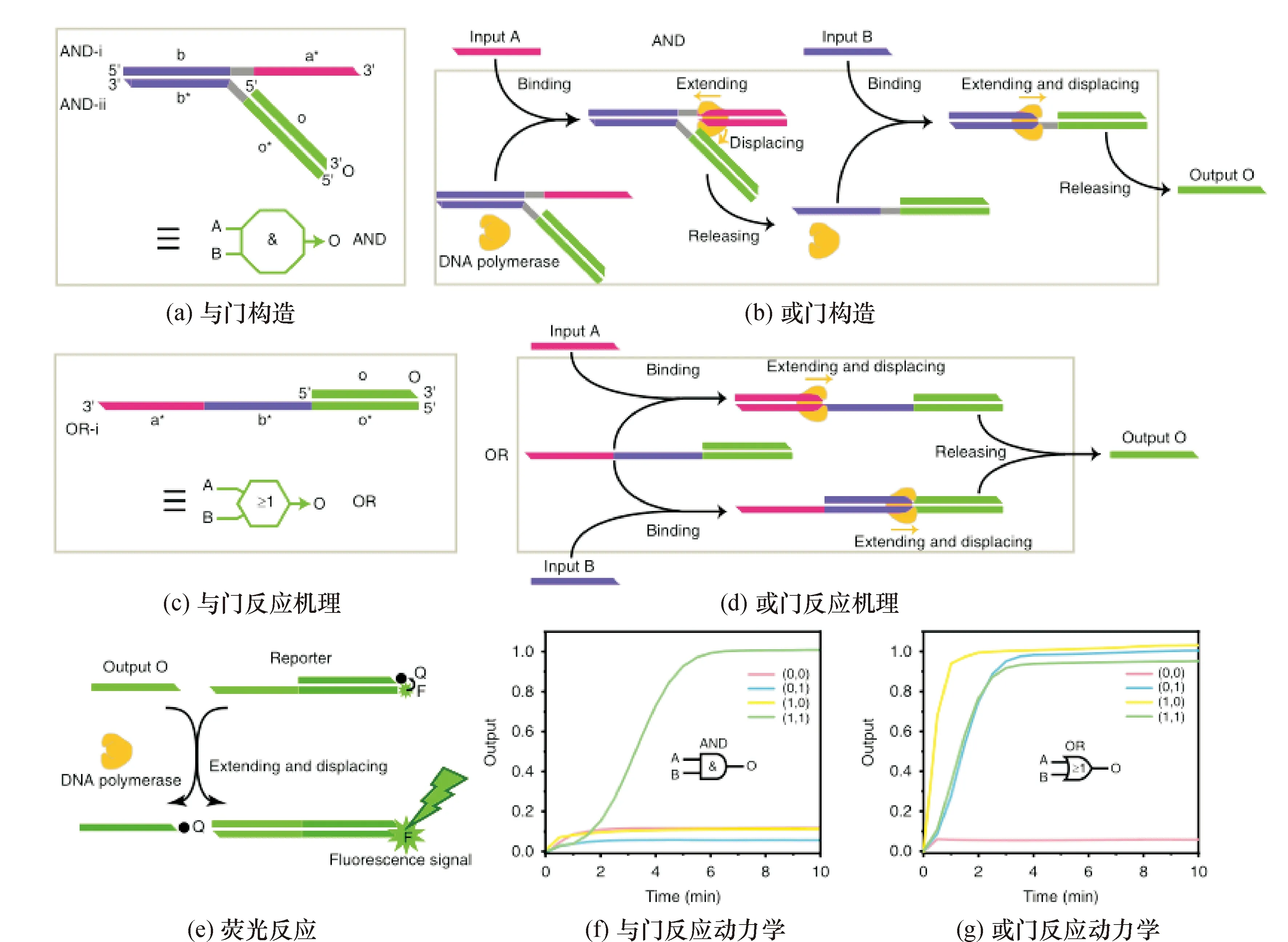
图2 DNA聚合酶与或门的构建及其性能
在实现了基本的与、或、非门之后,Su等[10]将这些基本组件组装成了全加法器和4∶1多路复用器.①全加法器.这个组装出来的全加法器由45条不同的DNA链组成,计算时间为20 min;②4∶1多路复用器.通过将两个和门集成到一个3输入的和门来获得更紧凑的结构,这个结构包含14种DNA和39条不同的DNA链,其反应时间约为20 min.之后在这两种逻辑器件的基础上引入了额外的集成门,制备了一个由27种DNA和74条DNA链组成的1比特的算数逻辑单元(Arithmetic and Logic Unit,ALU).虽然制备的ALU在其中一个输出中具有较高的泄漏,但仍能清晰地分辨出该输出值.随后,他们对其应用进行了进一步地扩展:①通过应用核酸适体和核酶,其他分子也可以转化为DNA输入物[13];②该系统的输出可以直接用于控制DNA分子机器,例如,DNA巡检器[14]和DNA货物分类机器人[15];③类似于功能性核酶[16]可以通过与体外转录结合,很容易地实现RNA输出装置.
1.2 基于链置换的逻辑电路
DNA计算包含许多方向和领域的应用,内容纷繁复杂.DNA链置换[17-18]是一种操作简单且具有自主性[19]的生物技术,其出色的计算能力使其在DNA计算领域得到了广泛的应用.以DNA链置换为主要原理的DNA逻辑电路是DNA计算中很多应用的研究基础.DNA逻辑电路是由各种DNA逻辑门组合而成的,DNA逻辑门是一种将若干输入经过逻辑运算转化为输出的设备,组成电路的基本逻辑门主要包括YES(是门)、AND(与门)、OR(或门)、NOT(非门)、NAND(与非门)、NOR(或非门)、XOR(异或门)和XNOR(同或门)等.这些逻辑门是实现DNA计算的重要组成部分.因此,如何构建不同的逻辑门,以及如何利用这些逻辑门进行组合、连接、级联成具有不同功能的DNA电路,以实现不同的复杂计算是DNA计算的一个很重要的研究领域.
过去的DNA逻辑电路研究主要是利用一次性使用的DNA逻辑电路,但是这种电路存在无法重复使用的短板,Eshra等[20]提出了一种可再生DNA逻辑电路的思路.这是一个基于可再生DNA发夹的基序(Motif),利用基序实现布尔逻辑门.可再生新电路使得样品的制备更加方便.在这项工作中,主要的突破点是设计了基于发夹的门和提取器(图3).
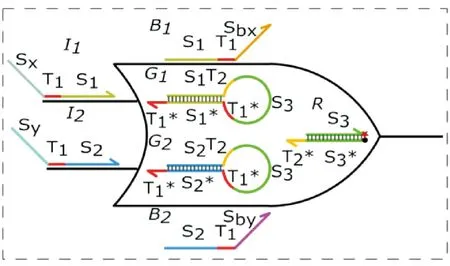
图3 可再生发夹基元的计算
通过荧光光谱法(Molecular fluorescence spectroscopy)和聚丙烯酰胺凝胶电泳(Polyacrylamide Gel Electrophoresis,PAGE)验证了2输入或门逻辑电路,而且报告了所有可能的输入组合的实验结果.使用了相同的发夹型门链来连续执行三次计算,更好地验证了其可再生性.通过添加荧光报告器,还证明了可多层电路一起工作.如果构建复杂的逻辑电路,则需要实现与门(AND)逻辑,尚需进一步的工作来实现.再生发夹基序的成功设计为大规模无酶再生DNA逻辑电路做了一个很好的奠基.但是,这个设计在门的恢复阶段产生的输出和抽取存在泄漏这一现象会造成在几个周期以后的计算链难以重置,这是目前存在的一个需要突破的难点.
Geng等[21]提出氧化石墨烯(GO)作为一种理想纳米材料用于基本的逻辑运算,以此来实现大规模的复杂计算.他们首次用了GO和分类单链DNA之间的相互作用,构建了一个智能化平台.通过这个智能平台对DNA碱基序列进行切割、编码,并对输入端之间的作用进行编程,完成了6位平方根和9位立方根的逻辑电路(图4).这个工作是通过多个逻辑器件来进行多个并行操作高效地集成到高阶逻辑网络中,类似于电子中的无线集成[22-23].如果要完成更大规模的6位平方根和9位立方根逻辑电路,则需要通过DNA杂交、支点介导、DNA链置换(Toehold-mediate DNA Strand Displacement,TDSD)和DNA纳米材料来实现.
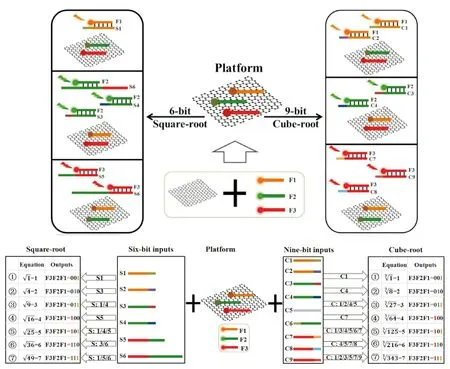
图4 平方根和立方根杂交反应机理
如果可以把握好DNA与GO的相互作用,由GO的独特性质设计其输出信号,可以为大规模的生物计算提供可能.以前的工作大多是DNA杂交、催化核酸和酶来操作逻辑电路,这大大地限制了生物计算的反应环境.例如,之前利用DNA杂交和TDSD,实现了一个10位平方根逻辑电路[24],但存在一定的局限性.这次通过智能反应平台的编程,在同一个系统上实现了6位平方根和9位立方根逻辑电路,与之前的工作相比,不仅减少了反应时间,而且还降低了复杂度[25].这个智能系统提供了简单的方法和强大的数据库,开拓了生物计算在数学方面的应用和可编程性器件的视野.
在化学反应电路中,基础逻辑门具有很低的重复利用率,造成了很大的浪费,因此,孙军伟等[26]以此为出发点,利用使能控制端思想以及DNA链置换的方法将基本逻辑门进行组合,设计了与或、与非或非和异或同或3种可重复利用的DNA组合逻辑门.通过实验利用这3种组合逻辑门实现了6种不同的逻辑运算功能,并且利用这3种组合逻辑门构造了一个4输入3级联的分子逻辑电路(图5).使用Visual DSD软件进行仿真验证,结果表明,设计的电路通过正确的输入信号是可以实现正确的输出结果的,真值表见表1.这种设计简化了运算的复杂性,是在逻辑电路上的一种新的尝试,为DNA计算提供了新的思路.
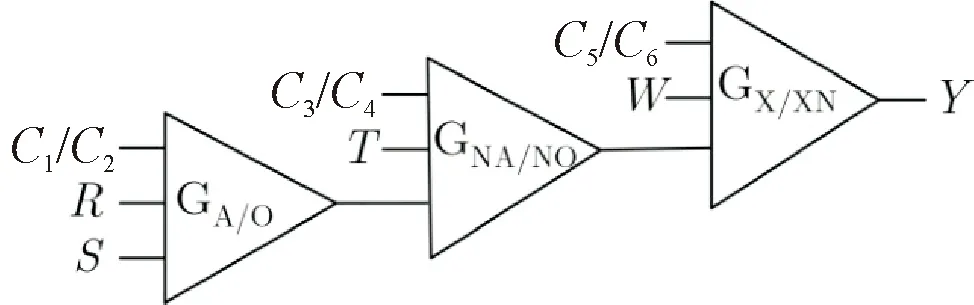
图5 三级联分子电路
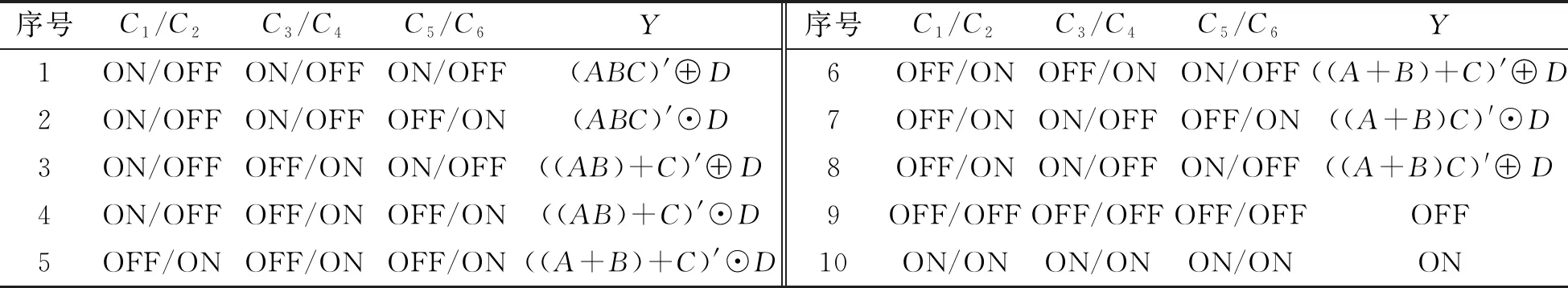
表1 4输入3级联组合逻辑电路真值表
建立在丰富的DNA计算和链置换电路的基础上,王宾等[27]展示了分子系统如何表现出类似大脑的自主行为.通过使用神经元来实现逻辑运算AND、NAND和OR并将其级联构建了一个基于DNA链置换的赢家通吃(Winner-Take-All, WTA)神经网络(图6),利用WTA神经网络解决了复杂的线性不可分问题.WTA神经网络使用了更少的DNA链,因为其不需要进行信号的放大和恢复.从网络形式上来说,文中实现的是前馈网络,其具有比反馈网络更快的反应速度,而且与Wang等[28]的双轨电路输入相比,本文所采取的单轨输入使电路更加简单,反应更加迅速,从而简化了电路结构,提高了工作效率,降低了成本.同时在Visual DSD[29]中也获得了理想的实验结果.又以此为基础构造了一个3人表决器(图7),以进一步探究神经元级联的可扩展性,然后,基于DNA链置换实现了科学家的分类(图8).Genot等[30]实现的电路14个输入中只有两个可以分类,完成率为14%,而本文所用到的方法,尽管使用了更多的输入链,但其能够将14个输入中的13个进行正确分类,分类效率达到了92%,远远超过了前者.
图6 WTA神经网络实现XOR
图7 3人表决器的WTA简化结构图
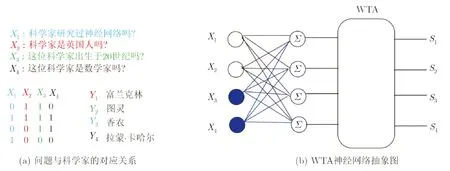
图8 分类电路的结构
2 DNA自组装算法
DNA计算的另一种方式是通过自组装来实现的.自组装是指通过小部件自动组装成大型复杂结构的过程.自组装算法即通过给系统嵌入一定的算法逻辑,使系统自动完成一系列简单的生长任务.Doty等[31]在2012年展示了DNA自组装算法的原理,同时该原理通过多达22种瓦片(Tile)类型的DNA纳米技术得到了实验证明.
Woods等[32]在这方面进行了更加深入的研究,并于2019年构建了一种普遍性的DNA自组装系统以实现各种算法.该系统包含了355个单链瓦片的瓦片集合,通过设计迭代布尔电路模型,让初始构建的算法系统在得到输入后自动增长成纳米管,根据增长后最终得到的周期性值或固定值确定结果.为了实现迭代布尔电路模型,他们将4个设计好的单链瓦片通过碱基互补配对组合成一个抽象瓦片,然后根据抽象电路模型中的逻辑门真值表设计对应的抽象瓦片,形成初始种子,之后初始种子根据输入值以及碱基互补配对的规则进行自组装并得到最终计算结果.为了证明这一构思,Woods等人用该系统构筑了针对各种算法的电路,比如复制、排序、识别回文和奇偶校验等实验,所得结果整体错误率小于1/3 000.
3 DNA计算的应用
近几年DNA计算的应用在不同领域都有了一定的研究成果,本文从三个方面进行概述.
3.1 DNA计算与图像加密
在互联网时代,数据安全越来越受到人们的关注与重视,如何才能保证数据的安全传输成为近些年研究的热点.数字图像作为数据载体的一种重要方式,对于其加密方法也已经有了诸多研究.但是,彩色数字图像具有很大的信息量,且其各像素间相关性很强,对计算机的计算速度和存储容量的要求较高,所以传统的加密算法很难再继续运用在数字图像中.
众所周知,DNA计算拥有高度并行性、存储量大和计算能耗低等优点,这无疑对密码学领域具有相当大的吸引力.赵凤[33]在此基础上基于混沌置乱和DNA计算等技术提出了一种对彩色图像进行加密的新的方法,整体流程见图9.
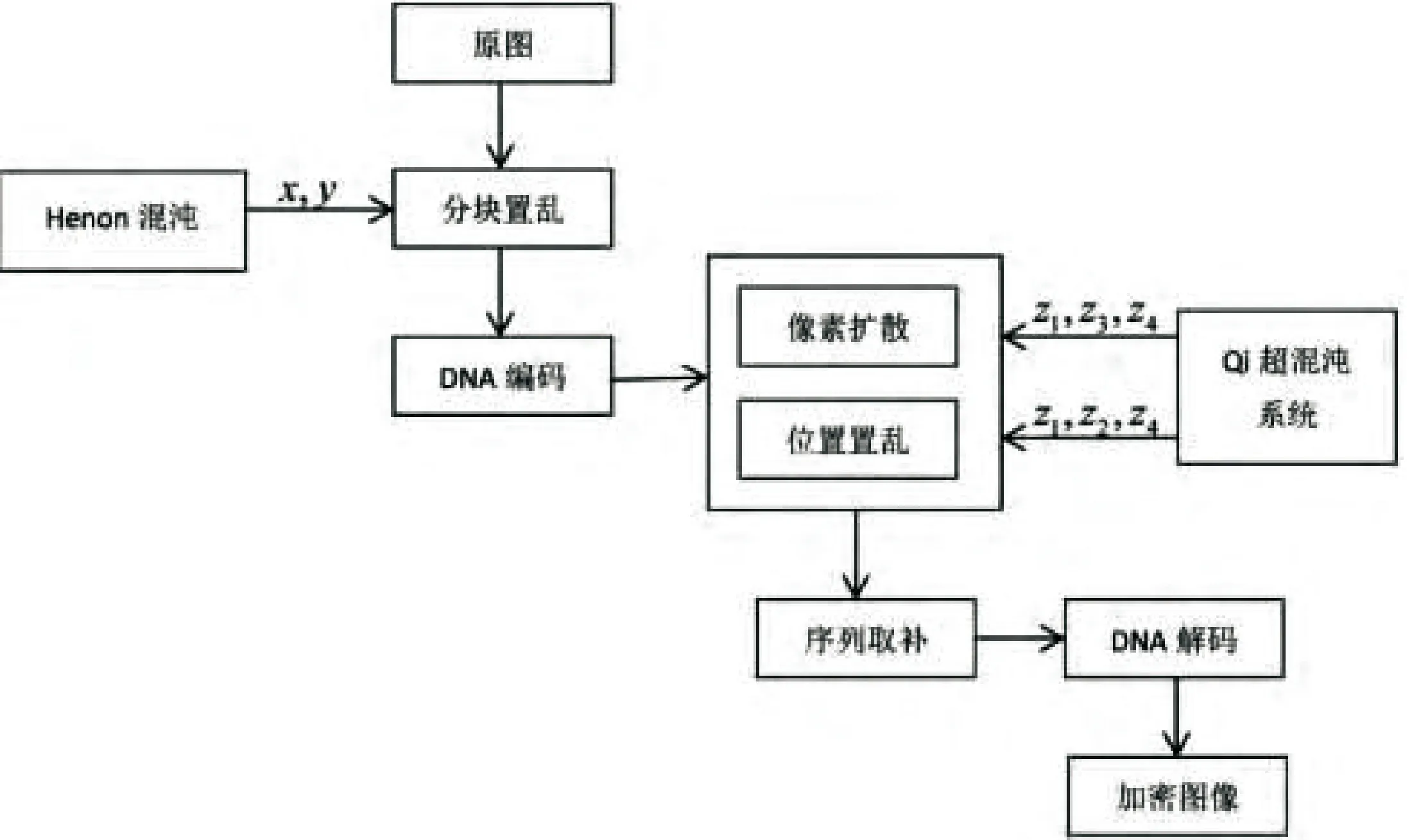
图9 加密方案结构图
首先将原图分块处理,利用Hénon映射将分块后的子图置乱,然后将置乱后的子图分离成三个通道进行DNA编码,使用Qi超混沌产生的随机序列对DNA序列进行深度位置置乱和像素扩散,最后将序列逐项取补后解码得到加密图像.
为了验证加密方法的效果对算法进行了仿真实验,仿真结果见图10.加密后的图像是无法获取任何信息的,而解密后的图像与原图几乎没有差别,说明此加密算法是有效的,且对明文攻击和穷举法攻击等具有较好的抵御效果,从而具有较高的安全性.

图10 加密、解码仿真结果
在云计算领域中,使用DNA计算来进行数据的加密也是一项十分重要的研究内容.许多研究者对云计算环境中的数据安全性做过许多的研究与尝试.Tanaka等[34]在2005年提出了一种利用DNA完成的公钥系统(Public Key System by using DNA, PKSDNA),解决了密钥的分配问题,但是却增加了数据访问时间,并且这种方案没有办法抵御穷举攻击.2016年,Tuncer等[35]通过使用DNA异或(DNA-Exclusive OR, DNA-EXOR)操作,提出了一种概率秘密共享方案(Probabilistic Secret Sharing Scheme, PSSS).在PSSS方案中使用了基于DNA的真值表,但是PSSS容易受到侧信道攻击.2017年,Sheela等[36]通过使用混合混沌移位变换、混沌图谱和DNA编码规则,提出了一种基于DNA的密码系统(Novel Cryptosystem based on DNA, NCDNA).该系统具有针对广泛控制参数的特殊混沌行为.但是,缺点是这个密码系统无法防止内部攻击.同年,Wang等[37]提出了一种基于DNA的可逆数据隐藏方案,用于加密用户的机密或敏感数据,但该方案没有提供用于保护用户数据的强大安全技术.Wang等[38]在2018年提出了一种基于耦合的图格和DNA序列的加密方案,但是,此方案的加密过程很缓慢.2019年,Wang等[39]提出了一种基于重组DNA技术和DNA序列双层数据隐藏(Double Layer Data Hiding, DLDH)方案.在此方案中,将消息或数据隐藏在使用映射规则生成的伪DNA序列中,然后使用重组DNA技术将其隐藏在活生物体(DNA质粒)中.DLDH可以防止密码猜测攻击,但同时也增大了系统的开销.2020年,Namasudra等[40]提出了一种基于DNA计算的新的数据访问控制模型,以解决过去研究中存在的一些问题.在这个模型中,通过云盘服务提供者(Cloud Service Provider, CSP)根据数据大小和数据类型维护一个临时表,从而能更加快速地访问数据.通过使用基于1 024位DNA的密码或密钥来加密用户数据,从而提高了数据的安全性.同时,此模型在云环境中可以防止内部攻击、穷举法攻击、分布式拒绝服务攻击以及侧通道攻击等.实验证明比其他已有方案效果要更加优越.
3.2 医疗应用
肿瘤转移是临床医学面临的一个艰巨问题,因为其存在着很多miRNAs和酶的异常表达.DNA纳米系统可以用于哺乳动物细胞中的RNA或酶反应的可视化[41-43].Bai等[44]通过DNA纳米计算技术跟踪活细胞中的生物分子,通过数字输出识别肿瘤转移的过程.其工作是做了细胞内熵驱动的多价DNA电路实现多位计算,该多价DNA电路也具有体外性能.通过细胞内端粒酶[45-47]、miR-21和miR-31(microRNAs)的复合DNA组装构建了一个电路系统,用低温电子显微镜(Cryo-EM)和凝胶电泳进行分析提出一种多臂结构,该多臂(Substrate Probe, SP)也被证明可以组装和拆卸.这三个生物分子能进行信号放大,进而通过各自触发DNA链进行置换反应,信号输出被编码成不同的细胞多位二进制码,从而可以区分出癌细胞的类别.实现了一种可用于临床的多价细胞分析芯片(图11),为临床医学分析作出了一定的贡献.
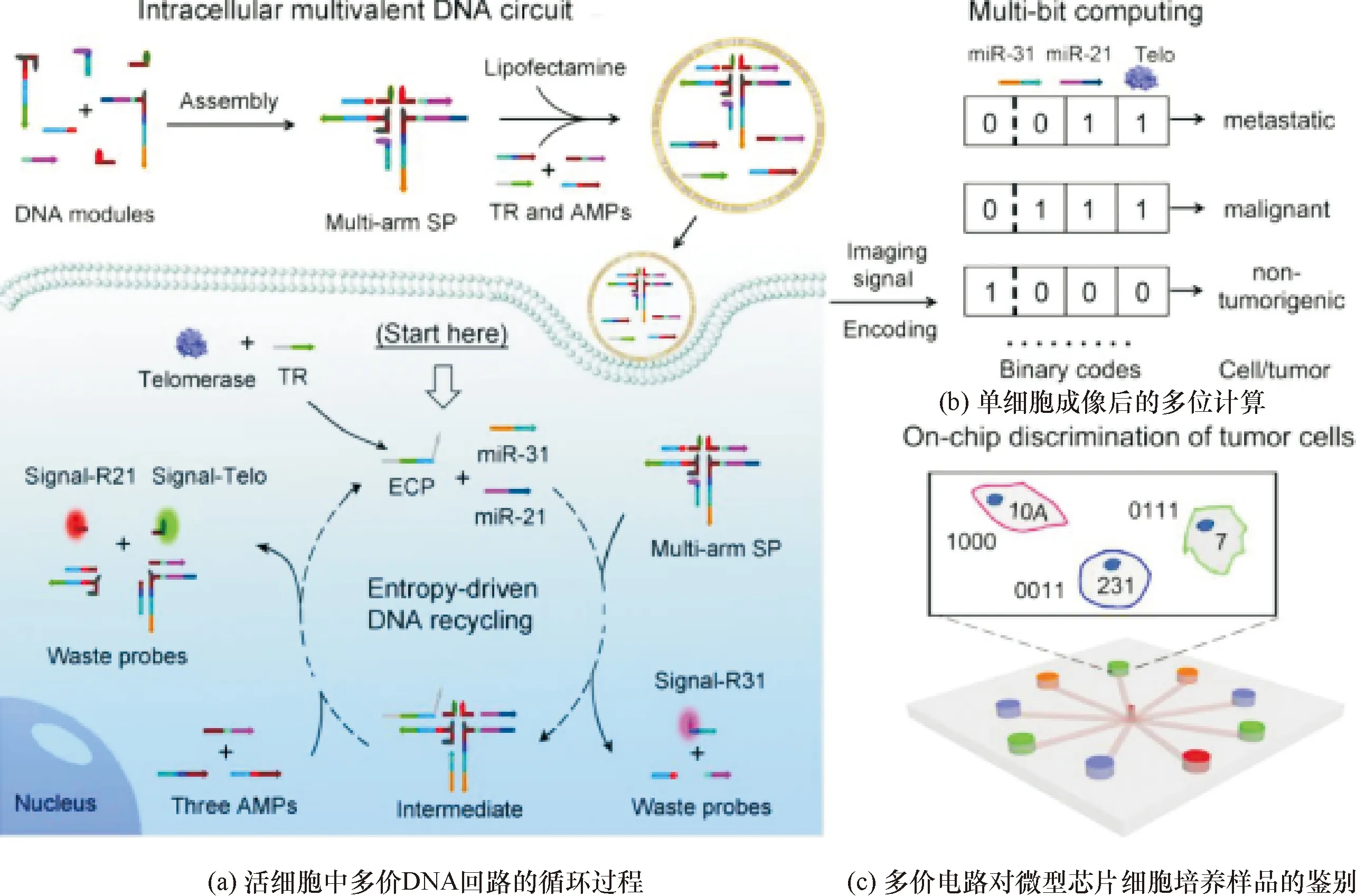
图11 多位DNA计算用于细胞类型和肿瘤进展的示意性描述
生物医学中探针杂交也被广泛应用.运用DNA纳米技术结合实验和模拟策略来促进核酸杂交的设计操作.目前,大部分的技术平台都是运用杂交探针和引物进行序列的识别、鉴定和组装.但这存在一定的局限性[48-51].最近介绍的一种DNA均衡器门(DNA Equalizer Gate, DEG)方法[52]是用于模拟核酸杂交探针和生物传感器的设计(图12),可以识别双链DNA中单核苷酸变体的监测窗口.通过热力学驱动理论模型定量地模拟DEG的性能,并在硅片和实验上验证了DEG对扩大检测窗口和提高序列选择性具有良好效果.DEG不仅在实验上得到了验证,而且还通过仿真得到了定量和精确的预测.
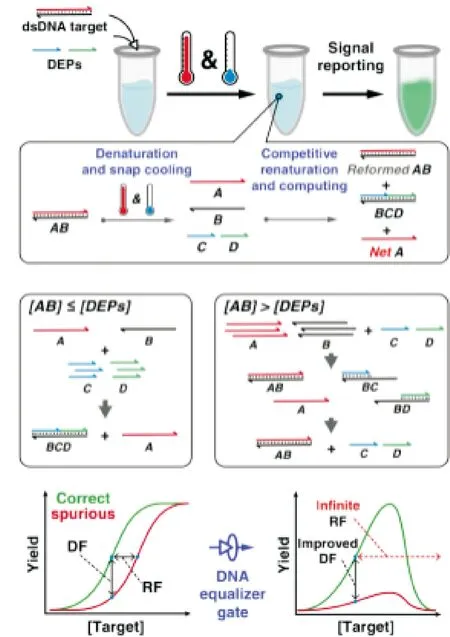
图12 DNA均衡器门(DEG)示意图
DEG为DNA纳米技术的研究做了铺垫,也为核酸杂交探针的实际应用作出了贡献.DEG的优点在于通过二甘醇将检测信号与目标浓度之间的定量关系转化为一个不对称的单峰函数.杂散目标的信号还是受DEG影响,进而抑制正确目标的量化.它还是一个简单免清洗的无酶程序.DES与现在策略最大的不同是其不需要经过复杂的程序生成dsDNA结构域[53],它巧妙的设计与上游进行PCR和下游检测探针完全兼容,信号泄漏少.同时也验证了DEG对于鉴别碱基突变有一定的效果,如单核苷酸a到G的突变,DEG在基因突变方面有一定的贡献,它也成为在临床环境中进行分析基因的合适工具.
3.3 CRN和DSD仿真软件
生命的基本成分——脂质、核酸和蛋白质都可以通过化学合成.化学合成分子之间的简单相互作用可以实现许多行文,包括数十到数百个化学合成分子的系统.目前,最广为人知的形式化学语言模型是抽象化学反应网络(Chemical Reaction Network, CRN),它可以将化学反应系统中的每个化学反应解释成从抽象的反应物多重集合到抽象产物的多重集合映射.目前,该模型已经可以对域级DNA链位移(Domain-level Strand Displacement, DSD)系统进行编程.为了能够更方便地对域级DNA纳米技术的反应网络进行枚举和模拟,Spaccasassi等[54]开发了Visual DSD的软件.该软件可以支持有限种类的DNA结构(例如,没有发夹环,没有分支的结构),以及这些结构之间内置的一套共同的预期反应规则.利用Visual DSD可以解释一种名为Logic DSD的编程语言[54-55],该语言已被用于枚举和模拟更广泛的DNA相关系统.然而,Logic DSD可能要求用户对系统有广泛的先验知识,以便为枚举制定适当的反应规则,并为模拟提供反应速率.
Badelt等[56]提出了三种算法:①DSD反应网络的枚举算法;②用总体慢反应来表示给定CRN的凝聚算法;③DNA域级系统的近似速率模型.之后,他们用这些算法和模型开发了另一款仿真软件——Peppercorn.该软件提供了一套详尽的无假结核酸二级结构空间内的分子结构域级反应.这类二级结构的绝大多数构象在空间上是可以构筑的,并且可以通过标准核酸结构预测软件使用成熟的DNA和RNA热力学能量模型[57]来很好地建模[58-60].此外,Peppercorn还提供了基于构象变化的生物物理学的近似动力学模型.
Badelt等[56]通过将软件枚举和仿真数据与实际的实验进行对比,得出了以下的结论:①Peppercorn可以为这些体系[61-65]列举出一个有效的反应网络;②所列举的反应可以为实验结果提供良好的估算率,以此表明选择了合理的单个反应途径概率;③模型无法计算准确的特别系统完成时间,因为它不补偿反应之前预期的完成时间和获得有用的结果;④在某些情况下,可能需要用户自行选择不同的反应条件或添加反应参数.
然而,编程好的CRN有一个基本问题,即用CRN语言编码的分子都在同一个空间中,要想避免两个或两个以上目标分子发生反应后产生副产物是很困难的.因此,Qian等[66]在2014年提出了表面CRN的概念.简单地说,表面CRN是类似CRN化学的简化模型,在原本分子相互作用的基础上加上了几何空间的限制.到目前为止,Clamons等[67]将表面CRN模型进一步完善,并用该模型实现了同步和异步确定的细胞自动机模拟,三种在局部区域内强制同步的新技术,连续活动的布尔逻辑电路,群体机器人的行为以及Python模拟器.
相较于CRN来说,表面CRN可以防止副反应的发生,这大大提高了CRN的可扩展性.虽然像充分混合的CRN一样,表面CRN可能没有捕捉到可能发生在被化学物质包围的表面上所有可能出现的特性,但它们仍捕捉到了表面化学的许多重要特征.此外,由于表面CRN可以大量平行存在,数十亿到数万亿的微小表面可以在一个试管中漂浮,因此,每个表面可以同时进行不同的计算.
4 总结与展望
虽然DNA计算已经在逻辑电路、计算模型研究及DNA计算应用研究等方面取得了丰厚的成果,但也存在着根本性问题:①计算速度慢;②大部分电路的通用性低;③设计相关计算电路复杂而繁琐等问题仍未得到彻底的解决.
未来DNA计算的研究将会有一些新的突破,例如,探索更加符合实现快速紧凑级联电路的实现方法;实现自组装算法模型通用性的扩大;在完善仿真软件等方面进行更深入的研究.同时,相信伴随着DNA纳米技术在结构上的发展以及DNA计算的研究,现存的问题也会得到进一步解决,在生命科学、计算机技术、医学等领域得到突破,将人类社会带入一个全新的纪元.

