SARS-CoV-2非结构蛋白NSP14的生物信息学分析
许勇,李静,王红萍,张忠东,王娟,胡倩,赵娜娜
安徽省医学科学研究院,安徽 合肥 230061
从2019年12月至今,新型冠状病毒病(Corona virus disease 2019,COVID-19)已在全球蔓延,感染人数超过400万人,逾28万人死亡,对全世界人民的健康生活造成了重大影响[1]。新型冠状病毒属于β属冠状病毒,有包膜,颗粒呈圆形或椭圆形,常为多形性[2]。2020年2月11日,因与SARS-CoV同属于β冠状病毒属的Sarbecovirus亚属,国际病毒命名委员会将其命名为严重急性呼吸系统综合征冠状病毒2型(severe acute respiratory syndrome coronavirus2,SARS-CoV-2),与SARS-CoV是姊妹病毒[3]。
和SARS-CoV等其他冠状病毒类似,SARSCoV-2的遗传物质为RNA,基因组全长约29 kb,共编码9860个氨基酸残基,其5'端约2/3的区域由2个大的开放读框ORFa和ORFb组成,编码约16种非结构蛋白(nonstructural protein,NSP),后1/3区域依次编码刺突(spike,S)蛋白、小包膜(envelope,E)蛋白、膜(membrane,M)蛋白、核衣壳(nucleocapsid,N)蛋白和一些辅助蛋白[4-5]。在SARS-CoV中,非结构蛋白NSP14全长527残基,包括3'-5'核酸外切酶和N7鸟甘甲基转移酶(N7-MTase)2个功能结构域,对病毒RNA的复制转录很重要,是抗冠状病毒治疗的潜在药物靶点[6]。
由于SARS-CoV-2出现的时间较短,实验室研究尚需时间,运用生物信息学方法对病毒的基因组、蛋白质等性质进行分析预测,了解病毒的感染机理,为疫苗、药物开放奠定理论基础是一种重要的研究方法[4,7]。Chan等通过基因组序列的比对分析,对SARS-CoV-2基因组的构成和编码蛋白质进行初步预测,发现SARS-CoV-2与蝙蝠中类似SARS的冠状病毒密切,而它自身的一些独特序列具有进一步研究的价值[4]。Baruah等运用生物信息学手段对SARS-CoV-2表面糖蛋白的B细胞和T细胞抗原表位进行预测,为疫苗的研发奠定理论基础[8]。目前未见针对SARS-CoV-2中NSP14的生物信息学研究相关报道。
本研究基于NSP14的氨基酸序列,运用生物信息学相关软件分析和预测NSP14蛋白的性质和结构,为新型冠状病毒药物筛选提供理论依据。
1 材料与方法
1.1 材料
SARS-CoV-2中NSP14蛋白的氨基酸序列从NCBI官网下载(YP_009725309)。
1.2 方法
采用NCBI中的Blastp工具比对SARS-CoV-2与SARS-CoV中NSP14蛋白序列的同源性;采用ProtParam分析理化性质[9];采用PredictProtein[10]对NSP14蛋白的二级结构、二硫键和功能基序进行预测;采用SWISS-MODEL[11-12]进行同源建模;采用MolProbity软件中的Ramachandran[13]对建模结果进行评估;采用CastP[14]预测活性区域。
2 结果
2.1 NSP14氨基酸序列的比对
通过Blastp比对发现,SARS-CoV-2与SARSCoV的NSP14氨基酸序列一致率达95%,阳性率达98%(包括氨基酸种类不同但性质相似的位点),说明2种病毒的NSP14具有很高的同源性。如图 1所示,第 31、128、176、220、228、259、260、307、319、374、396、437和 493位氨基酸残基不同且性质差异较大。
2.2 NSP14的理化性质分析
如表1,NSP14由527个氨基酸残基组成,含量最高的残基是缬氨酸(8.3%),最低的是色氨酸(1.9%)。NSP14的相对分子质量为59 815.67,理论等电点为7.80,不稳定系数为28.85,总平均亲水系数为-0.134。综上所述,NSP14是稳定的亲水性蛋白质。
2.3 NSP14的二级结构组成、二硫键和保守基序预测
经PredictProtein预测,NSP14的二级结构中含量最高的是无规卷曲(45.83%),其次为α螺旋(35.87%),延伸链的含量最低(18.30%),蛋白质中不含二硫键。NSP14中包含N-糖基化位点、cAMP或cGMP依赖蛋白激酶磷酸化位点、蛋白激酶C磷酸化位点、酪蛋白激酶Ⅱ磷酸化位点和N-豆蔻酰化位点等5个保守基序(表2)。
2.4 NSP14的三级结构建模和活性区域预测

图1 SARS-CoV-2与SARS-CoV中NSP14的氨基酸序列比对
SWISS-MODEL建模是以5c8s.1的B链为模板,总体评价得分0.98,Qmean为-3.14,主要配体包括鸟苷-P3-腺苷-5',5'-3磷酸、S-腺苷-L-高半胱氨酸、Mg和Zn,功能预测为鸟嘌呤-N7-甲基转移酶。Ramachandran评估结果表明同源建模显示氨基酸位点都位于最佳区域内的占94.8%(496/523),氨基酸位点位于允许区域内的占99.2%(519/523),说明该三级结构模型准确可靠。如图2所示,蛋白质中包含4个长的α螺旋、2个短的α螺旋和比较明显的5组β折叠。CastP预测发现NSP14有2个活性区域,分别由34个和41个氨基酸残基构成,总面积和体积分别为1469.712 Å2和1708.967 Å3(图3)。
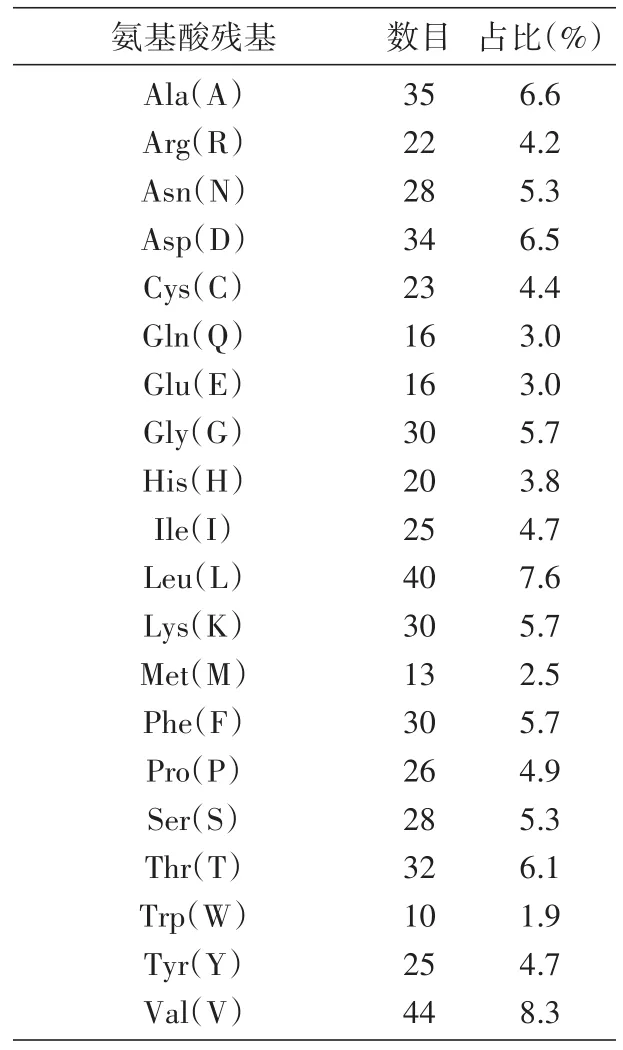
表1 SARS-CoV-2的NSP14氨基酸组成
3 讨论

图2 SWISS-MODEL对NSP14三级结构的预测

表2 SARS~CoV~2 NSP14的保守基序
大多数冠状病毒基因组RNA的5'端具有甲基化的帽子结构,该结构与真核细胞mRNA的帽子结构相似,使病毒RNA能够逃避宿主天然免疫系统的识别[15]。SARS-CoV的非结构蛋白NSP14的1~287 aa为N端核糖核酸外切酶结构域,288~527 aa为N7-甲基转移酶结构域,后者在其RNA加帽过程中发挥重要作用[6,16]。SARS-CoV-2的NSP14与SARS-CoV的NSP14氨基酸序列的一致率达到95%,CastP预测SARS-CoV-2的NSP14同样包含2个活性区域,说明SARS-CoV-2的NSP14具有与SARS-CoV的NSP14类似的功能。SARSCoV-2的NSP14的无规卷曲含量最高,为其2个活性区域提供了结构基础,同时使得该蛋白质的稳定性可能较差[17]。
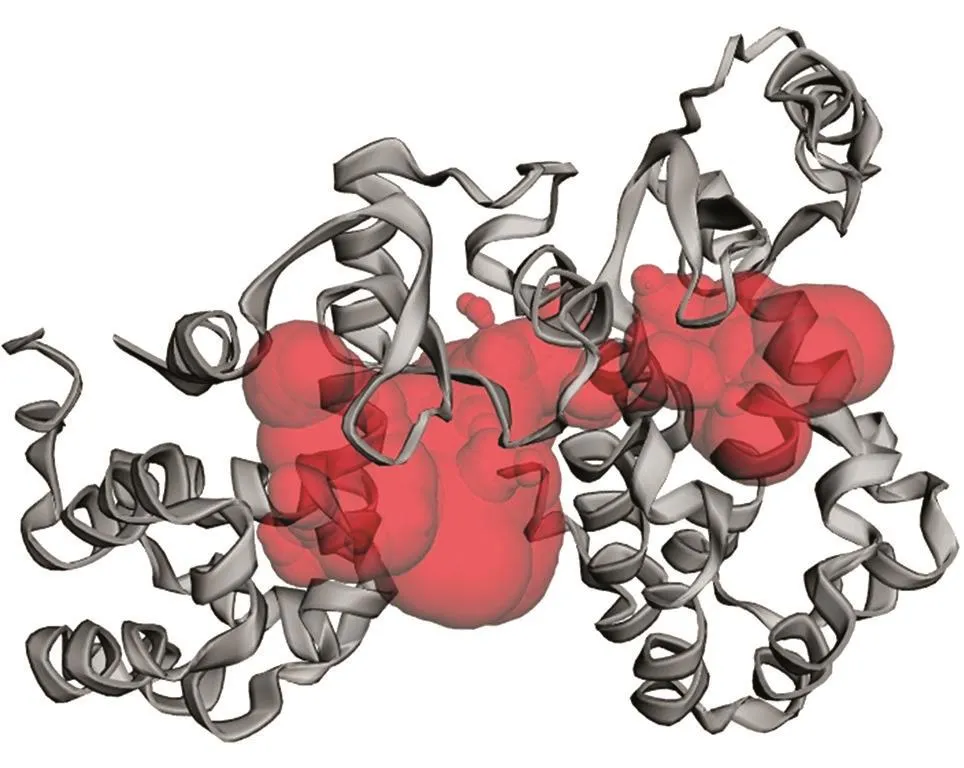
图3 CastP对NSP14活性区域的预测
冠状病毒NSP14的N端核糖核酸外切酶结构域属于核糖核酸外切酶DEDD家族,可能与病毒基因组RNA的修复有关,这可能使SARS-CoV-2的基因突变率较其他RNA病毒要低[6]。NSP14的N端核糖核酸外切酶活性使冠状病毒能抵抗包括利巴韦林等在内的许多核苷类似物的抑制作用,并能部分降低其对瑞德西韦的敏感性[18]。
SARS-CoV NSP14的 288~301 aa盘绕形成一条错综复杂的环结构[16],331~338 aa被认为是底物结合的关键位点[6],破坏这些结构会导致N7-甲基转移酶活性的丧失。SARS-CoV-2的NSP14中288~301 aa与 SARS-CoV 的 NSP14高度一致,只有293、294位点替换成了性质相似的氨基酸,而331~338 aa完全一致,说明SARS-CoV-2的NSP14具有较好的N7-甲基转移酶活性。
SARS-CoV-2的NSP14的176和374位点较SARS-CoV的NSP14有较大改变,导致其多了一个可能的N-糖基化位点和磷酸化位点。糖基化和磷酸化都是主要的翻译后修饰方式,对蛋白质功能有重要影响,这些位点的改变是否会对SARS-CoV-2的NSP14功能造成较大影响,需要进一步研究。
综上所述,SARS-CoV-2的NSP14对其抵抗利巴韦林等抗病毒药物、逃避免疫系统识别起到重要作用,以NSP14酶活性为靶点进行药物筛选研究可能具有重要意义。

