凡口铅锌矿酸性底泥可培养微生物资源的探索分离
房保柱, 王怡欢, 张 堃, 廖俊杰, 欧阳欣, 岳 秀, 肖 吉, 李文均*
(1.中山大学 生命科学学院, 广东 广州 510275;2.广东轻工职业技术学院 生态环境技术学院, 广东 广州 510300)

1 材料与方法
1.1材料
1.1.1 样品采集 样品采自广东省韶关市仁化县凡口铅锌矿(N25°2′57.5″,E113°39′34.1″),如图1所示。凡口铅锌尾矿5个采样点采集表层5 cm的底泥样品各10份,每个样品分别由5个金属钻孔获得的样品(呈圆周分布)混合而成。样品采集后,立即放入已灭菌的玻璃血清瓶中,置于冰盒中保存,送回实验室于4 ℃冰箱中保存,备用。
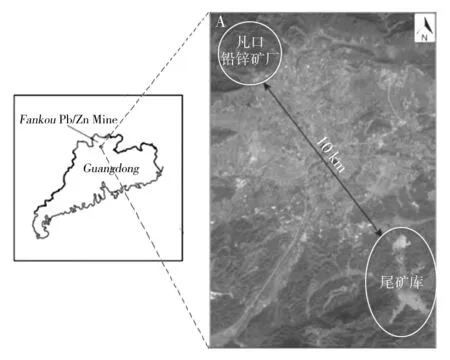
图1 凡口铅锌矿示意图Fig.1 The location of Fankou Lead-Zinc Mine, China
1.1.2 培养基 通过查阅文献选出13种微生物分离用培养基(表1)。经过筛选与优化确定3种培养基用于微生物的纯培养:①改良ISP 2培养基(3号培养基)[15](g/L),加入酪氨酸 0.4 g,pH 3.0;②改良FeO(9号培养基)固体培养基,加入连四硫酸钾0.76 g,pH 2.5;③改良9K(12号培养基)培养基[16]。所有培养基均加入100 mL采样点酸性废水原液过滤液(滤膜孔径0.22 μm)。
1.1.3 仪器与设备 电感耦合等离子体发射光谱仪(inductively coupled plasma optical emission spectrometry,ICP-OES;Optima 2100DV;珀金埃尔默,马萨诸塞州,美国);原子荧光谱法(atomic fluorescence spectrometry,AFS;AFS-820;Gi-Tian,北京,中国);光电子能谱(X-ray photoelectron spectroscopy,XPS);Star Prep Gel Extraction Kit胶回收试剂盒(GenStar北京康润诚业生物科技有限公司);pMDTM18-T Vector Cloning Kit(Takara Bio 宝生物工程(大连)有限公司);便携式pH计(HACH,5055)。
1.2 方法


表1 13种分离培养基组成
注:A、C液 121 ℃灭菌20 min, B液过滤,混合后倒平板
1.2.2 微生物的分离纯化 样品预处理:无菌条件下,称取底泥样品2.0 g于200 mL PBS缓冲液(pH 3.0)的三角瓶,放入数粒玻璃珠,28 ℃、200 r/min振荡1 h,并用PBS缓冲液(pH 3.0)分别稀释至10-2、10-3浓度。取200 μL经预处理的样品涂布于分离培养基上。分别置于20、28、37、45 ℃四组不同培养温度,10-2、10-3两组浓度梯度,在各温度的恒温培养箱中培养21 d。根据菌落的质地、形态和颜色、边缘凹凸、是否产生可溶性素及其颜等形态特征,进行初步归类。对每个分离培养基上生长的不同类群单菌落转接于培养基平板上,培养直至获得纯菌落。将初步归类为藻类的藻株接种到其对应的液体培养基中,进行摇瓶培养,200 r/min,设置光照和避光两组对照,在各温度下培养14 d并观察其生长状况。
1.2.3 微生物的分子生物学鉴定 ①菌体/藻体的收取:细菌/真菌的菌体收取:收集新鲜菌体,放入无菌的10 mL离心管中,置于液氮中快速冷冻,-80 ℃备用;藻体收取:在无菌环境下,将三角瓶中的菌液转移到无菌的50 mL离心管中,4 ℃、8 000 r/min离心10 min,去上清,用PBS缓冲液漂洗2次,再次离心去除上清。将收集好藻体的离心管放入液氮中快速冷却,然后置于-80 ℃备用。②DNA 的提取:细菌的基因组提取采用简化的酶小量法提取[19]。提取基因组DNA加入50 μL的1×TE,溶解后,-20 ℃保存。真菌及藻类:采用液氮研磨并配合CTAB法提取[15]。加入50 μL的1×TE 溶解基因组DNA,放置-20 ℃保存,备用。③PCR扩增:PCR扩增引物分别用于细菌、真菌及藻类的DNA扩增(表2)。④生物标记基因的确定:使用Star Prep Gel Extraction Kit胶回收试剂盒进行胶产物回收,胶回收产物利用pMDTM18-T Vector Cloning Kit进行连接转化,挑取白色克隆子,送上海生工生物工程有限公司测序。
1.2.4 生物标记基因的系统发育分析 利用EzBioCloud(http://eztaxon-e.ezbiocloud.net/)和NCBI数据库的BLAST对获得的基因序列进行比对分析,寻找与目的序列相似性最高的已知分类地位的菌/藻株作为比较对象[20-21],使用软件MEGA 7.0进行聚类分析,通过邻接法(Neighbor-Joining)构建系统进化树[22-23]。用于检验支持率的重复抽样次数为1 000次。

表2 细菌的16S rRNA、真菌的ITS、藻类的 18S rRNA基因引物序列
2 结果与分析
2.1 样品的理化特征

表3 凡口铅锌矿底泥样品理化特征
2.2 凡口铅锌矿可培养微生物多样性
利用多种分离培养基对10份底泥样品进行微生物分离,共分离得到细菌92株,真菌31株,藻类3株。去重复后最终获得41株细菌、8株真菌、1株藻,分别对其进行16S rRNA、18S rRNA和ITS的基因序列测定,上传EzBioCloud网站进行比对。结果显示,有41株不同种细菌分布在放线菌门、变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)的8个纲11个目18个科18个属,分别为酸硫杆状菌属(Acidithiobacillus)3株、产碱菌属(Alcaligenes)2株、芽胞杆菌属(Bacillus)4株、短波单胞菌属(Brevundimonas)1株、科恩氏菌属(Cohnella)1株、代尔夫特菌属(Delftia)1株、戈登氏菌属(Gordonia)2株、白蚁菌属(Isoptericola)1株、微杆菌属(Microbacterium)13株、微球菌属(Micrococcus)4株、栖水菌属(Enhydrobacter)1株、拟诺卡氏菌属(Nocardiopsis)2株、假单胞菌属(Pseudochrobactrum)1株、嗜冷芽胞杆菌属(Psychrobacillus)1株、氧化硫杆菌属(Sulfobacillus)1株、冢村氏菌属(Tsukamurella)1株(表4);有8株真菌分布在2个门4个纲5个目5个科5个属,分别为四球菌属(Teratosphaeria)2株、红酵母属(Rhodotorula)1株、青霉属(Penicillium)1株、外瓶霉属(Exophiala)2株、隐球菌属(Cryptococcus)2株(表5);此外,S2样品分离获得1株Galdieria属的红藻,与藻株Galdieriasulphuraria最相近,其 18S rRNA基因的相似性为93.73%。

表4 原核微生物16S rRNA基因相似性比对结果

续表4

表5 部分菌株ITS基因相似性比对结果
以上分离结果显示,通过前期不同培养基、温度和稀释梯度的培养,不同样品表现出了不同的分离效果。其中,样品S1、S2、S8的微生物资源多样性较高,且分离效果较好。在所选择培养基中,改良ISP 2培养基和改良9号培养基的培养效果更好,分离得到菌株多样性较为丰富。
2.3 凡口铅锌矿可培养微生物系统进化分析
将所分离微生物的16S rRNA、ITS或18S rRNA基因序列进行BLAST比对分析,选取与每个种类菌株相似性最大的典型菌株的16S rRNA、ITS或18S rRNA基因序列,利用软件MEGA 7.0进行聚类分析,构建系统发育树。
结果如图2所示,18个属中的细菌主要包含放线菌门的戈登氏菌属(Gordonia)、微杆菌属(Microbacterium)、微球菌属(Micrococcus)、拟诺卡氏菌属(Nocardiopsis)、冢村氏菌属(Tsukamurella)、白蚁菌属(Isoptericola)(图3);其次变形菌门的产碱菌属(Alcaligenes)、代尔夫特菌属(Delftia)、酸硫杆状菌属(Acidithiobacillus)、栖水菌属(Enhydrobacter)、短波单胞菌属(Brevundimonas)和假单胞菌属(Pseudochrobactrum)(图4);以及厚壁菌门的芽胞杆菌属(Bacillus)、葡萄球菌属(Staphylococcus)、嗜冷芽胞杆菌属(Psychrobacillus)和科恩氏菌属(Cohnella)等;除此之外,分离获得少量真菌(图5)和藻类的纯培养,进一步说明了凡口铅锌矿可培养微生物种类存在多样性。
如图6所示,藻SYSU C17050在Galdieria属的分支上稳定,与Galdieriasulphuraria的相似性最高,Blast的结果显示两者之间的相似度仅为93.7%,其准确的分类地位需要进一步研究。
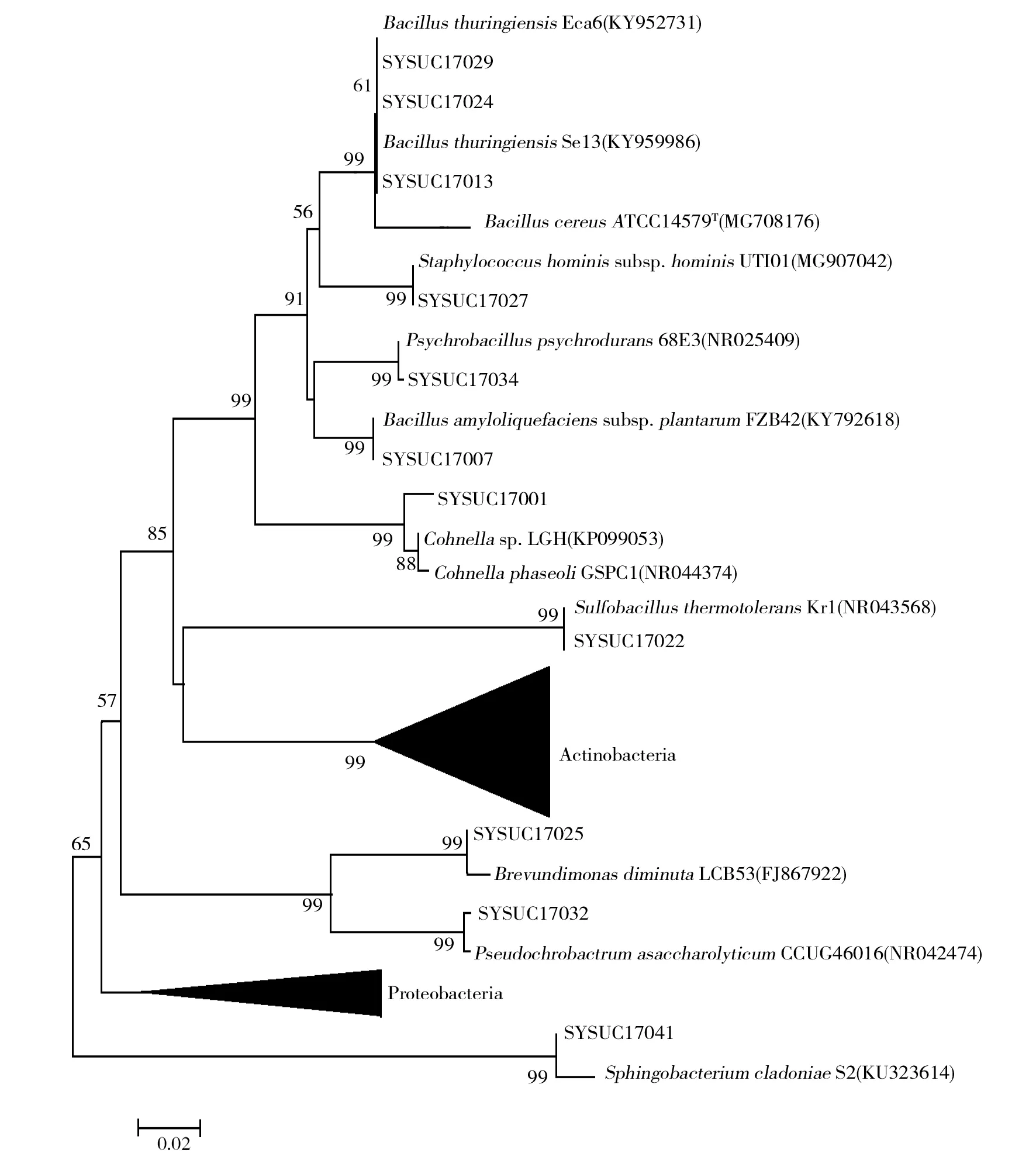
图2 基于16S rRNA 基因序列的邻位连接法构建凡口铅锌矿可培养细菌系统发育树Fig.2 Phylogenetic tree of culturable microbial community in Fankou Pb/Zn Mine, constructed by using the Neighbor-Joining method based on the 16S rRNA gene sequences

图3 基于16S rRNA 基因序列的邻位连接法构建的可培养放线菌门系统发育树Fig.3 Phylogenetic tree of culturable Actinobacteria community in Fankou Pb/Zn Mine, constructed by using the Neighbor-Joining method based on the 16S rRNA gene sequences
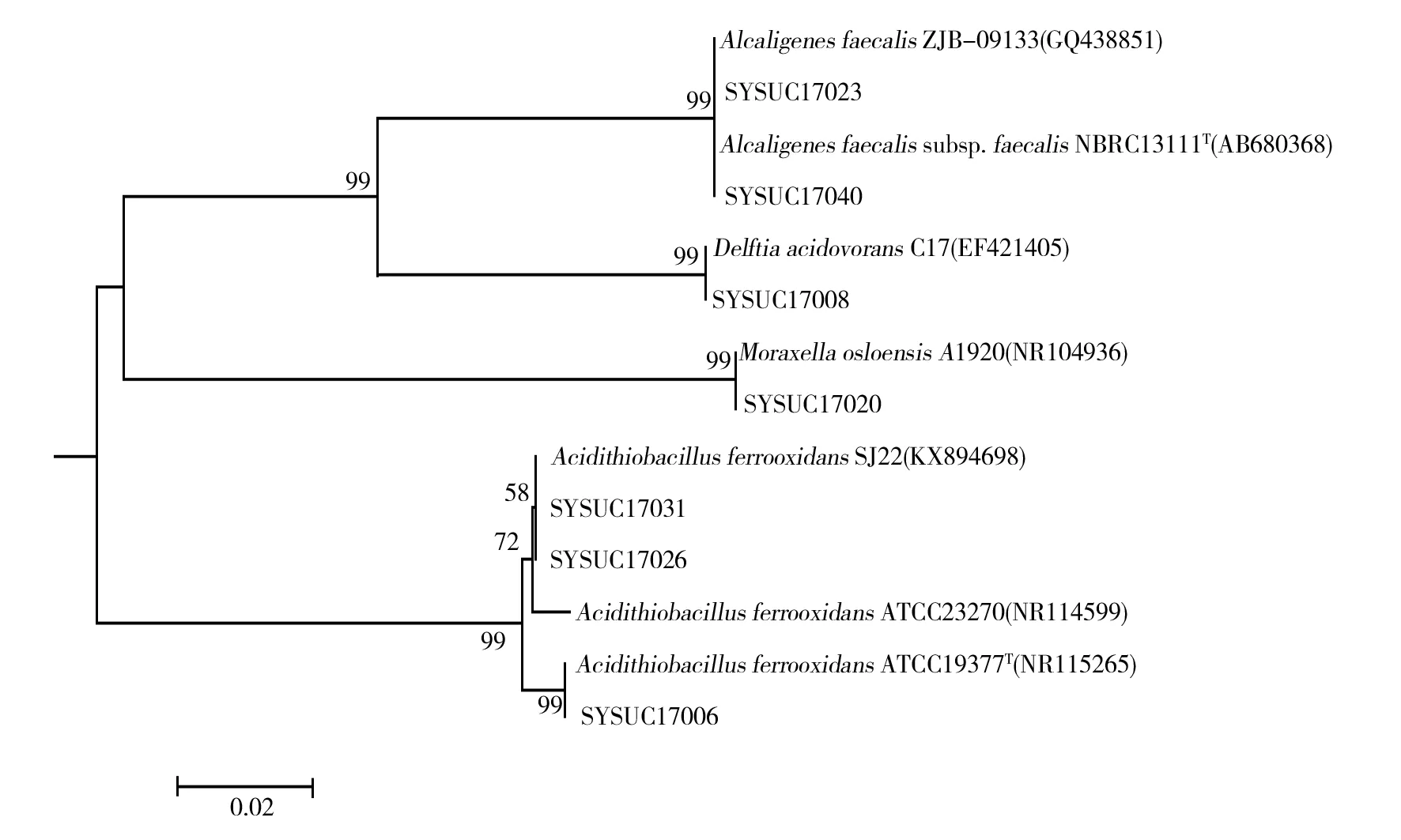
图4 基于 16S rRNA 基因序列的以邻位连接法构建凡口铅锌矿可培养变形菌门系统发育树Fig.4 Phylogenetic tree of culturable Proteobacteria community in Fankou Pb/Zn Mine, constructed by using the Neighbor-Joining method based on the 16S rRNA gene sequences

图5 基于 ITS基因序列的以邻位连接法构建凡口铅锌矿可培养真菌系统发育树Fig.5 Phylogenetic tree of culturable fungi community in Fankou Pb/Zn Mine, constructed by using the Neighbor-Joining method based on the ITS gene sequences

图6 基于 18S rRNA 基因序列的以邻位连接法构建凡口铅锌矿可培养藻类SYSU C17050系统发育树Fig.6 Phylogenetic tree of culturable alga SYSU C17050 in Fankou Pb/Zn Mine, constructed by using the Neighbor-Joining method based on the 18S rRNA gene sequences
3 讨 论
凡口铅锌矿作为中国最大的铅锌矿,在生产过程中产生大量尾矿等废弃物,由于地处亚热带气候地区,因降雨量大,温度高等特点,加速了矿山酸性废水污染的大量产生,而这类特殊酸性污染环境中存在丰富的嗜酸或耐酸的微生物资源。本研究选取广东凡口铅锌矿的多份底泥样品,结合优化已有的分离培养基对此类生境中可培养微生物资源的多样性进行探索性研究,为矿山酸性废水的生物修复提供有效的微生物资源基础。
通过多种分离培养基对嗜酸或耐酸的微生物进行全面分离,共获得细菌92株,真菌31株,藻类3株。根据形态或分子标记基因去重复,最终获得41株细菌、8株真菌、1株藻。比对结果显示,有41株不同种细菌分布在放线菌门、变形菌门和厚壁菌门的8个纲11个目18个科18个属;8株真菌分布在2个门4个纲5个目5个科5个属;1株来自Galdieria属的红藻。分离结果表明,凡口铅锌矿极端环境的可培养微生物类群丰富多样,有极大的微生物资源开发潜力。
分离得到的细菌中,微杆菌属、微球菌属和酸硫杆状菌属所占比例较高,其中酸硫杆状菌属为矿环境典型微生物。酸矿环境中的其他典型属如酸硫杆状菌属和Teratosphaeria属的菌株也在本研究中获得纯培养。酸性矿山废水属于酸度高、重金属含量高的极端生境,对于微生物而言,它们必须要有特殊的适应机制以适应生存。其中嗜酸氧化亚铁硫杆菌(Acidithiobacillusferrooxidans)为典型的适应酸矿环境的微生物菌株。一般而言,三价铁(Fe3+)是低pH环境下最主要的氧化剂[24],Fe2+的氧化又可以生成Fe3+,是一个动态循环的过程。Fe2+氧化的具体代谢途径在A.ferrooxidans中已经得到了充分研究[25]。许多研究表明,微生物具氧化还原性无机硫的作用[26-28]。在本研究的样品中,还原性无机硫化物主要以FeS和FeS2两种形式存在,而这两种形式的硫化物都不能被微生物直接利用,必须要经过环境中Fe3+(和 H+)的氧化,从而生成可以被微生物直接利用的产物[29]。耐温嗜酸氧化硫杆菌(Sulfobacillusthermotolerans)的基因组中含有大量与硫或还原性无机硫化物相关的基因[30-31],这也有可能是S.thermotolerans能够适应如此极端的生境的原因之一。
本次分离得到的藻株SYSU C17050,与其最相似藻株Galdieriasulphuraria的相似度达到93.7%,通过系统进化树的构建,藻株SYSU C17050与Galdieria属亲缘相似度最近,可能归属于同一个科。G.sulphuraria是一株温泉红藻,能够在低pH、高温、多重金属离子的极端环境下存活,并且表现出广泛的代谢多样性[32]。在自然情况下,G.sulphuraria存在于火山岩硫磺热泉、硫质土壤及一些人为干预的环境中[33]。在一些砷、铝、镉、汞等重金属含量极高的生境中,G.sulphuraria通常占总生物量的90%和几乎所有的真核生物量[34-35]。因此,本次分离得到的藻株 SYSU C17050是研究凡口铅锌矿中真核藻类极端环境适应性和新陈代谢灵活性的绝佳材料。

