放线菌分类地位的变迁及其系统学研究最新进展
李文均, 焦建宇
(1.中山大学 生命科学学院 有害生物控制与资源利用国家重点实验室,广东 广州 510275;2.中国科学院新疆生态与地理研究所 中国科学院干旱区生物地理与生物资源重点实验室,新疆 乌鲁木齐 830011)
典型的放线菌是一类革兰阳性、高(G+C)mol%含量的细菌,因其菌落呈放线状而得名。放线菌作为与人类生产和生活极为密切的微生物类群,其强大的代谢活性为人们所共识[1-2]。随着大量免疫抑制剂、抗恶性细胞增殖、杀虫剂、抗生素[3]等医疗实用组分在放线菌自然代谢产物中的发现,放线菌已经成为新药发现的主要来源[4]。放线菌作为细菌域最大的谱系之一,其门下物种繁多。随着放线菌类群的逐渐增多,对放线菌系统学进行研究,解析物种之间的相互关系显得十分必要。本文以放线菌系统学研究进展为主线,主要介绍放线菌系统学的研究方法、发展简史、最新进展及未来的发展方向,为放线菌资源研究和开发利用提供重要的理论基础。
1 放线菌系统学的研究方法
从方法学的角度来讲,放线菌系统学的研究方法与其他原核微生物一样,经历了三个阶段。
第一阶段是建立在形态学基础上的,主要根据基内菌丝和气生菌丝的有无,基丝的颜色,色素的有无及颜色,孢子的有无、形状及表面结构等形态学特征来对放线菌的各个类群进行区分。大部分放线菌都能产生基内菌丝,基内菌丝再分化为气生菌丝,而后形成孢子[5-6],但有些放线菌不产生气生菌丝因而也没有孢子;部分放线菌的基内菌丝呈单支,有的会形成分支,还有的基内菌丝会很发达[7];有些放线菌孢子直接在基内菌丝形成,如Micromonospora、Micropolyspora和Thermoactinomycetes;而更多类群的孢子是由气生菌丝分化形成,如Streptomyces。不同类群的放线菌,其孢子的形态存在差别,如平滑的、疣状的、多毛的或者褶皱的等等[8]。放线菌之间所产孢子链的长度不一样,如Micromonospora、Salinispora、Thermomonospora、Saccharomonospora和Promicromonospora只产生单个孢子,而Microbispora的孢子成对出现;Actinomadura、Saccharopolyspora、Sporicthya和一些Nocardia能产生较短的孢子链;Streptomyces、Nocardioides、Kitasatospora、Streptoverticillium和一些Nocardia孢子链上的孢子数可以达到100多个;而Frankia则能形成孢子囊。此外,放线菌还能产生各种颜色的色素,如红色、蓝色、橙色、橘色、棕色等,色素的产生会由于菌株的不同,培养基的不同以及培养时间的不同而发生不同的变化[9],色素是区别放线菌类群的一个重要指标,同时也是提高自身生存能力和竞争能力的有效策略。
第二阶段建立在细胞化学特征上。这阶段微生物的细胞化学成份如肽聚糖、脂肪酸、极性脂、醌以及细胞水解液糖组分被用来区分放线菌类群。脂肪酸的类型在不同属之间有明显差异,细菌脂肪酸链的碳原子数在2~90之间变化,但通常只有C10到C24的脂肪酸具有系统分类学意义。目前脂肪酸的鉴定主要借助于美国MIDI公司研制和开发的商品化Sherlock微生物鉴定系统。全细胞水解液糖组成对属以上级别放线菌的划分具有重要意义,根据全细胞水解液糖的不同,放线菌可被划分为五种不同的类型。醌型主要有甲基萘醌和泛醌两种类型,大多数细菌含有其中的一种,或者两者都有,但放线菌类群普遍只有甲基萘醌[10]。肽聚糖对放线菌属以上级别的划分具有重要的意义,四肽链上第3位氨基酸的种类及肽桥中甘氨酸的有无都是用来对放线菌进行区别的重要指标。
第三阶段则是建立在分子特征上。这一阶段主要是根据16S rRNA基因分析、DNA的碱基组成、特征性核苷酸及DNA-DNA杂交结果对放线菌类群进行区分。基于这些分子特征,很多之前根据形态学特征进行分类的放线菌被重新分类。然而近年来随着测序技术的飞速发展,越来越多的微生物有了基因组序列,基因组序列的出现又为微生物系统学带来了新的挑战与机遇。
经过以上三阶段的发展,放线菌系统学从理论和方法手段上都实现了质的飞跃。现代放线菌系统学形成了以基因组信息或16S rRNA基因构建的系统发育基础之上,将表型研究、细胞化学特征研究和分子特征研究结合在一起,使得放线菌系统学研究越来越完善,越来越规范化。
2 放线菌系统学发展简史
自1875年Cohn首次从人的泪腺中分离并报道1株丝状病原菌——链丝菌以来,放线菌被人类所认识和利用的研究历史已有近145年。因绝大多数放线菌具有发育良好的菌丝体,19世纪以前人们曾将放线菌归于真菌。随着科学的发展及新技术的应用,人们的认识逐渐深入,才将放线菌列入细菌。克拉西里尼科夫(Krassil′nikov)首先将放线菌列入植物界、原生植物门、裂殖菌纲。后有人认为把无真正细胞核的放线菌归于植物界不妥,因此将其列入动物界和植物界之外的原生生物界(Protista)内。1968年Murray提出原核生物界(Prockaryotae)和真核生物界(Eucaryotae)之后,放线菌被归于原核生物界。1978年,Gibbens和Murray根据细胞壁的有无和细胞壁的性质建议将原核生物界分为薄壁菌门(Gracilicutes),包括革兰阴性细菌;厚壁菌门(Tenericutes),包括革兰阳性细菌;疵壁菌门(Mendosicutes),包括无肽聚糖细胞壁的细菌;柔膜菌门(Mollicutes),包括无细胞壁的枝原体类细菌,而放线菌被包括在厚壁菌门中。在1989年出版的《伯杰氏系统细菌学手册》(Bergey′s Manual of Systematic Bacteriology)中,放线菌被划分在原核生物界,厚壁菌门,分枝菌纲(Thallobacteria),放线菌目(Actinomycetales)。1987年,Woese通过对500多种生物的16S rRNA基因序列的系统发育学分析,提出了著名的生命三域学说,即真细菌域(Eubacteria)、古细菌域(Archaebacteria)和真核生物域(Eucaryota)。1990年,Woese等人通过rRNA及RNA聚合酶(RNA polymerase)分子结构特征和序列的比较发现核苷酸分子的结构和序列比表型更能揭示生命的进化关系,将地球上的生命分为三个基本类群,正式建立了三域分类系统,并将生物分类的最高等级命名为域(domain)。生命三域分别为古菌域(Archaea)、细菌域(Bacteria)和真核生物域(Eucarya),每个域包含两个或多个界(kingdom),而放线菌当时所属的厚壁菌门归于细菌域。
放线菌作为细菌域最大的微生物类群之一[11],门下物种繁多、分类系统不稳定,导致建立适用于放线菌门层级的分类系统是一个十分具有挑战性的任务。1997年,Stackebrandt等[12]首次对放线菌纲下的物种以层级分类的方式进行系统地整理,并将放线菌纲分为5个亚纲,分别是Acidimicrobiadae、Actinobacteridae、Coriobacteridae、Rubrobacteridae和Sphaerobacteridae。随后,在2004年将Sphaerobacteridae亚纲从放线菌纲中剔除,并归类到绿弯菌门下[13]。5年后,随着放线菌物种的逐渐增多,放线菌的分类单元开始快速扩展,Zhi XY[14]等在基于16S rRNA基因数据的基础上,对放线菌类群进行重新整理,将另外放线菌的4个亚纲细分为5个目,其中包括Acidimicrobiales、Actinomycetales、Bifidobacteriales、Coriobacteriales和Rubrobacterales。随着Nitriliruptoridae亚纲在2010年提出,此时,放线菌纲下共有5个亚纲[15]。在之后的几年里,《伯杰氏系统细菌学手册》简化了微生物的系统层级关系,将亚纲、亚目提升到了纲和目的分类级别,并于2012年将放线菌纲下的亚纲(Actinobacteridae、Acidimicrobiadae、Coriobacteridae、Nitriliruptoridae和Rubrobacteridae)提升到了纲的水平(Actinobacteria、Acidimicrobiia,Coriobacteriia、Nitriliruptoria和Rubrobacteria);此时,放线菌纲Actinobacteria仅包括先前Actinobacteridae下的类群[16]。此外,4个科(Conexibacteraceae、Patulibacteraceae、Solirubrobacteraceae和Thermoleophilaceae)从Rubrobacteria纲剥离出来,被再分类到一个新的纲Thermoleophilia。至此,放线菌门下纯培养的类群包括Acidimicrobiia、Actinobacteria、Coriobacteriia、Nitriliruptoria、Thermoleophilia和Rubrobacteria6个纲(见图1)。
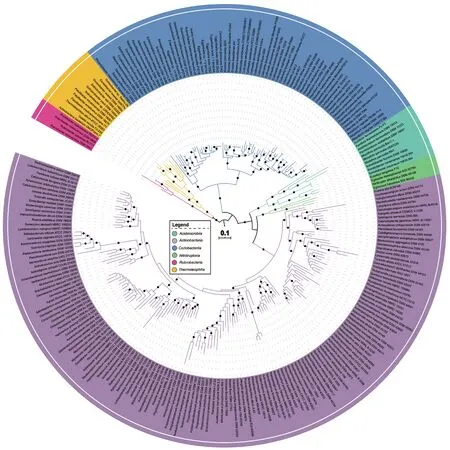
图1 基于基因组数据构建的放线菌系统进化树Fig.1 Phylogenetic tree of Actinobacteria based on genomic information
随着测序技术的发展,基因组信息逐渐成为推断物种之间进化关系、更新原核微生物分类系统不可缺少的重要证据[17]。近年来,越来越多的物种基于基因组系统学分析方法被再分类,例如Gupta等[18]将Mycobacterium再分类,重新归类出了Mycobacillus、Mycobacteroides、Mycolicibacter和Mycolicibacterium四个新属;Montero-Calasanz等[19]将Klenkia属从Geodermatophilus剥离出来;Sen等[20]同样通过基因组信息,将“Frankiales”目分类为包括Frankiales在内的4个新目(Frankiales、Acidothermales、Geodermatophilales和Nakamurellales);Nouioui等[21]基于基因组信息进行的系统发育分析,将100多个放线菌物种进行再分类,并提出了2个新目、8个新科、15个新属。这些研究,显示出了基于基因组序列进行的系统进化分析对于解决放线菌重分类问题的应用前景。尽管越来越多的放线菌物种通过基因组系统学分析被再分类,然而到目前为止,仍有大量放线菌没有可用的基因组序列,也就限制了基于基因组数据对放线菌部分类群的再分类应用。例如,由于Friedmenniellaendophytica和Friedmenniellaluteola没有基因组序列,导致Friedmenniella属下的所有物种被再分类到Microlunatus属的过程中,F.endophytica和F.luteola既没有被鉴定为Friedmenniella属的模式菌株,也没有被再分类到Microlunatus属。然而这并不是个例,同样的情况出现在Lechevalieriarhizosphaerae、Lysinimicrobiumsediminis、Verrucosisporaendophytica、Verrucosisporasonchi和Zimmermannellaalba等。基因组数据的缺失将导致这些物种在现有的分类系统中被逐渐遗忘。此外,尽管一些类群如Actinocatenispora、Aquipuribacter、Gaiella、Phytomonospora和Ornithinicoccus不论在系统发育位置还是表型上都与近缘物种差异明显,但由于没有适当的基因组信息的支撑,很难将它们再分类到一个适宜的分类地位上。近年来,以基因组数据为基础对原核微生物类群进行系统学分析已经逐渐成为不可阻挡的势头,然而目前已测序的放线菌模式菌株仅占全部放线菌模式菌株的不到50%。尽管Genomic Encyclopedia of Bacteria and Archaea (GEBA)项目和一些研究单位正在对合格发表的微生物类群基因组进行测序,然而想要获得全部物种的基因组数据仍需数十年的努力。目前来讲,很难完全依赖基因组数据将放线菌进行重新整理,构建完整放线菌系统学蓝图。
3 放线菌系统学研究的最新进展
近年来,通过基因组系统学的方法已经开始对放线菌类群进行重新分类[20-21],然而由于缺乏基因组数据,部分物种并未被再分类到相关类群。例如Friedmanniella、Lechevalieria、Lysinimicrobium和Verrucosispora分别被重新整合到Microlunatus、Lentzea、Demequina和Micromonospora下,但由于属内部分物种缺少基因组数据,因而并未被完全重新分类。近期,Salam等[22]通过16S rRNA基因及基因组系统进化分析,将放线菌物种进行再分类,将放线菌门下的物种类群重新整理为46个目、79个科、425个属(表1)。其中包括新目16个、新科10个。最主要的再分类类群集中在Micrococcales目和Frankiales目等。此外,本研究通过系统进化关系和相对进化距离对Gaiellales目进行再分类,将其作为目的分类级别归类于Thermoleophilia。同时,根据16S rRNA基因系统进化树,对先前遗漏的分类单元进行重新合并。此外,本研究提议将放线菌纲的拉丁文Actinobacteria这一不合命名法规的名称,依据ICNP中的法则8将其修订为Actinomycetia。放线菌系统学的更新见表1。

表1 放线菌门属及其以上等级分类

续表1-1

续表1-2

续表1-3

续表1-4

续表1-5

续表1-6

续表1-7

续表1-8
4 放线菌系统学研究的发展方向
微生物系统学的过去时是一部由方法学的革新而推动分类系统演变的科学史,其未来也将伴随着方法学的更新和物种认知的加深而逐步发展。基因组学发展至今,测序技术已经从早期的一代测序技术Sanger测序[23],步入到新一代测序技术的时代:包括了第二代测序技术(Next-Generation Sequencing, NGS),以及目前已投入使用的基于单细胞测序为特点的第三代DNA测序技术[24]。新一代测序技术的发展,已将人们带到了真正高通量、低成本的测序时代。这些技术的发展,从根本上改变了人类对于生命蓝图的认识方式,并且推动了基因组学的分支学科及其他相关学科的兴起。放线菌物种基因组的逐渐覆盖及积累,将引领着放线菌系统学研究逐渐步入后基因组时代,并有望通过基因组数据对放线菌系统学的研究方法提供新的理论或方法。
随着宏基因组和单细胞测序的普及,越来越多的未培养微生物类群被发现。2016年,Jilian F. Banfield课题组通过现有数据库的基因组以及宏基因组分装得到的基因组数据,通过16个核糖体蛋白序列,构建生命之树,极大地扩展了我们对微生物多样性的认知[25]。之后,Parks等人通过将1 500多个宏基因组进行分析,获得了近8 000个UBA (Uncultivated Bacteria and Archaea)基因组草图,并以此为基础,重新构建系统进化树,从基因组层面上扩展了生命之树[26]。这些研究,在刷新我们对生命之树认知的同时,同样也扩展了放线菌物种多样性。近年来,越来越多的科研工作者提议针对未培养微生物类群重新构建一个新的分类系统,以此对其基因组进行更加深入的了解[27]。随着组学时代的到来,越来越多的未培养放线菌类群基因组从环境样品直接获得,基因组数据的增加极大程度地扩展了其物种多样性,然而很少有研究关注未培养放线菌类群的进化地位和生物学功能。那么未培养放线菌物种进化地位是如何的、是否有我们尚未认知的高级分类单元、是否有一些新奇特的代谢功能,以及如何通过多组学的研究方式指导未培养放线菌类群进行富集分离?这些问题都将是未来值得探讨的重点。