副地衣芽孢杆菌FA6全基因组测序及比较基因组学分析
赵 帝 吴山功 丰文雯
(1. 中国科学院水生生物研究所农业部水产养殖病害防控重点实验室, 淡水生态与生物技术国家重点实验室,武汉 430072; 2. 中国科学院大学, 北京 100049)
地衣芽孢杆菌(Bacillus licheniformis)属于枯草芽孢杆菌近缘种, 是芽孢杆菌属中具有重要应用价值的菌种之一, 在工业发酵、农业生产、医药卫生、食品保健、水产养殖等领域应用广泛[1]。尽管同种属细菌的不同菌株表型非常相似, 但基因组的差异往往很大[2]。随着研究的深入和技术的进步,不同菌株可以聚类成2支, 这2支的菌株在基因组特征和和表型方面存在细微差异, 因此地衣芽孢杆菌的不同菌株被分为两个种, 地衣芽孢杆菌或新种副地衣芽孢杆菌(B. paralicheniformis)[3,4]。副地衣芽孢杆菌的细胞呈杆状, 是一种革兰氏阳性细菌, 其可以分泌高活性的胞外产物, 包括蛋白酶、脂肪酶和碳水化合物水解酶[3]。其中, 碳水化合物水解酶类主要包含淀粉酶、果胶酶、葡聚糖酶和纤维素酶, 这些酶有助于降解植物性饲料中的复杂的碳水化合物, 从而促进养殖动物对营养物质的吸收, 提高植物性饲料的利用率[5]。然而, 目前关于副地衣芽孢杆菌在水产养殖中研究和报道还很少。
草鱼(Ctenopharyngodon idellus)是全球水产养殖产量最大的一个品种。在中国, 草鱼的养殖产量占水产养殖总产量的18.2%以上[6,7]。草鱼是草食性鱼类, 主要以水生植物为食[8], 目前的研究表明草鱼消化植物性食物需要依靠肠道微生物的作用[9]。本课题组前期从草鱼肠道内分离到1株细菌, 16S rRNA基因序列相似性鉴定表明该菌株与和地衣芽孢杆菌DSM 13的相似性最高(99.5%), 且与地衣芽孢杆菌的生理特性基本相似, 因此最初命名为地衣芽孢杆菌FA6[10], 初步研究发现其可以在肠道内长期定植, 能够降解淀粉和纤维素等多种碳水化合物。为深入研究地衣芽孢杆菌FA6可能的益生机制, 有必要解析其基因组序列信息。本研究通过三代测序技术对地衣芽孢杆菌FA6进行了基因组测序, 进而深度挖掘其基因组信息, 推测地衣芽孢杆菌FA6可能的益生作用机制, 从而为该菌株在水产养殖中的应用提供了理论依据。同时, 本研究通过比较基因组学分析[11], 将地衣芽孢杆菌FA6与其他不同来源的4株芽孢杆菌菌株进行比较, 研究结果对于理解地衣芽孢杆菌FA6的适应性进化具有重要意义。
1 材料与方法
1.1 菌株培养及基因组DNA提取
将菌株接种于LB液体培养基(胰蛋白胨10 g/L,酵母提取物5 g/L, NaCl 10 g/L), 37℃有氧培养18h,在600 nm的波长下用紫外分光光度计测定吸光度值为1.376。菌液在7000 r/min下离心10min, 收集菌体重量约5 g, 使用QIAamp®DNA Stool Mini Kit(Qiagen, Germany)试剂盒提取基因组DNA, 步骤按照试剂盒说明书进行。最后, 用超微量分光光度计(Nanodrop 8000, Thermo)测定DNA的浓度。
1.2 基因组测序、组装与注释
细菌DNA浓度检测合格后送武汉菲沙基因信息有限公司行全基因组测序。采用全基因组扩增(Whole-genome Amplification, WGA)策略构建不同插入片段的文库, 使用Qubit 3.0和 Agilent 2100对文库质量进行检测。检测合格后基于PacBio测序平台对这些文库进行测序, 使用SMRT LINK 5.0软件进行数据处理, 采用HGAP软件[12]和Canu软件[13]对纯三代数据进行组装。采用Glimmer version 3.02软件[14]对细菌基因组进行基因结构、开放阅读框(Open Reading Frame, ORF)及蛋白基因的预测。编码蛋白质的基因通过与非冗余蛋白数据库(Non-redundant, NR)、直系同源基因簇数据库(Cluster of Orthologous Groups of proteins, COG)和基因本体论(Gene Ontology, GO)数据库[15]进行BLASTp比对,筛选条件为e-value小于等于1e-5, 获取比对得分最高的条目, 完成蛋白序列功能注释。全基因组的rRNA和tRNA分别使用RNAmmer version 1.2[16]和tRNAscan-SE[17]进行预测或扫描。
1.3 比较基因组学分析
地衣芽孢杆菌FA6的基因组序列上传至NCBI(https://www.ncbi.nlm.nih.gov/)的GenBank中, 序列号为CP033198。比较基因组学用于对地衣芽孢杆菌FA6株的基因组序列与已完成测序的近缘芽孢杆菌序列比较分析, 这些菌株包括副地衣芽孢杆菌ATCC 9945a(GenBank: CP005965)、副地衣芽孢杆菌14DA11(GenBank: CP023168)、地衣芽孢杆菌ATCC 14580(GenBank: CP000002)和地衣芽孢杆菌CBA 7132(GenBank: CP021970), 4株细菌的基因组序列及注释信息下载自GenBank数据库。统计5株芽孢杆菌基因组的基本特征, 使用Mauve软件进行基因组共线性分析[18], 设置局部共线区(Locally collinear blocks, LCBs)的最小权重值为143, 其他程序采用默认参数。蛋白编码基因的京都基因组和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路注释采用KAAS自动化注释系统完成[19]。此外, 基因组预测的ORFs用于搜索dbCAN(http://csbl.bmb.uga.edu/dbCAN/)[20], 进行碳水化合物活性酶(Carbohydrate-Active Enzymes,CAZy)家族注释和分析(http://www.cazy.org/), 参数使用E值<10-5, 覆盖率coverage > 0.35。
2 结果
2.1 基因组装与注释
菌株FA6的基因组序列与副地衣芽孢杆菌ATCC 9945a的基因组相似度最高(99.99%), 而与地衣芽孢杆菌DSM 13的基因组的相似度仅为96.01%,因此本研究将其更名为副地衣芽孢杆菌FA6。副地衣芽孢杆菌FA6的基因组由1条环状染色体组成,大小为4450579 bp, GC含量为45.9%。基因组包含4760个预测的蛋白质编码序列(Coding sequence,CDS), 占整个基因组的88.37%, 平均长度为826 bp。此外基因组包含24个核糖体rRNA、81个转运tRNA以及278个非编码ncRNA, 基因组圈图见图 1。
CDS进一步与COG数据库比对, 进行同源基因注释分类, 根据COG分类标准将基因划分为25类,以英文大写字母(A—Z)表示每一类的代码(图 2)。共有3438个蛋白获得COG功能注释, 其中参与糖类转运与代谢(G, Carbohydrate transport and metabolism)、转录(K, Transcription)、氨基酸代谢及转运(E, Amino acid transport and metabolism)、核糖体结构及合成(J, Translation, ribosomal structure and biogenesis)以及无机离子转运和代谢(P, Inorganic ion transport and metabolism)的基因丰度较高, 其数量分别为384、369、346、239和221个。将CDS编码对应的蛋白序列与GO数据库比对, 获得注释信息和功能聚类。GO数据库将蛋白序列分为三大类:生物过程(Biological process)、分子功能(Molecular function)和细胞组分(Cellular component)。这三大类又进一步分为多种过程, 注释结果显示归类为代谢过程(Metabolic process)、细胞内过程(Cellular process)、催化活动(Catalytic activity)、黏合(Binding)和细胞组分(Cell part)的基因丰度最高。

图 1 副地衣芽孢杆菌FA6基因组圈图Fig. 1 Circle map of the genome of B. paralicheniformis FA6
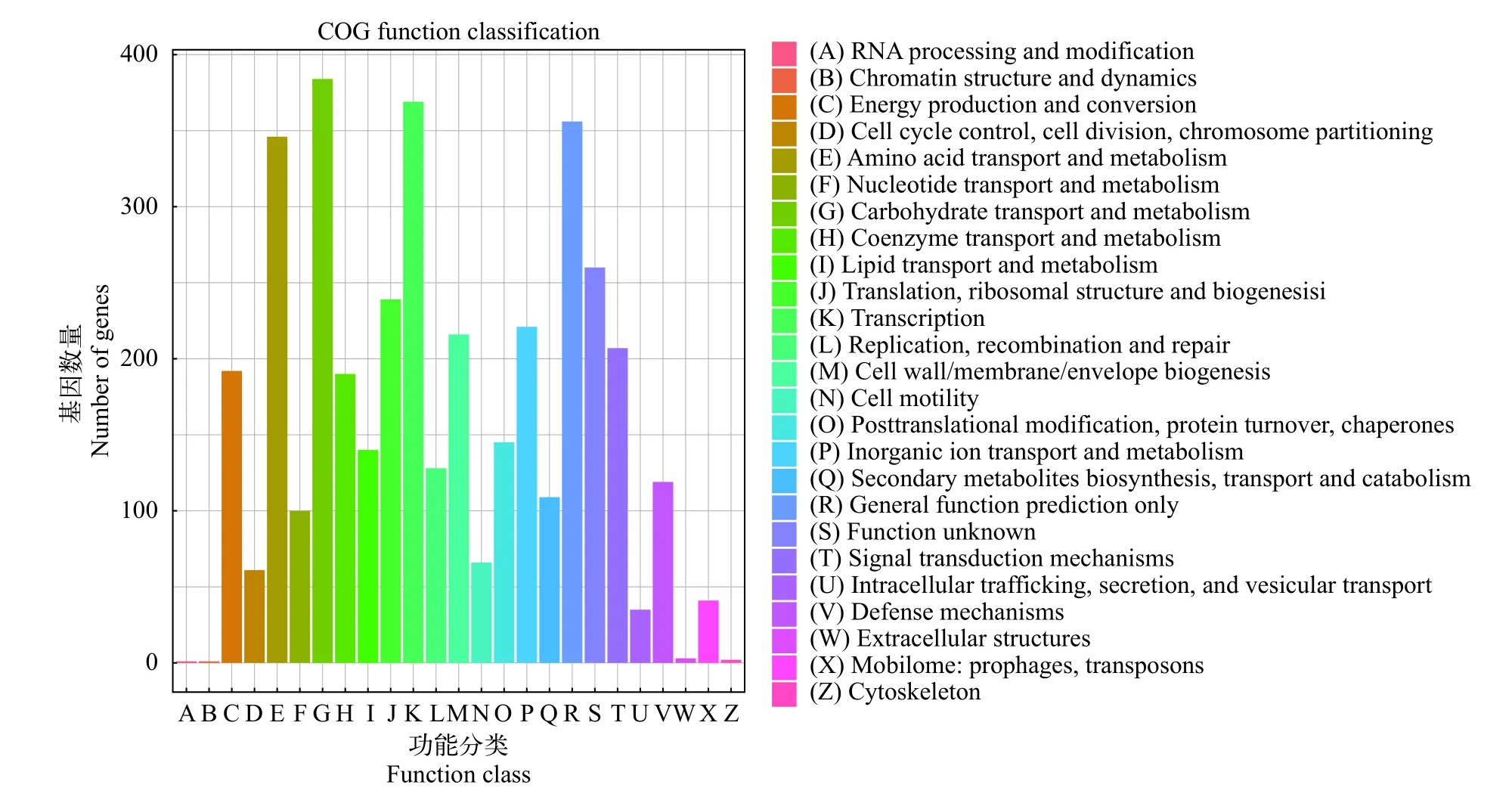
图 2 副地衣芽孢杆菌FA6基因组的COG功能分类Fig. 2 Gene distribution based on cluster of Orthologous groups of B. paralicheniformis FA6
根据基因组的注释信息, 副地衣芽孢杆菌FA6基因组含有128个蛋白酶基因, 32个脂肪酶基因, 1个次级胆汁酸合成相关基因, 以及72个糖苷水解酶酶基因, 这些基因与食物降解相关。此外, 副地衣芽孢杆菌FA6基因组含有7个编码羊毛硫抗生素(Lantibiotics)相关基因, 包括2型羊毛硫抗生素合成(Type 2 lantibiotic biosynthesis)LanM家族蛋白、羊毛硫抗生素转运蛋白(Lantibiotic transporter)LanT、羊毛硫抗生素保护ABC转运蛋白通透酶(Lantibiotic protection ABC transporter permease)MutG和MutE、羊毛硫抗生素保护ABC输出ATP结合蛋白(Lantibiotic protection ABC exporter ATP-binding protein)、羊毛硫抗生素合成蛋白LanM(Lantibiotic biosynthesis protein LanM)和羊毛硫抗生素转运ATP结合蛋白(Lantibiotic transport ATP-binding protein)SrtF相关基因。
2.2 五株芽孢杆菌基因组基本特征
5株芽孢杆菌菌株的基因组大小和GC含量相似, 基因组大小在4.21—4.54 Mb, GC含量在45.8%—46.2%(表 1)。同一种细菌的不同菌株的基因组特征更加接近, 副地衣芽孢杆菌FA6在基因组大小和GC含量方面更接近其他副地衣芽孢杆菌菌株。比较5株菌的rRNA和tRNA的数量, 结果显示, 来源于土壤的副地衣芽孢杆菌ATCC 9945a和地衣芽孢杆菌ATCC 14580的RNA数量相同, 均含有21个rRNA和72个tRNA; 来源于肠道或食物的3株细菌(副地衣芽孢杆菌FA6、副地衣芽孢杆菌14DA11和地衣芽孢杆菌CBA 7132)的RNA数量相同, 均含有24个rRNA和81个tRNA。
2.3 基因组共线性分析
采用Mauve软件对5株芽孢杆菌的全基因组进行比对分析, 共得到41个LCBs(图 3)。5株地衣芽孢杆菌的基因组间共线关系较好, 但是存在少量的插入、缺失、倒位和易位等基因重排事件。其中, 相对于其他菌株, 副地衣芽孢杆菌FA6的基因组起始端的3个LCBs, 总长度约45 kb, 出现倒位和易位的现象。
2.4 蛋白编码基因KEGG注释比较
对5株芽孢杆菌蛋白编码的基因进行KEGG注释(表 2), 副地衣芽孢杆菌FA6、副地衣芽孢杆菌ATCC 9945a、副地衣芽孢杆菌14DA11、地衣芽孢杆菌ATCC 14580和地衣芽孢杆菌CBA 7132分别有1209、1206、1167、1171和1184个代谢相关的基因映射在118个代谢通路上; 分别有284、275、272、266和263个与环境信息处理相关的基因映射在21个代谢通路上。

表 1 副地衣芽孢杆菌FA6与4株芽孢杆菌全基因组序列基本特征比较Tab. 1 General genomic features of Bacillus paralicheniformis FA6 as compared to 4 other Bacillus strains

图 3 五株芽孢杆菌基因组共线性分析Fig. 3 Synteny block of Bacillus spp. genome
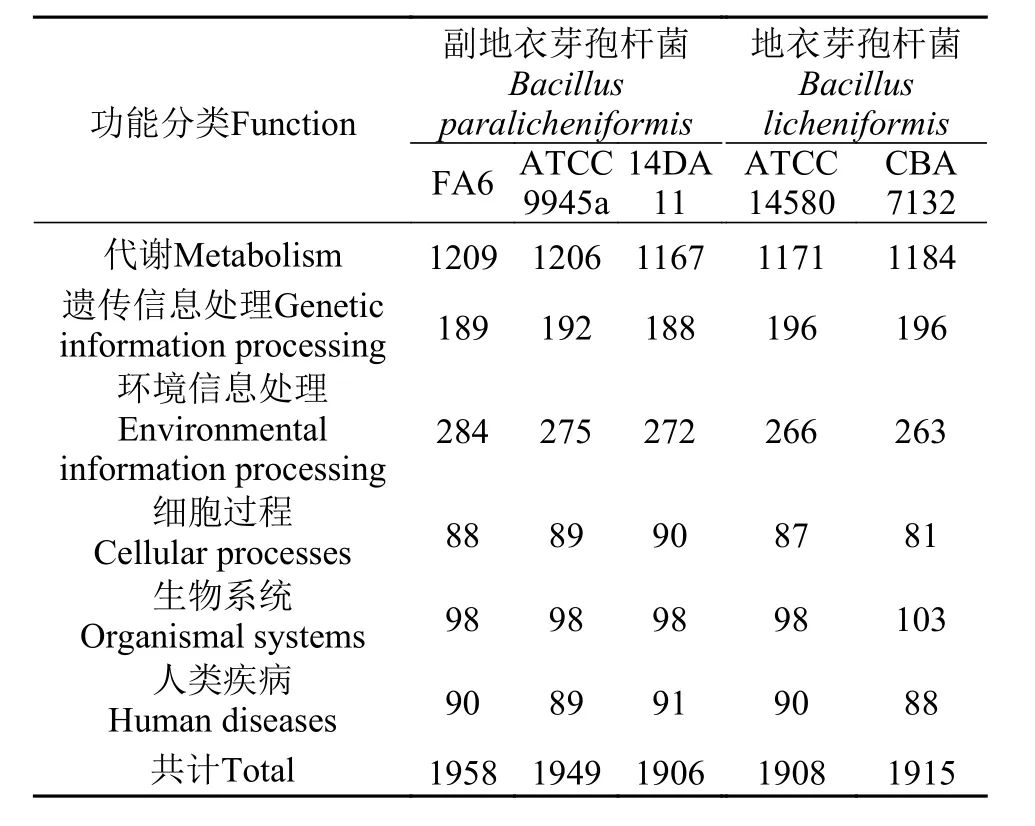
表 2 五株地衣芽孢杆菌的KEGG功能注释Tab. 2 KEGG annotation of 5 Bacillus strains
2.5 碳水化合物活性酶(CAZy)基因分析
5株芽孢杆菌的基因一共有67个CAZy家族, 包括5个碳水化合物结合结构域家族(Carbohydrate-Binding Module, CBM)、11个碳水化合物酯酶家族(Carbohydrate Esterases, CE)、30个糖苷水解酶家族(Glycoside Hydrolase, GH)、12个糖基转移酶家族(Glycosyl Transferase, GT)。副地衣芽孢杆菌FA6、副地衣芽孢杆菌ATCC 9945a、副地衣芽孢杆菌14DA11、地衣芽孢杆菌ATCC 14580和地衣芽孢杆菌CBA 7132的CAZy基因数量分别为180、127、131、132和138个; 植物多糖降解酶基因数量分别为34、32、28、27和28个(表 3); 淀粉酶基因(GH13家族)数量分别为5、4、4、4和4个。植物多糖降解酶基因包括纤维素酶、半纤维素酶、脱支酶和寡糖降解酶基因, 这些酶类基因在副地衣芽孢杆菌基因组中的数量分别是5、7、1和21个, 其中纤维素酶基因又包含4个内切纤维素酶基因(3个GH5和1个GH9基因), 1个外切纤维素酶基因(GH48)。
此外, 比较分析发现副地衣芽孢杆菌FA6在CBM50、GH4、GH23、GH28和GT2等家族的基因数量明显多于其余4株菌。其中, CBM50和GH23均与降解肽聚糖和几丁质相关。此外, 一些基因仅在副地衣芽孢杆菌FA6的基因组出现, 包括1个CE11基因、1个CE15基因和1个GT30基因。
3 讨论
本研究采用三代测序技术, 获得了副地衣芽孢杆菌FA6的全基因组序列。副地衣芽孢杆菌FA6基因组由一条环状染色体组成, 大小为4450579 bp, 整体GC含量为45.9%。比较5株芽孢杆菌(副地衣芽孢杆菌FA6、副地衣芽孢杆菌ATCC 9945a、副地衣芽孢杆菌14DA11、地衣芽孢杆菌ATCC 14580和地衣芽孢杆菌CBA 7132)的基因组特征, 发现与地衣芽孢杆菌菌株相比, 菌株FA6基因组特征, 如基因组大小和GC含量, 更接近于副地衣芽孢杆菌菌株。基因组共线性分析显示5株芽孢杆菌的共线性关系较好, 但是不同菌株基因组之间存在倒位和易位等染色体变异事件。染色体的易位可能导致某些基因的融合, 进而导致物种某些功能的改变[21],这可能是同种属不同菌株出现功能差异的原因。KEGG代谢通路比较分析发现5株芽孢杆菌中环境信息相关的基因数量差异较大, 而本研究中5株芽孢杆菌的来源差异很大, 说明菌株基因组的进化可能是为了适应生存环境。

表 3 五株芽孢杆菌GH家族植物多糖降解酶基因比较Tab. 3 Comparison of predicted GH profiles targeting plant structural polysaccharides in 5 Bacillus strains
羊毛硫抗生素是在革兰阳性细菌的核糖体上合成的一类作用于细胞膜上的热稳定小分子抗菌肽, 对革兰氏阳性菌有抑菌活性, 对革兰氏阴性菌基本无抑制作用[22]。近年来, 抗菌肽作为抗菌药物的替代品引起了广泛关注。副地衣芽孢杆菌FA6基因组中含有多种编码羊毛硫抗生素相关的基因,如LanM、LanT、MutG和MutE等蛋白基因, 具有修饰和转运功能, 这对于羊毛硫抗生素的合成开发具有重要意义。同时, 副地衣芽孢杆菌FA6可能可作为抗生素的替代品, 用于预防革兰氏阳性细菌引起的细菌病。
在天然水体中, 草鱼主要以水草为食, 水草细胞壁主要成分是纤维素和半纤维素。纤维素和半纤维素必须在纤维素酶和半纤维素酶的作用下被水解成单糖才能被动物吸收利用[23,24]。然而, 草鱼的基因组不含纤维素酶和半纤维素酶基因[25], 而草鱼肠道微生物含有大量的纤维素酶和半纤维素酶基因[26], 因此草鱼消化植物性食物需要依靠肠道微生物。与其他芽孢杆菌相比, 副地衣芽孢杆菌FA6具有较多的纤维素酶和半纤维素酶基因, 能够更好地降解植物细胞壁。淀粉是大多数高等植物细胞中一种重要的多糖[27,28], 副地衣芽孢杆菌FA6基因组中有5个淀粉酶基因, 而其他芽孢杆菌只有4个, 因此副地衣芽孢杆菌FA6能够更好的利用利用植物细胞中的淀粉类多糖。在比较的5株芽孢杆菌中, 副地衣芽孢杆菌FA6含有21个植物寡糖降解酶基因, 是最多的一株。以上这些结果说明副地衣芽孢杆菌FA6具有较为完整的植物糖类水解酶体系, 高度适应植物性成分, 反映了该菌株在草鱼肠道中的适应性进化[29]。此外, 副地衣芽孢杆菌FA6基因组中含有大量与氨基酸代谢、脂肪代谢相关的基因, 说明FA6有良好的食物降解能力。因此, 副地衣芽孢杆菌FA6可能可以作为益生菌添加到饲料中, 帮助宿主消化食物成分、提高植食性食物的利用效率。

