利用微卫星标记分析军曹鱼养殖群体的遗传多样性*
李伟强 陈 刚 马 骞 黄建盛 施 钢 潘传豪 周 晖 谢瑞涛,2 张健东 汤保贵
利用微卫星标记分析军曹鱼养殖群体的遗传多样性*
李伟强1陈 刚1①马 骞1①黄建盛1施 钢1潘传豪1周 晖1谢瑞涛1,2张健东1汤保贵1
(1. 广东海洋大学水产学院 湛江 524088;2. 广东恒兴集团有限公司 湛江 524000)
利用12个微卫星标记对北海(BH)、陵水(LS)、硇洲(NZ)、徐闻(XW)和三亚(SY) 5个军曹鱼()养殖群体进行遗传多样性分析。结果显示,12对微卫星引物在5个军曹鱼养殖群体中共检测到129个等位基因。各养殖群体的平均等位基因数为3.833~6.750,平均有效等位基因数为2.284~3.645,平均观测杂合度和平均期望杂合度分别为0.481~0.635和0.533~0.681,平均多态信息含量为0.463~0.630;哈迪-温伯格平衡检测结果显示,各群体在多个微卫星位点上均显著偏离平衡(<0.05);军曹鱼养殖群体间的遗传分化指数(st)为0.055~0.150,遗传距离()为0.240~0.635,其中,BH和NZ的st最大(0.150),最远(0.635);AMOVA分析表明,军曹鱼养殖群体的84%遗传变异来自于个体之间;基于Nei´s遗传距离构建的UPGMA系统进化树显示,BH和SY聚为一支,LS和XW聚为一支,两支聚为一支后与NZ聚为一支。研究结果将为军曹鱼种质资源保护和改良等提供科学的数据参考。
军曹鱼;养殖群体;微卫星;遗传多样性
军曹鱼(),是一种暖水性远洋洄游鱼类,广泛分布于地中海、大西洋和印度–太平洋(东太平洋除外)的热带或亚热带海域(麦贤杰等, 2005)。因其肉质细嫩,味道鲜美且无肌间刺,生长速度快等优点,已成为最具养殖潜力的海水鱼之一(李刘冬等, 2002; Nazar, 2013)。20世纪80年代末到90年代初,台湾最先开始军曹鱼人工养殖,并突破了军曹鱼的人工育苗等技术(Liao, 2004; Zhou, 2006)。1996年,广东、海南等地开始了军曹鱼海上网箱养殖,随后,叶富良等(2002)成功完成军曹鱼的全人工繁育,军曹鱼逐渐成为广东、海南等地重要的海水养殖对象。2017年,我国军曹鱼养殖总产量为43657 t,比2016年产量增加了17.78%,在全国海水养殖鱼类中,军曹鱼产量位居第7 (农业农村部渔业渔政管理局, 2018)。现有军曹鱼种质资源的保护和遗传改良工作,对促进我国军曹鱼养殖业健康快速发展具有重要意义。
微卫星标记(Microsatellite)是继限制性酶切片段长度多态性(RFLP)之后的第二代遗传标记。由于其在真核生物基因组中分布广泛,遵循孟德尔分离、呈共显性遗传等优点,而被广泛应用在遗传连锁图谱构建、亲缘关系鉴定和种群遗传学等领域(Dewoody, 2000; 孙效文等, 2008)。微卫星标记在鱼类遗传多样性研究中被广泛应用(张小谷等, 2006),其在军曹鱼群体遗传结构方面的研究也有诸多报道。国外学者曾利用微卫星标记对波斯湾和阿曼海(Aliabadi, 2008)、泰国湾(Phinchongsakuldit, 2013)、美国东海岸(Darden, 2014)、阿拉伯海和孟加拉湾(Divya, 2019)等地军曹鱼野生群体的遗传多样性和遗传结构进行了分析。结果表明,在天然水域中军曹鱼群体具有较高的遗传多样性。刘丽等(2008)、王中铎等(2010)分别对中国南海湛江海域野生军曹鱼群体和海南、广东和福建的军曹鱼养殖群体进行遗传多样性分析。结果显示,军曹鱼养殖群体与天然群体遗传结构特征基本一致,均具有较高遗传多样性水平。
为进一步做好军曹鱼的选育工作,本研究选取广东、广西、海南的5个军曹鱼养殖群体为研究对象,利用12个多态性微卫星标记开展群体遗传多样性分析,旨在阐明军曹鱼养殖群体遗传结构现状,为军曹鱼良种选育、种质资源保存和利用等提供科学依据。
1 材料与方法
1.1 材料来源
军曹鱼养殖群体样品包括北海养殖群体(BH) 30尾、陵水养殖群体(LS) 30尾、硇洲养殖群体(NZ) 30尾、徐闻养殖群体(XW) 20尾和三亚养殖群体(SY) 64尾,共计174尾。将各群体的所有个体运回广东海洋大学东海岛养殖基地进行养殖和保种,通过剪取少量军曹鱼尾鳍,保存在95%的乙醇中,于–40℃储存,用于基因组DNA提取。
1.2 实验方法
1.2.1 基因组DNA的提取 实验采用Mamiatis等(1985)的“酚–氯仿”法提取军曹鱼基因组DNA,用1.5%的琼脂糖凝胶电泳检测,并利用微量核酸分析仪测定其浓度,–40℃保存备用。
1.2.2 微卫星引物的筛选 军曹鱼微卫星引物参考Renshaw等(2005),共35对,由生工生物工程(上海)股份有限公司合成。以SY的48尾军曹鱼基因组DNA为模板进行扩增,扩增产物采用8%非变形聚丙烯酰胺凝胶电泳检测和硝酸银染液染色观察,根据条带情况共筛选出12对多态性引物。
1.2.3 多态性微卫星引物扩增和分型检测 在初筛的12对微卫星引物的正向引物5′端进行M13尾巴修饰(5′-AGGGTTTTCCCAGTCACG-3′或5′-GAGCGGATAACAATTTCACAC-3′),并合成相同序列的M13通用引物(5′端用FAM或HEX荧光基团标记)。以2个军曹鱼基因组DNA为模板优化PCR扩增条件,参照Schuelke (2000)三引物法对174个军曹鱼基因组DNA进行PCR扩增。PCR反应体系为20 μl:10 × PCR Buffer 2.5 μl,dNTP (10 mmol/L) 0.5 μl,酶(5 U/μl) 0.5 μl (RR001Q,TaKaRa),M13正向引物(10 umol/L) 0.1 μl,反向引物(10 umol/L) 0.6 μl,M13通用引物(10 μmol/L) 0.6 μl,DNA模板(10~50 ng) 1 μl,无菌去离子水14.2 μl。PCR扩增程序:94℃变性4 min;94℃变性30 s,引物各自退火温度下复性30 s,72℃延伸 30 s,27个循环;72℃延伸8 min。PCR产物经检测后,根据条带情况,将产量高且特异性强的PCR产物送至广州天一辉远基因科技有限公司进行基因分型。
1.3 数据统计及分析
等位基因数(a)、有效等位基因数(e)、Shannon指数()、基因流(m)、近交系数(is)、遗传距离()和遗传相似度()采用PopGene3.2软件完成;运用Cervus 3.0.7软件(Kalinowski, 2007)计算微卫星标记观测杂合度(o)、期望杂合度(e)和多态信息含量(PIC)等遗传学参数;Genepop version 4.0.7软件进行哈迪温伯格(Hardy-Weinberg, HWE)检测;FSTAT软件计算军曹鱼群体的遗传分化指数(st);采用ARLEQUIN3.1软件对各军曹鱼群体分子方差分析(AMOVA)。并基于Nei´s遗传距离和非加权配对算术平均法(Unweighted pair group method with arithmetic mean,UPGMA),使用MEGA6软件构建群体间的系统进化树。
2 结果
2.1 位点多态性和群体遗传多样性
根据聚丙烯酰胺凝胶和硝酸银染色后的条带图,最终筛选出12对微卫星引物用于本研究(表1),群体PCR扩增产物经检测后送往广州天一辉远基因科技有限公司进行基因分型。12个微卫星位点的多态性参数的统计结果见表2。12对微卫星在5个军曹鱼养殖群体中共检测到129个等位基因,其中,Rca1-D08位点的等位基因数最多(a=16),Rca1B-A10和Rca1B-D09位点最少(a=6);观测杂合度(o)为0.218~0.805,平均值为0.569,期望杂合度(e)为0.514~0.844,平均值为0.710;多态信息含量(PIC)为0.451~0.826,平均值为0.671;12个位点在5个军曹鱼养殖群体中的近交系数(is)范围为–0.234~0.475。
表1 12个微卫星位点信息
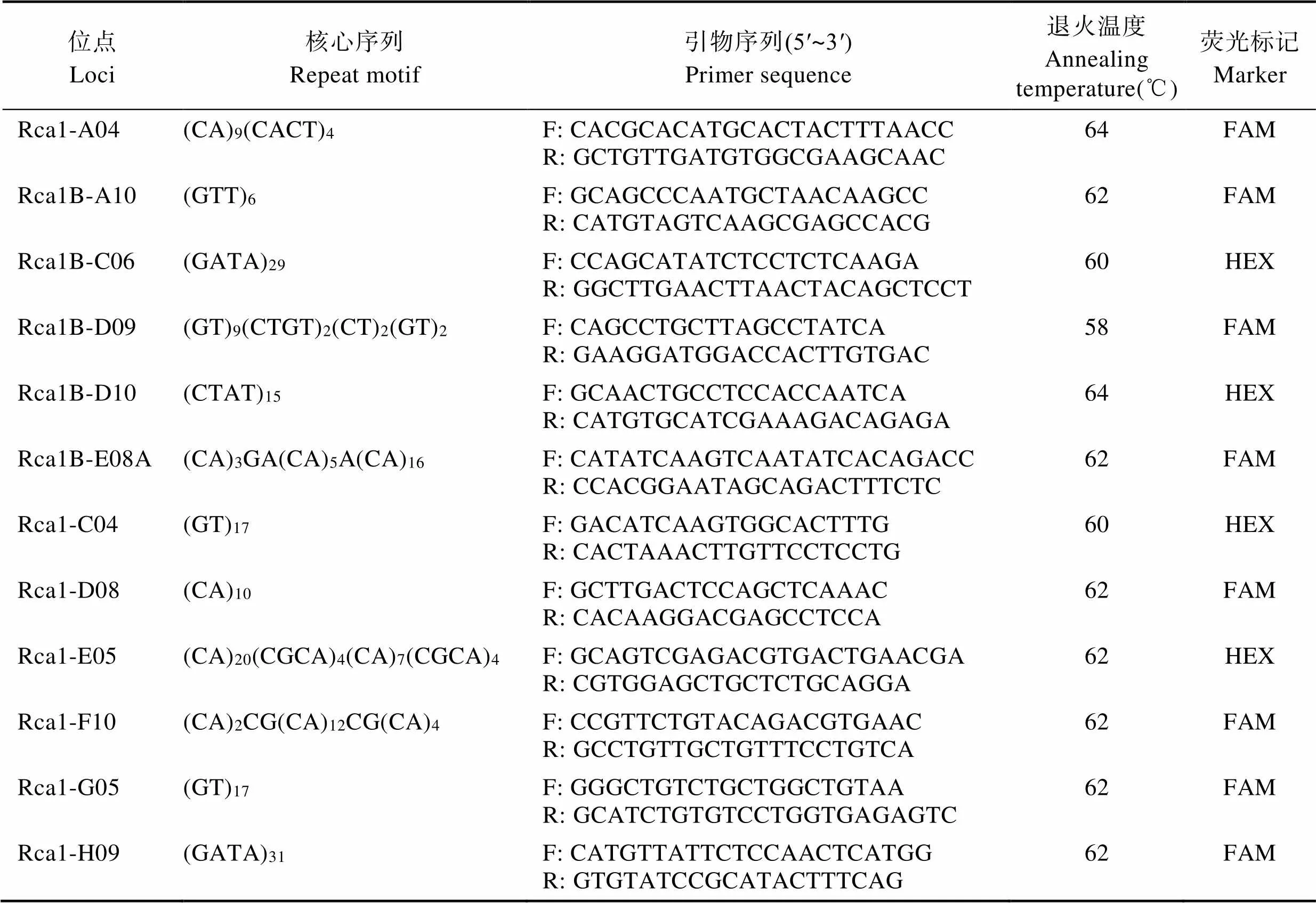
Tab.1 Information of 12 microsatellite loci in this study
表2 12个位点上的遗传多样性参数
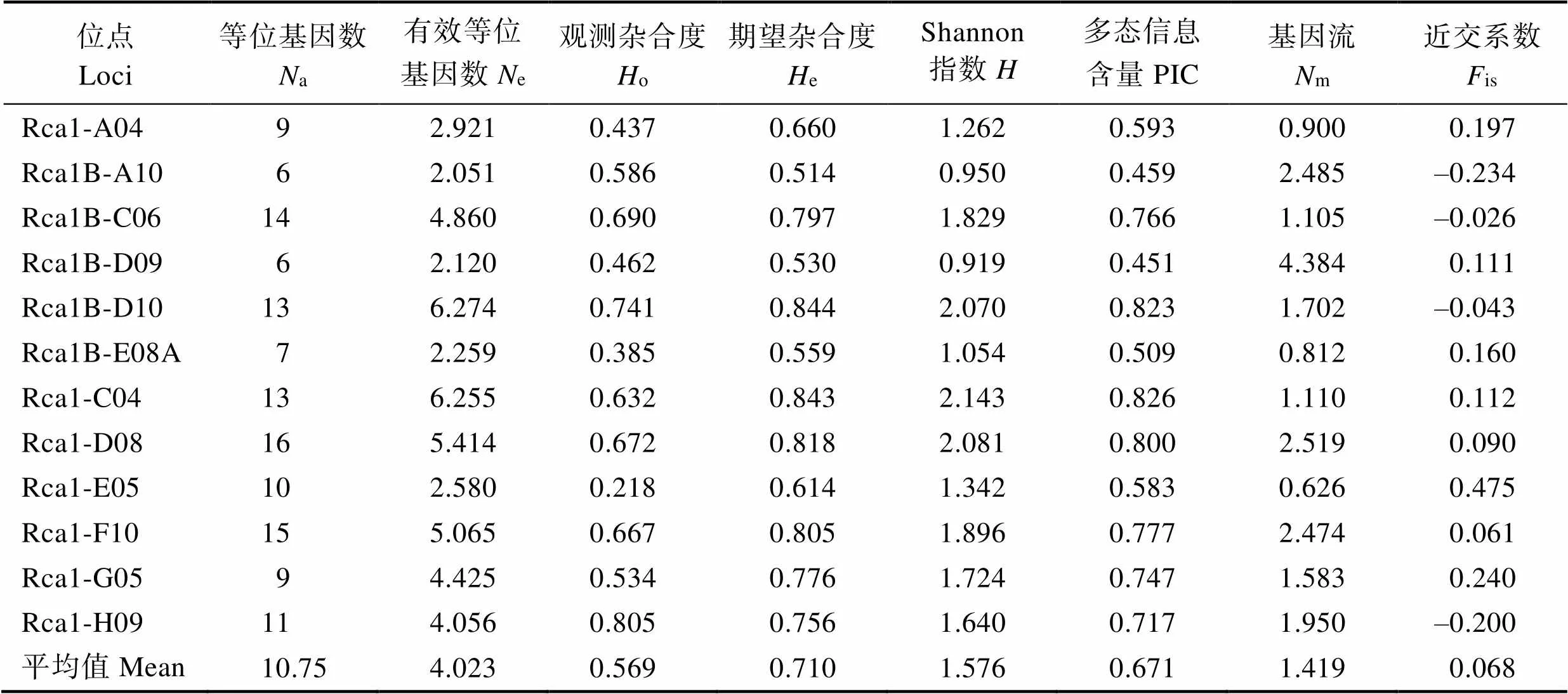
Tab.2 Genetic diversity indices of 12 microsatellite loci in five cultured population
表3是5个军曹鱼养殖群体在12个位点上的遗传多样性参数。其中,5个养殖群体的平均等位基因数依次为6.167、4.167、3.833、6.083和6.750;平均有效等位基因数依次为3.253、2.525、2.284、3.578和3.645;平均期望杂合度依次为0.611、0.541、0.533、0.664和0.681;平均观测杂合度依次为0.519、0.575、0.481、0.558和0.635;平均多态信息含量依次为0.568、0.474、0.463、0.605和0.630。
表3 5个军曹鱼养殖群体在12个位点的遗传多样性参数
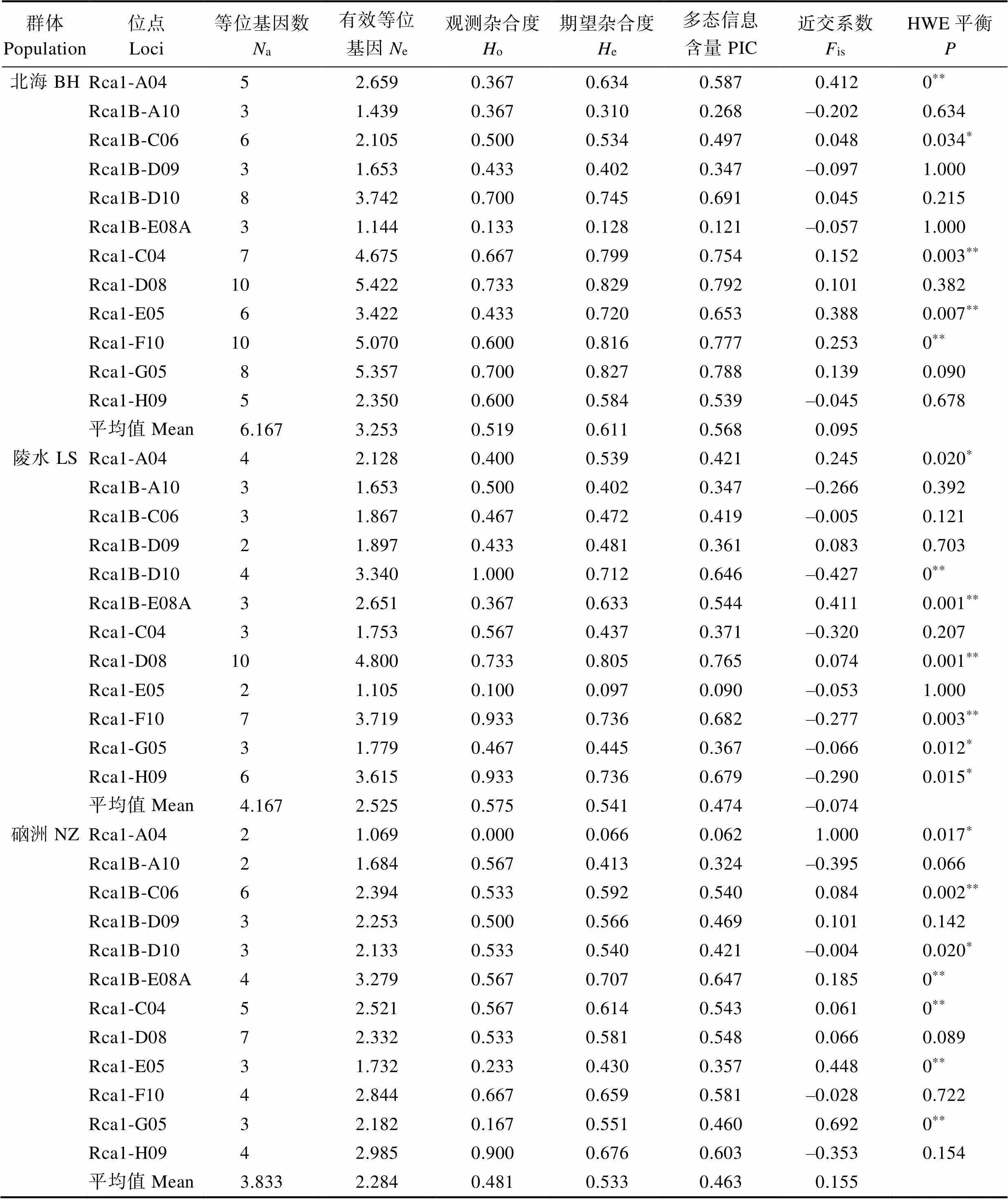
Tab.3 Genetic diversity indices of 12 microsatellite loci in different cultured population
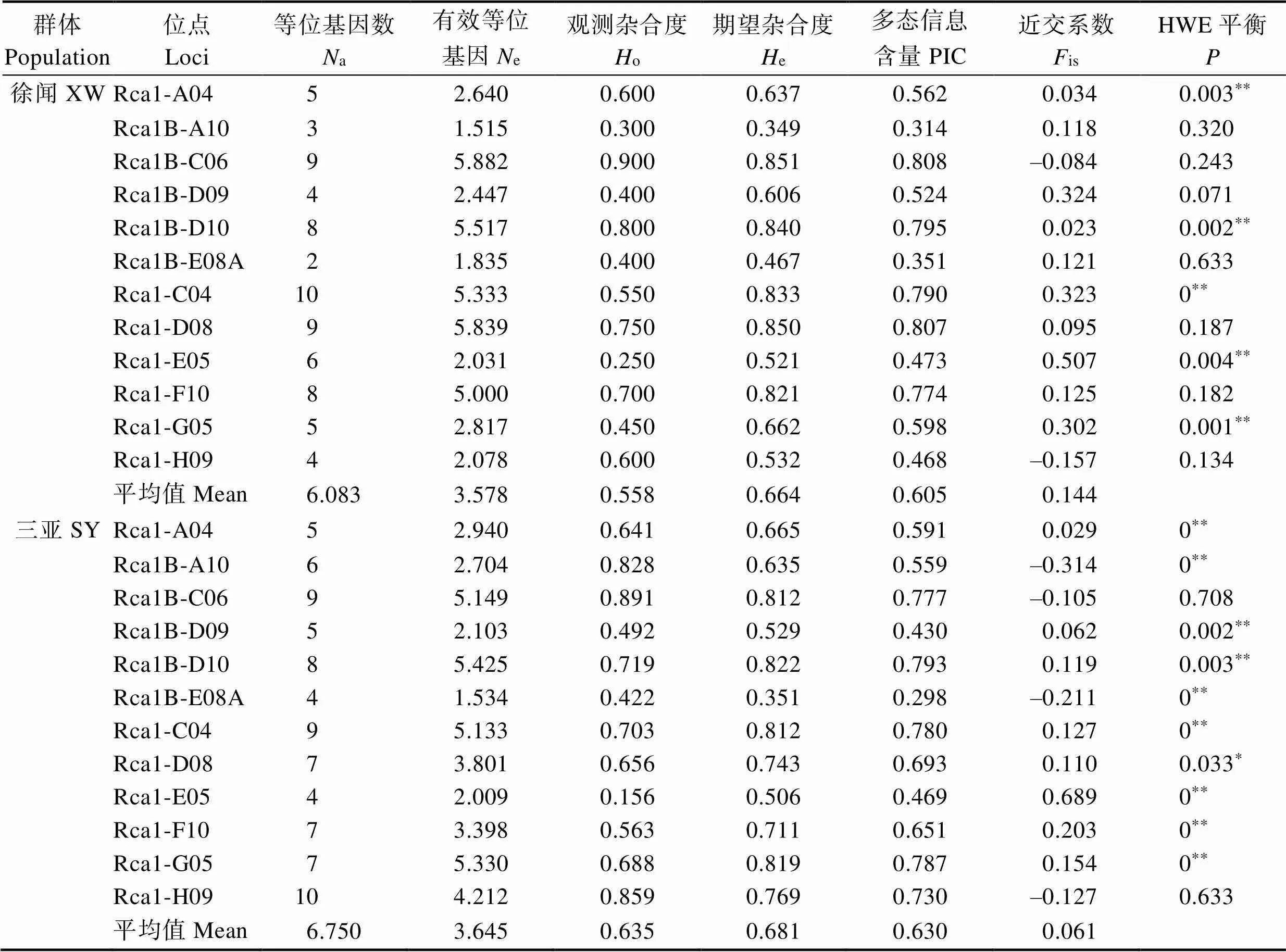
续表
注:*为显著偏离HWE平衡,<0.05;**为极显著偏离HWE平衡,<0.01
Note: * indicated significant deviation from HWE balance,<0.05; ** indicated significantly deviated from HWE balance,<0.01
2.2 军曹鱼养殖群体间的遗传分化
5个军曹鱼养殖群体间的st有显著差异(<0.05) (表4)。st的值在0.055~0.150之间,表明群体间遗传分化程度为中等。其中,BH和NZ的st值最大为0.150,说明这2个群体间存在中等程度偏上的遗传分化。AMOVA分析表明(表5),在5个军曹鱼养殖群体中,84%的变异来自于群体内个体之间,只有16%的变异来自群体之间。
表4 5个军曹鱼群体两两间的遗传分化指数(st)
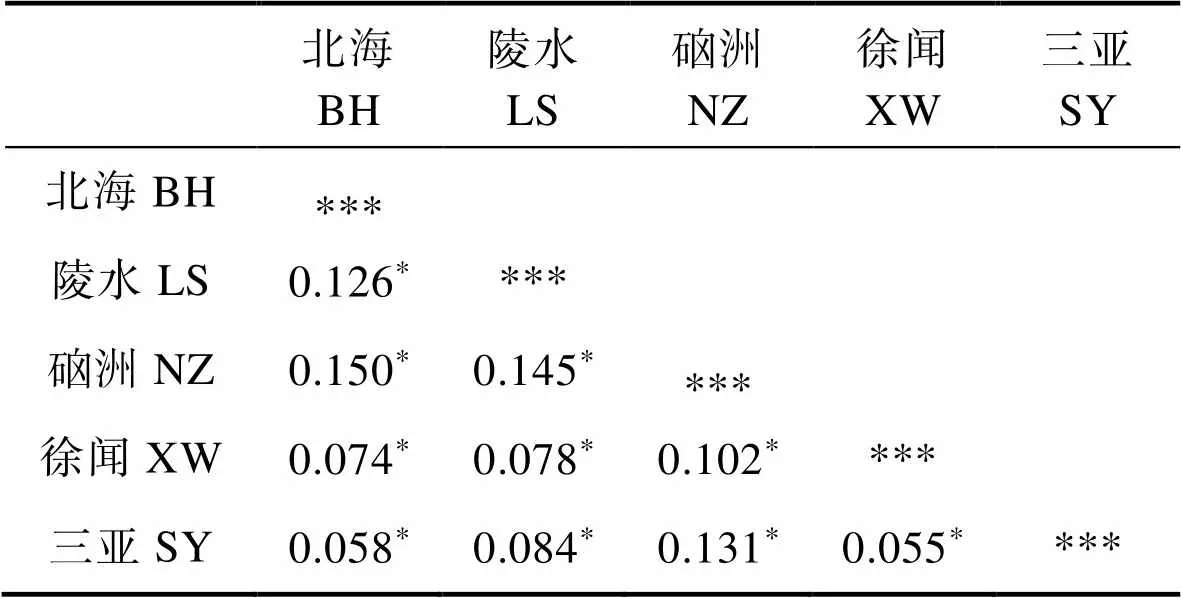
Tab.4 Genetic differentiation index (Fst) between two population of R. canadum
注:*表明差异显著(<0.05)
Note: * indicated significant difference (<0.05)
5个军曹鱼养殖群体间的遗传相似度()和遗传距离()如表6所示,为0.530~0.787,为0.240~0.635。根据Nei´s遗传距离构建的UPGMA系统进化树,如图1所示,BH和SY聚为一支,LS和XW聚为一支,两支聚为一支后又和NZ聚为一支。
3 讨论
遗传多样性研究可为水产动物种质资源的保存及遗传改良等提供理论依据(崔朝霞等, 2011)。a、e、o和e等参数可以反映群体遗传多样性大小,在一定范围内,各参数数值越高,说明群体的基因丰富度越高,其遗传多样性也越高;反之,参数数值越低,则基因丰富度和遗传多样性越低(叶香尘等, 2019;司飞等, 2017)。本研究中,5个军曹鱼养殖群体的平均等位基因数为3.833~6.750,平均有效等位基因数为2.284~3.645,平均观测杂合度为0.481~0.635,平均期望杂合度为0.533~0.681,各参数平均值均小于王中铎等(2010)和刘丽等(2008)对养殖群体和野生群体的研究结果。在选取的12个位点中,o>e的位点,BH和SY各有4个、NZ有3个,XW和LS各有2个,除LS的is为–0.0741外,其他4个养殖群体的is均为正值。结果表明,5个军曹鱼养殖群体的遗传多样性低于王中铎等(2010)和刘丽等(2008)的研究群体,但仍具有较高的遗传多样性,其中,SY遗传多样性最高;5个养殖群体在12个微卫星位点上存在杂合子缺失或过剩、无效等位基因的现象。养殖群体遗传多样性的降低可能与近10年军曹鱼的人工选育有关,经过多代的人工选择、近亲交配等,导致军曹鱼养殖群体的遗传多样性呈现不同程度的降低,这与马冬梅等(2018)和樊佳佳等(2019)对华南鲤()和大口黑鲈()不同世代选育群体的研究结果相似。
表5 5个军曹鱼养殖群体的AMOVA分析

Tab.5 AMOVA analysis in the five cultured population of R. canadum
表6 5个军曹鱼养殖群体的遗传相似度和遗传距离
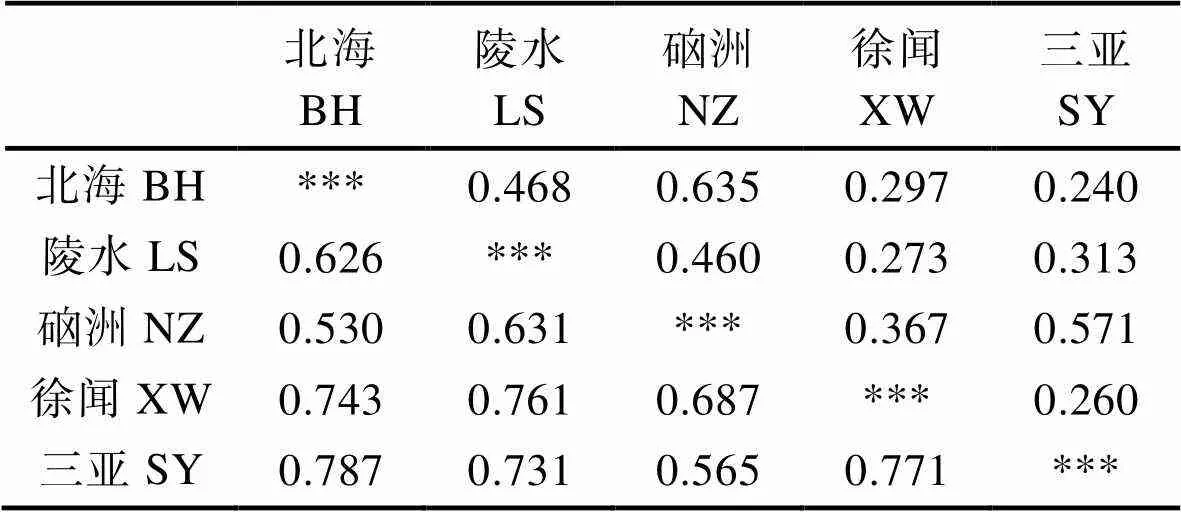
Tab.6 Genetic identity and genetic distance of 5 cultured population of cobia
注:对角线以上为遗传距离,对角线以下为遗传相似度
Note: Genetic distance is above diagonal, genetic identity is below diagonal
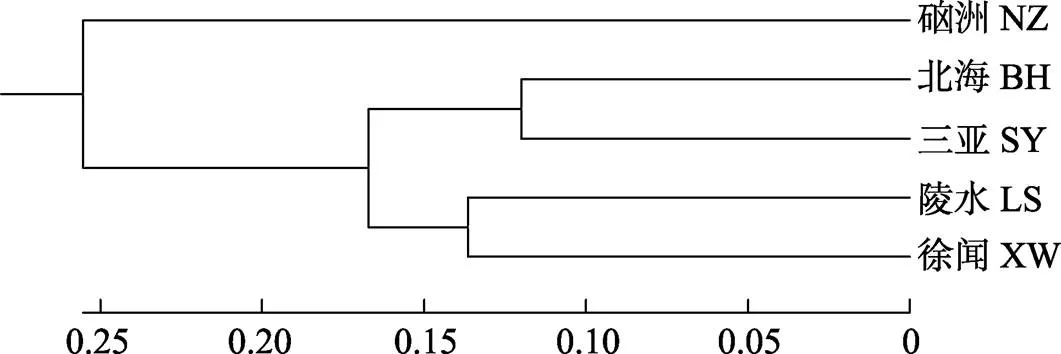
图1 基于Nei´s遗传距离构建的5个军曹鱼群体的UPGMA系统进化树
哈迪温伯格检测结果表明,BH、XW、LS、NZ、SY分别有5、5、7、7和10个微卫星位点偏离了哈迪温伯格平衡。平衡偏离可能是受近亲交配等因素影响,军曹鱼养殖群体的基因频率发生了较大的变化。连续多代的人工选育,导致军曹鱼选育群体中纯合子的增加,杂合子的缺失,使其养殖群体遗传结构的稳定性遭到破坏。
st是反应群体间遗传分化程度的重要参数 (陈锦豪等, 2019)。在本研究中,军曹鱼养殖群体间的st值介于0.055~0.150之间,根据Wright(1978)对st的划分,除了BH和NZ (st=0.150),其他群体间均存在中等程度的遗传分化(0.150≥st≥0.05)。AMOVA分析结果显示,84%的变异来自于群体内个体之间,只有16%的变异来自群体之间。由于人为因素等条件的影响,5个军曹鱼养殖群体多数具有中等程度的遗传分化,与王中铎等(2010)的研究群体相比,群体分化程度变大。
遗传距离()和遗传相似度()可以用来衡量群体间的遗传关系(宋炜等, 2017; 崔蕾等, 2012)。本研究中,SY和BH间的最小(0.240),最大(0.787),亲缘关系最近;NZ和BH间的最大(0.635),最小(0.530),亲缘关系最远。基于Nei´s遗传距离采用UPMGA法对5个军曹鱼群体进行聚类分析,结果显示,BH和SY聚为一支,LS和XW聚为一支,两支聚为一支后又和NZ聚为一支。
结果表明,5个军曹鱼养殖群体多数呈中等程度遗传分化,并存在着不同程度的杂合子缺失等现象,与王中铎等(2010)的研究结果相比,5个养殖群体的遗传多样性有不同程度的降低,但并未产生显著性差异,且群体间分化程度较之前加大,各群体仍具有较高的遗传多样性。尽管如此,在军曹鱼人工选育过程中仍需通过引进外来亲本、收集野生群体等手段扩大亲本群体数量(王军等, 2018),并辅以科学的选育技术,以确保军曹鱼在人工选育多代后,仍保持较高的遗传多样性,这对军曹鱼种业发展及健康养殖具有重要意义。
Aliabadi MAS, Gilkolaei SR, Savari A,Microsatellite polymorphism in Iranian populations of cobia (). Biotechnology, 2008, 7(4): 775–780
Bureau of Fisheries and Fisheries Law Enforcement, Ministry of Agriculture and Rural Affairs. China fishery statistical yearbook. Beijing: China Agriculture Press, 2018, 41–46 [农业农村部渔业渔政管理局. 中国渔业统计年鉴. 北京: 中国农业出版社, 2018, 41–46]
Chen JH, Zheng JB, Wang PP,. Analysis for genetic diversity of broodstock populations of breeding. Journal of Fisheries Research, 2019, 41(1): 1–10 [陈锦豪, 郑锦滨, 王攀攀, 等. 凡纳滨对虾养殖亲本群体遗传多样性分析. 渔业研究, 2019, 41(1): 1–10]
Cui L, Xie CX, Li YH,. Analysis of genetic diversity among four different channel catfish populations by using microsatellite markers. Journal of Huazhong Agricultural University, 2012, 31(6): 744–751 [崔蕾, 谢从新, 李艳和, 等. 斑点叉尾鮰4个群体遗传多样性的微卫星分析. 华中农业大学学报, 2012, 31(6): 744–751]
Cui ZX, Zhang H, Song LS,. Genetic diversity of marine animals in China: A summary and prospectiveness. Biodiversity Science, 2011, 19(6): 815–833 [崔朝霞, 张峘, 宋林生, 等. 中国重要海洋动物遗传多样性的研究进展. 生物多样性, 2011, 19(6): 815–833]
Darden TL, Walker MJ, Brenkert K,. Population genetics of cobia (): Implications for fishery management along the coast of the southeastern United States. Fishery Bulletin, 2014, 112(1): 24–35
Dewoody JA, Avise JC. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. Journal of Fish Biology, 2000, 56(3): 461–473
Divya PR, Linu J, Mohitha C,. Deciphering demographic history and fine-scale population structure of cobia,(Pisces: Rachycentridae) using microsatellite and mitochondrial markers. Marine Biodiversity, 2019, 49(1): 381–389
Fan JJ, Bai JJ, Li SJ,Analysis on genetic diversity of three breeding populations of largemouth bass using formulated feeds. Progress in Fishery Sciences, 2019, 40(4): 57–64 [樊佳佳, 白俊杰, 李胜杰, 等. 驯食配合饲料的大口黑鲈3个选育世代的遗传多样性分析. 渔业科学进展, 2019, 40(4): 57–64]
Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099–1106
Li LD, Chen BS, Feng J,. Analysis and evaluation in nutritive value of(linnaeus). Journal of Tropical Oceanography, 2002, 21(1): 76–82 [李刘冬, 陈毕生, 冯娟, 等. 军曹鱼营养成分的分析及评价. 热带海洋学报, 2002, 21(1): 76–82]
Liao IC, Huang TS, Tsai WS,. Cobia culture in Taiwan: Current status and problems. Aquaculture, 2004, 237(1–4): 155–165
Liu L, Liu CW, Liang N. A genetic analysis of population of cobia,around Zhanjiang waters of South China Sea with microsatellite markers. Journal of Tropical Oceanography, 2008, 27(6): 57–61 [刘丽, 刘楚吾, 梁宁. 利用微卫星DNA标记对南海湛江海域军曹鱼群体遗传多样性的分析. 热带海洋学报, 2008, 27(6): 57–61]
Ma DM, Su HH, Zhu HP. Genetic diversity and genetic structure analysis of different selective breeding generations inusing microsatellite markers. Acta Hydrobiologica Sinica, 2018, 42(5): 887–895 [马冬梅, 苏换换, 朱华平, 等. 华南鲤选育群体不同世代遗传多样性与遗传结构的微卫星分析. 水生生物学报, 2018, 42(5): 887–895]
Mai XJ, Huang WJ, Ye FL,. Reproductive biology and artificial breeding of marine fish. Beijing: China Ocean Press, 2005, 182–190 [麦贤杰, 黄伟健, 叶富良, 等. 海水鱼类繁殖生物学和人工繁育. 北京: 海洋出版社, 2005, 182–190]
Mamiatis T, Fritsch EF, Sambrook J,Molecular cloning: A laboratory manual, New York: Cold Spring Harbor Laboratory. Engineering in Life Sciences, 1985, 5(1): 104
Nazar AKA, Jayakumar R, Tamilmani G,. Sea cage farming of cobia. In: CMFRI manual customized training book. Publisher: Director, CMFRI, Kochi, India, 2013, 177–182
Phinchongsakuldit J, Chaipakdee P, Collins JF,. Population genetics of cobia () in the Gulf of Thailand and Andaman Sea: Fisheries management implications. Aquaculture International, 2013, 21(1): 197–217
Renshaw MA, Pruett CL, Saillant E,. Microsatellite markers for cobia,. Gulf of Mexico Science, 2005, 23(2): 248–252
Schuelke M. An economic method for the fluorescent labeling of PCR fragments. Nature Biotechnology, 2000, 18(2): 233– 234
Si F, Sun ZH, Yu SS,. Analysis of the genetic variability of the captured population of Japanese flounderin the coastal releasing area of Qinhuangdao. Progress in Fishery Sciences, 2017, 38(6): 18–24 [司飞, 孙朝徽, 于姗姗, 等. 秦皇岛近海增殖放流区牙鲆()回捕群体的遗传多样性分析. 渔业科学进展, 2017, 38(6): 18–24]
Song W, Meng YY, Jiang KJ,. Analysis of genetic diversity among seven wildpopulations by using microsatellite marker. Journal of Fisheries of China, 2017, 41(1): 31–39 [宋炜, 孟永永, 蒋科技, 等. 棘头梅童鱼七个野生群体遗传多样性的微卫星分析. 水产学报, 2017, 41(1): 31–39]
Sun XW, Zhang XF, Zhao YY,. Development and application of microsatellite markers in aquatic species. Journal of Fishery Sciences of China, 2008, 15(4): 689–703 [孙效文, 张晓锋, 赵莹莹, 等. 水产生物微卫星标记技术研究进展及其应用. 中国水产科学, 2008, 15(4): 689–703]
Wang J, Wang QY, Kong J,. SSR analysis on genetic diversity in breeding and wild populations ofs. Progress in Fishery Sciences, 2018, 39(2): 104– 111 [王军, 王清印, 孔杰, 等. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 104–111]
Wang ZD, Chen TM, Guo YS,. A genetic analysis of cultured populations of cobia () with microsatellite markers. Journal of Guangdong Ocean University, 2010, 30(3): 16–21 [王中铎, 陈铁妹, 郭昱嵩, 等. 军曹鱼全人工繁殖群体遗传特征的SSR分析. 广东海洋大学学报, 2010, 30(3): 16–21]
Wright S. Evolution and the genetics of populations Volume 4: Variability within and among natural populations. Chicago: University of Chicago Press, 1978, 439–459
Ye FL. Artificial reproduction technology of cobia. Scientific Fish Farming, 2002(12): 12–13 [叶富良. 军曹鱼人工繁殖技术. 科学养鱼, 2002(12): 12–13]
Ye XC, Wei LJ, Liang K,. Genetic diversity analysis in Changfeng silver carp and Guangxi local silver carp. Genomics and Applied Biology, 2019, 38(1): 100–108 [叶香尘, 韦玲静, 梁克, 等. 广西本地鲢和长丰鲢群体遗传多样性分析. 基因组学与应用生物学, 2019, 38(1): 100– 108]
Zhang XG, Tong JG, Xiong BX. Applications of microsatellite markers on studies of genetics and breeding of fishes. Journal of Agricultural Biotechnology, 2006, 14(1): 117– 121 [张小谷, 童金苟, 熊邦喜. 微卫星标记在鱼类遗传及育种研究中的应用. 农业生物技术学报, 2006, 14(1): 117–121]
Zhou QC, Wu ZH, Tan BP,. Optimal dietary methionine requirement for juvenile cobia (). Aquaculture, 2006, 258(1–4): 551–557
Genetic Diversity in Five Cultured Population of Cobia () Using Microsatellite Markers
LI Weiqiang1, CHEN Gang1①, MA Qian1①, HUANG Jiansheng1, SHI Gang1, PAN Chuanhao1, ZHOU Hui1, XIE Ruitao1,2, ZHANG Jiandong1, TANG Baogui1
(1. Fisheries College of Guangdong Ocean University, Zhanjiang 524088; 2. Guangdong Hengxing Group Co., Ltd., Zhanjiang 524000)
Cobia,,the only species in the family Rachycentridae, is a candidate for cage culture in tropical and subtropical waters. Taiwan was the first to cage cobia in the early 1990s, and culturing of cobia has also been developed in Southeast Asia and other areas. Understanding the genetic diversity of cultured populations is important for the sustainable development and management of aquaculture.In the present study, 12 polymorphic microsatellite loci were selected to investigate and assess genetic diversity in five cultured populations of cobia from Beihai (BH), Lingshui (LS), Naozhou (NZ), Xuwen (XW), and Sanya (SY). As a result, 129 alleles were detected in the five populations. The mean number of alleles was between 3.833 and 6.750, the mean number of effective alleles ranged from 2.284 to 3.645, the mean of observed heterozygosity and expected heterozygosity was between 0.481 and 0.635, and 0.533 and 0.681, respectively, and the mean polymorphism information content ranged from 0.463 to 0.630. The population deviated significantly from a Hardy-Weinberg equilibrium at multiple microsatellite loci (<0.05). Analysis of genetic differentiation indicated that thestrange was from 0.055 to 0.150 and the genetic distance () range was from 0.240 to 0.635. BH and NZ had the highestst(0.150) and the highest(0.635). The results of an analysis of molecular variance showed that 84% of the genetic variations were within cultured populations, and 16% were among cultured populations. A phylogenetic analysis using the unweighted pair group method with arithmetic mean and based on Nei’s genetic distance showed that one cluster comprising BH and SY, and the other cluster comprising LS and XW formed a branch, which was then clustered with NZ. These results provide data for further protection and improvement of the germplasm resources of cobia.
; Culture population; Microsatellite; Genetic diversity
S917.4
A
2095-9869(2020)02-0113-08
陈 刚,教授,E-mail: cheng@gdou.edu.cn;马 骞,副教授,E-mail: mfm_0624@163.com
2019-06-17,
2019-08-20
* 国家海水鱼产业技术体系–军曹鱼种质资源与品种改良岗位(CARA-47-G08)和南方海洋科学与工程广东省实验室(湛江)资助项目(ZJW-2019-06)共同资助[This work was supported by National Technical System of Marine Fish Industry– Germplasm Resources and Variety Improvement Positions of Cobia,(CARA-47-G08), and Southern Marine Science and Engineering Guangdong Laboratory, Zhanjiang(ZJW-2019-06)]. 李伟强,E-mail: lwqclj@126.com
10.19663/j.issn2095-9869.20190617001
http://www.yykxjz.cn/
李伟强, 陈刚, 马骞, 黄建盛, 施钢, 潘传豪, 周晖, 谢瑞涛, 张健东, 汤保贵. 利用微卫星标记分析军曹鱼养殖群体的遗传多样性. 渔业科学进展, 2020, 41(2): 113–120
Li WQ, Chen G, Ma Q, Huang JS, Shi G, Pan CH, Zhou H, Xie RT, Zhang JD, Tang BG. Genetic diversity in five cultured population of cobia () using microsatellite markers. Progress in Fishery Sciences, 2020, 41(2): 113–120
CHEN Gang, E-mail: cheng@gdou.edu.cn; MA Qian, E-mail: mfm_0624@163.com
(编辑 马璀艳)

