磷胁迫下羊草的响应及磷响应相关基因表达分析
万东莉,侯向阳,丁勇,任卫波,王凯,李西良,万永青
磷胁迫下羊草的响应及磷响应相关基因表达分析
万东莉1,侯向阳1,丁勇1,任卫波1,王凯1,李西良1,万永青2
(1中国农业科学院草原研究所/农业部草地生态与修复治理重点实验室,呼和浩特 010010;2内蒙古农业大学生命科学学院/内蒙古自治区植物逆境生理与分子生物学重点实验室,呼和浩特 010011)
【】磷是植物生长发育所必需的大量营养元素,研究无机磷酸盐(inorganic phosphate,Pi)胁迫下羊草的响应,筛选不同浓度Pi胁迫下的内参基因,并分析Pi响应相关基因的表达,为羊草Pi胁迫响应的分子机制研究提供基础数据。以羊草幼苗为材料,进行不同浓度Pi处理培养,对羊草的株高和根长进行检测,利用钒钼黄比色法检测羊草植株中Pi的含量。根据羊草转录组数据选取7个候选内参基因,从NCBI核酸序列数据库选取1个候选内参基因,对羊草不同处理材料进行总RNA提取和cDNA合成,利用qRT-PCR对候选内参基因的表达进行检测,并通过geNorm、NormFinder和Bestkeeper软件对其稳定性进行评估。根据筛选获得的较稳定内参基因,对Pi响应基因的qRT-PCR检测数据进行相对定量分析。表型观测结果显示,无论是低Pi还是高Pi胁迫都使得羊草的生长减缓;株高对低Pi或缺Pi胁迫较为敏感,根长对高Pi胁迫较为敏感;并随着处理浓度的增加羊草中Pi的含量也增加。qRT-PCR溶解曲线分析显示8个候选内参基因均具有单一的溶解峰,基因表达谱分析表明8个候选内参基因的CT值范围是17.16—26.61,其中,的表达丰度最高为17.16—20.22,的表达丰度最低为23.28—26.61,CT值变异系数最小的为(2.09%),最大的为(6.8%)。综合geNorm、NormFinder和Bestkeeper稳定性排名,通过计算几何平均数,获得8个候选内参基因综合稳定性排名,其中排名靠前的3个基因分别为、和,排名最后的2个基因为和。分别以、和作为内参基因,qRT-PCR相对定量表达分析结果显示,与对照相比,的表达受低Pi或者缺Pi诱导;的表达受高Pi诱导;而的表达同时受低Pi和高Pi下调。低Pi和高Pi胁迫均使羊草生长受阻,羊草地上组织与地下组织对Pi胁迫响应的模式不同。筛选出3个表达较为稳定的内参基因、和。和分别参与了羊草对低Pi和高Pi的应答响应,同时参与了羊草对低Pi和高Pi的响应进程。
羊草;磷胁迫;qRT-PCR;内参基因;磷响应基因
0 引言
【研究意义】羊草()隶属禾本科(Gramineae)赖草属,是欧亚大陆草原区东部草甸草原及干旱草原上的重要建群种之一。在中国内蒙古、东北三省、河北、山西和新疆等地广泛分布[1]。近半个世纪以来,由于过度放牧等原因,中国北方草原大面积退化,羊草等植物出现小型化,草原群落生产力锐减和优势种更替[2]。磷(phosphorus,P)是草原生产力的限制因子之一[3-4]。虽然通过施用磷肥可以提高退化草地土壤肥力、促进羊草等草原植物的生产力[4-5],但是磷是不可再生资源,并且过多施用磷肥会造成环境问题[6]。因此,通过分子改良获得羊草磷高效利用植物,是促进草原生产力恢复和提高的一个重要途径。【前人研究进展】磷是植物生长发育所必需的营养元素,不但是核酸、磷脂和ATP等生命大分子的重要组成成分,也是植物体内许多新陈代谢进程的关键调节子[7]。磷储量低和植物磷利用效率低是一个全球性的问题[6,8-9]。内蒙古天然草原土壤中全磷和有效磷含量的平均值分别为0.33 mg·g-1和3.53 mg·kg-1,其中,有效磷含量与全国土壤水平接近,但低于全球土壤水平[10]。中国草地植物磷的含量平均值为1.9 mg·g-1[11],内蒙古天然草地植物叶片磷的含量平均值为1.51 mg·g-1[10],低于英国草地植物的磷含量2.7 mg·g-1[12]。总体上,内蒙古草地和草地植物普遍处于缺磷状态[3]。过度放牧干扰下,典型草原严重退化样地(冷蒿+糙隐子草群落),与围封恢复样地(羊草+大针茅群落)相比,土壤全磷含量减少[13]。土壤中添加丛枝菌根真菌(arbuscular mycorrhizal fungi,AMF),能促进羊草根围土壤有效磷含量、对磷的吸收以及地上生物量的提高,有利于退化羊草草地的修复[14-15]。磷限制条件下植物会从形态、生理、生化和分子水平激发一系列的适应响应,例如减少植物生长,改变根系统体系结构,分泌有机酸、磷酸酶和和核酸酶以增加磷的获取[16]。基因表达模式的研究是现代分子生物学的基石之一。通过基因表达分析有助于深入了解复杂的生物过程,增加对环境响应信号和代谢途径的理解[17]。实时荧光定量PCR(quantitative real-time reverse transcription PCR,qRT-PCR)作为一个高效的基因表达检测工具,在生物学研究的各个领域广泛应用[18]。但是基因表达数据需要利用内参基因进行标准化处理[18]。内参基因的稳定表达是精确的标准化靶基因表达的关键,筛选不同条件下的合适内参基因是精确分析qRT-PCR结果的必要步骤[19]。在羊草中通过对不同组织中内参基因的稳定性分析,分别获得了根、茎、叶和穗中相对稳定的内参基因,可用作羊草不同组织qRT-PCR检测的内参基因[20]。【本研究切入点】目前,关于Pi处理条件下,羊草的内参基因筛选、以及Pi响应相关基因的表达研究尚缺乏。另外,在模式植物拟南芥、水稻和玉米等作物中对低磷响应的生理、分子适应机制研究较多[8-9,16,21-22]。在羊草中已有关于低磷响应的生理生化水平研究[23],但分子水平上的研究鲜见报道。【拟解决的关键问题】本研究通过对羊草进行不同浓度的Pi处理,从表型、生理水平上对羊草的响应情况进行分析。同时,对不同浓度Pi水平下羊草内参基因进行qRT-PCR检测,基于合适的内参基因筛选,对磷响应相关基因的表达进行标准化分析,为羊草Pi胁迫响应的分子机制研究提供理论基础和试验支持。
1 材料与方法
1.1 植物培养和处理
表型鉴定和分子试验分别于2014—2015年和2018年在内蒙古自治区植物逆境生理与分子生物学重点实验室进行,所用材料为羊草吉生4号种子。将羊草种子播种于装有营养土和蛭石(V﹕V=1﹕2)的培养钵中,并置于植物温室培养,温度为23℃—25℃,光照为16 h光照/8 h黑暗。待种子发芽后,将幼苗移入Hoagland培养中培养2 d,然后将幼苗移到分别含有0、0.01、0.33、5和30 mmol·L-1KH2PO4的Hoagland培养液中进行Pi胁迫处理培养,以含1 mmol·L-1KH2PO4作为对照,每隔4 d更换一次培养液并测量根长和株高(除25—31 d间隔6 d外),每种处理至少测量20株羊草。生长35 d后,分别取根和地上组织样品用于Pi含量检测。
基因表达检测:羊草培养和处理方法同前面所述,共设5个Pi浓度梯度(0、0.01、1、5和30 mmol·L-1),生长35 d左右,每个处理取3株羊草混合作为RNA提取检测的样品,并迅速放于液氮速冻后于-80℃冰箱保存。3次独立生物学重复。
1.2 植物Pi含量检测
将经过不同浓度Pi处理的羊草不同组织烘干并研成粉末,利用钒钼黄比色法对Pi的含量进行检测。
1.3 总RNA提取和cDNA合成
取0.05—0.1 g羊草样品,利用植物RNA提取试剂盒MiniBEST Plant RNA Extraction Kit(TaKaRa)提取总RNA,具体方法按照说明书进行操作。通过吸光率A260/280和琼脂糖凝胶电泳分别对RNA的纯度和完整性进行检测。取2 µg总RNA,利用反转录试剂盒PrimeScriptTMRT reagent Kit with gDNA Eraser(TaKaRa)进行cDNA合成,具体步骤按产品说明书进行。将反转录得到的cDNA原液稀释10倍作为基因表达检测的模板。
1.4 内参基因选择和引物设计评价
试验共用到8个qRT-PCR候选内参基因,其中7个基因序列(包括、、、、、和)来源于羊草转录组数据库,序列来自于NCBI(Genbank登录号HM623326.1)。采用生物软件Primer Premier 5设计qRT-PCR检测特异引物(表1),引物序列参考文献[20,24]。引物设计参数为:引物退火温度59℃—61℃,引物序列长度18—25 bp,扩增产物长度80—200 bp,GC含量40%—60%。
引物扩增效率:取不同Pi处理下的cDNA样品等量混合,按照10倍的梯度稀释5个浓度梯度,并以此为模板,按照1.5方法进行qRT-PCR反应后计算扩增效率。
1.5 实时荧光定量PCR
利用TaKaRa的SYBR Premix Ex Taq II对Pi处理下内参基因进行qRT-PCR检测。qRT-PCR反应总体系为20 µL,包括cDNA模板5 µL、SYBR Premix Ex Taq II 10 µL、正和反向引物各0.8 µL(10 µmol·L-1)和DEPC水3.4 µL。qRT-PCR反应程序为95℃ 30 s;95℃ 5 s,60℃ 30 s,72℃ 15 s,40个循环。溶解曲线:以0.5℃逐渐增加,从70℃加热至95℃,然后95℃恒温15 s。每个样品3个技术重复,每个试验进行3次独立生物学重复。
1.6 内参基因稳定性分析
利用软件geNorm(version 3.5)[25]、NormFinder(version 0.953)[26]和BestKeeper(version 1)[27]对内参基因的稳定性进行分析评估。具体方法参考文献[28]。geNorm基于对数转换表达率的标准差,确定每个内参基因与其他所有内参基因配对变异的平均值,作为内参基因的表达稳定性M值(average expression stability values),通过逐步排除M值最高的基因,对被测基因的表达稳定性进行排名,M值越低稳定性越高,M值小于1.5则默认为可用作内参基因[25]。NormFinder基于方差分析计算每个基因的表达稳定值(stability value),对候选内参基因的稳定性进行排名,稳定值越低则稳定性越高[26]。BestKeeper通过比较CT(cycle threshold)值的标准偏差(standard deviation,SD)和变异系数(coefficient of variance,CV)来评估内参基因的稳定性,SD值越高基因表达越不稳定[27]。geNorm和NormFinder将qRT-PCR检测获得的CT值换算为2-ΔCT值作为分析的输入数据,其中,ΔCT=相应样品CT值-最小CT值。BestKeeper直接将CT值作为输入数据进行分析。

表1 基因和qRT-PCR引物特性
1.7 磷响应相关基因表达分析
、和来源于羊草转录组数据库,利用qRT-PCR对不同Pi处理下相关基因的表达进行检测。选用获得的较稳定和不稳定的内参基因,利用2-ΔΔCT法[29]对磷响应相关基因的相对表达量进行分析。每个样品3个技术重复。qRT-PCR引物序列列于表1。
2 结果
2.1 羊草对不同浓度Pi处理的响应
通过对羊草进行不同浓度Pi处理,结果显示,对照(1 mmol·L-1Pi)条件下,羊草的株高和根长均随着处理时间的增加而增长;与对照相比,低Pi(0和0.01 mmol·L-1)和高Pi(5和30 mmol·L-1)处理都使得羊草的生长受阻(图1)。在低Pi或者缺Pi条件下,株高在处理17 d时显著低于对照,根长在处理的25 d显著低于对照,并且均随着处理时间的延长差异显著增强,与根长相比,株高对低Pi或缺Pi较为敏感。在高Pi处理条件下,株高分别在5和30 mmol·L-1Pi处理的25 d和13 d显著低于对照,并且随着处理时间的延长差异显著增强;根长分别从5和30 mmol·L-1Pi处理的13 d和9 d开始显著低于对照,并且一直持续至处理结束(除5 mmol·L-1Pi处理21 d外),与株高相比,根长对高Pi更为敏感。在地上组织中,与高Pi处理相比,株高对低Pi较为敏感;而在地下组织中,与低Pi相比,根长对高Pi(30 mmol·L-1)较为敏感。表明羊草地上、地下组织对Pi处理响应的模式不同。0.33 mmol·L-1Pi处理条件下,与对照相比,株高和根长都没有显著性差异,表明0.33—1 mmol·L-1Pi浓度范围是羊草正常生长的合适浓度。
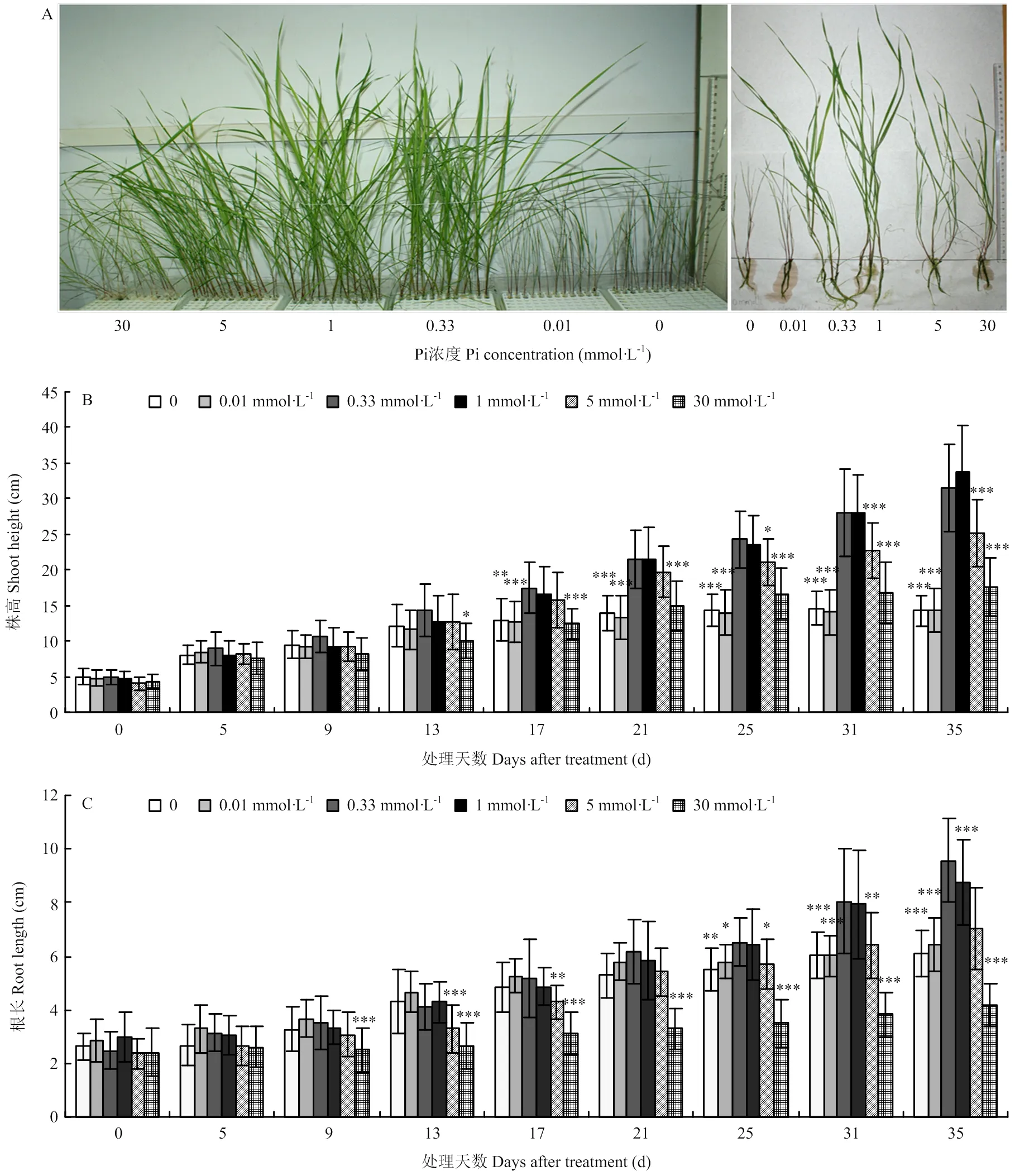
A:表型;B:株高检测;C:根长检测。*、**和***分别代表在P<0.05、P<0.01和P<0.001水平差异显著。下同
不同浓度Pi处理条件下,羊草根和叶中Pi含量随着处理浓度的增加均呈上升趋势(图2)。与对照相比,低Pi和高Pi处理下,除30 mmol·L-1Pi处理的根外,根和叶片中Pi的积累达到差异显著水平。与对照相比,0.33 mmol·L-1Pi处理差异不显著。表明不同浓度Pi处理下羊草的表型与Pi的含量相关。
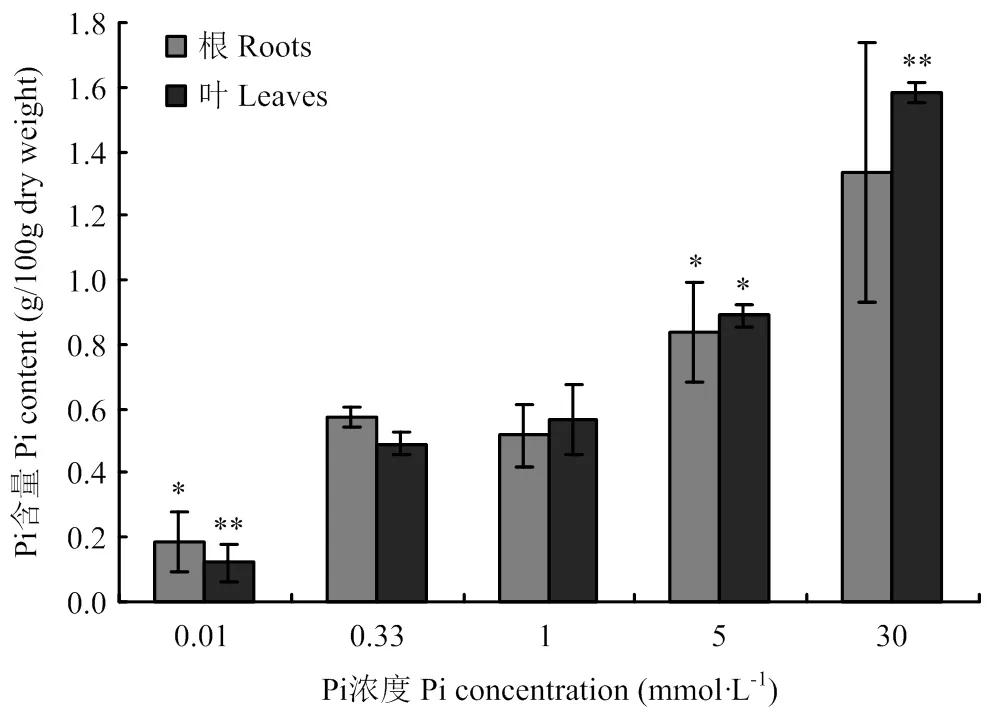
图2 不同浓度Pi处理下羊草组织中Pi的含量
2.2 候选内参基因的引物特异性和扩增效率分析
根据前期转录组数据,筛选出7个表达稳定的基因(表达水平变化小于2倍),同时从NCBI获得1个基因作为候选内参基因。利用Primer Premier 5软件设计基因特异引物,以不同浓度Pi胁迫下羊草组织cDNA为模板进行qRT-PCR,对8个候选内参基因的引物扩增效率进行分析,其范围在95.0%—107.2%(表1)。qRT-PCR溶解曲线分析显示,8个候选内参基因均具有单一的溶解峰(图3),表明候选基因引物特异性较好。
2.3 Pi胁迫条件下候选内参基因的表达谱分析
根据qRT-PCR结果中的CT值,对候选内参基因的表达谱进行分析。结果(图4)表明,在所有样品中,8个候选内参基因的CT值范围是17.16—26.61,其中,大多数基因的CT值范围在18.84—23.95。在所有基因中,的表达丰度最高,为17.16—20.22,其次为,其CT值范围为18.84—21.78;的表达丰度最低,为23.28—26.61,其次为和,CT值范围分别为22.26—23.95和20.35—26.30。通过对候选内参基因在所有样品中的CT值进行平均数(mean)、标准偏差SD和变异系数CV分析(表2),结果显示,和变异系数最大,分别为6.8%和6.6%,稳定性较差;的变异系数最小,为2.09%,稳定性较好;其他基因的变异系数居中。
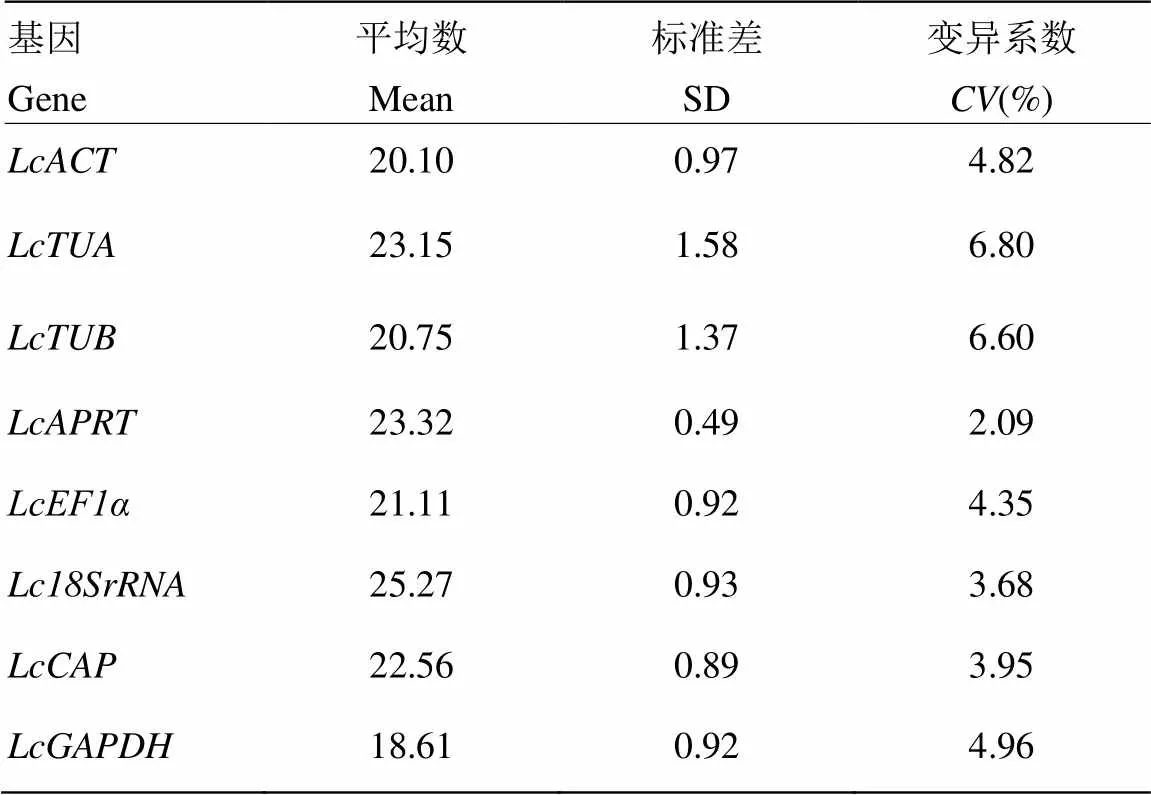
表2 候选内参基因的CT值
2.4 Pi胁迫条件下候选内参基因的稳定性分析
利用geNorm、NormFinder和Bestkeeper软件,对候选内参基因的表达稳定性进行分析。geNorm分析结果显示(表3),在不同浓度Pi处理条件下,所有基因的M值均小于1.5,和的M值最低(0.42),稳定性最高,然后依次为(0.49)、(0.52)、(0.54)和(0.60),(0.68)和(0.78)的M值最高,稳定性最差。geNorm通过分析配对变异系数(pairwise variation,V)评估标准化过程中合适的内参基因数目,Vn/n+1的值小于0.15作为阈值,判断是否需要再增加一个内参基因。V值分析结果显示,V2/3的值(0.16)大于0.15(图5),表明2个内参基因不足以进行标准化过程分析,需要引进第三个内参基因,V3/4及其他V值均小于0.15,即3个内参基因可满足对靶基因精准的标准化分析。

图3 候选内参基因的溶解曲线
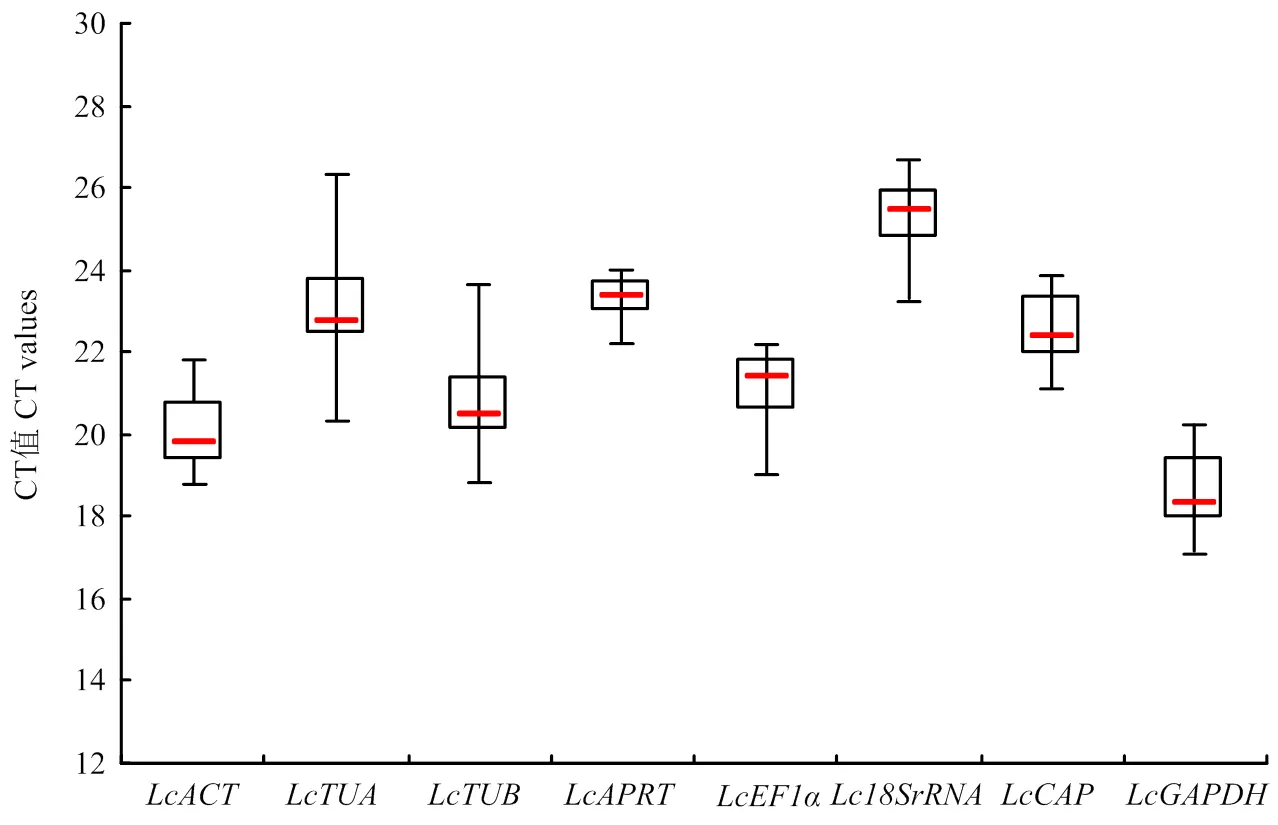
方框代表CT值的第一和第三四分位值;红线代表中位数;短横线代表最大值和最小值

图5 候选内参基因配对变异系数分析
NormFinder分析结果显示(表3),排名最靠前的基因为,紧随其后的3个基因为、和,最不稳定的2个基因为和。
根据BestKeeper分析结果(表3),稳定性较好的3个基因为、和,其次为和,和的SD值最高最不稳定。
综上所述,通过计算几何平均数对8个候选内参基因排名进行综合评价(表4),其中,排名靠前的3个基因为、和,并且在3个软件稳定性分析结果中排名均在前四名,表明稳定较好,最适合作为内参基因。排名最后的2个基因为和,这与稳定性分析结果基本相符,表明最不适合作为内参基因。
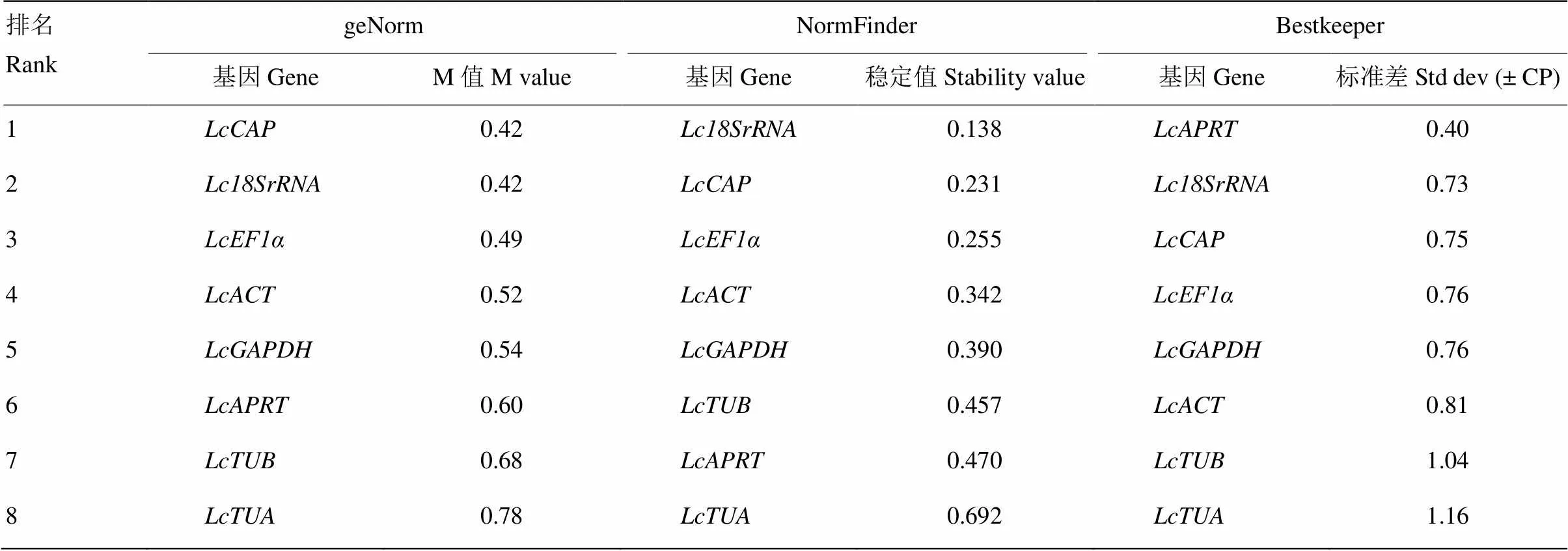
表3 候选内参基因的稳定性分析
2.5 Pi处理条件下磷响应相关基因表达分析
选用稳定性较好的、、以及++分别作为内参基因,对不同Pi处理下和进行qRT-PCR分析(图6)。结果显示,利用不同内参基因分析得到的表达模式类似。与对照相比,的表达受低Pi或者缺Pi诱导;的表达受高Pi诱导;而的表达同时受低Pi和高Pi下调。而以稳定性较差的作为内参基因时,、和的表达模式与以稳定性较好的内参基因不同。表明筛选的内参基因比较可靠。

图6 不同浓度Pi处理下磷响应基因表达分析

表4 候选内参基因稳定性整体排名
3 讨论
3.1 不同浓度Pi处理对羊草生长的影响
磷是植物生长发育所必需的大量无机营养元素之一,Pi缺乏条件下植物会从形态、生理和分子水平产生一系列反应以保持磷稳态[7,16]。Li等[23]对缺Pi条件下羊草的生理特性分析结果表明,Pi缺乏致使叶片的生长、生物量积累和净光合速率显著降低。本研究中通过进行不同浓度的Pi胁迫处理,对羊草的响应和候选内参基因进行了分析。研究结果表明,低Pi(或缺Pi)条件下,羊草的茎和根生长均受到明显的抑制,这与Li等[23]研究结果一致。同时,高浓度Pi条件下,羊草的生长也受到了抑制,表明外源施加较高浓度的Pi使得羊草体内Pi过量积累,产生P毒性症状(P-toxicity symptoms)。P毒性症状在不同植物物种间的差异不是很大,表现为生长减缓、叶片失绿坏死、细胞器损伤和细胞活力丧失等特征,但是在不同物种间出现P毒性症状的Pi浓度不同[30-33],本研究中Pi浓度达到5 mmol·L-1时羊草的生长减缓,表明产生了P毒性症状,当Pi浓度达到30 mmol·L-1时少数羊草植株出现了死亡的现象,表明P毒性症状加重。但是羊草中低Pi响应及P毒性症状的分子机制如何?是否与其他物种类似?值得进一步研究验证。
3.2 不同浓度Pi处理条件下羊草内参基因稳定性分析
内参基因筛选是qRT-PCR结果准确分析的关键步骤。本研究利用软件geNorm、NormFinder和Bestkeeper,对8个候选内参基因在不同浓度Pi处理下的表达稳定性进行了分析。geNorm分析结果显示,8个候选内参基因的M值均小于默认阈值1.5[25],表明均可以作为内参基因;配对变异系数V2/3的值大于0.15,V3/4的值小于0.15,表明理想的内参基因数目为3个。由于每个软件评估的策略不同,因此分析的结果稍有不同。通过计算不同软件排名的几何平均数,对内参基因进行整体排名,排名靠前的3个基因为、和,其在geNorm和NormFinder中的排名均为前三名之内,在Bestkeeper排名为前四名之内。在所有软件排名中均为最后一名,表明其稳定性最差,不适合作为内参基因。
作为传统的内参基因,和在不同的植物中被广泛应用,但是在不同条件下的表达不同[34]。在桂花(Lour.)不同品种、发育阶段和低温胁迫材料中,不适合作为内参基因[34];在草莓(×)的不同组织和发育阶段,的表达稳定性较低[35];而在水稻中,不同环境胁迫下,的表达稳定性最高[36]。在高羊茅(Schreb.)中,不同的逆境胁迫条件下是最不稳定的内参基因[37];而在板蓝根()中,是在所有试验样品中最稳定的内参基因之一[38]。胡宁宁等[20]对羊草不同组织部位内参基因筛选结果显示,和分别是根和茎中较为稳定的内参基因。作为新的内参基因,编码的是环化酶相关蛋白,在不同条件下其表达稳定性不同,例如在柠条锦鸡儿()中,在干旱、冷和高pH胁迫下稳定性较好,而在NaCl胁迫下稳定性较差[19];在大针茅()中,JA处理下的表达稳定性较差[28]。本研究通过综合分析显示,、和稳定性最好,但是geNorm、NormFinder和Bestkeeper软件在分析过程均没有考虑到基因的表达丰度,根据2.3中分析结果,的CT值较高、表达丰度较低,因此,综合考虑可优先选用和作为内参基因。
3.3 不同浓度Pi处理下磷响应基因表达分析
Wu等[7]通过拟南芥基因芯片分析显示,在Pi饥饿72 h以内,6 172个基因中的1 800多个基因有响应;通过RNA印记杂交进一步证实,一个在叶片中的表达受Pi饥饿诱导。Mission等[21]利用Affymetrix基因芯片技术,对低Pi胁迫下拟南芥的22 810个基因表达模式进行了分析,共发现612和254个基因的表达分别受低Pi诱导和抑制,其中包括11个受低Pi诱导表达的。与拟南芥中部分的表达模式不同,本研究检测的2个中,的表达受高Pi诱导,的表达同时受低Pi和高Pi下调,表明这两个基因参与了羊草对Pi胁迫响应的进程,并且主要与高Pi胁迫应答响应有关。编码的是SPX-EXS蛋白,主要负责将Pi从根运输到地上组织,其表达受低Pi诱导[6,39]。本研究的表达特异的受低Pi诱导,表明其参与了羊草对低Pi的应答响应,但是是否参与Pi从根到地上组织的运输还不清楚,有待于进一步试验验证。
4 结论
低Pi和高Pi胁迫均使羊草生长受阻,但地上组织与地下组织对Pi胁迫响应的模式不同,随着处理浓度的增加羊草中Pi的含量增加。筛选出3个表达较为稳定的基因、和,可作为Pi处理下羊草基因表达分析的内参基因。和分别参与了羊草对低Pi和高Pi的应答响应,同时参与了羊草对低Pi和高Pi的响应进程。
[1] 刘公社, 李晓霞, 齐冬梅, 陈双燕, 程丽琴. 羊草种质资源的评价与利用. 科学通报, 2016, 61(2): 271-281.
LIU G S, LI X X, QI D M, CHEN S Y, CHENG L Q. Evaluation and utilization of Leymus chinensis germplasm resources., 2016, 61(2): 271-281. (in Chinese)
[2] 王炜, 梁存柱, 刘钟龄, 郝敦元. 羊草+大针茅草原群落退化演替机理的研究. 植物生态学报, 2000(4): 468-472.
WANG W, LIANG C Z, LIU Z L, HAO D Y. Mechanism of degradation succession in+steppe community., 2000(4): 468-472. (in Chinese)
[3] 乌恩, 夏庆梅, 高娃, 邢旗. 内蒙古天然草地磷素营养问题及其解决途径. 内蒙古草业, 2006(3): 4-7.
WU E, XIA Q M, GAO W, XING Q. Phosphorus nutrition problems and solutions in natural grassland of Inner Mongolia., 2006(3): 4-7. (in Chinese)
[4] 杜青峰. 内蒙古退化典型草原植被和土壤对氮磷肥配施的响应[D]. 重庆: 西南大学, 2017.
DU Q F. The responses of degraded typical steppe vegetation and soil to nitrogen and phosphorus fertilizations in Inner Mongolia[D]. Chongqing: Southwest University, 2017. (in Chinese)
[5] 董晓兵, 郝明德, 郭胜安, 石学军, 马甜, 刘公社. 氮磷肥配施对羊草干草产量、养分吸收及品质影响. 草地学报, 2014(6): 1232-1238.
DONG X B, HAO M D, GUO S A, SHI X J, MA T, LIU G S. The effects of nitrogen fertilizer and phosphate fertilizer rates on the yield, nutrient uptake and quality of., 2014(6): 1232-1238. (in Chinese)
[6] WANG F, DENG M J, XU J M, ZHU X L, MAO C Z. Molecular mechanisms of phosphate transport and signaling in higher plants., 2018, 74: 114-122.
[7] WU P, MA L G, HOU X L, WANG M Y, WU Y R, LIU F Y, DENG X W. Phosphate starvation triggers distinct alterations of genome expression inroots and leaves., 2003, 132(3): 1260-1271.
[8] POIRIER Y, BUCHER M. Phosphate transport and homeostasis in., 2002, 1: e0024.
[9] XU L, ZHAO H, WAN R, LIU Y, XU Z, TIAN W, RUAN W, WANG F, DENG M, WANG J, DOLAN L, LUAN S, XUE S, YI K. Identification of vacuolar phosphate efflux transporters in land plants., 2019, 5(1): 84-94.
[10] 耿燕, 吴漪, 贺金生. 内蒙古草地叶片磷含量与土壤有效磷的关系. 植物生态学报, 2011, 35(1): 1-8.
GENG Y, WU Y, HE J S. Relationship between leaf phosphorus concentration and soil phosphorus availability across Inner Mongolia grassland., 2011, 35(1): 1-8. (in Chinese)
[11] HE J S, WANG L, FLYNN D F B, WANG X P, MA W H, FANG J Y. Leaf nitrogen: phosphorus stoichiometry across Chinese grassland biomes., 2008, 155(2): 301-310.
[12] THOMPSON K, PARKINSON J A, BAND S R, SPENCER R E. A comparative study of leaf nutrient concentrations in a regional herbaceous flora., 1997, 136(4): 679-689.
[13] MA W, LI J, JIMOH S O, ZHANG Y, GUO F, DING Y, LI X, HOU X. Stoichiometric ratios support plant adaption to grazing moderated by soil nutrients and root enzymes., 2019, 7: e7047.
[14] 甄莉娜, 王润梅, 周凤, 李侠, 魏玉荣, 张英俊. 不同施磷水平下AM真菌对羊草生长的影响. 中国草地学报, 2015(6): 56-61.
ZHEN L N, WANG R M, ZHOU F, LI X, WEI Y R, ZHANG Y J. Influence of AM fungi on the growth ofunder phosphorus applying at different rate., 2015(6): 56-61. (in Chinese)
[15] 钱洁鑫. 丛枝菌根对严重退化羊草典型草原植被修复效应的研究[D]. 呼和浩特: 内蒙古农业大学, 2018.
QIAN J X. Research on the restoration effect of arbuscular mycorrhiza in severely degradedtypical steppe[D]. Huhhot: Inner Mongolia Agricultural University, 2018. (in Chinese)
[16] SECCO D, JABNOUNE M, WALKER H, SHOU H, WU P, POIRIER Y, WHELAN J. Spatio-temporal transcript profiling of rice roots and shoots in response to phosphate starvation and recovery., 2013, 25(11): 4285-4304.
[17] EXPOSITO-RODRIGUEZ M, BORGES A A, BORGES-PEREZ A, PEREZ J A. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process., 2008, 8: 131.
[18] UDVARDI M K, CZECHOWSKI T, SCHEIBLE W R. Eleven golden rules of quantitative RT-PCR., 2008, 20(7): 1736-1737.
[19] YANG Q, YIN J, LI G, QI L, YANG F, WANG R. Reference gene selection for qRT-PCR inKom. under different stress conditions., 2014, 41(4): 2325-2334.
[20] 胡宁宁, 郭慧琴, 李西良, 孔令琪, 武自念, 张继泽, 常春, 万东莉, 臧辉, 任卫波. 羊草不同组织实时定量PCR内参基因的筛选. 草业科学, 2017(7): 1434-1441.
HU N N, GUO H Q, LI X L, KONG L Q, WU Z N, ZHANG J Z, CHANG C, WAN D L, ZANG H, REN W B. Selection of reference genes for quantitative real-time PCR ofin different tissues., 2017(7): 1434-1441. (in Chinese)
[21] MISSON J, RAGHOTHAMA K G, JAIN A, JOUHET J, BLOCK M A, BLIGNY R, ORTET P, CREFF A, SOMERVILLE S, ROLLAND N, DOUMAS P, NACRY P, HERRERRA-ESTRELLA L, NUSSAUME L, THIBAUD M C. A genome-wide transcriptional analysis usingAffymetrix gene chips determined plant responses to phosphate deprivation., 2005, 102(33): 11934-11939.
[22] SUN Y, MU C, CHEN Y, KONG X, XU Y, ZHENG H, ZHANG H, WANG Q, XUE Y, LI Z, DING Z, LIU X. Comparative transcript profiling of maize inbreds in response to long-term phosphorus deficiency stress., 2016, 109: 467-481.
[23] LI L, YANG H, PENG L, REN W, GONG J, LIU P, WU X, HUANG F. Comparative study reveals insights of sheepgrass () coping with phosphate-deprived stress condition., 2019, 10: 170.
[24] 万永青, 张杰, 侯向阳, 王照兰, 马玉宝, 万其号, 万东莉. 羊草DHN3基因的克隆及其逆境响应的表达分析. 西北植物学报, 2018, 38(9): 1598-1604.
WAN Y Q, ZHANG J, HOU X Y, WANG Z L, MA Y B, WAN Q H, WAN D L. Cloning of LcDHN3 inand its expression under abiotic stresses., 2018, 38(9): 1598-1604. (in Chinese)
[25] VANDESOMPELE J, DE PRETER K, PATTYN F, POPPE B, VAN ROY N, DE PAEPE A, SPELEMAN F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes., 2002, 3(7): research0034.1–research0034.11.
[26] ANDERSEN C L, JENSEN J L, ORNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets., 2004, 64(15): 5245-5250.
[27] PFAFFL M W, TICHOPAD A, PRGOMET C, NEUVIANS T P. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: Best Keeper-Excel-based tool using pair-wise correlations., 2004, 26(6): 509-515.
[28] WAN D, WAN Y, YANG Q, ZOU B, REN W, DING Y, WANG Z, WANG R, WANG K, HOU X. Selection of reference genes for qRT-PCR analysis of gene expression induring environmental stresses., 2017, 12(1): e0169465.
[29] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta CT) method., 2001, 25(4): 402-408.
[30] SHANE M W, LAMBERS H. Systemic suppression of cluster-root formation and net P-uptake rates inat elevated P supply: a proteacean with resistance for developing symptoms of 'P toxicity'., 2006, 57(2): 413-423.
[31] SHANE M W, MCCULLY M E, LAMBERS H. Tissue and cellular phosphorus storage during development of phosphorus toxicity in(Proteaceae)., 2004, 55(399): 1033-1044.
[32] DEL-SAZ N F, ROMERO-MUNAR A, CAWTHRAY G R, PALMA F, AROCA R, BARAZA E, FLOREZ-SARASA I, LAMBERS H, RIBAS-CARBO M. Phosphorus concentration coordinates a respiratory bypass, synthesis and exudation of citrate, and the expression of high-affinity phosphorus transporters in., 2018, 41(4): 865-875.
[33] LI Q, FU L, WANG Y, ZHOU D, RITTMANN B E. Excessive phosphorus caused inhibition and cell damage during heterotrophic growth of., 2018, 268: 266-270.
[34] ZHANG C, FU J, WANG Y, BAO Z, ZHAO H. Identification of suitable reference genes for gene expression normalization in the quantitative real-time PCR analysis of sweet osmanthus (Lour.)., 2015, 10(8): e0136355.
[35] ZHANG Y T, PENG X R, LIU Y, LI Y L, LUO Y, WANG X R, TANG H R. Evaluation of suitable reference genes for qRT-PCR normalization in strawberry (x) under different experimental conditions., 2018, 19: 8.
[36] JAIN M, NIJHAWAN A, TYAGI A K, KHURANA J P. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR., 2006, 345(2): 646-651.
[37] YANG Z, CHEN Y, HU B, TAN Z, HUANG B. Identification and validation of reference genes for quantification of target gene expression with quantitative real-time PCR for tall fescue under four abiotic stresses., 2015, 10(3): e0119569.
[38] QU R, MIAO Y, CUI Y, CAO Y, ZHOU Y, TANG X, YANG J, WANG F. Selection of reference genes for the quantitative real-time PCR normalization of gene expression infortune., 2019, 20(1): 9.
[39] STEFANOVIC A, RIBOT C, ROUACHED H, WANG Y, CHONG J, BELBAHRI L, DELESSERT S, POIRIER Y. Members of the PHO1 gene family show limited functional redundancy in phosphate transfer to the shoot, and are regulated by phosphate deficiency via distinct pathways., 2007, 50(6): 982-994.
Response and the expression of Pi-responsive genes inunder inorganic phosphate treatment
WAN DongLi1, HOU XiangYang1, DING Yong1, REN WeiBo1, WANG Kai1, LI XiLiang1, WAN YongQing2
(1Institute of Grassland Research, Chinese Academy of Agricultural Sciences/Key Laboratory of Grassland Ecology and Restoration, Ministry of Agriculture, Hohhot 010010;2College of Life Sciences, Inner Mongolia Agricultural University/Inner Mongolia Key Laboratory of Plant Stress Physiology and Molecular Biology, Hohhot 010011)
【】Phosphorus is the nutrient elements that is essential for plant growth. It would provide the basic data for molecular mechanism investigation ofresponding to inorganic phosphate (Pi) treatment, via analyzing the response under Pi stress in, and selection of the reference genes for qRT-PCR analysis, along with relative expression analysis of Pi-responsive genes. 【】Usingseedlings as materials, the length of shoots and roots were measured, and the contents of Pi were examined via vanadium molybdenum yellow colorimetric method following different concentration of Pi treatments. Based on transcriptome data of, as well as NCBI nucleic acid sequences database, 7 and 1 candidate reference genes were selected, respectively. Total RNA was isolated from each sample after different concentration of Pi treatment. Expression level of candidate reference genes was examined through qRT-PCR, and the stability was calculated by geNorm, NormFinder and Bestkeeper. According to the most stable reference genes selected, the relative expression of Pi-responsive genes was analyzed according to the qRT-PCR results.】The phenotype observation showed that both lower and higher concentration of Pi stresses reduced the growth of, and the shoot growth was more sensitive to low Pi (or Pi deficiency) stress, whereas root length was more sensitive to high Pi stress. In addition, the accumulation of Pi was enhanced along with the increased concentration of Pi treatments. The melting curve analysis showed that single peak of every candidate reference gene was observed, and gene expression profiles indicated that their CT values ranged from 17.16 to 26.61, among whichexhibited the highest expression abundance with CT values ranged form 17.16 to 20.22, andshowed the lowest expression level with CT values ranged form 23.28 to 26.61.(2.09%) showed the smallest expression variation, and(6.8%) had the largest expression variation. Finally, according to the stability ranking of geNorm, NormFinder and Bestkeeper, the comprehensive stability ranking of 8 candidate reference genes were obtained via calculation geometric mean, among which the top three stable genes were,and, and the most unstable two genes wereand. Using,andas reference genes, qRT-PCR results showed that, compared with the control, the expression ofwas induced by low Pi or complete deprive of Pi,was induced by high Pi, while the expressions ofwas induced by both low and high Pi treatments.【】Both lower and higher concentration of Pi stresses blocked thegrowth, and different responsive patterns were observed between the shoots and roots. Three relatively stable expression genes,andwere selected and could be used as reference genes for qRT-PCR analysis under Pi stress.andwere involved in the response ofto low Pi and high Pi treatment, respectively, whilewas participated in the responsive process of both low Pi and high Pi treatments.
; Pi stress; qRT-PCR; reference gene; Pi-responsive gene

10.3864/j.issn.0578-1752.2019.23.003
2019-06-04;
2019-09-19
内蒙古自治区自然科学基金(2019MS03002)、中央级公益性科研院所基本科研业务费项目(1610332016018,1610332018001)、内蒙古农业大学高层次人才引进科研启动项目(NDYB2018-61)
万东莉,E-mail:wandongli@caas.cn。
万永青,E-mail:wyq176@imau.edu.cn
(责任编辑 李莉)

